借助生物信息学方法对肝癌基因标志物的筛选
窦承贤,郭 菲,梁晋涛,李桂银,2
(1.桂林电子科技大学生命与环境科学学院,广西壮族自治区桂林 541004;2.广东石油化工学院化学学院,广东茂名 525000)
肝癌是发生于肝脏部位的恶性肿瘤,有着多因素、多步骤的复杂发病因素,早期诊断具有很大难度,目前依旧缺乏有效的早期检测标志物,肝癌晚期患者在治疗后仍会出现生存期较短等问题,因此有必要从基因层面寻找有效的早期标志物.近年来,基因组学研究处于高速发展时期,多项研究发现一些基因的上调或下调与肝癌发生具有显著性关系,已确定的肝癌标志物有P53[1]、CYP1A1[2](细胞色素P4501A1)、ALDH2[3](乙醛脱氢酶2)、EPHX1[4](环氧化物水解酶)等.此外,Luo 等[5]在一项肝癌预后生物标志物的检测实验中,通过Oncomine、HPA 等数据库的表达数据,发现SSRP1是一种新的肝癌预后标志物.Shen 等[6]研究MITD1 预后价值时发现,MITD1 在肝癌患者中可作为一个独立的预后因素并且起着关键作用,还阐明了细胞分裂和免疫环境的变化如何促进肝癌的发展.
本研究采用生物信息学的研究方法,从GEO 数据库中选择GSE36376、GSE14520、GSE25097 等3 组数据,以一定阈值筛选出差异表达基因(differentially expressed genes,DEGs),将3 组差异表达基因的交集定义为公共差异表达基因. 通过富集分析和蛋白质互作网络分析得到枢纽基因并进行生物信息学分析,证实这些枢纽基因可以作为有效的肝癌基因标志物预测肝癌的早期发生.
1 材料与方法
1.1 肝癌数据集
GEO 数据库(https://www.ncbi.nlm.nih.gov/geo/)包含了多物种的数据信息,数据资源全面[7].从GEO数据库选择3个基因芯片数据集,分别为GSE36376、GSE14520 和GSE25097.每个数据集中都有正常和肿瘤对照的样本,具体信息见表1.
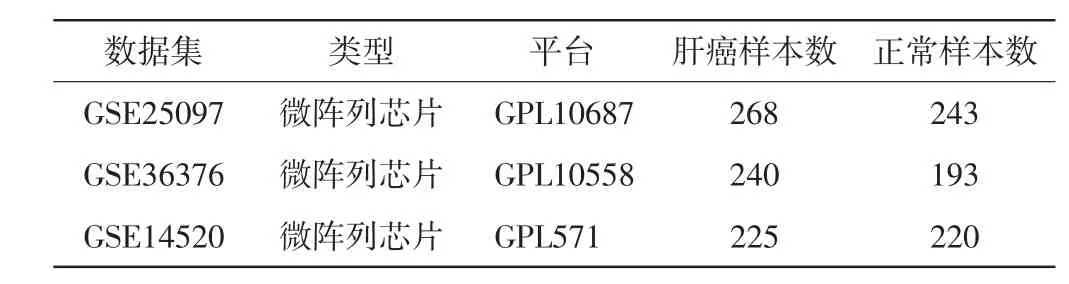
表1 基因数据集的详细信息Tab.1 Gene dataset details
1.2 差异表达基因的获取
使用GEO2R (https://www.ncbi.nlm.nih.gov/geo/geo2r/)工具获取3个数据集中肝癌与非肝癌组织间差异显著的基因[8],将结果进行进一步处理,去掉某些基因名为空的数据,以|log2(Fold change)|≥1 且P<0.01 为阈值,利用R 软件的“ggplot2”和“VennDiagram”包绘制火山图和韦恩图.
1.3 差异表达基因的富集分析
使用DAVID(https://david.ncifcrf.gov/tools.jsp)工具进行基因本体论(Gene Ontology,GO)和京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)富集分析[9],并且认为P<0.05 时为显著富集,具有统计学意义.
1.4 蛋白互作网络构建和枢纽基因选择
通过STRING(https://string-db.org/)建立蛋白互作网络(Protein-Protein Interaction,PPI)[10].设置交互评分的最低阈值为0.4,评估不同基因间的相互关系与相互作用.使用Cytoscape 软件的CytoHubba 插件,基于最大集团中心性(maximal clique centrality,MCC)筛选出枢纽基因.
1.5 枢纽基因的验证
使用GEPIA2(http://gepia.cancer-pku.cn/index.html)工具分别对枢纽基因进行总生存率的预后分析和对照样本间的表达分析[11],研究枢纽基因的预后及其在肝癌患者与正常人之间的差异表达.
1.6 枢纽基因KEGG 通路重分析与人类蛋白质图谱数据库中的数据挖掘
使用DAVID 在线工具对枢纽基因进行KEGG 分析,研究枢纽基因可能参与调控的通路.从蛋白质图谱数据库(Human Protein Atlas,HPA)[12]中检索关键调控基因的免疫组化染色数据,检测肝癌组织和正常组织对照中蛋白水平的表达.
1.7 肝癌病理分期分析
使用GEPIA2 在线工具对关键基因的主要肝癌病理分期进行分析,基于单向方差分析得到F值和P值,用小提琴图显示分析结果.
2 结果与分析
2.1 差异表达基因的获取
本研究利用GEO2R 在线工具分析GSE36376、GSE14520、GSE25097 这3个基因芯片数据,从而确定肝癌组织与正常组织间的差异表达基因.GSE25097包含1 872个DEGs(上调675个,下调1 197个);GSE36376 包含697个DEGs(上调430个,下调267个);GSE14520 包含1 289个DEGs(上调617个,下调672个),图1 为差异表达分析的火山图.3个数据集取交集后的公共DEGs 共197个,包含69个上调和128个下调,如图2 所示.
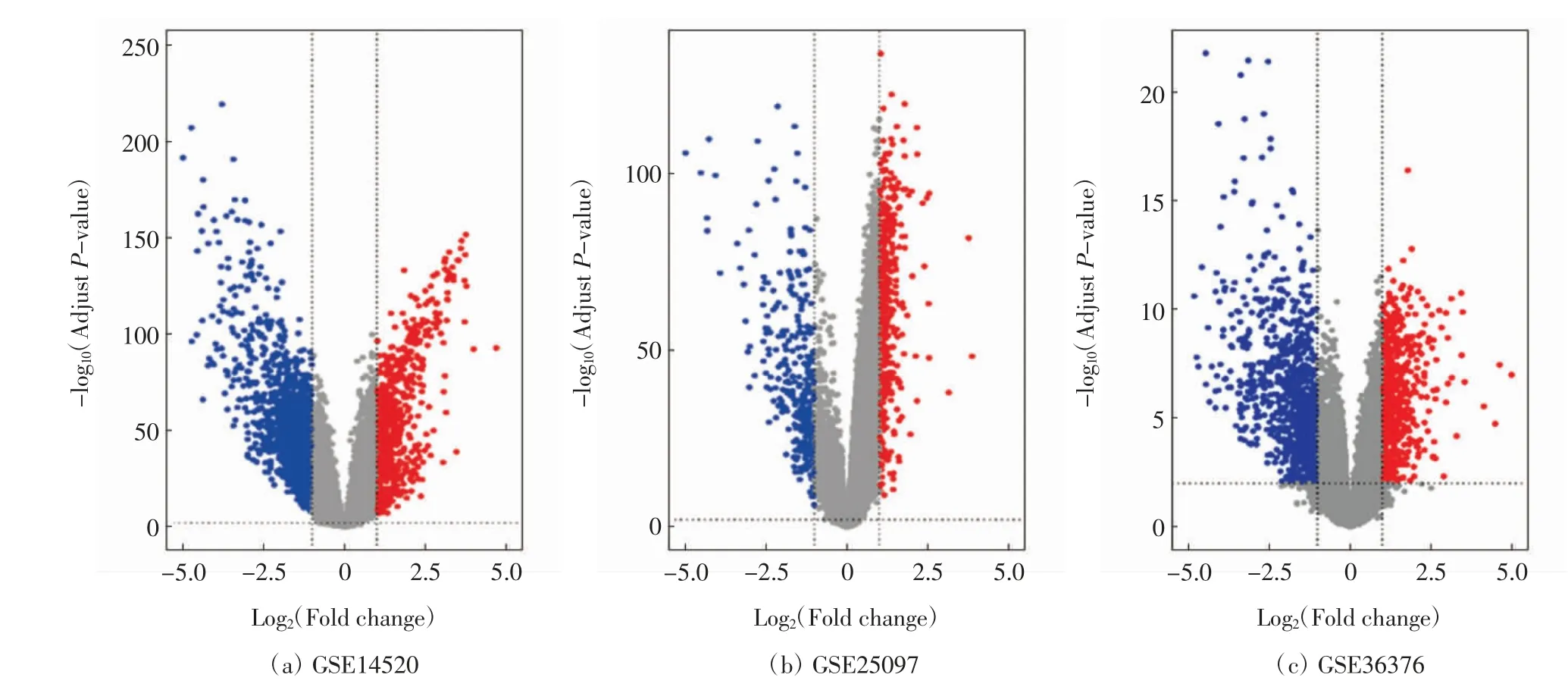
图1 差异表达基因的火山图Fig.1 Volcano map of DEGs

图2 筛选公共DEGs 的韦恩图Fig.2 Veen diagram for filter common DEGs
2.2 差异表达基因的功能富集分析
为了进一步研究DEGs 在肿瘤发生中的分子机制,通过GO 和KEGG 富集分析筛选了一系列富集途径,结果如图3 所示.GO 结果(图3(a)—3(c))显示有197个差异基因主要富集于:①生物过程(biological process,BP),包含异种生物代谢过程(xenobiotic metabolic process)、脂质代谢过程(cholesterol metabolic process)、药物代谢过程(drug metabolic process)等;②分子功能(molecular function,MF),包含同种蛋白结合(identical protein binding)、铁离子结合(iron ion binding)等;③细胞成分(cell component,CC),包含细胞外泌体(cytosol)、细胞外区域(extracellular exosome)和细胞外间隙(extracellular region)等. KEGG 分析(图3(d))表明:代谢途径(metabolic pathways)、药物代谢-细胞色素P450(drug metabolism-cytochrome P450)、视黄醇代谢(retinol metabolism)、酪氨酸代谢(tyrosine metabolism)和DNA 复制(DNA replication)是主要的富集通路,其中代谢途径最为显著.
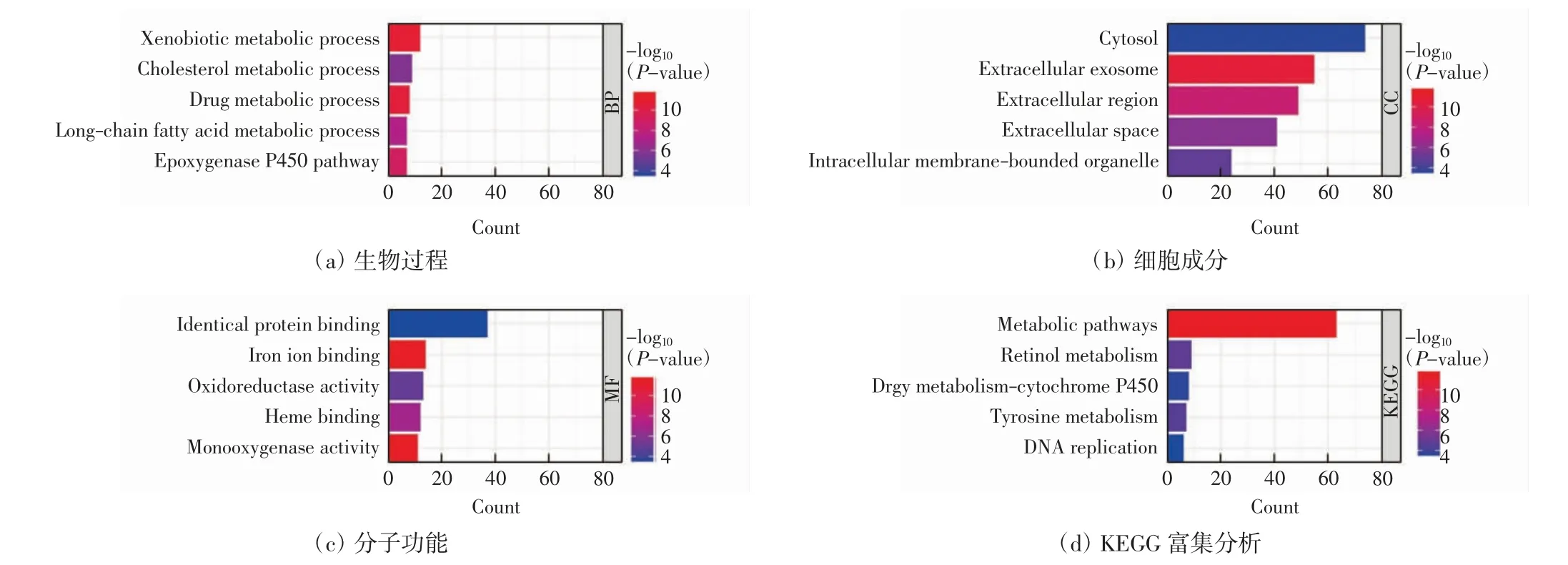
图3 197个差异表达基因的GO 和KEGG 富集分析Fig.3 GO and KEGG enrichment analysis of 197 DEGs
2.3 PPI 网络构建和枢纽基因筛选
结合STRING 工具和Cytoscape 软件构建PPI 网络,结果如图4(a)所示. 由图4(a)可知,该PPI 网络共有196个节点和764 条边. 选择MCC 计算出的8个得分最高的基因作为枢纽基因,分别为ASPM(assembly factor for spindle microtubules)、AURKA(aurora kinase A)、CDC20(cell division cycle 20)、FEN1(flap structure -specific endonuclease 1)、NUSAP1(nucleolar and spindle associated protein 1)、RACGAP1(rac gtpase activating protein 1)、RFC4(replication factor c subunit 4)、TOP2A(topoisomerase Ⅱalpha),这8个基因均为上调基因(图4(b)).

图4 PPI 网络和枢纽基因Fig.4 PPI network and hub genes
2.4 枢纽基因的验证
对8个枢纽基因进行总生存率的预后分析和差异表达分析,结果分别如图5 和图6 所示.由图5 可以看出,将样本以中位数为标准分为高表达组和低表达组,8个基因高表达时会伴随着总生存率的显著降低.进一步分析8个枢纽基因在肝癌患者与正常人之间的差异表达水平,结果显示,8个基因在肝癌样本中均为高表达(图6).
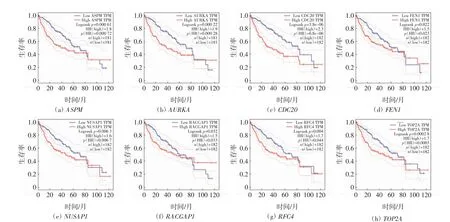
图5 枢纽基因生存分析与肝癌患者总生存曲线Fig.5 Hub genes survival analysis and overall survival curve of liver cancer patients
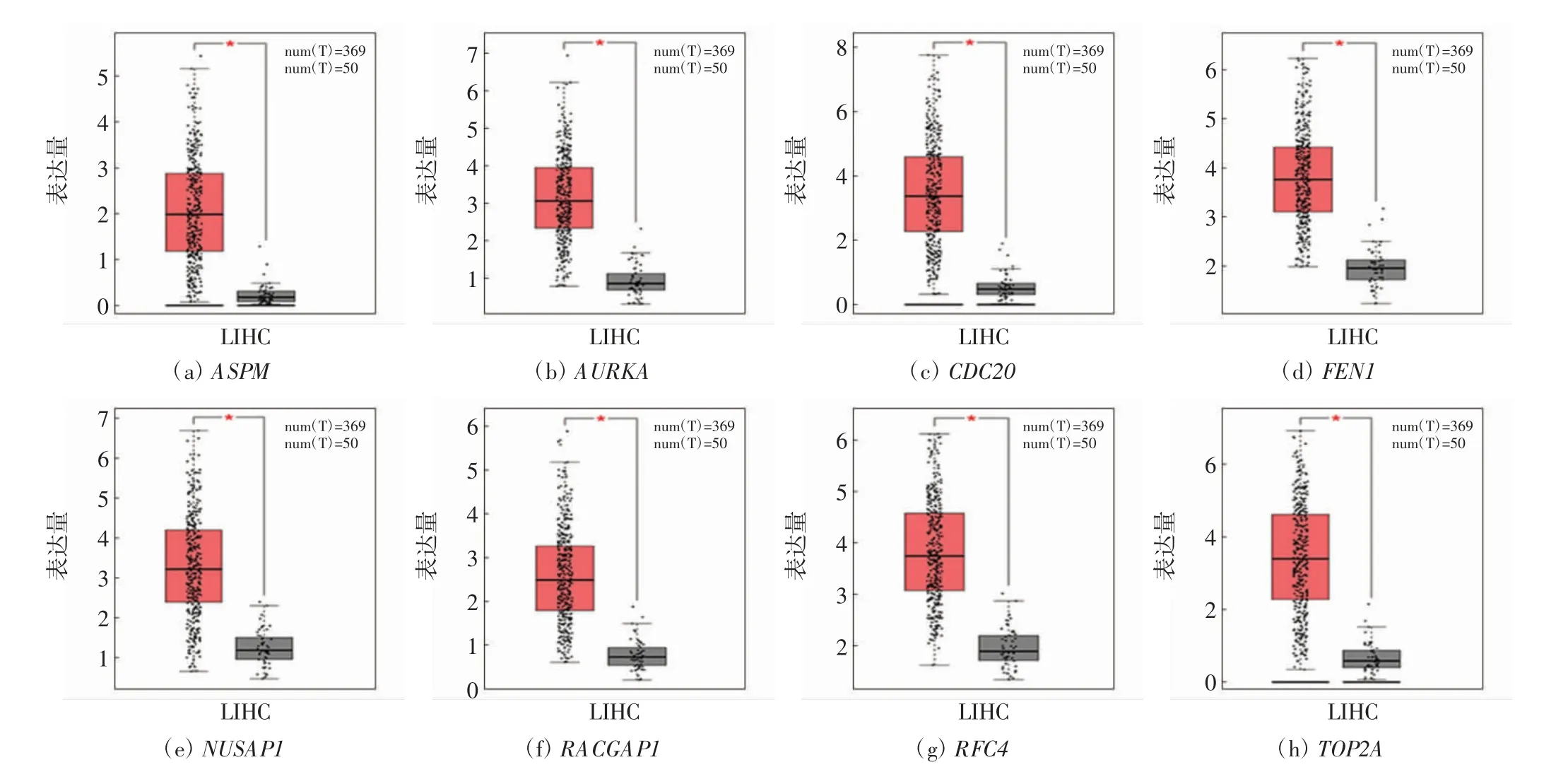
图6 枢纽基因在肝癌组织与正常组织中的表达Fig.6 Expression of hub gene in liver cancer and normal tissue
2.5 枢纽基因KEGG 通路重分析
对8个枢纽基因进行KEGG 重分析,研究这8个枢纽基因的富集通路,结果如表2 所示.由表2 可以看出,有4个基因在DNA 复制和卵母细胞减数分裂这2 条途径中富集,FEN1和RFC4这2个关键基因在DNA 复制通路中显著富集(P=0.017 6).
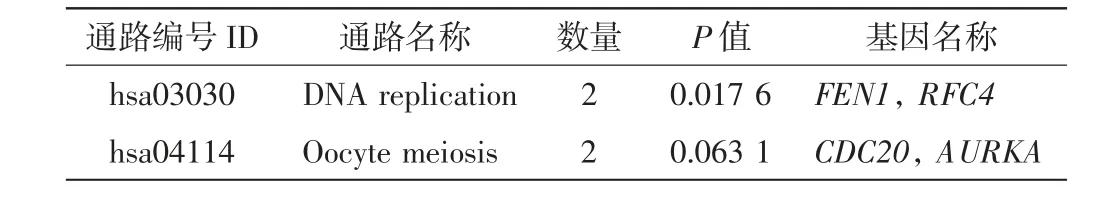
表2 枢纽基因的KEGG 重分析Tab.2 KEGG reanalysis of hub genes
2.6 蛋白表达分析
从人类蛋白质图谱数据库中通过检索HPA 数据库中的IHC 染色数据,在蛋白水平上研究RFC4和FEN1的表达,结果如图7 所示.由图7 可以看出,在肝癌组织中RFC4和FEN1均有中、高表达水平.

图7 HPA 数据库中肝癌和非癌性肝组织的免疫组化图像Fig.7 Immunohistochemical images of hepatocellular carcinoma and non cancerous liver tissues in HPA database
2.7 病理分期分析
使用GEPIA2 的“病理分期图”模块分析RFC4和FEN1的表达与病理分期之间的相关性,结果如图8所示.分析得到F值分别为7.23 和5.99,其对应的P值均小于0.05,表明这2个基因在肝癌各个分期的表达具有显著差异.
3 讨论与结论
近年来,多项研究通过生物信息学方法识别肝癌的预后生物标志物,然而肝癌的分子机制尚不完全清楚. 本研究综合生物信息学相关方法,利用GSE36376、GSE14520、GSE25097 数据集分析了一些有利于肝癌诊断和预测预后的生物标志物.通过3个基因数据集共筛选出了197个公共差异表达基因,GO 和KEGG 分析揭示了这些基因主要的富集途径,其中代谢途径是最显著的富集通路.代谢途径是完成代谢过程的一组相互衔接的酶促反应,肿瘤的起始和进展需要癌细胞的代谢重编程.癌细胞通过各种代谢途径自动改变其通量,以满足增加的生物能量和生物合成需求,并减轻氧化应激所需的癌细胞增殖和生存.癌症驱动基因突变与环境营养可用性共同控制着这些代谢途径的通量.当代谢产物异常积累时,也可促进肿瘤发生,异常代谢途径已被认为是癌症的标志之一,如Chen 等[13]报道了代谢途径增强可使P53 外显子产生突变进而导致肝癌预后不良.
利用Cytosacpe 软件的CytoHubba 插件从197个基因中筛选出了8个基因,分别为ASPM、AURKA、CDC20、FEN1、NUSAP1、RACGAP1、RFC4、TOP2A.既往研究显示,ASPM和TOP2A在结直肠癌[14-15]、胰腺癌[16]、膀胱癌[17-18]、肺腺癌[19-20]等多种癌症中异常表达,RFC4和FEN1与肿瘤的进展相关[21-22],AURKA的激活已被证明在多种癌症中发挥重要作用[23],CDC20、NUSAP1和RACGAP1均被报道了与肝癌的进展显著相关[24-26].
KEGG 重分析结果显示,RFC4和FEN1在DNA复制通路上富集.有研究证实,DNA 复制时发生错误是多种癌症发生的主要因素[27]. 基因组的稳定性直接关联到细胞是否发生癌变,其中,DNA 复制是最容易发生变化的过程,也是最容易致癌的过程.任何导致DNA 损伤高水平发生的条件也都会引发复制应激,这是基因组不稳定的来源之一,也是区分癌变前细胞与癌变细胞的一大标志.FEN1突变可能会引起单链DNA 断裂以及随后的DNA 复制叉瓦解,从而出现DNA 复制应激.持续的复制应激通常会引发P53 介导的衰亡进程或者细胞凋亡,用以预防肿瘤扩展.
RFC 家族在DNA 复制和DNA 修复中发挥重要作用.其中,编码RFC 复合体的第4 大亚基RFC4 也参与了这些生物过程.RFC4 全称为复制因子C 亚基4,位于第3 号染色体长臂上,可能参与了多引物DNA 模板的延伸. 有研究表明,RFC4基因与多种肿瘤不良预后显著相关,如Wang 等[28]提出DNA 拷贝数改变介导的RFC4表达上调与食管鳞状细胞癌的早期诊断和免疫逃逸有关,Zhang 等[29]发现RFC4在体内外均可促进口腔舌鳞癌的进展和生长. 在肝癌方面,Arai 等[30]发现敲除内源性复制因子C4 可降低肝癌细胞的生长并增强化疗敏感性;Chen 等[31]报道了人肝细胞癌中的复制因子C4 是一个与细胞增殖相关的强有力的预后因子.RFC4的解除调控可能有助于细胞增殖和肿瘤发生.
FEN1 全称为瓣结构特异性核酸内切酶1,位于第11 号染色体长臂上,主要功能为参与冈崎片段成熟、DNA 损伤修复、端粒稳定性维持与细胞凋亡DNA片段化,参与机体内多种DNA 代谢途径,作用极其重要.Wu 等[32]通过生物信息学分析发现,在乳腺癌组织中FEN1 蛋白水平和mRNA 表达明显高于正常组织,血清中FEN1 的水平随乳腺癌的发生而升高,而在术后患者中有所下降.Xu 等[33]也报道了FEN1 可调节polo 样激酶4(PLK4)的表达水平,从而促进三阴性乳腺癌细胞的迁移和侵袭.Shi 等[34]发现FEN1 的1个关键残基上的琥珀酰化参与了DNA 损伤反应,减少DNA 上存在的突变.Zhang 等[35]通过体外实验发现调节c-Myc、survivin 和G1/S-特异性周期蛋白-D1 沉默FEN1 可抑制细胞的增殖和迁移.
本研究借助生物信息学分析,从3个独立的基因芯片数据中,共鉴定出69个上调基因和128个下调基因.通过富集分析和蛋白质互作网络分析,筛选出8个枢纽基因,分别为CDC20、RACGAP1、ASPM、RFC4、FEN1、AURKA、NUSAP1、TOP2A.使用GEPIA2 在线工具进一步分析,8个基因的高表达都会伴随着肝癌总生存率的降低,并且8个基因在肝癌患者中均表现为高表达. 进一步对8个基因重新进行KEGG 通路富集,发现RFC4和FEN1这2个基因在DNA 复制通路中富集.通过人类蛋白质图谱数据库中的数据挖掘发现,在蛋白水平上肝癌患者的RFC4和FEN1基因的转录及翻译可能均表现为高表达,这还有待实验证明.本研究认为筛选出的8个基因可以作为肝癌诊断潜在的生物标志物,其中RFC4和FEN1可能是调控肝癌发生、发展和转移的关键基因.

