头颈部鳞状细胞癌血管生成预后模型的构建及验证
张钊银,王洪伟,苏萌,王珊,姚金光
(1. 右江民族医学院口腔医学院,广西 百色 533000;2. 右江民族医学院,广西 百色 533000)
研究显示[1],头颈部肿瘤是全世界第六大常见的恶性肿瘤,其中超过90%属于鳞状细胞癌,被称之为头颈部鳞状细胞癌(head and neck squamous cell carcinoma,HNSCC),其主要起源于口腔、鼻咽、口咽、下咽和喉部的黏膜上皮,包括吸烟、酗酒、HPV病毒感染等在内是其重要的危险因素[2-3]。由于其早期症状不明显,大多数HNSCC被发现时已属于晚期(Ⅲ期或Ⅳ期),其治疗后复发及转移风险高,5年生存率不足50%[4-6]。因此,迫切需要寻找有效的标志物来预测HNSCC预后和对药物治疗的反应。
血管生成是一种从现有血管系统的内皮生成新的血管,用于组织生长和伤口愈合的生物学过程[7]。研究表明[8],血管生成的调节是促血管生成因子和抗血管生成因子共同调控的,但在肿瘤组织中其调控失衡,导致病理性血管生成,并可通过分泌高水平的促血管生成因子,形成不成熟和可渗透血管为特征的异常血管网络,这会导致肿瘤灌注不良及缺氧反应,形成高度抑制性的肿瘤免疫微环境进而导致肿瘤免疫逃逸,最终使肿瘤更具侵袭性[9-10]。目前,抗血管生成药物已经在部分实体瘤中证明了其良好的应用价值,包括转移性结直肠癌、肾癌、晚期非小细胞肺癌等[11-14],证实了其能延长生存期。HUA Y H等[15]学者最新研究表明,抗PD-1单克隆抗体联合抗VEGF药物在复发/转移性HNSCC患者中安全有效,能延长总生存期,说明了抗血管生成药物在HNSCC的治疗中具有潜在的应用价值。然而,目前基于血管生成相关基因(angiogenesis related genes,ARGs)构建HNSCC预后模型少见报道。本研究旨在利用生物信息学寻找HNSCC中差异表达的预后相关ARGs,并构建预后模型,评估模型的预测价值及对治疗的反应,为HNSCC临床诊断和治疗提供新思路。
1 材料与方法
1.1 临床样本来源 收集2019年9月至2023年4月于右江民族医学院附属医院口腔科就诊且接受手术治疗的11例舌鳞状细胞癌患者的癌组织及对应的癌旁组织为研究对象,所有组织标本离体后放入RNA保存液并置于-80°长期保存,包括男性9例,女性2例,平均年龄为(58.90±6.80)岁,术前已排除放化疗等辅助治疗,术后经病理检查确诊。本研究经右江民族医学院附属医院伦理委员会审批(伦理审批编号:YYFY-LL-2023-116),所有患者均签署知情同意书。
1.2 数据来源 从UCSC Xena数据库(http://xena.ucsc.edu/)下载头颈部鳞状细胞癌(TCGA-HNSC-
C)的RNA-seq数据和生存数据,筛选样本编号结尾为01A(即原发肿瘤)的样本作为训练集,包含495例癌组织样本及44例正常组织样本。从GEO(https://www.ncbi.nlm.nih.gov/geo/)数据库下载GSE41613作为验证集,注释平台为GPL570,包含97例口腔鳞状细胞癌样本(见表1),其中494例带有完整生存数据。从GeneCards(https://www.genecards.org/)和MSigDB(https://www.gsea-msigdb.org/gsea/msigdb)数据库中获取ARGs,GeneCards中筛选关键词为“Angiogenesis”,筛选条件为Relevance score大于平均值(1.047),获得1 292个ARGs,MSigDB数据库的筛选关键词为“ANGIOGENESIS”,获取ANGIOGENESIS(Systematic name为“M14493”)数据集共48个ARGs,取上述两个数据集的并集并去除重复基因最终得到1 298个ARGs,获得数据时间为2023年3月20日。本研究所用数据都来自公共数据库,故无需伦理委员会审批。
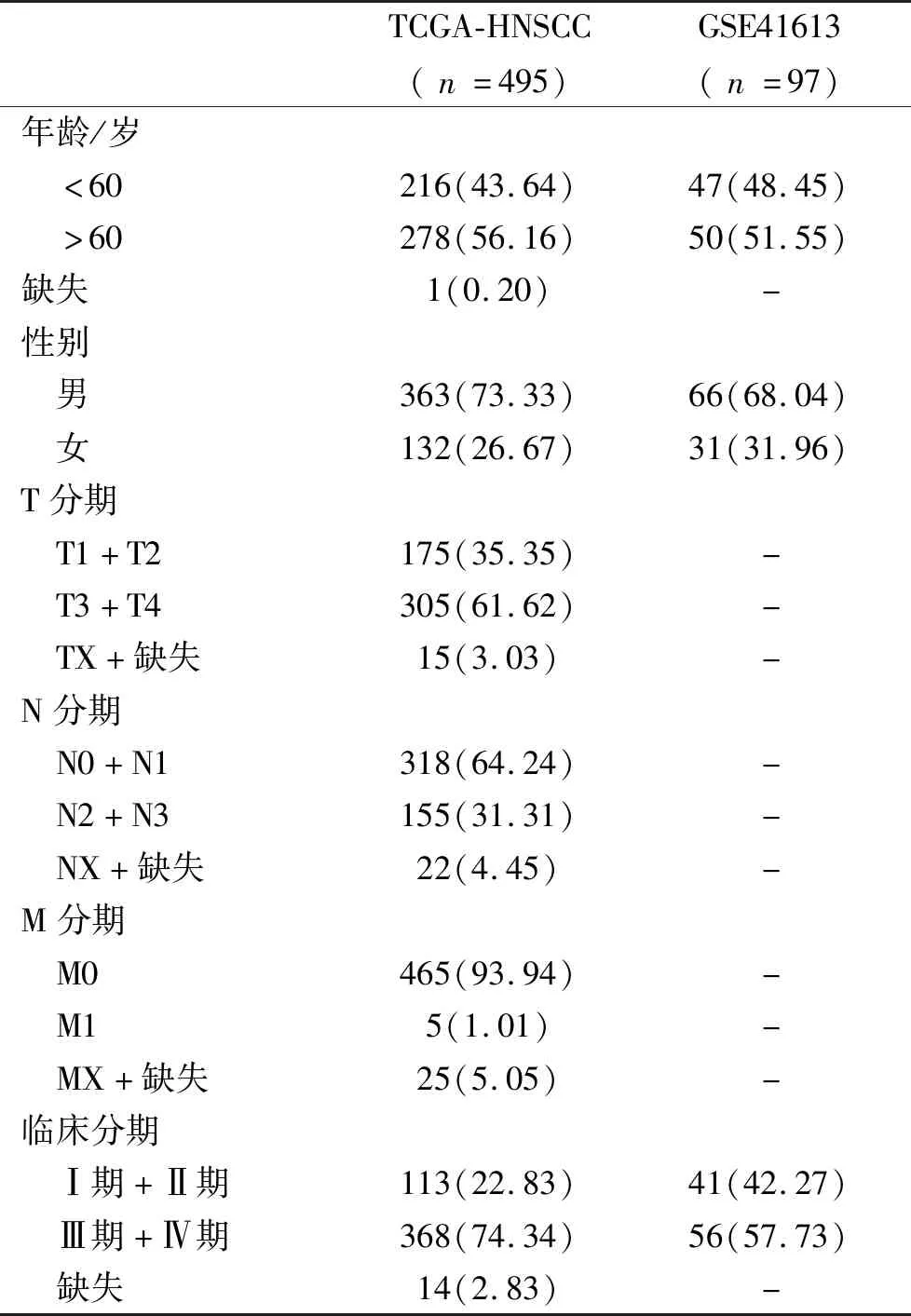
表1 TCGA-HNSCC和GSE41613临床基线表
1.3 差异表达ARGs的鉴定 首先利用R语言“DESeq2”包筛选出HNSCC的差异表达基因,以padj<0.05,|log2FoldChange|≥1作为标准筛选差异表达基因(differentially expressed genes,DEGs),并与ARGs取交集,获得差异表达的ARGs,并使用“ggthemes”包绘制差异基因火山图。
1.4 预后相关核心ARGs的鉴定 将HNSCC差异相关ARGs的表达数据与生存数据整合,使用“survival”包进行单因素Cox回归分析,以pvalue<0.05筛选获得预后相关ARGs。将筛选得到的预后相关ARGs上传至STRING(https://cn.string-db.org)数据库构建蛋白质-蛋白质互作用网络(protein-protein interacton,PPI),选择互作用评分为medium confident(0.4),并将互作用结果上传至Cytoscape软件(Version 3.9.0),使用Cytoscape中的“MCODE”插件筛选评分最高的模块基因作为预后相关核心ARGs,并进行可视化。
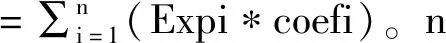
1.6 免疫浸润分析 使用ESTIMATE算法计算肿瘤组织中的基质评分(stromalscore)、免疫评分(immunescore)、以及基于前两者计算得出的ESTIMATE评分(ESTIMATEScore)和肿瘤纯度(tumorpurity),分析高低风险组肿瘤免疫浸润情况。分析高低风险组免疫检查点(immune checkpoint,IC)基因表达的差异。
1.7 药物敏感性敏分析 从药敏数据库GDSC(https://www.cancerrxgene.org/)中下载并整理药敏数据,基于药物半抑制浓度(IC50),使用“oncoPredict”包计算高低风险组对198种化疗药物的敏感性。
1.8 两个核心预后基因的RT-qPCR验证 对基于构建预后模型的预后相关核心ARGs进行生存分析,对两个预后基因进行RT-qPCR验证。使用Total RNA Kit I试剂盒(Omega Bio-Tek)提取总RNA,对提取的RNA进行质量检测。使用Hifair II 1st Strand cDNA Synthesis Super Mix[YEASEN,翌圣生物科技(上海)股份有限公司]逆转录试剂盒说明书将总RNA逆转录成cDNA,以cDNA为模板,使用Hieff qPCR SYBR Green Master Mix试剂[YEASEN,翌圣生物科技(上海)股份有限公司]并根据说明书进行RT-qPCR反应测定,使用β-ACTIN作为内参基因,使用2-△△Ct法进行定量分析计算相对表达量。RT-qPCR引物序列:PLAU上游引物:5′-TAACGATCCCCAGTTTGGCAC-3′;下游引物:5′-GGTCAGCAGCACACAGCATTT-3′;VEGF-C上游引物:5′-CACGAGCTACCTCAGCAAGA-3′;下游引物:5′-GCTGCCTGACACTGTGGTA-3′;β-ACTIN上游引物:5′-CCTGGCACCCAGCACAAT-3′;下游引物:5′-GGGCCGGACTCGTCATAC-3′,引物由生工生物工程(上海)股份有限公司合成。
1.9 统计学方法 使用R语言(version 4.2.3)及GraphPad Prism 9进行数据分析和图形绘制。针对两组数据的比较,对符合正态分布的数据使用t检验,而非正态分布的数据则使用WilCox秩和检验。P<0.05为差异具有统计学意义。
2 结果
2.1 差异表达ARGs的鉴定 利用R语言“DESeq2”包从HNSCC患者以padj<0.05,|log2FoldChange|≥1作为标准筛选出4 769个DEGs,并通过火山图展示(见图1A),与从GeneCards和MSigDB获得的1 298个ARGs取交集,获得414个差异表达的ARGs,并通过韦恩图进行展示(见图1B)。
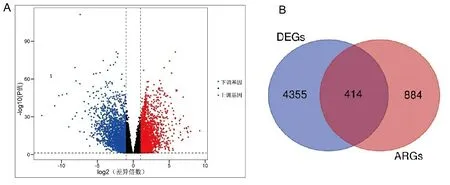
图1 HNSCC差异基因火山图和差异表达ARGs韦恩图
2.2 预后相关核心ARGs的鉴定 利用单因素Cox回归分析,以pvalue<0.05筛选得到72个预后相关ARGs(见图2A)。在STRING数据库上构建了72个节点基因及379条边数的PPI网络,利用Cytoscape中的“MCODE”插件筛选得到23个预后相关核心ARGs(见图2B)。
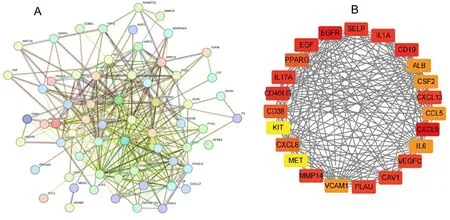
注:A.72个预后相关ARGs的PPI蛋白互作网络,圆圈表示蛋白质,圆圈之间连线表示蛋白质间相互作用,连线越多代表相互作用越强;B.“MCODE”插件筛选出的23个预后相关核心ARGs,颜色深浅与评分呈正相关,颜色越深,评分越高。
2.3 预后风险评分模型的构建和验证 对上述得到的23个预后相关核心ARGs进行进行LASSO回归分析(见图3A、图3B),得到14个与预后显著相关的ARGs,并展示了这14个基因单因素Cox回归分析结果及coef值(见表2)。根据coef值计算风险评分,具体公式为:风险评分=IL6表达×0.033491083+CCL5表达×(-0.030218342)+IL1A表达×0.006785821+ALB表达×0.233992094+PLAU表达×0.077462404+EGFR表达×0.07040458+CD19表达×(-0.136937929)+CD40LG表达×(-0.175847268)+KIT表达×(-0.122645187)+VEGFC表达×0.070554947+IL17A表达×(-0.229373238)+PPARG表达×0.312332183+CD38表达×(-0.117538852)+EGF表达×0.18141696,根据风险评分的中位值将样本分为高风险组和低风险组,作为训练集。使用这14个基因相同的coef值并根据基因在验证集GSE41613中的表达量计算风险评分(计算方法同训练集),并根据风险评分的中位值将样本分为高风险组和低风险组。生存分析显示在HNSCC训练集中高风险组的生存时间低于低风险组(P<0.001),见图4A,同时在验证集中高风险组的生存时间同样低于低风险组(P=0.01),见图4B,说明高风险组患者相对于低风险组生存时间更短、死亡率更高。ROC曲线显示验证集中1年、3年、5年的ROC曲线下面积(AUC)分别为(1年=0.675、3年=0.688、5年=0.644),见图4C,在验证集中ROC曲线下面积(AUC)分别为(1年=0.645、3年=0.654、5年=0.647),见图4D,说明模型具有较强的预测效力。
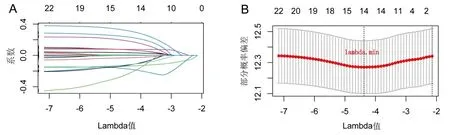
注:A.LASSO 系数分布特征;B.LASSO模型中最佳参数(lambda.min)的选择。
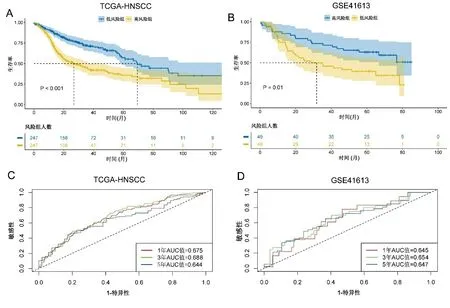
注:A.训练集TCGA-HNSCC高低风险组生存曲线;B.验证集GSE41613高低风险组生存曲线;C:训练集TCGA-HNSCC:1年、3年、5年的ROC曲线; D.验证集GSE41613:1年、3年、5年的ROC曲线。
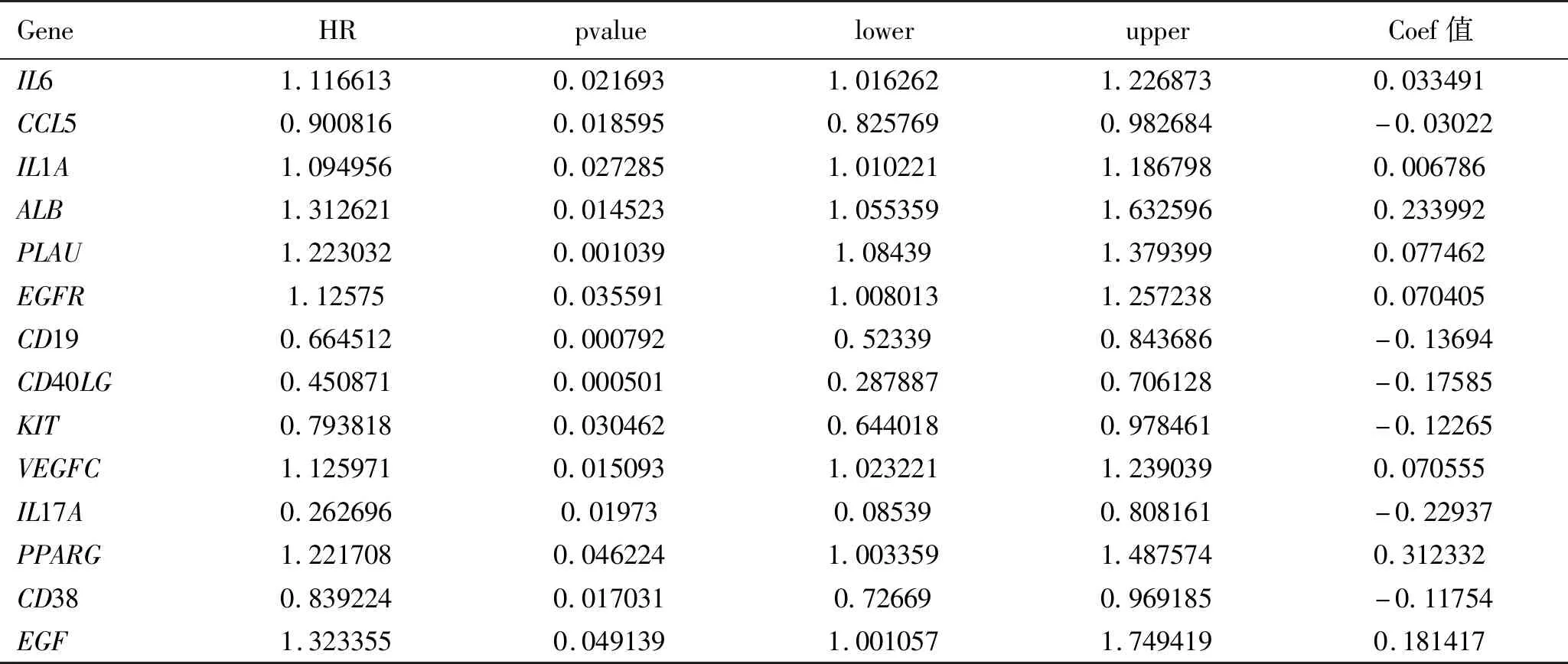
表2 14个与预后显著相关的ARGs单因素Cox回归分析结果及coef值
2.4 免疫浸润分析 进一步的免疫浸润分析显示,在ESTIMATE算法中,高低风险组的肿瘤基质评分无显著差异,低风险组的肿瘤免疫评分和ESTIMATE评分高于高风险组(P<0.001),高风险组具有更高的肿瘤纯度(P<0.001),如图5A。低风险组高表达PDCD1、CD274、CTLA4、LAG3、HAVCR2、TIGIT等6种免疫检查点基因(P<0.05),如图5B。
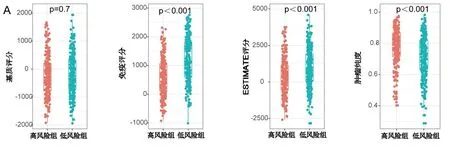
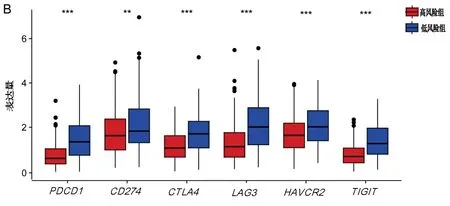
注:A.高低风险组基质评分、免疫评分、ESTIMATE评分、肿瘤纯度的差异;B.高低风险组PDCD1、CD274、CTLA4、LAG3、HAVCR2、TIGIT 6个免疫检查点基因的表达差异。**P<0.01,***P<0.001。
2.5 药物敏感性分析 基于药敏数据库GDSC,使用R语言“oncoPredict”包计算高低风险组对198种化疗药物的敏感性(基于IC50)。结果显示低分险组患者对包括顺铂(cisplatin)、5-氟尿嘧啶(5-fluorouracil)、吉非替尼(gefitinib)等HNSCC常用化疗药物IC50值更低(P<0.05),见图6A~图6C),低分险组患者对这些药物更敏感。而高风险组对包括Tozasertib、NU7441、SB216763等药物IC50值更低(P<0.05),见图6D~图6F,高分险组患者对这些药物更敏感。


注:A~F.依次为高低风险组对顺铂、5-氟尿嘧啶、吉非替尼、Tozasertib、NU7441、SB216763的敏感性(IC50),药物IC50越低说明药物处理更具敏感性。****P<0.0001。
2.6 核心预后基因的RT-qPCR验证 对基于构建预后模型的14个预后相关核心ARGs进行生存分析,发现PLAU、VEGF-C是影响预后的两个关键基因,PLAU(P=0.007),VEGF-C(P=0.023),见图7A、图7B)。RT-qPCR结果表明,与对应癌旁组织相比,PLAU,VEGF-C在舌鳞状细胞癌组织中的表达均显著上调(P<0.05),见图7C、图7D。
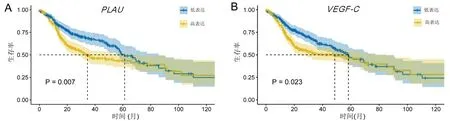
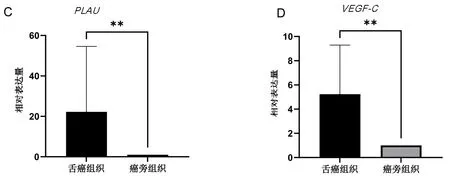
注:A.PLAU在TCGA-HNSCC的生存曲线;B.VEGF-C在TCGA-HNSCC的生存曲线;C.PLAU在舌鳞状细胞癌及对应癌旁中的表达情况;D.VEGF-C在舌鳞状细胞癌及对应癌旁中的表达情况。**P<0.01。
3 讨论
HNSCC通常被发现时已属于局部晚期,超过一半的患者会发生局部复发或远处转移[16],并且其复发和转移后的治疗效果差[17],开发可靠的预后和分子标志物并早期施以干预能大大改善HNSCC的生存状况。肿瘤血管生成是肿瘤治疗中一个很有吸引力的重要靶标,约90%的HNSCC高度表达血管生成因子,如VEGF[18],并且高表达VEGF预示着其较差的生存率[19-20]。基于抗血管生成药物的研究已经在HNSCC中进行,HOANG T等[21]研究表明,贝伐珠单抗(一种抗VEGF的单克隆抗体)在头颈部鳞癌细胞系和异种模型中能抑制肿瘤血管生成,显著增强放疗对肿瘤的抑制作用。因此,基于肿瘤血管生成相关基因的表达探究其与HNSCC治疗及预后的关系具有重要研究价值。在本研究中,从TCGA-HNSCC数据集中鉴定出了414个差异表达的ARGs。通过单因素Cox回归分析、构建PPI蛋白互作用网络结合Cytoscape的“MCODE”插件筛选核心模块及LASSO回归分析,最终得到14个与预后显著相关的ARGs并成功构建了预后模型,使用Kaplan-Meier生存分析及1年、3年、5年ROC曲线评估高低风险组的预测效能,结果显示风险评分具有良好的预测价值,且预后价值在GSE41613数据集都得到了验证,说明基于14个ARGs构建的预后模型能很好地预测HNSCC患者的预后。
免疫治疗目前已经成为治疗HNSCC患者的一个很有前景的治疗方法[22],肿瘤免疫浸润意味着免疫系统的初步识别,可能提示抗肿瘤免疫应答,肿瘤免疫浸润水平同时影响着免疫检查点抑制剂(ICIs)的抗肿瘤疗效[23-24]。基于ESTIMATE算法进行了高低风险组免疫浸润分析,结果提示低风险组具有更高的免疫评分,这提示低风险组相对于高风险组具有更高的免疫细胞浸润水平。针对免疫检查点的抗肿瘤研究已经在HNSCC中不断开展[25],本次研究结果提示低风险组高表达包括PDCD1、CD274、CTLA4等6个免疫检查点基因,这提示针对低风险组的免疫检查点抑制剂可能更容易取得疗效。通过免疫浸润分析结果发现,低风险组显示出“热”肿瘤免疫,显示出更强的免疫原性,而高风险组更多表现为“冷”肿瘤免疫,这对今后进一步深入认识血管生成与肿瘤免疫浸润的关系具有重要意义。对高低风险评分组的药物敏感性分析提示低风险评分组对目前HNSCC常用化疗药顺铂(cisplatin)、5-氟尿嘧啶(5-fluorouracil)、吉非替尼(gefitinib)更敏感。顺铂+5-氟尿嘧啶联合治疗是目前治疗HNSCC的传统标准治疗方案,而西妥昔单抗是第一个被批准用于HNSCC治疗的分子靶向药物[26],低风险评分的患者可能预示针对这3个HNSCC常用治疗药物更好的治疗效果。而高风险评分组则对Tozasertib、NU7441、SB216763 3种药物更敏感,由于高风险评分预示着患者更差的预后,故针对这3种药物的研究将更具前景。为了进一步寻找核心预后基因,本研究对建模的14个ARGs进行了生存分析,发现PLAU、VEGFC是其中影响预后的两个关键基因。已有的研究表明PLAU、VEGFC与HNSCC进展密切相关,CHEN G J等[27]研究表明PLAU能促进HNSCC的增值和上皮间质转化,本研究中选取HNSCC中的舌鳞状细胞癌组织进行RT-qPCR验证,结果表明PLAU在舌鳞状细胞癌中高表达于癌旁组织,但目前PLAU在舌鳞状细胞癌中的功能和机制少见报道,值得进一步深入研究。VEGF-C即血管内皮生长因子C,较早的研究表明VEGF-C广泛高表达于HNSCC血管与淋巴管组织,表明其参与了HNSCC的血管及淋巴管生成[28],SASAHIRA T等[29]研究表明VEGF-C在转移性舌癌细胞中的表达高于非转移性舌癌细胞,并且RT-qPCR结果也表明VEGF-C在舌鳞状细胞癌组织中显著上调,故VEGF-C很可能与HNSCC特别是舌鳞状细胞癌的复发转移密切相关。
综上所述,通过生物信息学方法构建了基于14个ARGs的HNSCC预后模型,该模型能有效预测HNSCC患者的预后和对药物治疗的反应,同时本次研究预测出了Tozasertib等3种针对高ARGs评分患者治疗敏感的药物。最后,利用RT-qPCR对其中的两个预后基因PLAU、VEGF-C进行了验证,这两个预后基因在HNSCC特别是在舌鳞状细胞癌中的机制值得进一步深入探究,但以上研究结果尚需进一步的实验验证及临床研究加以证明。

