基于PARMS技术的稻瘟病抗性基因分子标记研究及其在江苏太湖稻区的应用
宋云生,曹鹏辉,于雅洁,乔中英,朱勇良,谢裕林,袁彩勇,董明辉
(江苏太湖地区农业科学研究所,江苏 苏州 215000)
0 引言
水稻(Oryza sativaL.)是全球最为重要的粮食作物之一,尤其在我国其产量和稳定性对国家粮食安全和经济稳定具有至关重要的作用[1]。然而,水稻的产量和品质往往受到各种病害的严重威胁,其中稻瘟病(Magnaporthe oryzae)对水稻的影响尤为显著[2]。近年来,江苏太湖稻区的稻瘟病发生频率和严重程度均有上升的趋势[3],给当地水稻生产带来了重大压力,而抗病品种的利用是抵抗稻瘟病灾害风险最直接、最有效的方法,因此,对稻瘟病抗病育种的研究成为太湖稻区水稻研究的重要内容。
在稻瘟病的防治中,利用抗性基因对水稻品种进行筛选和改良是一种有效的策略[4]。随着基因工程和生物技术的快速发展,已经成功克隆并研究了一系列抗稻瘟病基因[5],如Pi1、Pi2、Pi9、Pia、Pib、Pik和Pita等。然而,由于不同水稻品种中抗性基因的分布、频率和效果存在较大差异[6],如何准确、高效地在育种材料中识别和利用抗性基因,已经成为当前学者们的重要研究方向之一。相比于传统的育种方法,分子标记辅助育种[7](Marker-Assisted Selection, MAS)有助于提高育种的效率和精确性,利用抗性基因的分子标记,可以在育种过程中对目标基因进行追踪和筛选,从而实现快速、准确的育种进程。以往多项研究[8-9]已开发了一系列的分子标记,如SSR、AFLP、CAPS、SNP等分子标记在抗性基因的定位、鉴定和辅助选择育种等方面取得了显著的成果。作为一种独特的SNPPCR分析技术,PARMS (Penta-Primer Amplification Refractory Mutation System,五引物扩增受阻突变体系)结合了1对通用荧光引物、1对SNP等位基因特异引物以及1条反向共用引物进行检测[10],该技术无须对PCR扩增产物进行耗时的DNA电泳分析,简化了操作过程,同时保持了较高的标记精确性,使得在大规模的育种项目中快速、准确地筛选和鉴定抗性基因成为可能。卿冬进等[11]采用PARMS技术成功开发了水稻Pigm基因的分子标记,通过对48份水稻亲本进行基因型鉴定,并结合田间抗性表型验证,证实了该技术的可靠性。伍豪等[12]研究表明,利用PARMS技术开发的分子标记可有效地运用于水稻抗病性分子标记辅助育种。然而,以往研究大多集中于单个抗性基因的分子标记开发和应用,对于多个抗性基因共同作用的研究相对较少,而且抗性基因的效果容易受到遗传背景和自然环境的影响[13]。因此,利用抗性基因进行水稻抗病性育种仍需要进一步研究和探索。
江苏太湖稻区水稻种植历史悠久,品种变异类型多样、抗性基因资源丰富[14],开发和利用分子标记对于提升该地区水稻的抗性育种水平具有重要的现实意义。本研究以课题组多年筛选培育的水稻高代材料为主体,针对江苏太湖稻区的稻瘟病种类和病原菌特性[15],基于PARMS技术对Pi2、Pib、Pita和Pik等4个基因的快速准确检测进行深入研究,分析以上抗性基因在供试水稻材料中的分布特征,并根据田间抗病性表现探讨其与抗性基因之间的关系,旨在为该地区抗稻瘟病水稻品种选育提供技术支持。
1 材料与方法
1.1 供试材料
供试水稻材料为江苏太湖地区农业科学研究所筛选培育的高代稳定材料206份。供试菌株为2021年江苏省农业科学院植物保护研究所提供的代表性稻瘟病菌株(2021-3、2021-43、2021-71、2021-150和2021-497)。由4个抗性基因分子标记开发的感病和抗病品种对照分别为嘉58和淮稻5号(Pi2)、南粳46和淮稻5号(Pib)、武运粳24和南粳46(Pita)、武运粳24和南粳46(Pik)。
1.2 供试水稻材料穗颈瘟抗性鉴定
穗颈瘟田间自然鉴定在江苏省常州市金坛试验基地进行。供试水稻材料和诱发品种(苏御糯、丽江黑谷)采用交错播种方式,在水稻分蘖阶段将稻瘟病菌样撒布在田间,于成熟期观察并记录穗颈瘟的发病情况,群体抗性分级标准如表1所示。水稻全生育期内不防治病害,虫害防治和水肥管理参照大田常规生产。
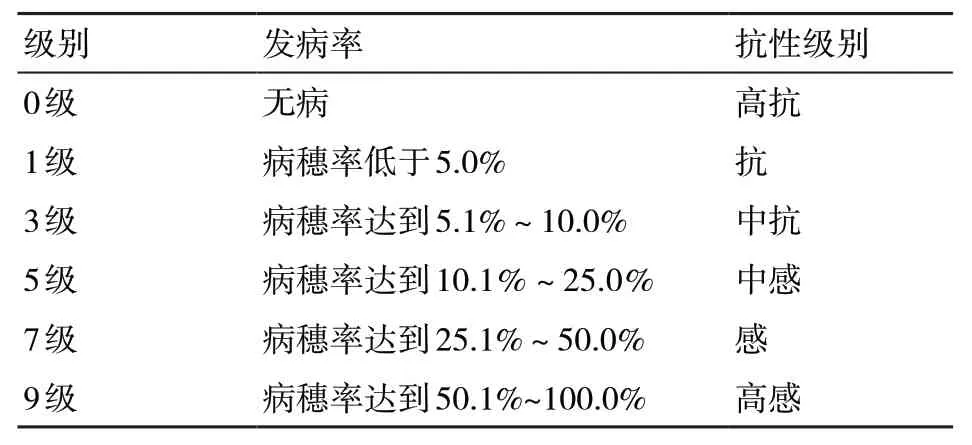
表1 水稻穗颈瘟发病群体抗性分级标准
1.3 水稻叶片DNA提取与PCR扩增
2021年8月对206份水稻材料叶片进行取样。取长、宽约1 cm的叶片置入深孔板中(96孔1.2 mL),注入100 μL 0.3 mol/L的氢氧化钠,进行50 Hz磨样1 min,研磨完成后以3000 r/min离心1 min,随后在沸水浴中处理1 min,加入400 μL 0.2 mol/L Tris-HCl混匀,再次进行沸水浴1 min,水浴完成后以3000 r/min离心1 min,取上清液稀释10~30倍,用于后续PCR扩增。参考卿冬进等[11]的研究方法对所选206份水稻材料进行PCR扩增,通过PARMS引物设计网站(http://www.snpway.com)设计等位基因特异引物和反向共用引物,详细的标记引物序列见表2,由武汉景肽生物科技有限公司负责合成引物。

表2 分子标记引物序列及荧光信号类型
1.4 等位基因型分型鉴定
PCR扩增完成之后,采用酶标仪(TECAN infinite M1000)读取扩增产物的荧光信号强度,使用snpdecoder 在线软件(www.snpway.com/snpdecoder)将荧光信号进行解析并转换,以获取清晰且直观的基因分型图。
1.5 数据处理
使用Excel 2016软件对试验数据进行处理和图表绘制。采用SPSS 21.0统计分析软件对水稻材料携带的抗性基因与其田间穗颈瘟抗性级别进行相关性分析。
2 结果与分析
2.1 分子标记开发
利用稻瘟病抗性品种基因序列与感病品种等位基因的序列差异,设计针对水稻抗稻瘟病基因Pi2、Pib、Pita和Pik的4组共显性SNP分子标记,相应的分子标记名称分别为SYS-Pi2、SYS-Pib、SYS-Pita和SYS-Pik,其引物序列及荧光信号类型见表2。对每个基因都设计了1对通用荧光引物、1对SNP等位基因特异引物以及1条反向共用引物,以确保扩增出目标基因的SNP位点。
其中,2条携带不同荧光信号的等位基因特异引物,将分别与PARMS master mix中带FAM和HEX的荧光信号相互匹配。根据引物序列对比分析可知,预测PCR扩增后,以上引物能够有效地扩增目标基因的SNP位点,并通过检测2种荧光信号将抗病品种与感病品种的基因型区分开,以实现这4组分子标记的特异性。
2.2 基因型检测
利用开发设计的4组荧光分子标记检测206份水稻材料的基因型,于PCR扩增后经荧光扫描、数据分析,得到FAM和HEX荧光信号的散点图(图1)。在供试水稻材料中,4组分子标记均能有效识别区分相应的SNP差异,表明分子标记开发成功。
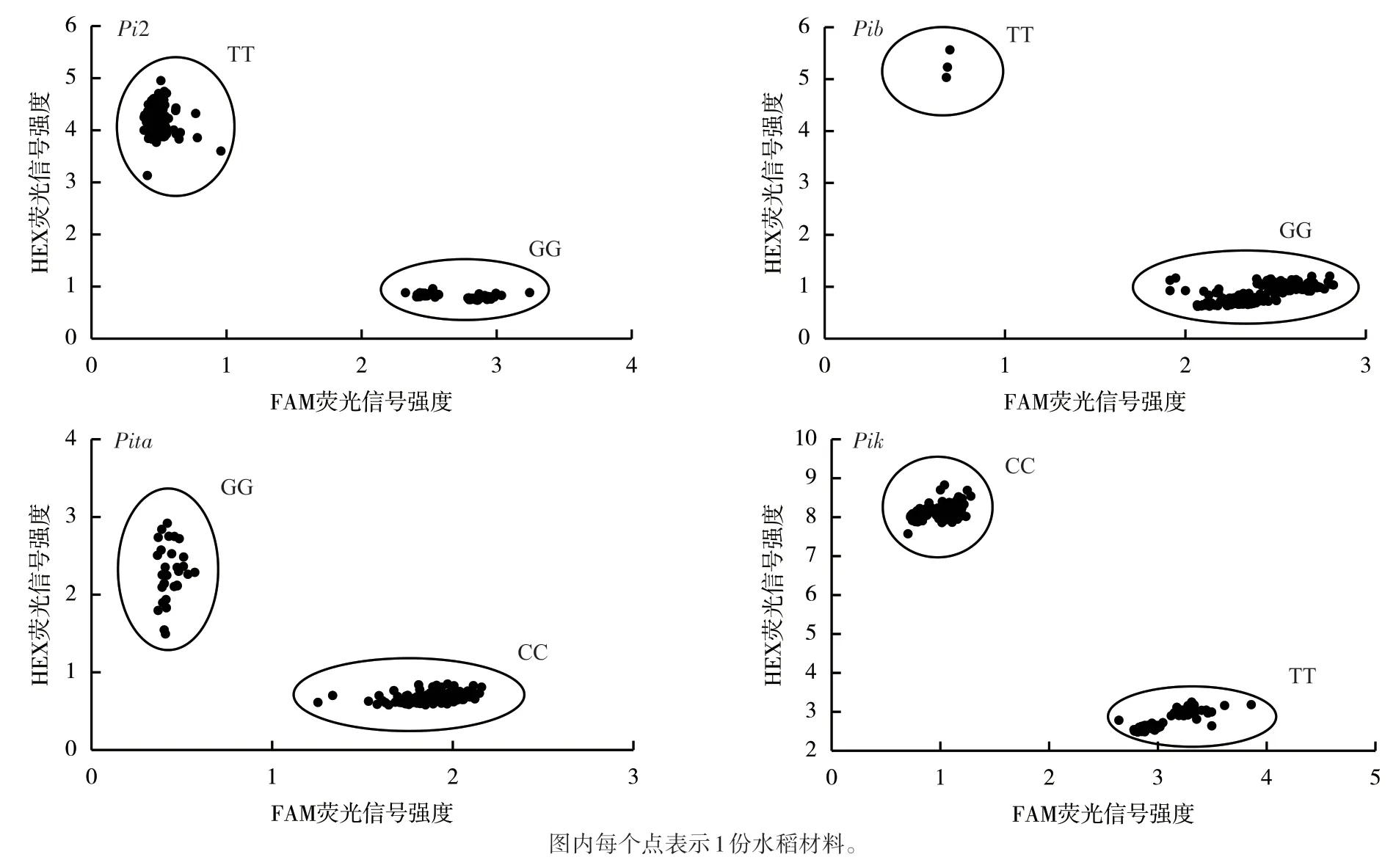
图1 分子标记对206份水稻材料的基因分型检测结果
根据图1结果,汇总稻瘟病抗性基因在供试材料中的分布如图2所示。结合引物设计原理分析,发现在206份水稻材料中Pib抗性基因的检出率最高,达到了98.06%(202/206);其次为Pita和Pik基因,检出率分别为85.44%(176/206)和40.29%(83/206);Pi2基因的检出率则较低,为26.70%(55/206);而未检出以上抗性基因的材料仅1份。以上结果表明,多年的育种选择在很大程度上提高了抗性基因存在的比例,其中Pib、Pita和Pik抗性基因在样本中具有较为广泛的分布,而Pi2基因的低检出率也提示了其在水稻材料中的稀缺性,这可能与水稻材料的遗传背景和种质资源有关。
进一步分析显示,在所有研究材料存在的基因组合中,同时存在4个抗性基因(Pi2+Pib+Pita+Pik)和3个抗性基因(Pi2+Pita+Pik)的材料数量较少,均仅有11份,占比均为5.34%(11/206)。而存在2个抗性基因和3个抗性基因的组合分别以(Pib+Pita)和(Pib+Pita+Pik)的材料较多,占比分别为84.47%(174/206)和34.47%(71/206)。这种情况反映出在太湖稻区,水稻更倾向于利用Pib、Pita和Pik基因提供对穗颈瘟的抗性,而较少依赖Pi2基因。
2.3 田间诱发抗病性鉴定
206份水稻材料田间诱发穗颈瘟发病情况统计结果显示(表3),穗颈瘟达到1级的有48份,表现为抗;3级的有90份,表现为中抗;5级的有47份,表现为中感;7级的有21份,表现为感;0级高抗和9级高感的材料均未发现。结合4个抗性基因存在情况发现,具有单一Pib基因的水稻材料在抗穗颈瘟上表现优异,中抗级别以上材料有4份,而Pi2、Pita和Pik基因则相对较弱,这突显了Pib基因在防控稻瘟病中的重要性。在复合基因型中,当同时存在2个基因“Pib+Pita”时,水稻材料对穗颈瘟的抗性表现出更强的稳健性,中抗级别以上材料有48份,反映出这2个基因在防治稻瘟病上的潜在协同作用。此外,三基因型“Pi2+Pita+Pik”的抗性级别偏向于较强,中抗以上材料达到47份,尤其是当4个抗性基因Pi2、Pib、Pita和Pik共存时,虽然样本量仅有11份,但其抗性表现较好,特别是在1级的抗性级别中占比最高,揭示了多抗性基因共存对稻瘟病的抵抗力显著增强。
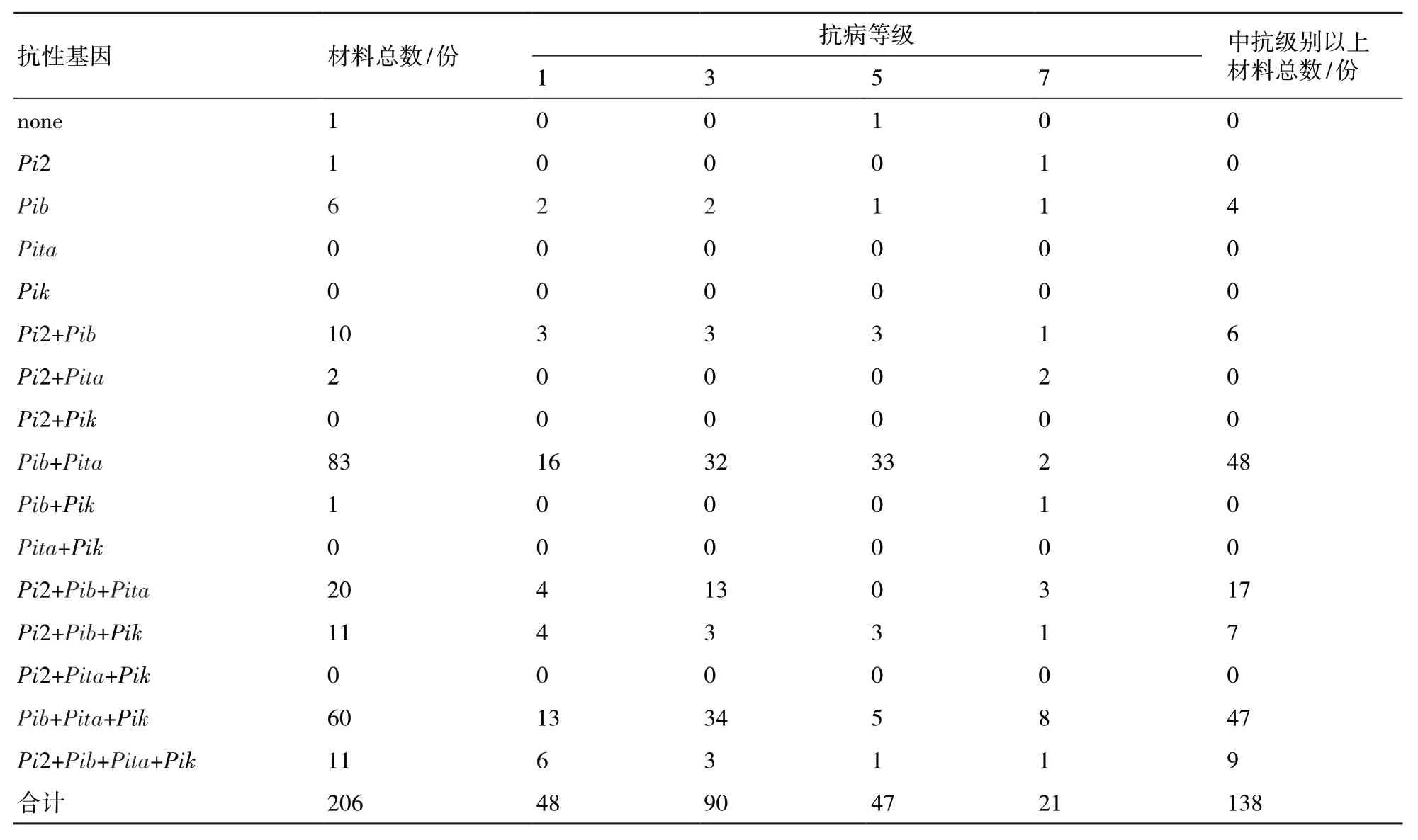
表3 不同抗性基因组合的田间抗病性鉴定结果
相关性分析表明,Pib基因的存在与穗颈瘟的抗性呈极显著相关(r=0.204**,P<0.01),表明带有Pib基因的水稻材料在抗穗颈瘟病方面可能有更好的表现。Pik基因与穗颈瘟抗性的相关性也达显著水平(r=0.149*,P<0.05),而Pi2和Pita基因与穗颈瘟抗性的相关性均不显著(r=0.086和r=0.135,P>0.05),这表明Pib和Pik基因可能对稻瘟病的抗性产生更大的影响。
3 讨论与结论
水稻抗稻瘟病的种质创新和优良品种的选育是稻瘟病防治的最有效、最经济的手段[16],其优势主要体现在减少了对化学药剂的依赖,不仅降低了对生态环境的破坏,还有效地减轻了农业生产者的成本负担。分子标记技术在抗病品种的选育过程中起到了重要的推动作用,可以迅速、高效地从大量水稻品种中筛选出携带抗性基因的优良品种[17-18]。本研究基于PARMS技术开发了针对抗稻瘟病基因的分子标记,并对江苏太湖稻区206份水稻育种高代稳定材料的基因型进行了准确鉴定,为了解抗稻瘟病基因的分布和作用,以及抗性基因与田间抗病性的关联,提供了高效的技术工具。通过应用分子标记对基因型的鉴定结果,有针对性地对水稻育种材料进行稻瘟病抗性筛选改良,可提高稻瘟病抗性选育效率。
水稻品种的抗病水平与其抗性基因种类密切相关[19],不同种植区域水稻中蕴含的稻瘟病抗性基因有所不同[20],深入了解抗性基因的分布情况是稻瘟病抗性品种选育的基础。本研究结果显示,Pib、Pita和Pik抗性基因在太湖稻区供试水稻材料中分布广泛,同时存在2个抗性基因(Pib+Pita)和3个抗性基因(Pib+Pita+Pik)的样本较多,而Pi2基因的检出率则较低。王小秋等[21]研究发现,在江苏粳稻品种中Pib和Pita基因的比例超过了50%,说明以上2个基因在稻瘟病抗性育种中应用广泛。范方军等[22]通过对近年来审定的江苏水稻品种进行稻瘟病抗性基因检测,发现大部分品种都包含Pib、Pita、Pik基因,多数材料聚合了多个基因,与本研究结果较为一致。本研究对基因型与田间自然诱发抗病性表现的关系进行了深入分析,探讨了基因间的协同作用。供试材料中具有单一Pib基因的水稻材料在抗穗颈瘟上表现优异,当Pib和Pita同时存在时,对稻瘟病的抗性显著增强,特别是4个抗性基因Pi2、Pib、Pita和Pik共存时,材料中达到1级抗性级别的占比最高,这与王军等[23-25]的研究结果一致,均强调了复合抗性基因在病害抗性增强中的关键作用。同时,本研究还发现Pib和Pik基因与穗颈瘟的抗性呈(极)显著正相关,表明Pib和Pik基因可能会对稻瘟病的抗性产生更大的影响,为进一步理解稻瘟病抗性的遗传机制提供了重要线索。与传统的PCR技术和序列标记位点(SNP)技术相比,PARMS技术在基因型鉴定中表现出更高的检测效率[26],近年来在水稻[11]、小麦[27]、玉米[28]、油菜[29]上被广泛应用。然而,本研究在操作中发现,当目标区域变异较小或重复序列较多时,PARMS技术的准确性会受到影响,在应用中还需注意进行相应优化。
尽管本文在抗稻瘟病基因分布和基因型与田间抗病性关系等方面取得了一些研究结果,但试验数据可能受到研究材料和技术选择的影响,仍存在一些局限性。由于研究聚焦于江苏太湖稻区,选择的水稻材料和抗性基因也主要是针对该地区,一方面可能会限制结论的泛化性,但另一方面也使本研究结果更具有针对性和实际应用价值。今后的研究将进一步考虑抗性基因的组合及其与抗病性的关系,进行更全面的抗性基因检测和更大样本的田间抗病性鉴定。

