基于全基因组测序的丁酸梭菌安全性评价
高应瑞 , 康福忠 , 孟铁健 , 刘珂飞 , 王调调 , 陈金艳 , 孙彤
1.天津生机集团股份有限公司,天津 300384;2.天津市农业生态环境监测与农产品质量检测中心,天津 300193
丁酸梭菌(Clostridium butyricum),又名酪酸菌、酪酸梭菌和丁酸梭状芽孢杆菌[1],在自然界中主要存在于动物和人类消化道、粪便以及环境土壤中[2]。丁酸梭菌是人体及动物肠道的重要益生菌,能够调节肠道微生物菌群,提高机体免疫力等,从而改善畜禽健康[3]。随着丁酸梭菌潜在应用价值的发现及逐渐增长的需求,对丁酸梭菌菌株相关制品的安全性进行评估已迫在眉睫,也亟需建立益生性丁酸梭菌的鉴定标准。
近年来,菌种的安全性日益受到相关行业人员和研究者的关注,而其对抗生素的耐药性、及产毒能力和致病因子的存在成为其安全性判断的重要依据。随着基因测序技术的不断发展,人们对微生物菌株安全性的认识也在不断深入。《益生菌的科学共识(2019年版)》中曾提出,对于新发现的菌株,应该通过全基因组测序对其生物信息进行分析与阐述,以此评估其耐药性、致病性等特征[4]。因此,本研究从分子生物学、基因组学对丁酸梭菌的安全性进行综合评价研究,基于基因测序技术的生物信息学分析以筛查菌种中与抗生素耐药、毒力和致病性基因,以期探究菌种潜在的安全性风险机制。
1 材料与方法
1.1 实验材料
1.1.1 实验菌种及培养基 本研究所用的丁酸梭菌分离自健康鸡肠道,并由生机集团实验室保存。所用的培养基为硫乙醇酸盐培养基。
1.1.2 仪器与设备 AW200SG厌氧培养箱购自ELECTROTEK依莱泰科公司;GI80TW立式自动压力蒸汽灭菌器购自致微(厦门)仪器有限公司;Qubit荧光定量仪购自美国Thermo Fisher Scientific公司;Covaris超声破碎仪购自苏州贝斯派生物科技有限公司;Agilent 2100 Bioanalyzer生物分析仪购自安捷伦公司;Qubit dsDNA HS Assay Kit 500 assays试剂盒购自Thermo Fisher公司,Agencourt AMPure XP-Medium kit试剂盒购自Bechman Coulter公司。
1.2 实验方法
1.2.1 培养基配制 蛋白胨15.0 g,酵母浸出粉5.0 g,葡萄糖5.0 g,硫乙醇酸钠0.5 g,L-胱氨酸盐酸盐0.5 g,氯化钠2.5 g,加入去离子水溶解、定容至1000 mL,并调整pH为7.4(固体培养基另需加入琼脂20 g),121 ℃下高压蒸汽灭菌培养基20 min。L-胱氨酸盐酸盐用无菌0.22 μm膜过滤后,无菌加入到灭菌后的培养基中。
1.2.2 菌株活化与培养 将实验室保存的丁酸梭菌甘油管用接种针穿刺于固体试管培养基上,37 ℃下培养18~20 h。待长出明显菌落后接种于试管液体培养液中,装液量10 mL,然后于37 ℃静止厌氧培养18 h。
1.2.3 菌株DNA提取 取少量待检菌体于无菌研磨器中,研磨成粉末状,后将其快速刮至10 mL离心管中,加入3.5 mL裂解液缓冲液1和100 μL蛋白酶K,并加入30 μL RNaseA,反复混匀数次,37 °C水浴30 min后,再加入1.2 mL裂解缓冲液2,反复混匀,55 °C水浴反应120 min。
1.2.4 基因组DNA过柱纯化 平衡Genomic-tip柱子,加入4 mL缓冲液QBT,待完全流出使用。将组织裂解好的样本管,室温离心10 min。将裂解后的上清液转移至无菌5 mL离心管中,室温离心5 min。将得到的上清溶液过Genomic-tip柱,使DNA被吸附在Genomic-tip柱吸附膜上,自然load流出柱子。加入QC wash buffer洗脱2次。用5 mL QF缓冲液洗脱吸附于膜上的DNA。用异丙醇沉淀洗脱下来的液体,反复混匀,-20 °C冰箱沉淀2 h以上。将用于沉淀的管从-20 °C拿出,离心10 min。用750 μL 75%酒精洗涤沉淀后转移至2.0 mL离心管。继续离心10 min,弃上清,室温速甩30 s,晾干备用。根据DNA沉淀的大小加入适量的EB溶液于样品管中,37 °C孵育30~60 min,直至样品完全溶解,后放入4 °C冰箱进入核酸质检环节。
1.2.5 DNA测序、质控与组装 基于PromethION测序仪全基因组DNA文库构建的基本操作。取一定量基因组DNA样品,用Pippin HT进行片段分选,筛选目的范围内的片段。配制反应体系,适温反应进行DNA末端修复和缺口(nick)修复。配制接头连接反应体系,适温反应使DNA与接头连接,然后对文库进行质检。激活处理芯片,选择合适孔数芯片进行上机测序。去除质量值不合格以及含N碱基数目总和超过10%的reads;去除adapter和duplication污染,得到Clean reads。测序得到原始数据为Polymerase reads,过滤掉测序接头及低质量等数据最终得到可用Subreads,并以.bam格式保存(proovread)。后对Subread进行自纠正或混合纠正,得到Corrected reads。基于Corrected reads采用多种软件分别组装,最后选择出最优组装结果。采用第二代小片段数据对其进行单碱基纠正,以得到最优序列。对组装结果进行序列成环判断、基因组序列及质粒序列区分[5-10]。
1.2.6 基因预测与注释 采用Glimmer软件进行组装结果的基因预测[10-12]。将预测得到基因的氨基酸序列与相关数据库:Gene Ontology(GO)[13-14]、Kyoto Encyclopedia of Genes and Genomes(KEGG)[15]、Cluster of Orthologous Groups of proteins(COG)[16]、Virulence Factor Database(VFDB)进行比对,以获取相关功能注释数据。所有的注释均使用Diamond软件以及各数据库协调判定完成。
1.2.7 耐药基因注释 通过与CARD和ARDB数据库相比较,发现了可能导致菌株耐药的基因。参照农业农村部发布的《直接饲喂微生物和发酵制品生产菌株鉴定及其安全性评价指南》要求,在序列比对时,设置序列相似性≥80%,序列覆盖度≥70%。
1.2.8 细菌致病菌毒力因子注释 通过与VFDB数据库比对分析,对菌株基因组中可能存在的毒力基因进行检测。参照农业农村部发布的《直接饲喂微生物和发酵制品生产菌株鉴定及其安全性评价指南》要求,在序列比对时,设置序列相似性≥80%,序列覆盖度≥70%。
1.2.9 比较基因组学 对测序菌株基因组序列、参考菌株基因组序列和基因集序列数据进行对比分析,得出测序菌株与参考菌株之间的结构差异、突变和菌株间亲缘进化关系。
2 结果与分析
2.1 丁酸梭菌全基因组概况
如图1所示,获得来自测序平台的有效数据CleanData后进行组装,得到最佳组装结果,然后通过单碱基纠正、序列环状、质粒库比对分析,结果显示丁酸梭菌基因组DNA总长度4709 567 bp,其中包含1条环状染色体DNA、1条环状质粒和2条线性质粒。环状染色体DNA大小3873 131 bp,G+C摩尔百分含量为28.86%,环状质粒DNA大小769297 bp,G+C含量为28.33%,线性质粒1 DNA大小49922 bp,G+C含量为28.26%,线性质粒2 DNA大小17217 bp,G+C含量为27.89%。基因组中预测到4285个基因、87个tRNA基因、44个rRNA基因和3个sRNA基因。
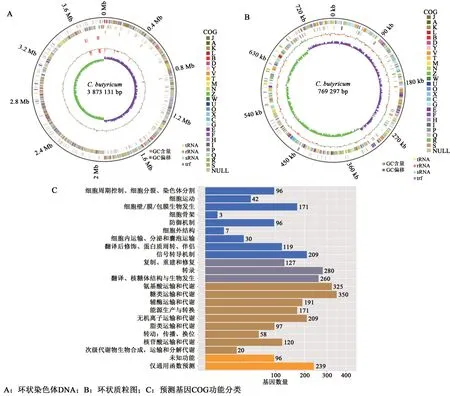
图1 丁酸梭菌的基因组完成图与预测基因COG功能分类图Fig. 1 Genome completion map and predicted gene COG functional classification map of Clostridium butyricum
通过COG对丁酸梭菌基因进行注释与功能分类,共得到2894个(67.54%)注释基因,分23个不同功能组(图1C),预测分组最多的为一般功能基因。丁酸梭菌基因组中注释基因数相对较多的为氨基酸代谢与转运、核糖体的生物合成、辅酶转运、脂代谢与转运、能量传递等。它们包含合成、运输和分解次级代谢产物的能力,且碳水化合物代谢相关基因占比最大。
2.2 耐药性分析
通过注释得到有关耐药基因的名称及其所耐受不同抗生素的种类信息。Antibiotic Resistance Genes Database数据库包含13293个基因,共377种类型、257种抗生素,124个门,3369个物种。The Comprehensive Antibiotic Resistance Database数据库包含细菌耐药性分子基础数据,包括基因、蛋白质以及突变。通过将丁酸梭菌基因组序列与数据库比对,均未检测到耐药基因存在。丁酸梭菌为益生菌类菌株,常运用于动物饲养以及调节人类胃肠道功能,但无已知耐药基因存在,这为今后在动物饲料添加剂的耐药性安全开发方面有重要的指导价值。
2.3 毒力基因分析
VFDB数据库主要对致病菌、衣原体和支原体致病因子进行注释分析。通过将丁酸梭菌的基因组序列与VFDB数据库进行比对,结果未检测到有毒力相关基因的存在,说明该株丁酸梭菌不含已知毒力基因,有助于今后在动物用饲料添加剂的毒力安全性评价方面提供指导。
2.4 比较基因组学分析
2.4.1 ANI分析 基于全基因组的相似性指标,例如平均核苷酸同一性(average nucleotide identity,ANI),有助于解决有关遗传不连续性或明确物种边界的问题。有文献分析的80亿个基因组对中有99.8%符合>95%的种内ANI值和<83%的种间ANI值。
ANI能够反映出不同的两个基因组之间共享的所有直系同源基因的平均核苷酸同一性,并且能够分辨出相同或进化关系密切的物种菌株。由于直系同源基因在比较基因组对之间可能有很大差异,虽然ANI并不严格地衡量核心基因组的进化相关性,但它能够密切反映在传统微生物学概念中用于定义物种的DNA-DNA杂交相关性,同时它考虑了细菌基因库的流动性或者细菌之间功能的共享。对菌株进行ANI分析,分析方案结果如图2所示,被测丁酸梭菌株与来源为健康鸡肠道中的丁酸梭菌株种间进化关系最为密切,ANI值为99.995361%,这一结果为今后丁酸梭菌应用于禽类饲料添加剂的开发奠定了基础。

图2 ANI热图Fig. 2 ANI heat map
2.4.2 物种进化 通过表型或基因型相似性和差异性绘制不同物种或菌株之间的分支图,同时可以显示不同物种之间的进化关系。树中的节点表示其分支最近的共同祖先,节点与节点之间线段的长度对应于进化的时间。使用TreeBeST构建系统发育树,从图3中可见,C. butyricum菌株与C. butyricum1菌株种间进化关系较近,这对后续禽类靶动物饲料开发具有重要的指导价值。
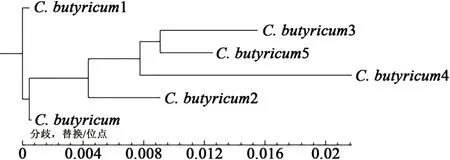
图3 系统进化树Fig. 3 System evolution tree
3 讨论
目前,国际上对益生菌的评价暂无统一标准,各国的安全性评价仍处于不断研究与规范中。近年来,随着高通量测序技术的迅速发展,基因组测序技术已成为微生物鉴定分类、追溯和功能预测的常用方法,因此,从遗传水平上评价菌株的安全性和危险因素成为可能。ARDB、CARD耐药基因数据库以及VFDB毒力因子数据库等为菌株从全基因组测序中筛查耐药、毒力与致病性等基因提供了重要的数据保障。本研究对丁酸梭菌的全基因组进行测序组装,通过与耐药性数据库ARDB和CARD进行数据注释,全面地掌握了菌株的耐药情况,检测比对结果中未检出有任何耐药基因的存在,同时,通过测序结果与毒力因子数据库VFDB比对,未检测到相关毒力因子基因的存在,以目前已经收录的数据库为基础,在基因组水平上证实了该菌株的安全性。对丁酸梭菌的耐药性、毒力和致病性方面的安全性研究,为该菌株在畜牧业中的实际应用奠定了良好的基础。
随着畜牧业减抗、禁抗时代的来临,丁酸梭菌的问世填补了我国“后抗生素时代”的空白,对于今后在动物医学临床上治疗多种动物肠道疾病、增加生产者收益、改善饲养环境、提高禽畜动物的产品安全品质,具有极其重要的意义。因此,菌株的安全性评价也受到越来越多的重视,这不仅要求在菌株分子水平上寻求突破口,今后在产品开发及应用中,还应增加临床耐药性及临床致病性等安全性评价内容,从而综合评价并指导其应用于产品开发。

