BDNF基因、同伴关系与青少年早期抑郁:基于动态发展视角*
曹衍淼 方惠慈 朱欣悦 纪林芹 张文新
基因、同伴关系与青少年早期抑郁:基于动态发展视角*
曹衍淼 方惠慈 朱欣悦 纪林芹 张文新
(山东师范大学心理学院, 济南 250014)
青少年抑郁是遗传基因与环境动态交互作用的结果, 但是现有研究忽视了抑郁遗传效应的发展动态性。本研究通过对1086名青少年(平均年龄12.32, 50%女生)进行3年的追踪, 分别从遗传效应的年龄差异以及遗传效应影响抑郁发展轨迹的角度, 考察基因与同伴关系对青少年抑郁的动态影响。结果显示:(1)在3个时间点上,基因与同伴拒绝交互影响青少年抑郁, 但其作用模式存在年龄差异:12岁时, MetMet基因型携带者对环境敏感性高于ValMet基因型携带者; 13岁时, MetMet和ValVal基因型携带者对环境的敏感性均高于ValMet基因型携带者; 14岁时, ValVal基因型携带者对环境的敏感性高于ValMet基因型携带者。(2)青少年早期抑郁呈线性增长趋势, 但是抑郁初始水平与增长速度无关。(3)基因与同伴拒绝交互预测青少年抑郁的初始水平, 相比ValMet基因型, 携带MetMet基因型的青少年在经历同伴拒绝后抑郁初始水平更高。(4)基因显著预测青少年抑郁增长速度, 相比ValMet基因型携带者, 携带MetMet和ValVal基因型的青少年抑郁增长速度更快。
青少年抑郁, BDNF基因, 同伴拒绝, 年龄差异, 潜变量增长模型
1 问题提出
近20年来, 抑郁的分子遗传研究取得了系列重要突破。既有研究探索了众多抑郁的风险基因及其与环境的交互作用机制, 揭示了基因通过内表型影响抑郁的潜在过程(如Cao et al., 2021; Caspi et al., 2003)。然而, 多数分子遗传研究仍然采用静态视角, 对抑郁遗传效应的动态发展问题关注不足。特别是在该领域一系列关键问题上, 譬如“抑郁的遗传效应是否随年龄增长发生变化?” “风险基因如何在抑郁发展中发挥作用”等, 已有研究尚不能给出确定的答案。本研究采用动态发展视角, 考察脑源性神经营养因子(brain-derived neurotrophic factor,)基因与同伴关系交互作用的年龄差异及其对青少年抑郁发展轨迹的影响。
1.1 BDNF基因、同伴关系与青少年抑郁
根据抑郁的神经营养假说(neurotrophin hypothesis), 中枢BDNF功能缺陷会导致海马和前额叶皮质神经元受损, 引发抑郁障碍(Martinowich et al., 2007)。此外, 抑郁的下丘脑−垂体−皮质醇假说(hypothalamic-pituitary-cortisol hypothesis)指出, 应激反应失调是引发抑郁的重要机制(Belmaker & Agam, 2008)。基因通过调节BDNF的表达, 不仅在突触可塑性和神经元连接性中发挥重要作用(Kowiański et al., 2018), 而且影响个体对压力的反应性(Notaras & van den Buuse, 2020), 因而是抑郁的“易感”基因。基因位于11号染色体p13区, 其第11号外显子区的Val66Met (rs6265)单核苷酸多态性与BDNF的细胞内转运和活性依赖性分泌密切相关。该多态性上G到A突变导致缬氨酸−蛋氨酸(Val-Met)替换, 使得Met(A)等位基因与BDNF分泌减少与活性降低相关(González-Castro et al., 2015)。基于上述神经生理基础, 诸多研究发现了基因与抑郁的关联(Hilt et al., 2007), 并且在抑郁相关内表型——杏仁核和海马体积、功能研究中获得了支持(Casey et al., 2009)。
在探索抑郁易感基因的同时, 研究者亦关注影响抑郁的环境风险, 尤其是青少年期的同伴关系。研究显示, 同伴关系(如同伴接纳和同伴拒绝)与抑郁的发生发展密切相关, 其在青少年期的重要性超过了其他环境风险(Rudolph et al., 2000)。同伴接纳是抑郁的保护性因素, 高水平的同伴接纳不仅是青少年自尊和归属感的重要来源, 并且与较低的皮质醇浓度有关, 因而能够降低抑郁风险(Baumeister & Leary, 1995; Blackhart et al., 2007)。同伴拒绝则会导致青少年形成消极的自我图式, 导致应激功能失调, 增加抑郁风险(Blackhart et al., 2007; Platt et al., 2013)。尽管同伴接纳和同伴拒绝具有中等程度的负相关, 但其对抑郁的影响和作用模式存在差异(Guyer et al., 2012; Hsu et al., 2015), 因此本研究分别考察同伴接纳和同伴拒绝在抑郁发展中的作用。
更重要的是,基因与环境交互影响抑郁。譬如, Gottfredson等(2015)对青少年的研究发现, 在经历同伴侵害后, 携带ValVal基因型的青少年比携带Met等位基因的青少年具有更高的抑郁水平。此外, Chen等(2013)研究显示,基因与压力性生活事件、母亲教养行为交互影响青少年抑郁, 相比Met等位基因携带者, Val等位基因携带者对积极或消极环境因素更加敏感, 表现出更少或更多的抑郁症状。有关儿童(Meyer et al., 2018)和成人(Aguilera et al., 2009)的研究虽然也发现了基因与环境的交互作用, 却显示Met等位基因携带者对环境的敏感性更高。譬如, Meyer等(2018)发现基因与消极教养对错误相关负波(error- related negativity, ERN, 一种抑郁的内表型)具有交互作用, 相比ValVal基因型携带者, 携带Met等位基因的幼儿在经历高消极教养后具有更高的ΔERN。尽管既有研究在抑郁的易感基因上(Val vs. Met)存在分歧, 但这些不一致的研究结果提示基因及其与环境的交互作用可能存在年龄差异。
1.2 BDNF基因及其与环境因素的交互作用:年龄差异的证据
遗传与环境交互作用的发展动态性问题是发展心理学和行为遗传学的重要理论问题。根据Lerner (1978)的天性与教养的动态交互观, 遗传基因与环境因素的交互效应会随年龄或发展阶段而动态变化。最近, Boyce等(2020, 2021)的“基因−环境-时间”交互理论框架(the interactive gene-environment- time framework), 进一步指出基因(DNA序列、表观遗传过程等)、环境(积极、消极经历等)和时间(年龄、发展阶段、关键期等)三个因素交互影响个体的心理健康适应结果。
近年来, 在抑郁、反思(rumination)、线索学习(cued learning)等多个领域的研究均发现基因的直接效应可能是随年龄变化的函数。譬如, Hilt等(2007)发现Val66Met多态性的ValVal基因型与青少年期起始的抑郁和更高的反思有关, 而ValMet基因型与成年期起始的抑郁有关。Casey等(2009)发现在童年期, 相比ValVal基因型携带者, Met等位基因携带者对条件刺激具有更长的反应时(线索学习的测量指标), 但在青少年时期, 两种基因型间的反应时差异消失。
据我们所知, 目前尚未有研究直接考察基因×环境对抑郁的年龄差异, 但是有关大脑皮质体积的研究为该年龄差异提供了支持(Casey et al., 2009)。研究显示, 在无压力环境下(控制组), Met等位基因和ValVal基因型携带者间的皮质体积差异随年龄增长逐渐减小, 在BDNF含量达到顶峰时, 其差异基本消失; 但是在压力环境下, BDNF在海马等关键脑区的含量降低, 并且使得Met等位基因携带者具有更小的皮质体积, 其与ValVal基因型携带者间的差距随年龄逐渐增大。类似的, Dalton等(2014)关于基因、基因累加遗传得分与家庭环境质量对抑郁的研究发现, 在15岁前, 累加基因得分与家庭环境的交互作用模式符合不同易感性模式; 在15岁后, 累加基因得分与环境的交互模式符合素质−压力模式。这些研究不仅为基因及其与环境交互作用的年龄差异提供了证据, 更重要的是, 这些研究提示青少年期可能是基因效应产生动态变化的关键时期。
关于脑内BDNF浓度的发展研究和表观遗传研究为理解基因效应在青少年期发生动态变化提供了启示。一方面, 在青少年期, 脑内BDNF浓度发生重要变化, 这可能是引发基因效应呈现年龄差异的重要原因。动物研究显示, 脑内BDNF含量呈倒U型发展模式, 在青少年期达到峰值(Ivanova & Beyer, 2001)。关于人类背外侧前额叶的BDNF mRNA含量变化的研究也显示从童年期到青少年期, BDNF表达持续增加(Webster et al., 2002)。Casey等(2009)进一步提示基因与脑内BDNF浓度共同影响BDNF功能。基于此, 在前青少年期, Met等位基因能够加剧较低的BDNF浓度导致的功能失调, 呈现风险性; 但是在青少年期, 脑内BDNF含量的升高反而使得Met等位基因的风险性消失。
另一方面, 青少年的环境经历也发生重要变化(Steinberg, 2022), 不同时期环境经历的差异也会导致遗传效应的动态变化(Kageyama et al., 2022; Suri et al., 2013)。动物研究显示, 不同年龄段的高果糖摄入会导致基因甲基化差异, 仅在童年期和青少年期的高果糖摄入会导致小鼠基因启动子区甲基化增加, 但是在成年早期和晚期小鼠中并未观察到DNA甲基化的变化(Kageyama et al., 2022)。此外, 早期消极经历对小鼠基因甲基化的短期和长期影响亦存在差异, Suri等(2013)的研究显示早期母子分离经历会导致成年早期小鼠的IV启动子区甲基化降低进而增加含量, 但是会增加成年中期小鼠的甲基化。基于动物表观遗传研究结果, 不同时期的环境经历或者早期环境经历对不同发展阶段基因甲基化水平存在差异性影响, 可能在不影响基因型的情况下, 使得基因在不同年龄阶段表现出不同的功能, 进而呈现出年龄差异的特点。
值得指出的是, 青少年早期可能是观察基因功能变化的关键期。一方面, 有关神经生理功能的研究显示, 相比其他发展阶段,基因调节恐惧相关神经环路的关键期是青少年早期(Dincheva et al., 2016), 并且从青少年早期到青少年中期, BDNF含量及其TrkB受体在眶额叶和内侧前额叶的表达水平发生明显的变化(Shapiro et al., 2017)。恐惧神经环路、眶额叶和内侧前额叶等均是与抑郁密切相关的关键脑区(Hare & Duman, 2020; Ho & King, 2021; Subramaniam et al., 2018), 因而青少年早期及其向青少年中期的过渡阶段是观察基因效应变化的关键时期。另一方面, 一项4~15岁的追踪研究发现,基因及其与母亲产前焦虑的交互作用模式对内化问题的影响在4~13岁时相对稳定, 但是在青少年中期(15岁)基因的作用模式与4~13岁时存在差异(O’Donnell et al., 2014)。这也提示,基因在童年期与青少年早期的效应差异可能没有青少年早期与青少年中期的差异显著。基于此, 本研究假设青少年早期是观测基因功能变化的窗口期。
1.3 BDNF基因与环境交互模式的发展动态性:素质−压力、优势敏感与不同易感模型
基因与环境的交互作用模式可能被三种理论模型所解释:“素质−压力” (diathesis-stress)、“优势敏感” (vantage sensitivity)与“不同易感性” (differential susceptibility)模型。“素质−压力”模型指出携带易感基因的个体对消极环境更加敏感, 因而在经历不利环境时才会产生心理或行为问题(Monroe & Simons, 1991); “优势敏感”模型则认为携带易感基因的个体对积极环境更加敏感, 在支持性环境中则会发展地更好(Pluess & Belsky, 2013); “不同易感性”模型则是前两种模型的整合, 认为所谓的易感基因事实上是一种遗传可塑性, 更具可塑性的个体对积极和消极环境均更加敏感, 因而相比不携带可塑性基因的个体, 具有可塑性的个体在积极环境中发展“更好”, 但在消极环境中发展“更坏” (Belsky et al., 2007)。
然而, 这三种理论模型并非相互对立。近期, 研究者指出个体的环境敏感性会受早期环境经历影响而产生变化, 携带环境敏感性基因的个体在生命早期对积极和消极环境均具有敏感性(即不同易感性模式), 但当这些个体在偏积极环境中成长时, 其对积极环境的敏感性则更显著(即优势敏感模式); 当其在偏消极环境中成长时, 则对消极环境的敏感性更强(即素质−压力模式; Pluess, 2015)。由此, 基因与环境的交互作用模式可能也会随个体发展而动态变化, 其变化模式取决于个体的环境经历。如前所述, Dalton等(2014)研究显示在15岁前后, 基因−环境交互作用从不同易感性模式向素质−压力模式变化。因此, 本研究在考察基因与环境交互作用年龄差异的基础上, 进一步检验其交互作用模式随年龄的动态变化。
综上, 青少年期的个体面临一系列生理、环境经历的变化, 这不仅增加了个体的抑郁易感性, 并且可能使得基因及其与环境交互作用模式呈现随年龄的动态变化。本研究目的之一是追踪童年期向青少年期过渡的个体, 考察基因与同伴接纳、同伴拒绝对青少年抑郁影响的年龄差异。基于脑内BDNF含量的倒U型发展模式, 本研究假设在青少年早期, Met等位基因对抑郁的易感性可能逐渐减弱, 而Val等位基因的易感性逐渐显现, 且其交互作用模式会随年龄产生动态变化。
1.4 BDNF基因与环境因素对青少年抑郁发展轨迹的影响
青少年早期不仅是遗传效应动态变化的时期, 也是青少年抑郁发展的关键期。纵向研究显示, 抑郁发端于童年期, 但是直到青少年早期(12岁左右), 抑郁发病率才开始迅速上升, 并在青少年中期(15岁左右)达到顶峰(Avenevoli et al., 2015; Hankin et al., 2015)。由此可见, 遗传效应动态变化与抑郁发展变化的关键期相重叠, 这提示青少年期遗传效应动态变化可能与抑郁的发展轨迹密切相关。
已有研究显示, 遗传基因及其与环境的交互作用既影响青少年抑郁的初始水平, 也可以解释抑郁、孤独感等情绪障碍随年龄的发展变化趋势(如Nelemans et al., 2021; van Roekel et al., 2011)。譬如, Nelemans等(2021)发现重性抑郁风险基因累加得分与父母批评交互影响青少年抑郁的初始水平, 也预测抑郁增长速度。具体表现为, 携带更多风险基因并且经历更多父母批评的青少年不仅具有更高的抑郁症状, 并且其抑郁增长速度更快。此外, Petersen等(2012)发现低转录效能基因型携带者比高转录效能基因型携带者在16~17岁时具有更高的抑郁加速发展模式, 而与其他年龄段的抑郁发展速度和加速度无关。据我们所知, 目前仅有一项研究考察基因与青少年孤独感发展轨迹的关系(Verhagen et al., 2014)。追踪研究发现, 孤独感不仅能够预测个体的抑郁风险, 并且与抑郁具有相似的易感因素(Lim et al., 2020), 由此可推测基因及其与环境的交互作用亦可能解释抑郁发展轨迹。
发展级联模型(developmental cascades)能够为理解基因与环境交互影响抑郁发展轨迹提供启示。发展级联模型指出, 多种发展系统间存在跨时间的交互作用和相互作用, 随着年龄的增长, 不同水平(如生理水平和行为水平)、同水平不同领域(如认知领域和社会性领域)以及不同系统(如家庭系统和同伴系统)或世代(如父母和子女)的心理社会因素间发生的多重相互作用并产生累积效应, 引起跨水平、跨领域甚至跨系统的扩散效应(Masten & Cicchetti, 2010)。根据发展级联模型, 在生命早期发展阶段, 携带易感基因型的个体会具有更高的抑郁初始水平(如Hilt et al., 2007); 其高水平的抑郁症状将进一步唤起其他影响情绪发展的消极经历, 如同伴侵害、同伴拒绝等(Beeson et al., 2020); 消极环境经历不仅会直接增加个体的抑郁症状, 并且会继续增加个体对消极环境的生物敏感性(Pluess, 2015), 个体在后续发展过程中则更易受到不利环境的消极影响, 增加抑郁的风险。由此, 随着时间的推移, 具有遗传易感性的个体在发展过程中抑郁的多种风险因素不断累积, 其与不携带遗传易感性的个体在消极经历、消极环境敏感性及抑郁水平间的差异也越来越大, 进而表现出更高的抑郁增长速度。
基于此,基因及其与环境的交互作用不仅会影响青少年抑郁的初始水平, 并且影响抑郁的发展速度。本研究目的之二是考察基因与同伴关系对青少年抑郁发展轨迹的影响。基于发展级联模型, 本研究假设, 携带风险基因的个体不仅具有更高的抑郁初始水平, 并且具有更高的抑郁增长速度, 且该差异在个体经历高风险环境时进一步扩大。
总体来说, 本研究基于动态发展视角, 考察基因与同伴接纳、同伴拒绝对青少年抑郁的动态影响, 主要探讨以下问题:(1)基因与同伴关系对青少年抑郁影响的年龄差异模式; (2)基因及其与同伴关系的交互作用对青少年抑郁初始水平和发展速度的影响。
2 研究方法
2.1 被试
本研究为国内一项儿童青少年发展大型追踪项目的子研究。由于本研究关注青少年早期这一遗传效应动态变化的关键阶段, 基于Steinberg (2022)对青少年期各阶段的划分, 选取该追踪项目中的青少年早期及其向青少年中期过渡的3个时间点(12~14岁)。被试为在山东省济南市14所小学整群抽取的六年级学生1090名, 在每年春季学期进行一次问卷测查, 连续追踪3年, 其中青少年抑郁和同伴关系在3个时间点均进行测评, 在第三年采集被试唾液样本进行基因分型。其中, 4人DNA含量过低无法分型而缺失基因数据。在1086名具有基因数据的被试中, 被试平均年龄12.32 ± 0.50岁, 男生和女生各半, 每年因转学、请假等流失被试分别为12人、8人和34人, Little’s MCAR检验不显著(χ2= 58.91,= 49,= 0.16), 且流失被试和具有全部数据的被试在性别(χ2= 3.61,= 1,= 0.06)、年龄((1063) = 0.27,= 0.78)、民族(χ2= 0.20,= 1,= 0.65)、基因型(χ2= 2.85,= 2,= 0.24)等人口学变量上亦不存在显著差异, 表明被试为随机缺失。
2.2 研究工具
2.2.1 抑郁
采用儿童抑郁量表(Children’s Depression Inventory, CDI; Kovacs, 1992)测量被试抑郁症状。该量表共包含27个项目, 要求被试报告近两周内的抑郁症状, 分别采用0、1、2记分, 均分越高则表明青少年抑郁症状越多。该量表在正常青少年群体中应用广泛(Zhang et al., 2015), 具有良好的测量学特性。本研究中3个时间点抑郁的Cronbach’s α系数均为0.88。
2.2.2 同伴关系
采用同伴提名法测评青少年的同伴拒绝和同伴接纳。要求被试写出班里最不喜欢的三位同学(消极提名)和最喜欢的三位同学(积极提名)。对每个被试的被提名次数进行班级内标准化, 以便于不同班级青少年提名分数的比较。消极提名的标准分是同伴拒绝的指标, 积极提名的标准分是同伴接纳的指标。
2.2.3 DNA提取与分型
采用Sequenom (San Diego, CA, USA)芯片基质辅助激光解吸/电离飞行时间(MALDI-TOF)质谱平台从唾液样本中提取DNA, 并对基因rs6265多态性位点进行基因分型。PCR引物为:forward 5’-TCAAGAGGCTTGACATCATTGG-3’; reverse5’-GCCGAACTTTCTGGTCCTCAT-3’。PCR反应条件:94°C 15 min; 94°C 20 s, 56°C 30 s, 72°C 1 min, 共45个循环; 最终72°C 3 min。随后经历单碱基延伸反应, 基因分型采用MassARRAY Typer 3.4软件系统进行分析。本研究所使用的检测平台和技术具有较高的可靠性(基因分型有效率 > 97%)。
2.3 研究程序
研究经过山东师范大学伦理委员会审核批准。首先, 项目组将问卷测查、唾液样品采集、DNA提取与分型流程等信息告知施测学校负责人、监护人和青少年, 获得三方的知情同意后进行了数据收集。其次, 以班级为单位, 由经过严格培训的教师和研究生担任主试, 采用现场问卷调查的方式测评青少年抑郁症状、同伴拒绝和同伴接纳, 施测结束后问卷当场收回。第三, 在第三次问卷测评结束后一周内以班级为单位采集被试唾液样本, 每人采集2 ml以上。
2.4 数据处理与分析
本研究中同伴关系(同伴接纳和同伴拒绝)与青少年抑郁在T1~T3三个时间点上进行追踪测评, 在T3时, 进行被试唾液采集并对基因进行分型。基于纵向数据, 本研究统计分析如下:
第一, 对T1~T3的同伴接纳、同伴拒绝和青少年抑郁进行描述统计、相关分析, 并对基因进行Hardy-Weinberg平衡检验。后续分析中对基因型进行虚拟编码, 以ValMet为参照组,1 = MetMet vs. ValMet,2 = ValVal vs. ValMet。
第二, 采用SPSS 23.0进行分层线性回归分析。分别以T1~T3的抑郁为因变量, 以性别为控制变量, 以基因型(以ValMet基因型为参照组形成的两个虚拟变量)、各时间点的同伴关系、基因型与各时间点同伴关系的交互项为预测变量, 在3个时间点考察基因与同伴关系对青少年抑郁的同时性影响并比较3个时间点上结果的差异, 若任一时间点的基因和环境的交互项显著, 则通过简单斜率检验进一步分析其交互作用模式。为进一步检验本研究发现的年龄差异效应, 参照Odgers等(2008)研究, 合并3个时间点的数据, 将时间点作为预测变量(T1 = 0, T2 = 1, T3 = 2), 考察基因、环境与时间点的三者交互作用。
第三, 采用再参数化回归(Widaman et al., 2012)检验基因与同伴关系的交互作用符合何种理论模型(“不同易感性”、“素质−压力”和“优势敏感”模型)。再参数化回归公式如下, D1代表ValMet基因型(1 = ValMet, 0 = MetMet和ValVal), D2代表MetMet基因型(1 = MetMet, 0 = ValMet和ValVal), D3代表ValVal基因型(1 = ValVal, 0 = MetMet和ValMet),代表同伴关系,代表性别,代表交叉点,代表截距,1、2、3分别代表三种基因型中同伴关系的回归系数,代表性别的回归系数。
第四, 采用Mplus 6.0, 构建无条件潜变量增长模型(Latent Growth Curve Modeling, LGCM)分析抑郁的一般发展趋势以及抑郁起始水平和发展速度是否存在显著的个体差异。
第五, 在无条件模型(Model 1)基础上, 逐次增加性别(Model 2)、基因型(Model 3)、T1同伴关系(Model 4)、基因与T1同伴关系的交互项(Model 5)为预测变量, 考察其对抑郁发展截距和斜率的预测作用。上述潜变量增长模型采用全息最大似然估计, 并分别构建同伴拒绝和同伴接纳模型, 以区分两者的差异。
第六, 进行敏感性分析, 检验结果的可靠性。其一, 采用同伴地位(同伴接纳−同伴拒绝)为环境指标重复上述检验。其二, 采用平行潜变量增长模型, 考察同伴关系的变化与青少年抑郁变化间的关系。
3 结果
3.1 描述统计
各变量均值、标准差和相关系数见表1。青少年抑郁呈上升趋势, 青少年抑郁与同伴接纳在不同时间点均呈显著负相关, 青少年抑郁与同伴拒绝在不同时间点均呈显著正相关, 抑郁、同伴接纳和同伴拒绝均具有中等程度的稳定性。重复测量方差分析显示, 青少年抑郁随年龄呈增长趋势((2, 1036) = 68.80,< 0.001), 同伴拒绝呈下降趋势((2, 1081) = 4.43,= 0.01), 但同伴接纳保持稳定((2, 1081) = 1.04,= 0.35)。独立样本检验显示, 在3个时间点上, 女生均具有更低的同伴拒绝水平(s (1083) ≥4.52,s < 0.001), 但在抑郁和同伴接纳水平上不存在性别差异(s (1072) ≤ 1.74,s ≥ 0.08)。基因型分布为MetMet (252人, 23.2%), ValMet (533人, 49.1%), ValVal (301人, 27.7%); 基因型分布符合Hardy-Weinberg平衡(χ2= 0.29,= 2,= 0.59)。方差分析显示, 不同基因型在各时间点同伴接纳、拒绝水平上均无显著差异(s (2, 1081)≤ 2.40,s ≥0.09)。
3.2 BDNF基因与同伴关系对青少年抑郁的交互作用及其年龄差异
以性别、基因、同伴关系以及基因与同伴关系交互项为预测变量, 分别对T1~T3的抑郁进行同时性预测(见表2和表3), 以检验基因与环境交互作用的年龄差异。结果显示, 在3个时间点上,基因型的主效应均不显著, 同伴拒绝均能显著正向预测青少年抑郁。基因型与同伴拒绝的交互作用对青少年抑郁的同时性影响均显著, 但是其交互作用模式存在差异。T1 (12岁)时, 相比ValMet基因型, 在MetMet基因型携带者中, 同伴拒绝对青少年抑郁的预测力更强; T2 (13岁)时, 相比ValMet基因型, 在MetMet和ValVal基因型携带者中, 同伴拒绝对青少年抑郁的预测力更强; T3 (14岁)时, 相比ValMet基因型, 在ValVal基因型携带者中, 同伴拒绝对青少年抑郁的预测力更强。在同伴接纳模型中, 在3个时间点上, 同伴接纳均能显著负向预测青少年抑郁, 但是基因型的主效应及其与同伴接纳的交互作用均不显著。

表1 描述统计与相关系数表
注:*< 0.05;**< 0.01;***< 0.001。

表2 BDNF基因与同伴拒绝对青少年抑郁的交互作用(T1~T3)
注:采用Benjamini–Hochberg校正后显著的结果加粗显示;进行虚拟编码, 以ValMet为参照组,1 = MetMet vs. ValMet,2 = ValVal vs. ValMet;+< 0.1;*< 0.05;**< 0.01;***< 0.001。

表3 BDNF基因与同伴接纳对青少年抑郁的交互作用(T1~T3)
注:采用Benjamini–Hochberg校正后显著的结果加粗显示;进行虚拟编码, 以ValMet为参照组, BDNF 1 = MetMet vs. ValMet, BDNF 2 = ValVal vs. ValMet;+< 0.1;*< 0.05;**< 0.01;***< 0.001。
进一步的简单斜率检验显示(见图1), T1时, 在MetMet (= 0.06,= 3.96,< 0.001)和ValVal基因型(=0.03,= 2.48,= 0.01)携带者中, 同伴拒绝显著正向预测青少年抑郁, 但在ValMet基因型中, 同伴拒绝对青少年抑郁的预测作用不显著(= 0.01,= 1.16,= 0.24)。T2时, 在MetMet (= 0.09,= 5.45,< 0.001)、ValVal (= 0.06,= 4.52,< 0.001)和ValMet基因型(= 0.02,= 2.06,= 0.04)携带者中, 同伴拒绝均显著正向预测青少年抑郁。T3时, 在MetMet (= 0.04,= 2.34,= 0.02)和ValVal基因型(= 0.07,= 4.08,< 0.001)携带者中, 同伴拒绝显著正向预测青少年抑郁, 但在ValMet基因型中, 同伴拒绝对青少年抑郁的预测作用不显著(= 0.004,= 0.41,= 0.68)。
同时性回归分析显示,基因与同伴拒绝交互作用模式在不同的年龄存在差异, 并且从简单斜率检验可知12~14岁, ValVal基因型和MetMet基因型对环境的敏感性存在较大的变化, 为进一步比较MetMet和ValVal对环境的敏感性差异随时间的变化。本研究以MetMet为参照组重编码基因, 参照Odgers等(2008)研究, 将时间点作为预测变量, 考察基因、环境与时间点的三者交互作用。结果显示,基因型(ValVal vs. MetMet(参照组))、同伴拒绝与时间点的三者交互作用显著(= 0.03,2.010.045), 这提示ValVal和MetMet基因型对环境的敏感性差异随年龄而变化。但是基因型(MetMet vs. ValMet(参照组))、同伴拒绝与时间点的三者交互(= −0.01,−0.810.42),基因型(ValVal vs. ValMet(参照组))、同伴拒绝与时间点的三者交互(= 0.02,1.580.11)未达到显著水平。
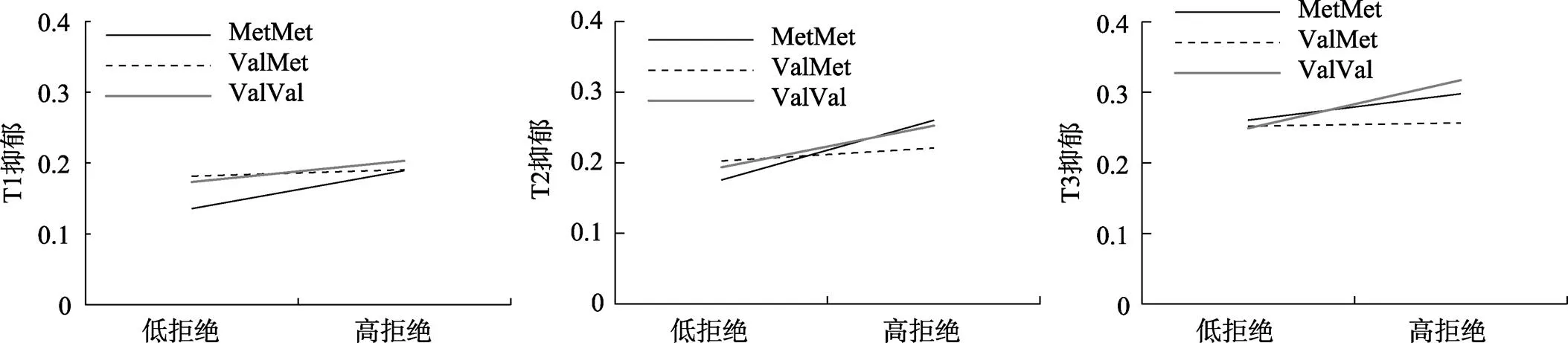
图1 BDNF基因与同伴拒绝对青少年抑郁的交互作用图(T1~T3)
3.3 基因与环境交互模式检验:素质−压力、优势敏感与不同易感模型
基于回归分析结果, 采用再参数化回归检验基因与同伴拒绝的交互作用符合何种理论模型。T1同伴拒绝与基因对抑郁的再参数化分析(表4)显示, 交叉点C及其95% CI均在同伴拒绝全距内([−0.74, 6.74]), 限定ValMet回归系数为0后, Model a与Model b在解释率上不存在显著差异, 且Model a的AIC和BIC值最小, 强不同易感模型为最优模型。T2同伴拒绝与基因对抑郁的再参数化分析(表5)显示, 交叉点C及其95% CI均在同伴拒绝全距内([−1.01, 6.01]), 限定ValMet基因型回归系数为0后, Model a与Model b在解释率上存在显著差异, 且Model 1的AIC值最小, 弱不同易感模型为最优模型。T3同伴拒绝与基因对抑郁的再参数化分析(表6)显示, 交叉点C及其95% CI超出同伴拒绝全距内([−0.80, 6.04]), 该基因−环境交互不符合不同易感模型, 优势敏感模型解释率与不同易感模型不存在显著差异, 且Model e的AIC和BIC更小, 强优势敏感模型为最优模型。

表4 BDNF基因与同伴拒绝对青少年抑郁的再参数化分析(T1)
注:*< 0.05;**< 0.01;***< 0.001。
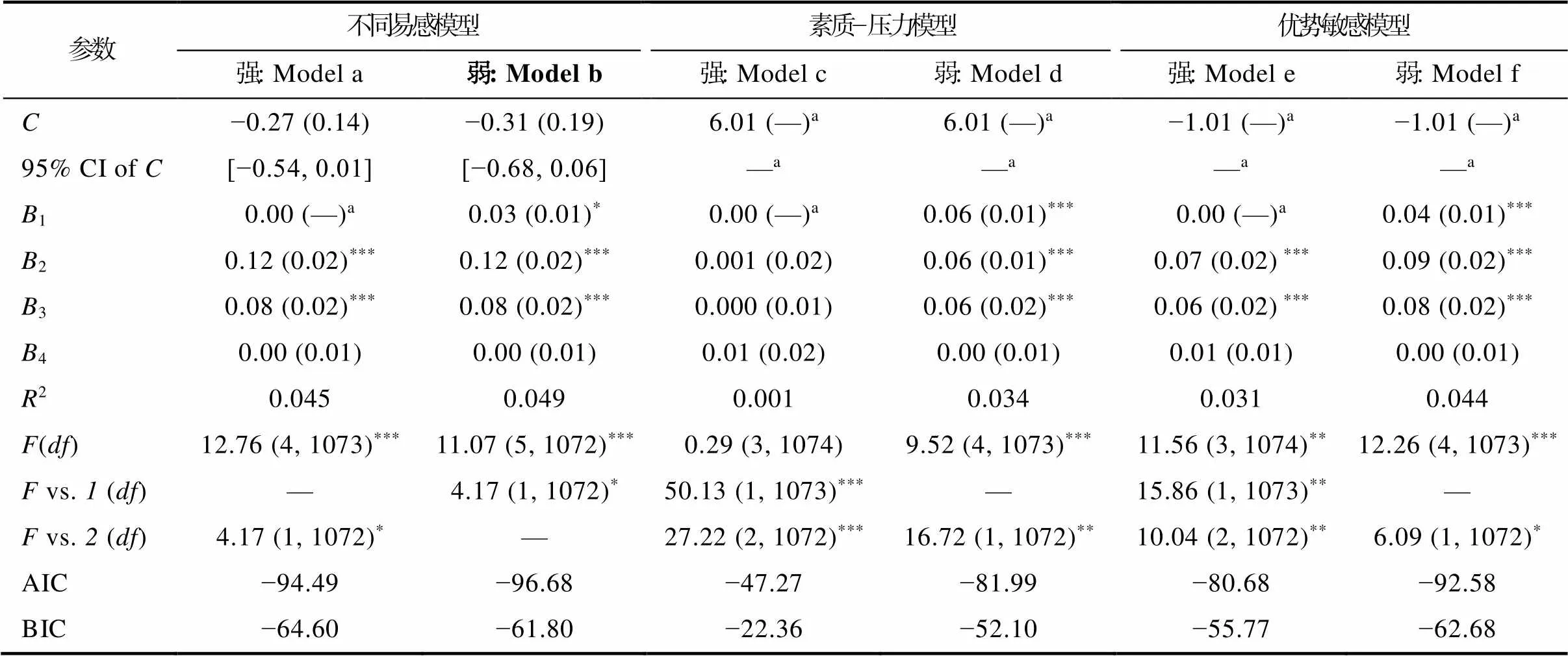
表5 BDNF基因与同伴拒绝对青少年抑郁的再参数化分析(T2)
注:*< 0.05;**< 0.01;***< 0.001。
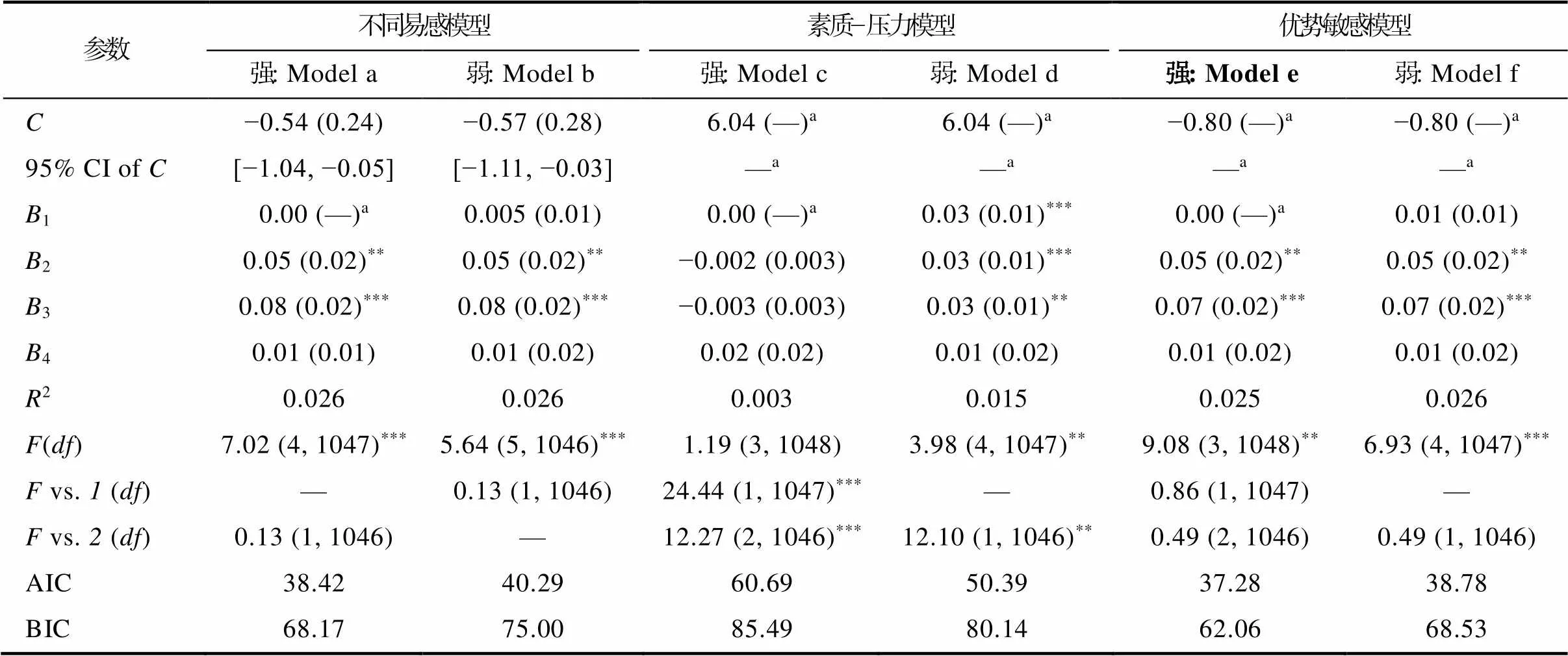
表6 BDNF基因与同伴拒绝对青少年抑郁的再参数化分析(T3)
注:*< 0.05;**< 0.01;***< 0.001。
3.4 抑郁的发展趋势:BDNF基因与同伴关系的交互作用
首先, 对青少年抑郁构建无条件潜变量增长模型, 如表7所示, 无条件模型拟合良好。从12至14岁, 青少年抑郁呈线性增长趋势, 截距(σ2= 0.03,< 0.001)和斜率(σ2= 0.01,< 0.001)的变异均呈现出明显的个体间差异。截距和斜率之间的相关不显著(= −0.16,= 0.053), 表明抑郁增长速度与初始水平无关。
在无条件模型基础上, 以截距和斜率为结果变量, 逐次增加性别(Model 2)、基因型(Model 3)、T1同伴关系(Model 4),基因与T1同伴关系的交互项(Model 5)等预测变量, 建立条件模型。结果显示,1 (MetMet vs. ValMet)和2 (ValVal vs. ValMet)对斜率的预测作用显著, 相比ValMet基因型, Met和Val纯合子携带者的抑郁增长速度更快(见图2)。
如表7和图3所示, 同伴拒绝模型(Model 5a)拟合良好,1与T1拒绝的交互项对截距的预测显著。如表7和图4所示, 同伴接纳模型(Model 5b)拟合良好, 但该模型未发现基因与接纳交互作用对截距或斜率具有显著预测作用。此外, 多群组比较显示, 条件模型无性别差异(Δχ2= 22.03, Δ=15,= 0.12)。

表7 BDNF基因与同伴关系对青少年抑郁发展轨迹的影响
注:在所有条件模型中均控制了性别, 表格中仅呈现了每个模型中新增变量的参数。进行虚拟编码, 以ValMet为参照组,1 = MetMet vs. ValMet,2 = ValVal vs. ValMet; 表中所有参数估计值均为标准化结果。
*< 0.05;**< 0.01;***< 0.001。

图2 不同BDNF基因型青少年抑郁的发展变化

图3 BDNF基因与同伴拒绝对青少年早期抑郁发展模式的影响(Model 5a)

图4 BDNF基因与同伴接纳对青少年早期抑郁发展模式的影响(Model 5b)
3.5 敏感性分析
第一, 采用同伴地位(同伴接纳 −同伴拒绝)为环境指标重复上述检验。同时性回归分析(见网络版附录表A1)显示, 在3个时间点上,基因型与同伴地位的交互作用对青少年抑郁的同时性影响均显著。12~13岁时, 相比ValMet基因型, 在MetMet基因型携带者中, 同伴拒绝对青少年抑郁的预测力更强; 14岁时, 相比ValMet基因型, 在ValVal基因型携带者中, 同伴拒绝对青少年抑郁的预测力更强。潜变量增长模型(见表7)显示, 同伴地位模型(Model 5c)拟合良好, 仅1与T1同伴地位的交互项对截距的预测显著。在T1~T2时,基因与同伴地位的交互作用更符合不同易感性模型, 而在T3时, 优势敏感模型能够很好地解释基因×环境交互作用模式(见网络版附录表A2~ A4)。
第二, 采用平行潜变量增长模型, 考察同伴关系的变化与青少年抑郁变化间的关系。结果显示(见网络版附录表A5), 同伴拒绝、同伴接纳和同伴地位的初始水平均显著预测抑郁的初始水平, 而非抑郁的增长速度, 同伴关系的增长速度与青少年抑郁的增长速度无关。多群组比较分析显示, 不同基因型携带者仅在同伴拒绝初始水平到青少年抑郁初始水平这一路径上存在显著差异(Δχ2= 16.01, Δ=2,< 0.001), 与上述分析结果相一致。
4 讨论
本研究基于动态发展视角, 考察了基因与同伴关系对青少年抑郁交互作用的年龄差异, 以及该基因及其与环境交互作用对青少年抑郁一般发展轨迹的影响。结果显示, 12~14岁,基因与同伴拒绝的交互作用均显著同时性预测青少年抑郁, 但是其交互作用模式存在年龄差异。具体表现为, 12岁时, MetMet基因型携带者对环境敏感性高于ValMet基因型携带者; 13岁时, MetMet和ValVal基因型携带者对环境的敏感性均高于ValMet基因型携带者; 14岁时, ValVal基因型携带者对环境的敏感性高于ValMet基因型携带者。此外,基因与同伴拒绝交互预测青少年抑郁的初始水平, 相比ValMet基因型携带者, 携带MetMet基因型的青少年在经历同伴拒绝后抑郁初始水平更高;基因显著预测青少年抑郁的增长速度, 相比ValMet基因型, MetMet和ValVal基因型携带者的抑郁增长速度更快。
各时间点的同时性回归分析均发现基因与同伴拒绝交互预测青少年抑郁, 携带易感基因的青少年在经历同伴拒绝时报告了更多抑郁症状。在12~14岁, 个体对环境的易感基因型从MetMet基因型逐渐转变为ValVal基因型。该研究结果能够为以往研究在基因与环境交互结果上的分歧提供启示。如前所述, 以成年人或儿童为被试的研究显示Met等位基因携带者具有更高的抑郁水平并且对环境更加敏感(Aguilera et al., 2009; Meyer et al., 2018), 而以青少年为被试的研究却显示Val等位基因携带者表现出对环境更高的敏感性(Chen et al., 2013)。针对该分歧, 已有研究经常提及但鲜少直接检验基因效应的年龄差异(Casey et al., 2009), 本研究则为这一发展动态性模式提供了支持。此外, 除基因外, 已有关于(Hankin et al., 2015)、基因(Zhang et al., 2015)的研究亦发现了类似的年龄差异。譬如, Zhang等(2015)的研究考察了基因与母亲教养行为影响抑郁的年龄差异, 在个体11和12岁时, 发现携带A1等位基因的个体对消极教养行为更加敏感, 但在13岁时, 该基因与教养的交互作用不显著。这提示基因及其与环境交互作用的发展动态性并不罕见, 尤其是在发展过渡的关键阶段(如本研究的青少年早期)更容易观测到基因及其与环境交互作用的变化。但是, 本研究基于较短的年龄跨度且在没有外部验证样本的情况下, 所观测到基因与同伴拒绝交互作用随时间的变化仍可能是一种假阳性结果, 未来仍需要大量研究重复验证该结果的可靠性。
如前所述,基因及其与环境交互作用随年龄的动态变化既可能与含量的发展变化有关, 也可能与环境因素的动态变化有关。第一, 来自动物和人类研究的发现均显示, 从童年期到青少年期,浓度呈增长趋势(如Ivanova & Beyer, 2001; Webster et al., 2002)。因而, 不同年龄阶段基底浓度使得基因功能存在动态变化。在童年晚期或前青少年期, 基底浓度较低, 高活性Val等位基因能够缓冲低浓度的风险性, 而低活性Met等位基因则表现出风险性; 而随着年龄增长基底含量上升, 使得Met与Val基因型在含量上的差异被掩盖, 因而低活性Met等位基因的风险性逐渐减弱并消失。更重要的是, 除了Met等位基因的风险性随年龄增长而消失, 本研究发现Val等位基因风险性逐渐显现出来。这提示功能与抑郁间的关系可能呈现非线性趋势。换言之, 过高或过低的功能均可能增加抑郁的风险性。因而, 不能仅仅依据基因活性判断等位基因是“好”或“坏”, 还需要考虑基因在BDNF功能与抑郁U型函数上所处的位置。第二, 在童年期和青少年期, 压力易感的神经生理机制差异亦可能影响基因与环境交互作用的年龄差异。研究显示, 海马区发育成熟时间早于杏仁核(Andersen & Teicher, 2008), 因而童年期海马对压力更加敏感, 青少年期压力易感脑区是杏仁核(Lupien et al., 2009)。暴露于压力环境会导致海马区BDNF含量降低, 而杏仁核BDNF含量上升(Chang et al., 2021)。更重要的是,在海马和杏仁核的功能截然相反, 海马区较低的BDNF含量与较高的抑郁水平有关(Duman & Monteggia, 2006), 而较高的BDNF含量会导致较大的杏仁核体积进而增加抑郁风险(Thomas et al., 2001)。基于此, 可推测在童年期, 低活性Met等位基因与压力共同导致海马体更低的BDNF含量, 导致抑郁风险增加; 而在青少年期, 高活性Val等位基因与压力共同导致杏仁核更高的含量, 增加抑郁风险。第三, 本研究被试处于“小升初”的生态过渡期, 青少年面临着从熟悉同伴群体过渡到陌生同伴群体, 这也是个体同伴地位重组的关键时期。本研究也发现随着年龄增长, 青少年同伴拒绝水平呈现下降趋势, 如前所述环境变化能够改变甲基化水平(Kageyama et al., 2022), 由此可推测环境变化引起的易感基因DNA甲基化会使该基因失效从而掩盖风险等位基因的消极影响, 呈现出基因与同伴拒绝交互作用的动态变化。
基因−环境交互作用的发展动态性不仅体现在易感基因型的变化上, 也体现在其基因与环境交互作用模式的变化上。再参数化分析显示, 在12~13岁时,基因与同伴拒绝的交互作用模式更符合不同易感模型, 即携带易感基因型(ValVal和MetMet基因型)的个体对积极和消极环境均更加敏感, 而表现出更低或更高的抑郁水平。在14岁时, 相比不同易感模型, 优势敏感模型能够更好的解释基因与同伴拒绝的交互模式。如前所述, 个体的环境敏感性会受早期环境经历影响而产生动态变化, 携带环境敏感性基因的个体在生命早期对积极和消极环境均具有敏感性(即不同易感性模式), 但当这些个体在积极或消极环境中成长时, 则其环境敏感性则会向优势敏感或素质压力模式发展(Pluess, 2015)。基于该理论假设, 本研究发现青少年同伴拒绝水平随年龄逐渐降低, 而同伴接纳水平保持稳定, 即个体的积极同伴经历更为显著, 因而在相对积极环境中成长起来的青少年, 其基因−环境交互作用模式呈现从不同易感性向优势敏感性的转变。值得指出的是,基因与同伴拒绝而非同伴接纳存在交互作用, 该基因−环境交互作用模式的转变可能受到同伴拒绝有限取值范围的影响, 因而本研究补充分析了同伴地位(同伴社会偏好)的结果, 亦发现14岁时优势敏感模型能够很好地解释基因−环境交互模式。这一结果也提示早期的环境经历可能会影响个体对未来环境的反应, 但是由于本研究仅在消极环境中发现了个体的环境敏感性从不同易感模式向优势敏感模型的变化, 应该对本研究结果保持谨慎, 未来研究应该进一步关注个体环境敏感性的发展变化, 尤其是早期环境经历对个体环境敏感性的塑造作用。
无论是易感基因型还是基因−环境交互模式随年龄的变化均为“天性与教养的动态交互观” (Lerner, 1987)和“基因−环境−时间”交互理论框架(Boyce et al., 2020, 2021)提供了支持。尽管在个体发展过程中DNA序列不会发生变化, 但是遗传基因的效应并非是静态的, 生理成熟因素、环境经历等均会通过复杂的相互作用机制影响基因功能的表达, 并且提示未来研究应该深入考察基因−环境的动态交互的基础理论问题。首先, 本研究仅采用年龄作为时间(发展)因素, 但是发展时程(timing, 如青春期发展阶段)、神经环路发展关键期(不同脑区的发展关键期存在差异)、世代变迁(如代际变迁、进化)等均是揭示基因−环境的动态变化的时间因素(Boyce et al., 2020)。不同类型的时间因素可能揭示了不同的动态发展机制, 譬如, 环境因素(如营养和社会情绪因素)会延长或缩短特定发展阶段(如青春期)所需的时间(Ellis et al., 2011), 使得发育阶段和年龄无关, 因而产生不同的基因−环境−时间交互模式。与此一致, Hankin等(2015)有关青少年抑郁的研究发现基因−环境−年龄存在交互作用, 但青春期发展阶段对基因−环境交互无调节作用。由此, 未来研究应该进一步扩展对时间(发展)因素的探讨以更全面描述“基因−环境−时间”交互理论框架。其次, 探索基因与环境交互作用在时间轴上的动态展开机制是未来研究的重点问题。根据Cole (2009)的递归发展(recursive development)模型, 表观遗传过程可能是解释遗传−环境交互效应发展动态性的关键机制。该模型指出某一时间点的环境因素可以通过中枢神经系统影响个体的行为表型, 并通过表观遗传机制影响RNA转录进而塑造出下一个时间点新的神经反应功能, 新塑造的神经反应功能对环境因素的反应则会与上一时间点存在差异, 进而导致新的行为表型和新的RNA转录过程, 以此循环递归产生动态变化。事实上, 这一模型与发展级联模型的观点相似, 均提示不同水平的影响因素跨时间的相互作用是解释个体动态发展的关键机制, 但该模型更明确的指出基因表达机制在其中的作用。由此, 未来研究应该在发展级联模型或递归发展模型框架下, 采用表观遗传研究范式, 在基因表达水平上, 揭示遗传−环境交互作用动态变化的潜在机制。
本研究并未发现基因与同伴接纳间存在显著的交互作用, 这可能与社会接纳和拒绝在脑内产生的不同影响有关。研究显示, 社会接纳会导致伏隔核的内源性阿片释放增加, 而社会拒绝则会导致调节压力、情绪和动机脑区的内源性阿片释放降低(Hsu et al., 2015)。同时, 动物研究显示注射δ-阿片受体激动剂(其效应类似于阿片含量增加)的大鼠额叶皮质、海马和基底外侧杏仁核中mRNA表达增加(Torregrossa et al., 2004)。由此, 可推测同伴接纳会增加内源性阿片含量, 通过增加mRNA表达, 提高脑内浓度而产生“天花板效应”, 使得Val和Met等位基因间的差异消失, 因而无法观察到基因−环境交互作用。此外, 这也可能与青少年对消极环境的敏感性高于积极环境有关。研究发现, 在9~17岁儿童青少年中, 面对同伴拒绝时个体瞳孔扩张反应(反映情绪调节脑区活动程度的指标)比面对同伴接纳时更大, 并且该差异随年龄增长而增加(Silk et al., 2012)。
与以往研究结果(Avenevoli et al., 2015)一致, 无条件模型显示青少年早期抑郁呈线性增长趋势。该时期抑郁的迅速增加可能源于青少年早期个体面临一系列生理、认知、社会角色过渡, 不仅使青少年具有更高的压力易感性, 并且伴随着压力性经历增多, 加剧了青少年罹患抑郁的风险(Hyde & Mezulis, 2020)。此外, 本研究还发现抑郁的初始水平与其发展速度并无显著相关。这提示, 无论个体进入青春期时的初始抑郁水平高还是低, 均可能具有更高或更低的抑郁增长速度。因此, 初始抑郁水平较低并不意味着个体具有低抑郁风险, 高抑郁初始水平也可能因保护因素而降低其抑郁的增长速度。更重要的是, 本研究显示青少年抑郁的初始水平和增长速度存在明显的个体差异,基因及其与环境的交互作用部分解释了该个体差异。
本质上, 青少年抑郁初始水平的影响因素与起始时间点的同时性预测因素相一致。即基因型(MetMet vs. ValMet)与同伴拒绝的交互作用显著预测抑郁的初始水平, 但基因主效应与抑郁初始水平无关, 对此结果不再赘述。然而, 与抑郁的初始水平不同,基因能够预测青少年抑郁的发展速度。这提示, 影响抑郁的遗传差异从童年晚期到青少年早期逐渐扩大。这一现象与双生子研究结果相似, 从童年期到青少年期, 抑郁的遗传率逐渐增加(Scourfield et al., 2003)。抑郁的遗传差异随着年龄逐渐扩大或显现的原因可能有两种:第一, 在青少年期出现了导致基因遗传效应增大的新兴神经生理过程或遗传基因表达。如前所述, 在青少年期浓度的新变化会改变基因功能, 使得个体的易感基因发生变化, 进而影响了抑郁的发展速度。这可能是一些发展敏感的“Clock”基因在青春期开始启动(Pickles et al., 1998), 不仅引起BDNF浓度变化导致基因效应差异逐渐扩大, 并且使得在青少年期观察到新兴的遗传效应, 导致抑郁遗传率的增加。第二, 在发展过程中, 生命早期较小的遗传差异会像滚雪球一样越来越大。根据发展级联模型, 在生命早期发展阶段, 个体所携带基因型的差异会导致其对消极环境的敏感性存在差异, 遭遇消极同伴经历会导致携带易感基因的青少年抑郁水平更高, 由此强化或增加了遗传差异。值得指出的是, 基因−环境交互作用并不影响抑郁的发展速度, 而是更多的表现出时间特异性。但是,基因与同伴拒绝交互作用随时间的波动不仅会对青少年抑郁产生暂时性的影响, 亦有可能导致抑郁在总体发展轨迹上出现明显的偏离。同时, 高水平的抑郁症状将进一步唤起其他风险因素, 导致具有遗传易感性的个体在发展过程中抑郁的风险因素不断累积, 其与不携带遗传易感性的个体之间的发展差异也越来越大, 进而表现出更高的抑郁增长速度。
综合回归分析和一般发展轨迹的结果, 本研究发现ValMet基因型呈现出的独特作用模式可能是一种杂合子优势效应(heterosis), 即携带ValMet基因型的个体不仅不易受消极环境的影响, 而且抑郁的增长速度更慢。对于该杂合子优势现象的一种可能解释是,与心理社会适应间呈“倒U型”趋势, 中等水平的含量有助于个体达到最优的心理适应状态, 并且ValMet基因型可能很好地平衡了ValVal和MetMet基因型在杏仁核和海马等脑区的不同功能, 使得个体更具发展优势。关于基因的杂合子优势效应, 已有抗抑郁药物治疗研究为此提供了支持。Tsai等(2003)发现经历4周的氟西汀抗抑郁治疗后, 相比纯合子携带者, ValMet基因型携带者表现出更好的抗抑郁疗效。Yoshida等(2007)也发现无论采用何种抗抑郁药物,G196A基因杂合子携带者均表现出更低的抑郁症状。近年来, 虽然越来越多的研究为Val66Met多态性的杂合子优势效应提供支持, 但是已有研究通常采用将ValMet基因型与MetMet或ValVal基因型进行合并的基因编码方式, 不同的编码方式会影响基因研究结果(Aliev et al., 2014; 张文新等, 2021), 因而可能掩盖了杂合子优势效应(Tsai, 2018)。值得指出的是, 这种杂合子优势效应在遗传研究中并不罕见, 甚至有研究者指出人类50%的基因具有杂合子优势效应(Comings & MacMurray, 2000)。然而, 本研究并未直接对不同基因型的神经生化功能进行直接检测, 仅基于当前观测结果而推测出的杂合子优势效应可能混淆了其他因素的影响。因此未来仍需要对本研究发现的ValMet独特作用模式进一步检验, 验证该效应的可靠性和普遍性。
为了提高基母材之间的焊接强度,通常会采用在其间增加过渡涂层的方法来增加强度,因此针对银铜焊料,选用电镀铜和银来进行基母材的表面处理,镀层厚度10 μm,再制作焊接试样进行试验。不同胎体配方的基母材电镀铜和银的焊接强度汇总表见表9、表10。
本研究具有一定的理论创新性和实践价值。一方面, 本研究突破已有的静态发展观, 考察基因及其与环境交互作用的发展动态性问题, 不仅为“基因−环境−时间”交互理论框架提供了实证支持, 并且提示未来研究应该将研究视角从单纯地将某一等位基因视为“风险基因”转化为意识到遗传基因呈现风险性或保护性依赖于其所处的发展阶段。另一方面, 本研究结果对于青少年抑郁的预防和干预实践具有一定的启示意义。本研究发现, 青少年早期是具有高可塑性的关键期, 而个体的环境经历可以塑造个体对未来环境的反应模式, 积极经历(甚至仅仅是消极环境的减少)能够增加个体对积极环境的敏感性, 使其更容易从积极环境中获益产生更好的发展适应结果, 由此预防和干预应该关注个体环境敏感性发展的关键期, 塑造良好的成长环境以降低青少年抑郁的风险。
本研究具有一些局限性。第一, 由于本研究关注青少年遗传效应变化的关键阶段, 仅选取了青少年早中期的3个时间点进行纵向分析, 不能全面刻画个体抑郁及其遗传效应的动态发展趋势, 尤其是抑郁的二次发展趋势和异质性亚组发展轨迹。同时, 仅关注青少年早中期的3个时间点, 也难以揭示从童年期到青少年期以及青少年期各阶段间的遗传效应的稳定性和差异性等关键问题。因此, 未来研究需要开展更长跨度的追踪研究, 更全面地描绘遗传−环境动态发展模式。第二, 本研究基于候选基因关联范式采用了基因的单个位点进行分析, 未开展多基因研究, 这可能限制了遗传效应的解释率。近年来, 伴随着对单基因和候选基因研究范式的质疑, 多基因风险得分和全基因组关联研究逐渐成为当前研究的主流范式, 但是现有多基因研究在提升遗传解释率方面亦存在不足, 且全基因组关联研究仍然需要通过富集分析确定基因的生物功能或生化通路, 这与候选基因研究基于生化通路确定遗传风险无本质差异。尽管如此, 未来研究仍需采用多基因范式进一步检验抑郁遗传效应的发展动态性问题。第三, 本研究缺乏外部验证样本。近年来, 伴随着基因×环境交互作用研究分歧的增多, 越来越多的研究者对遗传研究的可重复性问题提出了质疑, 并且指出应该采用外部验证样本进行重复检验(Dick et al., 2015)。但是本研究未能具有相同测量工具及相似被试特征的外部验证样本, 其结果的可重复性有待未来研究进行验证。此外, 本研究被试来自城市常态群体青少年, 该研究结果能否适用于临床样本或重性抑郁的青少年群体尚需未来研究的验证。最后, 基于以上局限性, 本研究所观测到的基因×环境交互作用年龄差异和杂合子优势效应虽然有间接的证据支持, 但仍存在假阳性风险, 需要谨慎对待和更多未来研究的支持。
Aguilera, M., Arias, B., Wichers, M., Barrantes-Vidal, N., Moya, J., Villa, H., van Os, J., Ibáñez, M. I., Ruipérez, M. A., Ortet, G., & Fañanás, L. (2009). Early adversity and 5-HTT/BDNF genes: New evidence of gene-environment interactions on depressive symptoms in a general population.,(9), 1425–1432. https://doi.org/10.1017/S0033291709005248
Aliev, F., Latendresse, S. J., Bacanu, S. A., Neale, M. C., & Dick, D. M. (2014). Testing for measured gene-environment interaction: Problems with the use of cross-product terms and a regression model reparameterization solution.,(2), 165–181. https://doi.org/10.1007/s10519- 014-9642-1
Andersen, S. L., & Teicher, M. H. (2008). Stress, sensitive periods and maturational events in adolescent depression.,(4), 183–191. https://doi.org/ 10.1016/j.tins.2008.01.004
Avenevoli, S., Swendsen, J., He, J. P., Burstein, M., & Merikangas, K. R. (2015). Major depression in the national comorbidity survey-adolescent supplement: Prevalence, correlates, and treatment.,(1), 37–44. https:// doi.org/10.1016/j.jaac.2014.10.010
Beeson, C. M., Brittain, H., & Vaillancourt, T. (2020). The temporal precedence of peer rejection, rejection sensitivity, depression, and aggression across adolescence.,(5), 781–791. https:// doi.org/10.1007/s10578-020-01008-2
Belmaker, R. H., & Agam, G. (2008). Mechanisms of disease: Major depressive disorder.(1), 55–68. https://doi.org/10.1056/NEJMra073096
Belsky, J., Bakermans-Kranenburg, M. J., & van Ijzendoorn, M. H. (2007). For better and for worse: Differential susceptibility to environmental influences.,(6), 300–304. https://doi.org/10.1111/j.1467-8721.2007.00525.x
Blackhart, G. C., Eckel, L. A., & Tice, D. M. (2007). Salivary cortisol in response to acute social rejection and acceptance by peers.,(3), 267–276. https:// doi.org/10.1016/j.biopsycho.2007.03.005
Boyce, W. T., Levitt, P., Martinez, F. D., McEwen, B. S., & Shonkoff, J. P. (2021). Genes, environments, and time: the biology of adversity and resilience.,(2), e20201651. https://doi.org/10.1542/peds.2020-1651
Boyce, W. T., Sokolowski, M. B., & Robinson, G. E. (2020). Genes and environments, development, and time.,(38), 23235– 23241. https://doi.org/10.1073/pnas.2016710117
Cao, Y., Chen, G., Ji, L., & Zhang, W. (2021). Inhibitory control mediates the associations between parenting practices and depressive symptoms in adolescents: The moderating role of catechol-O-methyltransferase (COMT) gene.,(10), 2079– 2095. https://doi.org/10.1007/s10964-021-01466-4
Casey, B. J., Glatt, C. E., Tottenham, N., Soliman, F., Bath, K., Amso, D., ... Lee, F. S. (2009). Brain-derived neurotrophic factor as a model system for examining gene by environment interactions across development.,(1), 108–120. https://doi.org/10.1016/ j.neuroscience.2009.03.081
Caspi, A., Sugden, K., Moffitt, T. E., Taylor, A., Craig, I. W., Harrington, H., McClay, J., Mill, J., Martin, J., Braithwaite, A., & Poulton, R. (2003). Influence of life stress on depression: Moderation by a polymorphism in the 5-HTT gene.,(5631), 386–389. https://doi.org/10. 1126/science.1083968
Chang, S. H., Yu, Y. H., He, A., Ou, C. Y., Shyu, B. C., & Huang, A. C. W. (2021). BDNF protein and BDNF mRNA expression of the medial prefrontal cortex, amygdala, and hippocampus during situational reminder in the PTSD animal model.,, 6657716. https://doi.org/10.1155/2021/6657716
Chen, J., Li, X., & McGue, M. (2013). The interacting effect of the BDNF Val66Met polymorphism and stressful life events on adolescent depression is not an artifact of gene- environment correlation: Evidence from a longitudinal twin study.,(10), 1066–1073. https://doi.org/10.1111/jcpp.12099
Cole, S. W. (2009). Social regulation of human gene expression.,(3), 132–137. https://doi.org/10.1111/j.1467-8721.2009.01623.x
Comings, D. E., & MacMurray, J. P. (2000). Molecular heterosis: A review.,(1–2), 19–31. https://doi.org/10.1006/mgme.2000.3015
Dalton, E. D., Hammen, C. L., Najman, J. M., & Brennan, P. A. (2014). Genetic susceptibility to family environment: BDNF Val66met and 5-HTTLPR influence depressive symptoms.,(6), 947–956. https://doi.org/10.1037/fam0000032
Dick, D. M., Agrawal, A., Keller, M. C., Adkins, A., Aliev, F., Monroe, S., Hewitt, J. K., Kendler, K. S., & Sher, K. J. (2015). Candidate gene–environment interaction research: Reflections and recommendations.,(1), 37–59. https://doi.org/10. 1177/1745691614556682
Dincheva, I., Lynch, N. B., & Lee, F. S. (2016). The role of BDNF in the development of fear learning.,(10), 907–916. https://doi.org/10.1002/da.22497
Duman, R. S., & Monteggia, L. M. (2006). A neurotrophic model for stress-related mood disorders.,(12), 1116–1127. https://doi.org/10.1016/j. biopsych.2006.02.013
Ellis, B. J., Shirtcliff, E. A., Boyce, W. T., Deardorff, J., & Essex, M. J. (2011). Quality of early family relationships and the timing and tempo of puberty: Effects depend on biological sensitivity to context.,(1), 85–99. https://doi.org/10.1017/ S0954579410000660
González-Castro, T. B., Nicolini, H., Lanzagorta, N., López-Narváez, L., Genis, A., Pool García, S., & Tovilla-Zárate, C. A. (2015). The role of brain-derived neurotrophic factor (BDNF) Val66Met genetic polymorphism in bipolar disorder: A case-control study, comorbidities, and meta-analysis of 16, 786 subjects.,(1), 27–38. https://doi.org/10.1111/ bdi.12227
Gottfredson, N. C., Foshee, V. A., Ennett, S. T., Haberstick, B., & Smolen, A. (2015). Genetic heterogeneity in adolescents’ depressive symptoms in response to victimization.,(5), 762–774. https://doi.org/10.1080/15374416.2014.910787
Guyer, A. E., Choate, V. R., Pine, D. S., & Nelson, E. E. (2012). Neural circuitry underlying affective response to peer feedback in adolescence.,(1), 81–92. https://doi.org/10. 1093/scan/nsr043
Hankin, B. L., Young, J. F., Abela, J. R. Z., Smolen, A., Jenness, J. L., Gulley, L. D., Technow, J. R., Gottlieb, A. B., Cohen, J. R., & Oppenheimer, C. W. (2015). Depression from childhood into late adolescence: Influence of gender, development, genetic susceptibility, and peer stress.,(4), 803–816. https:// doi.org/10.1037/abn0000089
Hankin, B. L., Young, J. F., Abela, J. R., Smolen, A., Jenness, J. L., Gulley, L. D., Technow, J. R., Gottlieb, A. B., Cohen, J. R., & Oppenheimer, C. W. (2015). Depression from childhood into late adolescence: Influence of gender, development, genetic susceptibility, and peer stress.,(4), 803–816. https:// doi.org/10.1037/abn0000089
Hare, B. D., & Duman, R. S. (2020). Prefrontal cortex circuits in depression and anxiety: Contribution of discrete neuronal populations and target regions.,(11), 2742–2758. https://doi.org/10.1038/ s41380-020-0685-9
Hilt, L. M., Sander, L. C., Nolen-Hoeksema, S., & Simen, A. A. (2007). The BDNF Val66Met polymorphism predicts rumination and depression differently in young adolescent girls and their mothers.,(1), 12–16. https://doi.org/10.1016/j.neulet.2007.09.053
Ho, T. C., & King, L. S. (2021). Mechanisms of neuroplasticity linking early adversity to depression: Developmental considerations.,(1), 1–13. https://doi.org/10.1038/s41398-021-01639-6
Hsu, D. T., Sanford, B. J., Meyers, K. K., Love, T. M., Hazlett, K. E., Walker, S. J., Mickey, B. J., Koeppe, R. A., Langenecker, S. A., & Zubieta, J. K. (2015). It still hurts: Altered endogenous opioid activity in the brain during social rejection and acceptance in major depressive disorder.,(2), 193–200. https://doi.org/10. 1038/mp.2014.185
Hyde, J. S., & Mezulis, A. H. (2020). Gender differences in depression: Biological, affective, cognitive, and sociocultural factors.,(1), 4–13. https://doi.org/10.1097/HRP.0000000000000230
Ivanova, T., & Beyer, C. (2001). Pre- and postnatal expression of brain-derived neurotrophic factor mRNA/protein and tyrosine protein kinase receptor B mRNA in the mouse hippocampus.,(1), 21–24. https:// doi.org/10.1016/s0304-3940(01)01905-x
Kageyama, I., Yamada, H., Munetsuna, E., Yamazaki, M., Ando, Y., Mizuno, G., Fujii, R., Nouchi, Y., Wakasugi, T., Sakakibara, T., Teshigawara, A., Ishikawa, H., Shimono, Y., Suzuki, K., Hashimoto, S., & Ohashi, K. (2022). Differential effects of excess high-fructose corn syrup on the DNA methylation of hippocampal neurotrophic factor in childhood and adolescence.,(6), e0270144. https://doi.org/10.1371/journal.pone.0270144
Kovacs, M. (1992).Toronto, Canada: Multi-Health Systems Inc.
Kowiański, P., Lietzau, G., Czuba, E., Waśkow, M., Steliga, A., & Moryś, J. (2018). BDNF: A key factor with multipotent impact on brain signaling and synaptic plasticity.,(3), 579–593. https://doi. org/10.1007/s10571-017-0510-4
Lerner, R. M. (1978). Nature, nurture, and dynamic interactionism.,(1), 1–20. https:// doi.org/10.1159/000271572
Lim, M. H., Eres, R., & Vasan, S. (2020). Understanding loneliness in the twenty-first century: An update on correlates, risk factors, and potential solutions.,(7), 793–810. https://doi.org/10.1007/s00127-020-01889-7
Lupien, S. J., McEwen, B. S., Gunnar, M. R., & Heim, C. (2009). Effects of stress throughout the lifespan on the brain, behaviour and cognition.,(6), 434–445. https://doi.org/10.1038/nrn2639
Martinowich, K., Manji, H., & Lu, B. (2007). New insights into BDNF function in depression and anxiety.,(9), 1089–1093. https://doi.org/10.1038/ nn1971
Masten, A. S., & Cicchetti, D. (2010). Developmental cascades.,(3), 491– 495. https://doi.org/10.1017/S0954579410000222
Meyer, A., Hajcak, G., Hayden, E., Sheikh, H. I., Singh, S. M., & Klein, D. N. (2018). A genetic variant brain-derived neurotrophic factor (BDNF) polymorphism interacts with hostile parenting to predict error-related brain activity and thereby risk for internalizing disorders in children.,(1), 125–141. https://doi.org/10.1017/S0954579417000517
Monroe, S. M., & Simons, A. D. (1991). Diathesis-stress theories in the context of life stress research: Implications for the depressive disorders.,(3), 406–425. https://doi.org/10.1037/0033-2909.110.3.406
Nelemans, S. A., Boks, M., Lin, B., Oldehinkel, T., van Lier, P., Branje, S., & Meeus, W. (2021). Polygenic risk for major depression interacts with parental criticism in predicting adolescent depressive symptom development.,(1), 159–176. https://doi.org/ 10.1007/s10964-020-01353-4
Notaras, M., & van den Buuse, M. (2020). Neurobiology of BDNF in fear memory, sensitivity to stress, and stress-related disorders.,(10), 2251–2274. https://doi.org/10.1038/s41380-019-0639-2
Odgers, C. L., Moffitt, T. E., Broadbent, J. M., Dickson, N., Hancox, R. J., Harrington, H., Poulton, R., Sears, M. R., Thomson, W. M., & Caspi, A. (2008). Female and male antisocial trajectories: From childhood origins to adult outcomes.,(2), 673–716. https://doi.org/10.1017/S0954579408000333
O’Donnell, K. J., Glover, V., Holbrook, J. D., & O’Connor, T. G. (2014). Maternal prenatal anxiety and child brain- derived neurotrophic factor (BDNF) genotype: Effects on internalizing symptoms from 4 to 15 years of age.,(4pt2), 1255–1266. https://doi.org/10.1017/S095457941400100X
Petersen, I. T., Bates, J. E., Goodnight, J. A., Dodge, K. A., Lansford, J. E., Pettit, G. S., Latendresse, S. J., & Dick, D. M. (2012). Interaction between serotonin transporter polymorphism (5-HTTLPR) and stressful life events in adolescents’ trajectories of anxious/depressed symptoms.,(5), 1463–1475. https:// doi.org/10.1037/a0027471
Pickles, A., Pickering, K., Simonoff, E., Silberg, J., Meyer, J., & Maes, H. (1998). Genetic “clocks” and “soft” events: A twin model for pubertal development and other recalled sequences of developmental milestones, transitions, or ages at onset.,(4), 243–253. https://doi. org/10.1023/a:1021615228995
Platt, B., Kadosh, K. C., & Lau, J. Y. (2013). The role of peer rejection in adolescent depression.,(9), 809–821. https://doi.org/10.1002/da.22120
Pluess, M. (2015). Individual differences in environmental sensitivity.(3), 138–143. https://doi.org/10.1111/cdep.12120
Pluess, M., & Belsky, J. (2013). Vantage sensitivity: Individual differences in response to positive experiences.,(4), 901–916. https://doi.org/10. 1037/a0030196
Rudolph, K. D., Hammen, C., Burge, D., Lindberg, N., Herzberg, D., & Daley, S. E. (2000). Toward an interpersonal life-stress model of depression: The developmental context of stress generation.,(2), 215–234.https://doi.org/ 10.1017/S0954579400002066
Scourfield, J., Rice, F., Thapar, A., Harold, G. T., Martin, N., & McGuffin, P. (2003). Depressive symptoms in children and adolescents: Changing aetiological influences with development.,(7), 968–976. https://doi.org/10.1111/1469-7610.00181
Shapiro, L. P., Parsons, R. G., Koleske, A. J., & Gourley, S. L. (2017). Differential expression of cytoskeletal regulatory factors in the adolescent prefrontal cortex: Implications for cortical development.,(5), 1123–1143. https://doi.org/10.1002/jnr.23960
Silk, J. S., Stroud, L. R., Siegle, G. J., Dahl, R. E., Lee, K. H., & Nelson, E. E. (2012). Peer acceptance and rejection through the eyes of youth: Pupillary, eyetracking and ecological data from the chatroom interact task.,(1), 93–105. https://doi.org/10.1093/scan/nsr044
Steinberg, L. (2022).(13thed.). New York: McGraw-Hill.
Subramaniam, P., Rogowska, J., DiMuzio, J., Lopez-Larson, M., McGlade, E., & Yurgelun-Todd, D. (2018). Orbitofrontal connectivity is associated with depression and anxiety in marijuana-using adolescents.,, 234–241. https://doi.org/10.1016/j.jad.2018.07.002
Suri, D., Veenit, V., Sarkar, A., Thiagarajan, D., Kumar, A., Nestler, E. J., Galande, S., & Vaidya, V. A. (2013). Early stress evokes age-dependent biphasic changes in hippocampal neurogenesis, BDNF expression, and cognition.,(7), 658–666. https:// doi.org/10.1016/j.biopsych.2012.10.023
Thomas, K. M., Drevets, W. C., Dahl, R. E., Ryan, N. D., Birmaher, B., Eccard, C. H., Axelson, D., Whalen, P. J., & Casey, B. J. (2001). Amygdala response to fearful faces in anxious and depressed children.,(11), 1057–1063. https://doi.org/10.1001/archpsyc. 58.11.1057
Torregrossa, M. M., Isgor, C., Folk, J. E., Rice, K. C., Watson, S. J., & Woods, J. H. (2004). The delta-opioid receptor agonist (+) BW373U86 regulates BDNF mRNA expression in rats.,(4), 649–659. https://doi.org/10.1038/sj.npp.1300345
Tsai, S. J. (2018). Critical issues in BDNF Val66Met genetic studies of neuropsychiatric disorders.,, 156.https://doi.org/10.3389/ fnmol.2018.00156
Tsai, S. J., Cheng, C. Y., Yu, Y. W. Y., Chen, T. J., & Hong, C. J. (2003). Association study of a brain-derived neurotrophic-factor genetic polymorphism and major depressive disorders, symptomatology, and antidepressant response.,(1), 19–22. https://doi.org/ 10.1002/ajmg.b.20026
van Roekel, E., Goossens, L., Scholte, R. H., Engels, R. C., & Verhagen, M. (2011). The dopamine D2 receptor gene, perceived parental support, and adolescent loneliness: Longitudinal evidence for gene-environment interactions.,(10), 1044–1051. https://doi.org/10.1111/j.1469-7610.2011.02424.x
Verhagen, M., van Roekel, E., & Engels, R. C. (2014). Involvement of the BDNF gene in loneliness in adolescence: A report of opposite gene effects in boys and girls.,(3), e92768. https://doi.org/10.1371/journal.pone. 0092768
Webster, M. J., Weickert, C. S., Herman, M. M., & Kleinman, J. E. (2002). BDNF mRNA expression during postnatal development, maturation and aging of the human prefrontal cortex.,(2), 139–150. https://doi.org/10.1016/s0165-3806(02)00540-0
Widaman, K. F., Helm, J. L., Castro-Schilo, L., Pluess, M., Stallings, M. C., & Belsky, J. (2012). Distinguishing ordinal and disordinal interactions.,(4), 615–622. https://doi.org/10.1037/a0030003
Yoshida, K., Higuchi, H., Kamata, M., Takahashi, H., Inoue, K., Suzuki, T., Itoh, K., & Ozaki, N. (2007). The G196A polymorphism of the brain-derived neurotrophic factor gene and the antidepressant effect of milnacipran and fluvoxamine.,(6), 650–656. https://doi.org/10.1177/0269881106072192
Zhang, W., Cao, Y., Wang, M., Ji, L., Chen, L., & Deater- Deckard, K. (2015). The dopamine D2 receptor polymorphism (DRD2 TaqIA) interacts with maternal parenting in predicting early adolescent depressive symptoms: Evidence of differential susceptibility and age differences.,(7), 1428–1440. https://doi.org/10.1007/s10964-015-0297-x
Zhang, W., Li, X., Chen, G., & Cao, Y. (2021). The relationship between positive parenting and adolescent prosocial behaviour: The mediating role of empathy and the moderating role of the oxytocin receptor gene.,(9), 976–991.
[张文新, 李曦, 陈光辉, 曹衍淼. (2021). 母亲积极教养与青少年亲社会行为: 共情的中介作用与OXTR基因的调节作用.(9), 976–991.]
敏感性分析1:
采用同伴地位(同伴接纳−同伴拒绝)为环境指标进行同时性回归分析和潜变量增长模型分析。如表A1所示, 同伴地位与同伴拒绝模型的结果相似, 均发现了T1~T3的基因−交互作用显著, 且基因−环境交互模式的年龄差异也与同伴拒绝模型一致。
表A1基因与同伴地位对青少年抑郁的交互作用(T1~T3)
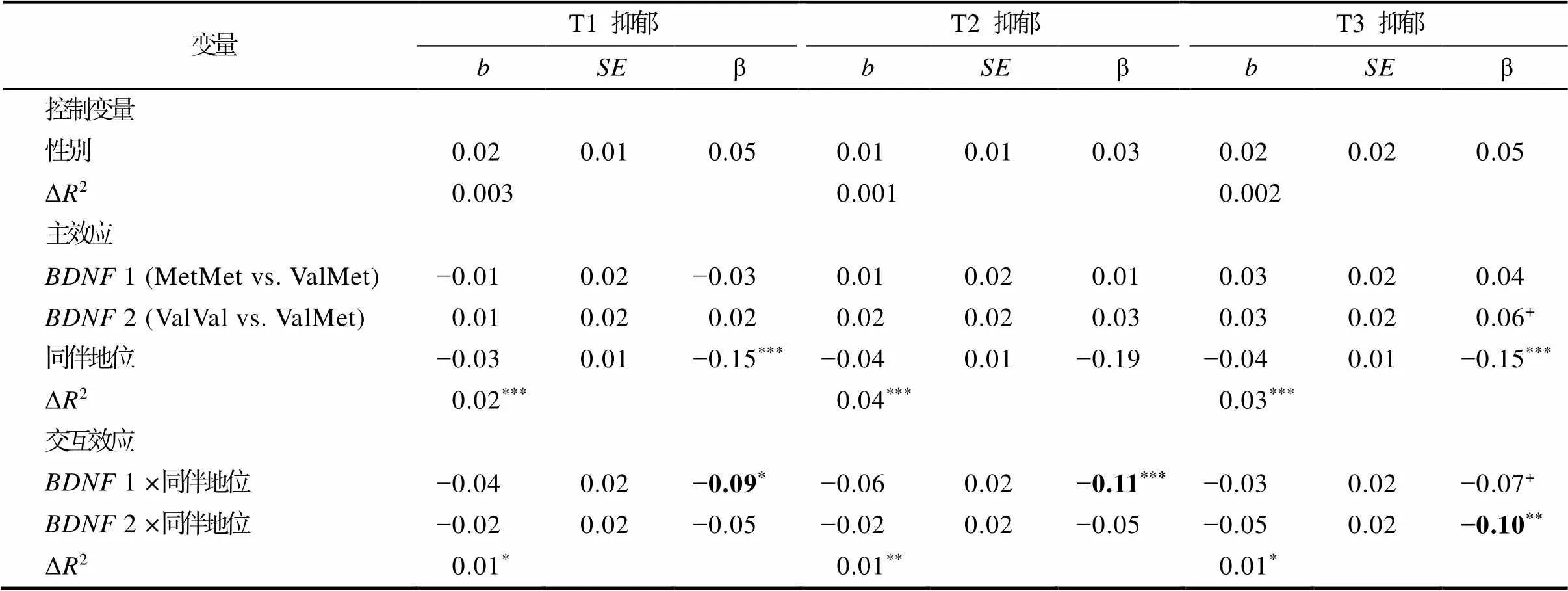
变量T1 抑郁T2 抑郁T3 抑郁 bSEβbSEβbSEβ 控制变量 性别0.020.010.050.010.010.030.020.020.05 ΔR20.0030.0010.002 主效应 BDNF 1 (MetMet vs. ValMet)−0.010.02−0.030.010.020.010.030.020.04 BDNF 2 (ValVal vs. ValMet)0.010.020.020.020.020.030.030.020.06+ 同伴地位−0.030.01−0.15***−0.040.01−0.19−0.040.01−0.15*** ΔR20.02***0.04***0.03*** 交互效应 BDNF 1 ×同伴地位−0.040.02−0.09*−0.060.02−0.11***−0.030.02−0.07+ BDNF 2 ×同伴地位−0.020.02−0.05−0.020.02−0.05−0.050.02−0.10** ΔR20.01*0.01**0.01*
注:*< 0.05;**< 0.01;***< 0.001。
采用再参数化回归(Widaman et al., 2012)检验基因与同伴地位的交互作用符合何种理论模型(“不同易感性”、“素质−压力”和“优势敏感”模型)。
T1同伴地位与BDNF基因对抑郁的再参数化分析(表A2)显示, 交叉点C及其95% CI均在同伴地位全距内([−07.88, 4.67]), 限定ValMet回归系数为0后, Model 1与Model2在解释率上存在显著差异, 且Model 2的AIC值最小, 弱不同易感模型为最优模型。
T2 同伴地位与BDNF基因对抑郁的再参数化分析(表A3)显示, 交叉点C及其95% CI均在同伴地位全距内([−7.22, 4.74]), 限定ValMet基因型回归系数为0后, Model 1与Model2在解释率上存在显著差异, 且Model 1的AIC值最小, 弱不同易感模型为最优模型。
T3 同伴地位与BDNF基因对抑郁的再参数化分析(表A4)显示, 强不同易感模型与弱优势敏感模型均比弱不同易感模型减少了一个参数估计, 但是解释率与不同易感模型不存在显著差异, 因此弱优势敏感模型亦能很好的解释基因−环境交互模式。
表A2基因与同伴地位对青少年抑郁的再参数化分析(T1)
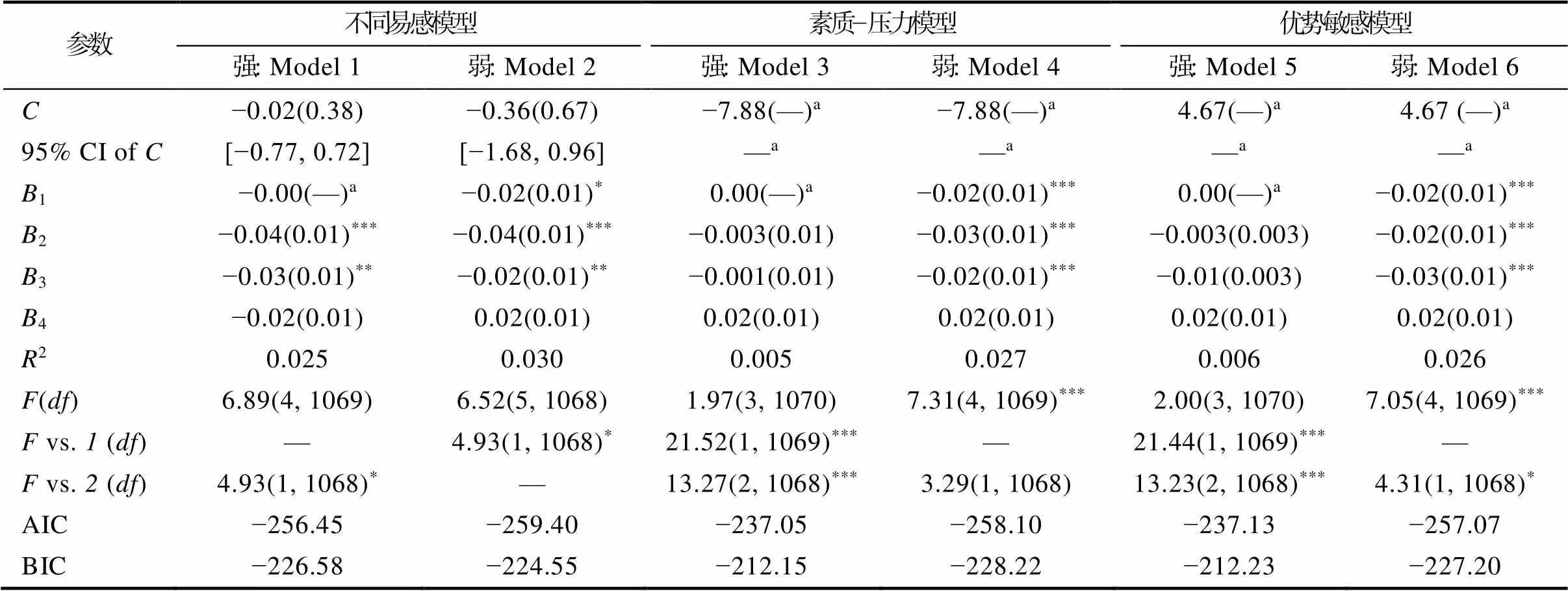
参数不同易感模型素质−压力模型优势敏感模型 强: Model 1弱: Model 2强: Model 3弱: Model 4强: Model 5弱: Model 6 C−0.02(0.38)−0.36(0.67)−7.88(—)a−7.88(—)a4.67(—)a4.67 (—)a 95%CI of C[−0.77, 0.72][−1.68, 0.96]—a—a—a—a B1−0.00(—)a−0.02(0.01)*0.00(—)a−0.02(0.01)***0.00(—)a−0.02(0.01)*** B2−0.04(0.01)***−0.04(0.01)***−0.003(0.01)−0.03(0.01)***−0.003(0.003)−0.02(0.01)*** B3−0.03(0.01)**−0.02(0.01)**−0.001(0.01)−0.02(0.01)***−0.01(0.003)−0.03(0.01)*** B4−0.02(0.01)0.02(0.01)0.02(0.01)0.02(0.01)0.02(0.01)0.02(0.01) R20.0250.0300.0050.0270.0060.026 F(df)6.89(4, 1069)6.52(5, 1068)1.97(3, 1070)7.31(4, 1069)***2.00(3, 1070)7.05(4, 1069)*** F vs. 1 (df)—4.93(1, 1068)*21.52(1, 1069)***—21.44(1, 1069)***— F vs. 2 (df)4.93(1, 1068)*—13.27(2, 1068)***3.29(1, 1068)13.23(2, 1068)***4.31(1, 1068)* AIC−256.45−259.40−237.05−258.10−237.13−257.07 BIC−226.58−224.55−212.15−228.22−212.23−227.20
注:*< 0.05;**< 0.01;***< 0.001。
表A3基因与同伴地位对青少年抑郁的再参数化分析(T2)

参数不同易感模型素质−压力模型优势敏感模型 强: Model 1弱: Model 2强: Model 3弱: Model 4强: Model 5弱: Model 6 C0.45(0.33)0.47(0.52)−7.22(—)a−7.22(—)a4.74(—)a4.74(—)a 95%CI of C[−0.20, 1.09][−0.55, 1.49]—a—a—a—a B10.00(—)a−0.02(0.01)**0.00(—)a−0.03(0.01)***0.00(—)a−0.03(0.01)*** B2−0.05(0.01)***−0.05(0.01)***−0.002(0.002)−0.03(0.01)***−0.01(0.004)*−0.03(0.01)*** B3−0.03(0.01)***−0.03(0.01)***−0.001(0.002)−0.03(0.01)***−0.01(0.004)*−0.03(0.01)*** B40.01(0.01)0.01(0.01)0.01(0.01)0.01(0.01)0.01(0.01)0.01(0.01) R20.0330.0400.0010.0330.0100.035 F(df)9.25(4, 1073) ***8.85(5, 1072)***0.46(3, 1074)9.20(4, 1073)***3.51(3, 1074)9.63(4, 1073)*** F vs. 1 (df)—7.05(1, 1072)**35.59(1, 1073)***—26.22(1, 1073)***— F vs. 2 (df)7.05(1, 1072)**—21.42(2, 1072)***7.27(1, 1072)**16.71(2, 1072)***5.57(1, 1072)* AIC−80.97−86.04−47.79−80.75−56.94−82.45 BIC−51.07−51.16−22.88−50.85−32.03−52.55
注:*< 0.05;**< 0.01;***< 0.001。
表A4基因与同伴地位对青少年抑郁的再参数化分析(T3)
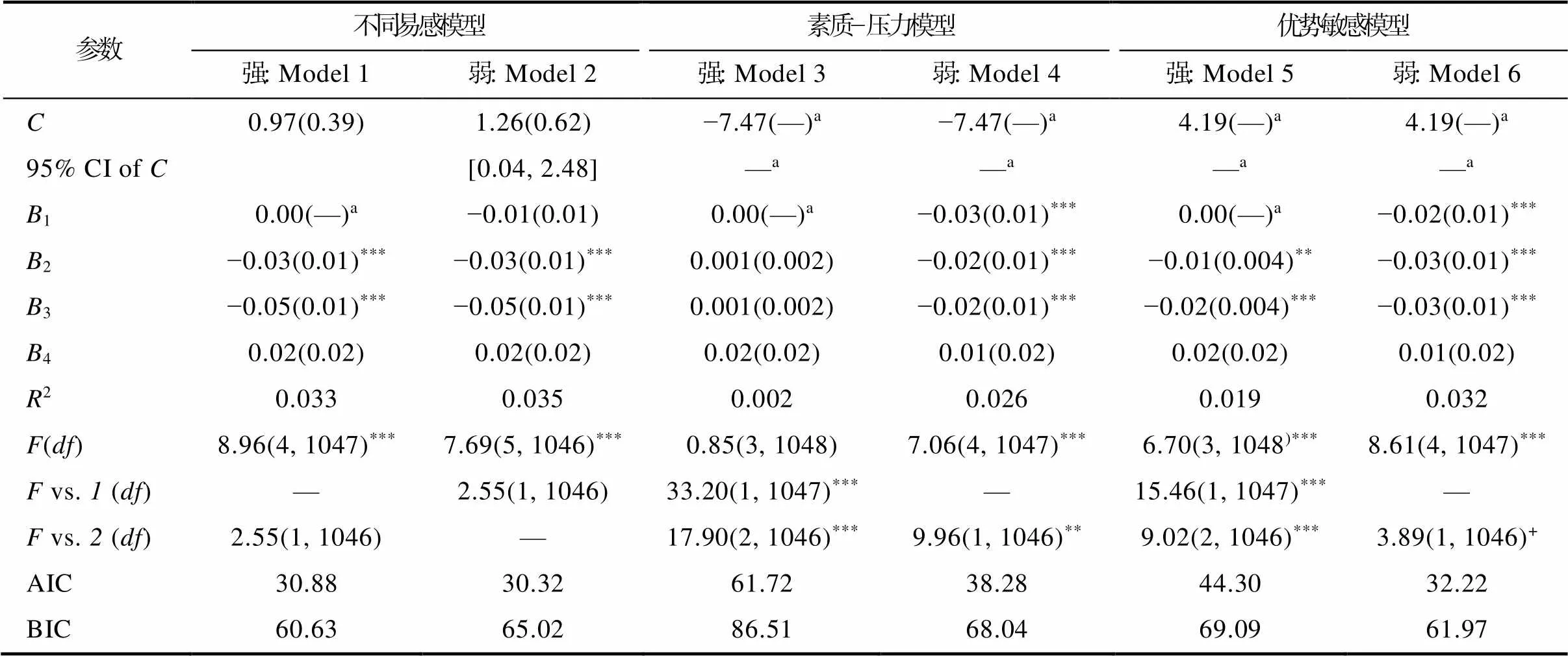
参数不同易感模型素质−压力模型优势敏感模型 强: Model 1弱: Model 2强: Model 3弱: Model 4强: Model 5弱: Model 6 C0.97(0.39)1.26(0.62)−7.47(—)a−7.47(—)a4.19(—)a4.19(—)a 95%CI of C[0.04, 2.48]—a—a—a—a B10.00(—)a−0.01(0.01)0.00(—)a−0.03(0.01)***0.00(—)a−0.02(0.01)*** B2−0.03(0.01)***−0.03(0.01)***0.001(0.002)−0.02(0.01)***−0.01(0.004)**−0.03(0.01)*** B3−0.05(0.01)***−0.05(0.01)***0.001(0.002)−0.02(0.01)***−0.02(0.004)***−0.03(0.01)*** B40.02(0.02)0.02(0.02)0.02(0.02)0.01(0.02)0.02(0.02)0.01(0.02) R20.0330.0350.0020.0260.0190.032 F(df)8.96(4, 1047)***7.69(5, 1046)***0.85(3, 1048)7.06(4, 1047)***6.70(3, 1048)***8.61(4, 1047)*** F vs. 1 (df)—2.55(1, 1046)33.20(1, 1047)***—15.46(1, 1047)***— F vs. 2 (df)2.55(1, 1046)—17.90(2, 1046)***9.96(1, 1046)**9.02(2, 1046)***3.89(1, 1046)+ AIC30.8830.3261.7238.2844.3032.22 BIC60.6365.0286.5168.0469.0961.97
注:*< 0.05;**< 0.01;***< 0.001。
敏感性分析2:
平行潜变量增长模型见表A5, 同伴拒绝、同伴接纳和同伴地位的初始水平均显著预测抑郁的初始水平, 而非抑郁的增长速度, 同伴关系的增长速度与青少年抑郁的增长速度无关。因此, 本研究正式分析仍采用T1同伴关系作为预测变量进行条件潜变量增长的预测变量。
表A5 同伴关系与青少年抑郁的平行潜变量增长模型

预测因素抑郁截距抑郁斜率χ2(df)CFIRMSEA Model A: 同伴拒绝截距0.20 (0.05)***−0.06 (0.05)16.29 (7)0.990.04 [0.01, 0.06] 同伴拒绝斜率0.09 (0.08) Model B: 同伴接纳截距−0.19 (0.04)***−0.01 (0.05)4.17(7)1.000.00 [0.00, 0.03] 同伴接纳斜率−0.14 (0.07) Model C: 同伴地位截距−0.23 (0.05)***0.03 (0.05)11.33 (7)0.990.02 [0.00, 0.05] 同伴地位斜率−0.15 (0.08)
注:表中所有参数估计值均为标准化结果。*< 0.05;**< 0.01;***< 0.001。
Associations among brain-derived neurotrophic factor gene, peer relationships,and depression across early adolescence: Dynamic genetic effects
CAO Yanmiao, FANG Huici, ZHU Xinyue, JI Linqin, ZHANG Wenxin
(School of Psychology, Shandong Normal University, Jinan 250014, China)
There has been a dramatic rise in gene–environment interaction (G × E) studies of depression over the last two decades. These studies are pivotal to understanding the etiology of depression and individual differences in environmental sensitivity. However, these studies rarely take into consideration how the genotype by environment interactions change over development and how the interactions work on the developmental trajectories of depression. The brain-derived neurotrophic factor (BDNF) gene is a good candidate for the investigation of the dynamic genetic effects on depression because it is involved in several age-related changes in behavior and brain maturation. On the one hand, the effect of the BDNF gene may depend on the basal BDNF level. The BDNF level peaks during adolescence, so the effect of the BDNF gene on depression may change during development. Moreover, peer experiences change may alter epigenetic modifications of the BDNF gene, which may change the pattern of gene–environment interactions. On the other hand, according to the developmental cascades model, the differences in genetic effects on depression may increase over time in that initial depressive symptoms may evoke poor peer experiences. Taken together, this study aimed to investigate the age differences in the G × E interaction on depression and the G × E effect on the developmental trajectories of depression.
One thousand and eighty-six adolescents (aged 11–12 years with a mean of 12.32, 50% girls) were followed up for three years. Saliva samples, self-reported depressive symptoms, and peer nomination were all collected. All of the measures showed good reliability. Concurrent hierarchical regression analyses and latent growth curve models (LGCMs) were conducted. We also completed re-parameterized regression and parallel LGCMs to understand the gene by environment interaction pattern and the dynamic association between peer relationships and depression.
The results showed that the BDNF Val66Met polymorphism significantly moderated the influence of peer rejection—but not peer acceptance—on youth depressive symptoms at three time points; however, the susceptible genotype changed over time. In particular, the effect of peer rejection on depression was stronger in MetMet compared to ValMet carriers at 12 years of age; the effect of peer rejection on depression was stronger in MetMet and ValVal compared to ValMet carriers at 13 years of age; the effect of peer rejection on depression was stronger in ValVal carriers compared to ValMet carriers at 14 years of age. LGCMs suggested that adolescents’ depression increased in a linear trajectory from 12 to 14 years of age. In addition, there were significant genotype differences in the change of depression over time, but this effect was not moderated by peer relationships.
These findings may move research in the field away from the simplistic notion of risk alleles, recognizing that an allele may be a risk factor during one period and a protective factor during another. Further, this study has progressed the conceptualization of how genes and the environment interact to influence the developmental trajectories of depression during early adolescence.
adolescent depression,, peer rejection, age differences, latent growth curve model
B844; B845
https://doi.org/10.3724/SP.J.1041.2023.01620
2022-10-28
*国家自然科学基金项目(32071073; 31900776; 31971008)资助。
张文新, E-mail: zhangwenxin@sdnu.edu.cn

