苜蓿miR156及其靶基因生物信息学分析
刘宝宝,孟桂智,刘祖江,贾晶莹,段红娟,马 云,蔡小艳
(宁夏大学农学院/宁夏回族自治区反刍动物分子细胞育种重点实验室,银川 750021)
【研究意义】畜牧生产中,苜蓿(Medicagosativa)作为奶牛重要的饲料来源之一,与产奶量和乳品质密切相关。而miR156家族作为苜蓿中表达最高的miRNA之一[1],探究和挖掘其生物信息和调控功能具有潜在的研究意义。【前人研究进展】微小RNA(microRNA,miRNA)是生物体内普遍存在的一种内源性的单链非编码RNA,长度大约为18~25 nt。最初在秀丽隐杆线虫(Caenorhabditiselegans,C.elegan)体内被发现[2]。其发挥作用的方式是通过miRNA与靶基因3’非翻译区(Untranslated regions,UTR)抑制性结合,抑制该蛋白合成或诱导该mRNA降解,从而对基因进行转录后表达调控[3]。miRNA参与包括细胞间信号传递,细胞增殖、分化、凋亡以及细胞代谢等多种生物进程[4-5]。植物miRNA与动物miRNA最大的区别在于植物miRNA 3’末端具有2’-O-甲基化修饰,甲基化修饰不仅能够增强miRNA的稳定性,还能保护甲基化修饰的miRNA不会被核酸外切酶降解[6]。甲基化修饰使植物miRNA经过加工、烹饪和储存等预处理后不会被降解,依然可以进入人和动物体内跨界调控发挥一定作用[7]。目前,植物跨界调控主要在抑制乳腺癌增长、促进细胞增殖、预防疾病等方面研究较多[8-10]。【本研究切入点】苜蓿在畜牧生产中是非常重要的经济作物,有“牧草之王”之称的紫花苜蓿产草量高,适口性好,营养价值高,不仅含有丰富的蛋白质、矿物质和维生素等营养成分,而且富含动物所需的必需氨基酸、微量元素和未知生长因子[11]。苜蓿以其干草和青贮形式饲喂奶牛在奶牛瘤胃中更容易消化[12]。在日粮中添加苜蓿饲喂荷斯坦奶牛,能够增加牛乳中乳蛋白率、提高消化率和产奶量[13]。因此,探究苜蓿中的非编码RNA将具有非常大的潜力。苜蓿中miR156家族成员较多,且在奶牛体内具有复杂的调控关系。明确其家族基因的序列组成及调控功能,是畜牧生产中利用苜蓿非编码RNA的关键所在。【拟解决的关键问题】本研究将利用生物信息学方法,对mtr-miR156基因家族成员进行成熟序列和茎环序列保守性分析、成熟序列染色体定位、茎环序列同源树分析和靶基因预测,为mtr-miR156在奶牛体内的调控研究以及苜蓿优良品种选育等提供参考依据。
1 材料与方法
1.1 数据来源
在植物miRNA数据库PmiREN(http://www.pmiren.com/)中搜索mtr-miR156数据信息,通过下载获得mtr-miR156家族全部成员的成熟序列、茎环序列和染色体定位信息。
1.2 序列比对和同源树分析
利用weblogo(http://weblogo.berkeley.edu/logo.cgi)在线网站对mtr-miR156家族成员的成熟序列和茎环序列进行多序列比对,并绘制序列保守性分析图。Logo图可以显示出序列中每个位置上碱基出现的频率和序列保守程度,其中参数均设置为默认值。通过DNAMAN软件对mtr-miR156家族10名成员的茎环序列进行比对分析并绘制同源树,其中参数均设置为默认值。
1.3 靶基因预测及功能富集分析
利用联川生物云平台(https://www.omicstudio.cn/index)、TargetScan、RNAhybrid在线网站预测获得mtr-miR156家族成员在牛体内的靶基因信息以及靶基因Geng Ontology(GO)功能注释和京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)通路富集分析。
2 结果与分析
2.1 mtr-miR156家族成熟序列保守性分析及其基因定位
利用PmiREN数据库共搜索到10个mtr-miR156家族成员(表1)。结果显示,除了mtr-miR156g和mtr-miR156j的长度分别为22和21 nt外,其余8个成员的长度均为20 nt。利用数据库对10个家族成员进行基因定位,发现10个成熟序列共分布在3条染色体上,分别为染色体1、3和4。其中有5个成员位于染色体3上,分别是mtr-miR156c、mtr-miR156d、mtr-miR156e、mtr-miR156f、mtr-miR156g,有3个成员位于染色体1上,分别是mtr-miR156a、mtr-miR156b、mtr-miR156j,有2个成员位于染色体4上,分别是mtr-miR156h、mtr-miR156i。利用DNAMAN软件对mtr-miR156家族成员成熟序列进行保守性分析(图1),结果表明,除mtr-miR156g和mtr-miR156j外,其余成熟序列中20 nt完全保守。

表1 mtr-miR156家族成熟序列及其基因定位

图1 mtr-miR156家族成熟序列的保守性分析
2.2 mtr-miR156家族成员茎环序列的保守性分析
对mtr-miR156家族茎环序列进行保守性分析(表2)发现,碱基长度有4个组别(mtr-miR156d、mtr-miR156f长度为81 nt;mtr-miR156h、mtr-miR156i长度为84 nt;mtr-miR156a、mtr-miR156b、mtr-miR1 56j长度为86 nt;mtr-miR156c、mtr-miR156e、mtr-miR156g长度为89 nt)。通过DNAMNA软件比对(图2),mtr-miR156家族成员中茎环序列高度保守区域与成熟序列完全重合。而其余序列保守性差可能是因为miR156家族成员合成过程在苜蓿的不同组织和不同发育阶段造成。

表2 mtr-miR156家族茎环序列
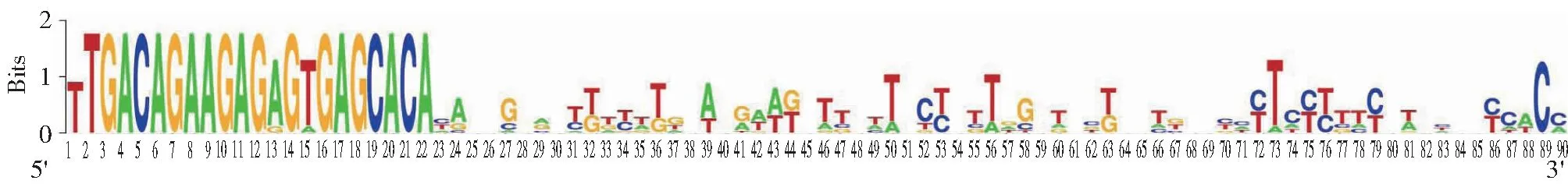
图2 mtr-miR156家族茎环序列的保守性分析
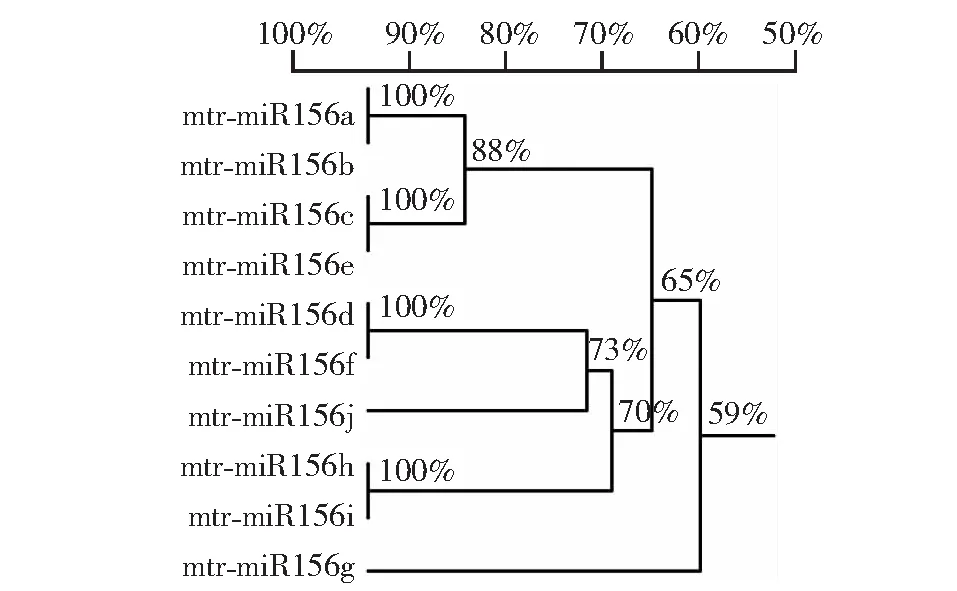
图3 mtr-miR156茎环序列同源树
2.3 mtr-miR156家族茎环序列的同源树分析
利用DNAMAN软件对mtr-miR156家族的茎环序列进行比对分析并绘制同源树(图3)。结果显示,mtr-miR156家族10个成员除miR156g外,共分为3个分支:mtr-miR156a、mtr-miR156b、mtr-miR156c、mtr-miR156e(分支Ⅰ);mtr-miR156d、mtr-miR156f、mtr-miR156j(分支Ⅱ);mtr-miR156h、mtr-miR156i(分支Ⅲ)。从进化树上看,mtr-miR156a和mtr-miR156b、mtr-miR156c和mtr-miR156e、mtr-miR156d和mtr-miR156f、mtr-miR156h和mtr-miR156i亲缘关系分别都达到100%,成员彼此之间亲缘关系最近,而mtr-miR156g与其他序列差异较大,与其他成员亲缘关系最远。进一步分析PmiREN数据库中成熟序列表达位置和RPM信息,发现mtr-miR156其他成员主要在植物的幼苗、嫩芽、叶中高表达。而mtr-miR156g在苜蓿的嫩芽和叶中极少表达,只有在苜蓿的幼苗中高表达,在苜蓿的种子中少量表达。结合同源树,分析mtr-miR156g在苜蓿不同组织中高表达可能会导致与其他成员成熟序列有所差异,亲缘关系较远。有研究表明,miRNA在同一物种的不同细胞或组织中表达量显著差异,比如miR171在拟南芥的花序和花组织中表达量高,而在茎和叶组织中不表达[14]。这也表明miRNA的表达模式具有分化的组织特异性和时序性,即miRNA水平在不同组织、不同发育阶段中有显著差异。
2.4 mtr-miR156a靶基因GO功能富集分析
miRNA与mRNA作用是通过miRNA成熟序列的种子区与mRNA的3’UTR结合,而mtr-miR156家族中种子区高度保守,因此使用mtr-miR156a在TargetScan、RNAhybrid和联川生物云平台进行牛体内靶基因预测。Gene Ontology(GO)功能富集分析(图4)发现,注释到生物学过程层面显著富集的功能为RNA聚合酶II转录正调控、转录调控-DNA-模板化、正调控GTRase活性,分别有44、36和34条靶基因富集。注释到细胞组成层面显著富集的功能为膜的组成部分、细胞质、细胞核,分别有204、194和177条靶基因富集。注释到分子功能层面显著富集的功能为ATP合成、锌离子结合、金属离子结合、分别有80、73和72条靶基因富集。
2.5 mtr-miR156a靶基因功能KEGG通路富集分析
KEGG通路富集分析发现(图5),在靶基因注释的全部KEGG通路中,最显著富集的通路是脂肪酸降解(P<0.001),其次是Ras信号通路的调节(P<0.001),其中靶基因数量最多的是Ras信号通路,其次是MAPK信号通路,分别是有35和30个。表明mtr-miR156a可能对牛体内脂肪酸降解、细胞生长、分化、蛋白质运输和分泌等都具有调控作用。
2.6 mtr-miR156a调控乳品质中蛋白质合成和脂类代谢相关靶基因预测
乳蛋白是人类营养优质蛋白质的重要来源,乳蛋白合成和分泌是由奶牛乳腺上皮细胞完成,主要由高丰度蛋白和低丰度蛋白构成,其中高丰度蛋白以酪蛋白为主,低丰度蛋白主要包括乳清蛋白、乳脂肪球膜蛋白及乳铁蛋白等[15]。乳蛋白作为牛乳中主要的营养成分之一,其中高丰度的酪蛋白由α-酪蛋白、β-酪蛋白、κ-酪蛋白共同完成,是决定牛乳品质的重要因素[16]。乳脂也是牛奶的重要成分,是一种高质量的天然脂肪,消化率高达98%[17]。乳脂乳蛋白率作为评判牛乳品质的重要指标之一,成为关注奶牛营养调控及分子调控的热点。为此,在预测到的所有靶基因中筛选出mtr-miR156a调控蛋白质合成和脂类代谢相关的靶基因,并在RNAhybrid在线网站上进行比对,最终筛选出符合自由能阈值的靶基因(表3),预测其靶基因在奶牛体内参与乳脂乳蛋白合成代谢,为后续研究mtr-miR156a调控奶牛乳脂乳蛋白合成代谢奠定基础。

图5 mtr-miR156a在牛体内靶基因KEGG通路富集分析

表3 mtr-miR156a调控蛋白质合成和脂类代谢相关靶基因预测
3 讨 论
在植物miRNA研究中,miR156是一种进化非常保守的miRNA[18],在拟南芥、苜蓿、大豆、水稻等多种植物中,均能检测到iR156,发现其对植物开花过程、生长发育、响应生物和非生物胁迫等都具有调控作用。miR156最早发现于拟南芥植物中,靶向squamosa启动子结合蛋白(Squamosapromoterbindingprotein-like,SPL)基因家族参与植物表型改变和调控开花过程[19-21],可以调节植物嫩芽从幼年期到成年期的过渡,伴随着植物营养形态的改变和生殖潜力的增加。植物开花易受外界因素的影响,温度、湿度等环境因素都会影响植物开花过程,延期开花会影响果实成熟和农作物产量,miRNA在植物开花过程中起重要调节作用。SPL家族是植物中特有的一类具有多功能的转录因子家族,具有高度保守的DNA结合区域SBP结构域[22]。在拟南芥中,SPL基因家族成员有17个,其中有11个基因受到miR156调控,参与调节开花过程、代谢调节以及非生物胁迫等多种调节功能[23]。研究发现,miR156可以靶向SPL3基因控制叶片中的开花基因(FloweringlocusT,FT)表达以调节环境温度响应性开花,在低温(16 ℃)条件下,过表达miR156会抑制拟南芥开花,而过表达SPL3基因则会促进拟南芥开花[24]。在大豆(Glycinemax)中,Gm-miR156b靶向SPL直系同源物,并负调控GmSPLs,从而上调大豆开花相关基因的启动子,延迟大豆开花时间[25]。在对SPL家族基因的研究中,发现其多个基因可以参与植物生长发育的调控。miR156通过靶向SBP/SPL基因参与调控植物的生长发育过程[26-27]。多年生低温短日照植物草莓(Fragaria×ananassa)中miR156通过靶基因FaSPL9对草莓的生长发育呈负调控作用,抑制草莓的开花及生长发育[28]。叶绿素是植物进行光合作用的主要色素,在高等植物中叶绿素主要有叶绿素a和叶绿素b,叶绿素含量越多,植物光合作用能力越强。在水稻中SPL14能够直接结合叶绿素b还原酶(Non-yellowcoloring1,NYC1)基因的启动子来抑制NYC1的表达,而miR156可以抑制SPL14的表达,即miR156-SPL14-NYC1基因作用在同一信号通路上,从而对叶绿素含量起到负调控作用,进而影响水稻的生长发育[29]。此外,miR156在转录水平还可以抑制SPL13,从而增加WD40-1基因表达来增强紫花苜蓿的抗旱能力[30]。
植物miR156除了可对植物的种子成熟过程、开花、抗旱能力进行调控外,还可以进入动物体内,被动物机体吸收,跨界调控动物细胞的生理功能。2012年首次研究发现,植物源外源性miRNA存在人体血清中,检测到miR168a和miR156a有较高的表达水平,其中大米来源的miR168a能够被消化道吸收进入肝脏靶向低密度脂蛋白受体衔接蛋白1(Low-densitylipoproteinreceptoradapterprotein1,LDLRAP1),导致小鼠血浆中LDL含量升高[31]。这一现象的发现,使研究人员对植物miRNA跨界调控的研究产生了兴趣,因此有更多的人开始对植物miRNA跨界调控动物基因表达机制进行深入研究。在之后几年的研究中,miR156a-5p在小麦[32]和玉米[33]等经济作物中高表达,小麦和玉米作为人类食品和动物饲料的重要来源,被人和动物大量摄食,其所含的miR156a-5p也可能随着饮食进入人和动物体内发挥跨界调控作用。在最近的研究报道中,植物miR156a-5p可以靶向通路上游基因Wnt家族成员10b(Wntfamilymember10b,Wnt10b)在细胞水平抑制Wnt/β-catenin信号通路活性,抑制肠道上皮细胞增殖,影响小鼠肠道发育[34]。在成年母猪体内也检测到了玉米来源的miR156a-5p,并发现玉米衍生的miR156a-5p可以穿过胃肠道并留存在母猪体内,表明外源性miRNA具有调节哺乳动物基因表达的潜力[35]。此外,在甘草水煎剂中发现的miR156,通过靶向Toll样受体(Toll-likereceptor,TLR)家族基因从而在小鼠和人体内抑制炎症、抑制细胞凋亡、促进细胞增殖、调节肠道菌群的组成以及肠道免疫系统的作用[36-38]。研究还发现,miR156可以靶向鲍曼不动杆菌(Acinetobacterbaumannii,AB),调节AB菌的生长,通过调节肠道菌群来调节肠道免疫;与对照组相比,miR156灌胃组能显著提高小鼠肠道内优势菌群的存活,不仅可以影响自身的肠道菌群和肠道免疫,还会影响同笼饲养互食粪便共培养未灌胃小鼠的肠道菌群和肠道免疫。miR156可以靶向Cd28和Cd79a基因并使之上调,激活脾脏T淋巴细胞和B淋巴细胞信号通路,促进小鼠脾脏免疫[39]。Cd28分子是一种广泛存在于T细胞表面的Ⅰ型跨膜蛋白受体,属于免疫球蛋白家族,参与共刺激信号通路[40],促进T淋巴细胞增殖,在免疫应答中发挥重要作用[41]。Cd79a是B细胞标志物,与B细胞受体结合后产生信号转导。研究表明,植物miR156a对人结肠腺癌细胞(Humancolorectaladenocarcinomacells,Caco-2)中JAM-A(Junctionadhesionmolecule-A,JAM-A)基因有直接靶向作用,miR156a可以抑制人Caco-2细胞中JAM-A的表达。而对小鼠主动脉内皮细胞中JAM-A基因的mRNA水平没有明显变化[42]。推测植物miR156在哺乳动物中与JAM-A潜在靶点不完全保守,说明植物miR156与JAM-A结合跨界调控可能具有种属性。连接粘附分子A(Junctionadhesionmolecule-A,JAM-A)是免疫球蛋白超家族跨膜粘附分子成员,主要存在于内皮和上皮紧密连接处,也存在于循环白细胞和血小板上[43]。它发挥着广泛的功能,包括调节细胞极性、细胞迁移、血管完整性和细胞旁通透性[44-48]。因此,植物miR156a抑制人体内JAM-A的表达,可能会增强内皮细胞的迁移,从而减少人体内动脉粥样硬化斑块的形成[49]。
本研究利用生物信息学方法对mtr-miR156基因家族的成熟序列和茎环序列进行保守性分析、同源树分析、成熟序列染色体定位、靶基因预测。发现mtr-miR156基因家族共有10个家族成员,其中8个成员成熟序列完全一致,其余2个成员成熟序列保守性较高。在茎环序列中,10个成员中位于成熟序列位置的碱基与成熟序列一致。通过对mtr-miR156基因家族茎环序列进行同源树分析发现,在进化过程中茎环序列碱基发生了突变和缺失,产生了茎环序列只有成熟序列区高度保守,而其他区域不保守。
4 结 论
到目前为止,PmiREN数据库收录共100多个物种1807个miR156家族成员。本研究通过对mtr-miR156基因家族预测到的靶基因进行GO功能富集分析和KEGG通路富集分析,发现mtr-miR156家族成员可能会通过靶向靶基因跨界调控奶牛体内脂肪酸、乳脂乳蛋白合成代谢及其他调控作用。该结果为mtr-miR156跨界调控研究提供理论基础,也对其他miRNA跨界调控的研究具有参考意义。

