茶树CsTMFs的克隆与表达分析
孙明慧 吴琼 刘丹丹 焦小雨 王文杰
(安徽省农业科学院茶叶研究所,合肥230001)
茶是起源于中国的世界三大无酒精饮料之一,由茶树(Camellia sinensis)叶片加工制备而成,具有抗炎、抗病毒和降血糖等多种生理功能[1]。茶树易受生物胁迫和非生物胁迫的威胁,影响茶树产量和质量。其中茶树对冷较为敏感,茶树喜温不耐寒,这也是制约茶树种植区扩大的重要原因之一[2]。随着全球气候变暖,干旱日益严重,制约茶叶生产[3]。低温会破坏细胞、限制植物生长,而干旱胁迫会降低茶叶产量、增加茶树死亡率,低温和干旱均会对茶树的次级代谢和生长发育带来不利影响[4]。
TATA元件调控因子(TATA element modulatory factor,TMF)在基因的转录调控中能特异结合TATA-box[5],最早在人体内发现,在动物中研究较多,主要参与基因的表达调控、蛋白降解、细胞内的膜运输等,并在精子发育[6-7]、肿瘤发生[8]、细胞的代谢应激中也发挥关键作用[9]。水稻中仅有1个与人类同源的TMF基因——OsTMF,双定位于细胞核及高尔基体,含有TMF-DNA-bd和TMF-TATA-bd 2个保守结构域[10]。冷胁迫处理下,OsTMF表达上调,水稻细胞壁结构成分果胶、纤维素含量发生改变,低温诱导OsTMF直接调控OsBRUP16、OsCesA4和OsCesA9显著上调,表明OsTMF通过调控水稻细胞壁的合成与降解响应低温胁迫,影响水稻对低温的敏感性[10]。低温处理OsTMF-OE植株,脱水反应元件结合蛋白1家族(dehydration-responsive clement binding protein 1,DREB1s)编码基因表达上调,维持ROS水平并促进脯氨酸的积累,揭示DREB1s基因在植物低温胁迫应答中起着正调控的作用[11-12]。
目前,TMF基因仅在拟南芥[13]、水稻等植物中获得鉴定,茶树中暂未报道。本研究通过对茶树全基因组CsTMF基因进行筛选与鉴定,得到2个CsTMF基因。对这2个CsTMF基因进行克隆和生物信息学分析;利用转录组数据与qPCR方法探索低温和干旱胁迫下CsTMF的表达模式,为茶树TMF基因的研究提供理论依据。
1 材料与方法
1.1 茶树CsTMF基因家族的筛选与鉴定
在NCBI数据库(https://www.ncbi.nlm.nih.gov/)搜索水稻OsTMF的cDNA序列(登录号:AK067-197),下载其氨基酸序列,用已鉴定的OsTMF氨基酸序列作为种子序列,在安徽农业大学茶树基因组数据库(http://tpdb.shengxin.ren/index.html)上进行BLASTP对茶树基因组进行序列比对搜索,得到茶树CsTMF成员氨基酸序列,同时通过在线数据库Interpro(Interprohttps://www.ebi.ac.uk/interpro/)对序列进行结构域分析,以确定其含有TMF-DNA-bd和TMF-TATA-bd完整保守结构域。
1.2 CsTMFs的克隆与测序
使用NCBI Primer-BLAST(http://www.ncbi.nlm.nih.gov/tools/primer-blast/)在线设计CDS全长引物(表1)。选取安徽省农业科学院茶叶研究所茶树品系比较试验园(以下简称品比园)福鼎大白茶成熟叶片进行RNA的提取,合成cDNA第一链。以cDNA为模板,利用高保真酶 KOD One PCR Master Mix(TOYOBO),进行PCR扩增。反应程序为98℃10 s;55℃ 5 s,68℃ 10 s,共35个循环。PCR产物经1%凝胶电泳检测,回收纯化,与pEASY-Blunt Cloning载体(TRANSGENBIOTECH)连接,转化,经卡那筛选后,选择阳性克隆送南京擎科生物科技有限公司测序。
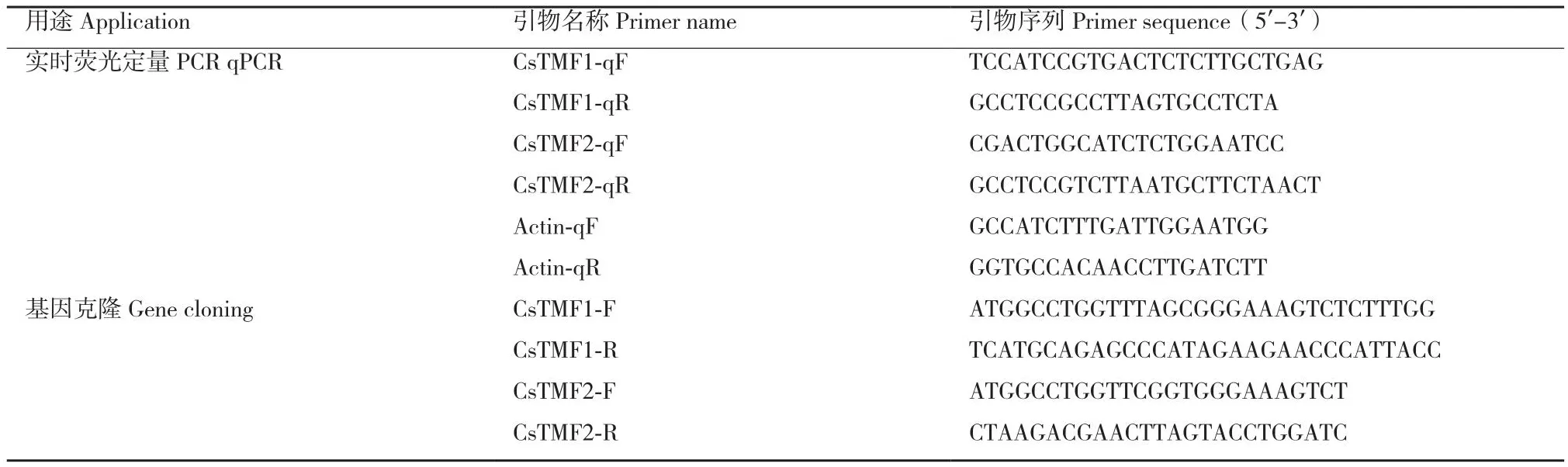
表1 PCR 引物信息Table 1 Primer sequences for PCR
1.3 茶树CsTMFs家族保守基序与结构预测
使用TBtools软件对测序所得的序列进行开放阅读框的查找及氨基酸序列的翻译;使用MEME(Multiple Expectation Maximization for Motif Elicitation)在线分析工具(http://meme.sdsc.edu/meme4_3_0/intro.html)对CsTMFs的蛋白保守基序进行分析,基序的最大数值设为10,其余参数均为默认值,通过在线数据库Pfam对motif进行结构域分析。使用Gene Structure Display Server(http://gsds.gao-lab.org/)绘制CsTMFs家族的基因结构图。
1.4 茶树CsTMFs生物信息学分析
使用在线软件Prot Param(http://web.expasy.org/ prot param)推导蛋白质的相对分子质量等理化性质;用在线软件SOPMA(https://npsa-prabi.ibcp.fr/)对CsTMF1进行蛋白二级结构预测;从基因组数据库中查找CsTMFs家族2个基因的位置信息,使用TBtools软件对目的基因进行染色体定位分析;通过WoLFPSORTII在线软件(https://www.genscript.com/tools/wolf-psort)进行亚细胞定位;通过STRING在线软件(https://string-db.org),以在OsTMF的互作网络为基础预测茶树CsTMF潜在的互作关系。运用NCBI数据库中(https://www.ncbi.nlm.nih.gov)BLAST程序下载多个物种的TMF蛋白序列,使用MEGA 7软件进行系统进化树的构建,采用邻接法(neighbor-joining)构建,Bootstrap检验值设置为1 000;并使用MAFFT version 7(https://mafft.cbrc.jp/alignment/software/adjustdirection.html)对不同植物的TMF同源蛋白进行序列比对,用在线软件Sequence Manipulation Suite:(https://groups.molbiosci.northwestern.edu/)进行颜色对齐。
1.5 茶树CsTMFs的差异表达分析
从公共数据库[14]下载CsTMFs在茶树芽、花、果实、嫩叶、成熟叶、老叶、根和茎中组织表达情况的转录组数据,使用TBtools软件绘制热图。
低温胁迫:选取品比园中舒茶早新鲜枝条为材料,进行(4±1)℃低温胁迫处理,分别于0、3、6、9、12、24、36和48 h摘取3-5个枝条第二叶,作为低温胁迫的样品,并设置对照组于人工气候室(24±2)℃环境下同时间点取样。
干旱处理:将稳定生长于人工气候室(24±2)℃的舒茶早枝条转移至PEG-6000溶液(浓度20%)进行干旱处理,分别于0、1、2、4、8、12、24和48 h摘取3-5个枝条第二叶,液氮速冻,-80℃保存备用,作为干旱胁迫样品。
使用NCBI Primer-BLAST在线软件(http://www.ncbi.nlm.nih.gov/tools/primer-blast/)设计CsTMF1与CsTMF2的荧光定量引物(表1),以茶树的β-actin(登录号KJ946252)为内参基因。进行qPCR反应。反应体系为SYBR Green I Master 10 μL、cDNA 2 μL、上下游引物各0.8 μL和ddH2O 6.4 μL。反应程序为95℃ 30 s;95℃ 5 s,60℃ 30 s,40个循环;95℃ 5 s,65℃ 60 s,95℃ 1 s,采用2-ΔΔCT法[15]进行定量数据分析。使用GraphPad prism 7软件进行作图,采用IBM SPSS Statistics 21单因素的方差分析对CsTMFs表达水平的差异显著性进行检验。
2 结果
2.1 CsTMF基因家族筛选与鉴定
以水稻OsTMF氨基酸序列为模板,在TPIA进行在线BLASTP比对,根据比对结果下载候选蛋白序列。通过Interpro(https://www.ebi.ac.uk/interpro/)对序列进行结构域分析,结果(图1)显示,有2个氨基酸序列包含TMF-DNA-bd(pfam12329)和TMFTATA-bd(pfam12325)保守结构域,获得茶树TMF同源氨基酸两个CSS0015824和CSS0047293,其中,CSS0015824与CSS0047293的相似度为86.4%。
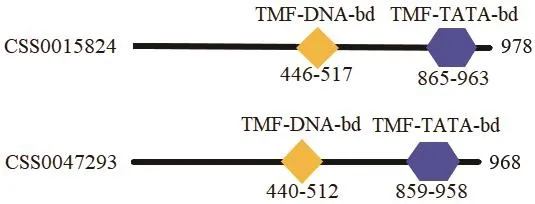
图1 CsTMFs蛋白的结构域分析Fig.1 Domain analysis of CsTMFs proteins
2.2 CsTMFs的克隆与测序
以福鼎大白茶叶样品的cDNA为模板进行扩增,获得2个3 000 bp条带,分别命名为CsTMF1和CsTMF2(图2)。经测序可知,CsTMF1序列全长3 052 bp,与茶树基因组预测的CsTMF1(GenBank:CSS0015824.1)序列相似度达99.6%,该序列包含2 934 bp开放阅读框,共编码977个氨基酸;CsTMF2序列全长2 908 bp,与茶树基因组预测的CsTMF2(GenBank:CSS0047293.1)序列相似度达99.9%,该序列包含2 904 bp开放阅读框,共编码967个氨基酸基,CsTMF1与CsTMF2的相似度为86.7%。
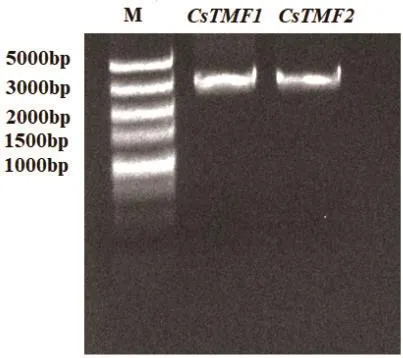
图2 CsTMFs扩增电泳图Fig.2 CsTMFs amplification electropherogram
2.3 茶树CsTMFs家族保守基序与结构预测
利用MEME软件识别2个CsTMF蛋白的10个保守Motif(图3-A,表2),可以看出2个CsTMF的保守基序分析结果较为相似,其中,Motif 5和Motif 7分别包含TMF的2个保守结构域。根据基因组数据绘制2个基因的结构图(图3-B),可以看出2个CsTMF基因均包含18个内含子。
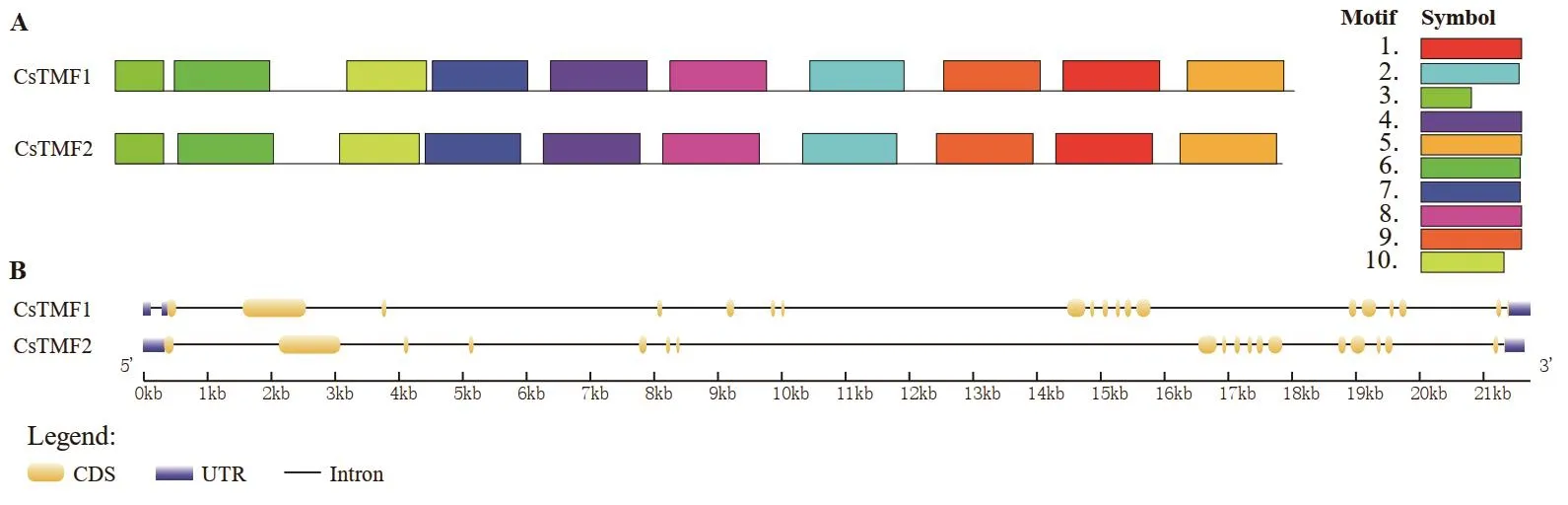
图3 CsTMFs保守基序(A)及基因结构(B)Fig.3 Conserved motifs(A)and gene structures(B)of CsTMFs members

表2 CsTMFs蛋白序列中10个保守基序的详细信息Table 2 Detailed information of 10 motifs in the CsTMFs proteins
2.4 CsTMFs蛋白质理化性质分析
通过在线软件Prot Param预测,CsTMF1蛋白的原子总数为15 282,分子式为C4660H7595N1367O1631S29,分子量为109.798 kD,氨基酸长度为978 aa,属于小分子蛋白;理论等电点为4.73,表明该蛋白质为酸性蛋白。带负电荷的残基总数(Asp+Glu):203,带正电荷的残基总数(精氨酸+赖氨酸):128。不稳定指数(II)为65.44,将蛋白质归类为不稳定。脂肪族指数:73.50;亲水性平均值(GRAVY):-0.840。
CsTMF2蛋白的原子总数为15 139,分子式为C4623H7530N1352O1605S29,分子量为108.662 kD,氨基酸长度为968 aa,属于小分子蛋白;理论等电点为4.78,表明该蛋白质为酸性蛋白。带负电荷的残基总数(Asp+Glu):196,带正电荷的残基总数(精氨酸+赖氨酸):125。不稳定指数(II)计算为62.85,将蛋白质归类为不稳定。脂肪族指数:75.56;亲水性平均值(GRAVY):-0.797。
2.5 CsTMFs二级结构预测、染色体定位和亚细胞定位预测
使用在线工具SOPMA进行蛋白二级结构预测,CsTMF1(图4-A)与CsTMF2(图4-B)中均包含大量的α-螺旋,分别占比68.20%和70.87%。CsTMF1包含随机线圈(23.01%),以及少量的延伸链(5.01%)和β-转角(3.78%)。CsTMF2包含随机线圈(19.52%),少量的延伸链(5.17%)和β-转角(4.44%)。
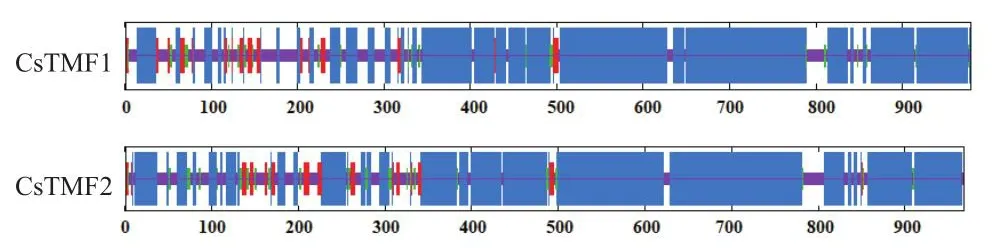
图4 CsTMFs蛋白二级结构预测Fig.4 Secondary structure prediction of CsTMFs protein
从基因组数据库中查找CsTMFs家族2个基因的位置信息,使用TBtools软件对目的基因进行染色体定位分析(图5),CsTMF1与CsTMF2分别位于第2和第15染色体上。
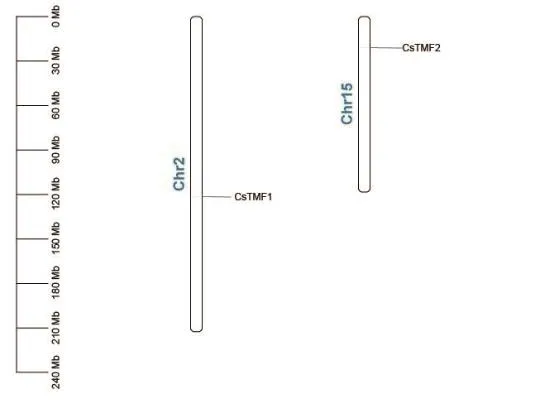
图5 CsTMFs家族染色体定位Fig.5 Chromosome location of CsTMFs family
使用WoLF PSORTII进行亚细胞定位,结果显示,CsTMF1有可能定位在细胞核上,CsTMF2可能定位在细胞核或者细胞质内。
2.6 CsTMFs互作网络模型预测
通过STRING在线预测了CsTMFs蛋白潜在的互作关系(图6),以水稻蛋白质数据库为参考,OsTMF主要与Ras相关蛋白Rab-6A、GTP结合蛋白Rab6相互作用,相关系数均为0.980。
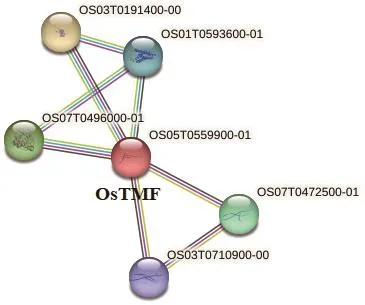
图6 STRING软件以水稻蛋白质数据库为参考预测CsTMFs蛋白互作网络模型Fig.6 Interaction network of CsTMFs based on Oryza sativa protein database by STRING software
2.7 CsTMFs系统发育关系分析
为了分析茶树CsTMFs与其他植物同源蛋白的进化关系,选取水稻、红猕猴桃、杨树、拟南芥、玉米等20个物种中与CsTMF同源性较高的蛋白序列,使用MEGA 7软件,采用邻接法(Neighbor-joining)进行系统进化树的构建(图7),茶树CsTMFs与红猕猴桃和中华猕猴桃TMF的亲缘关系较近,而与鸡血藤、大枣的TMF等亲缘关系较远。
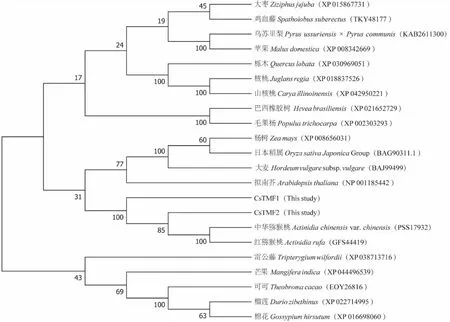
图7 茶树CsTMFs系统进化树分析Fig.7 Phylogenetic tree analysis of CsTMFs from tea plant and other plant species
2.8 不同物种TMF蛋白同源性比对
通过公共数据库下载5种不同物种的TMF蛋白序列(表3),使用MAFFT version 7对2个CsTMF蛋白和其他不同物种TMF蛋白进行序列比对(图8),6个不同物种中TMF-TATA-bd与TMFDNA-bd结构域仅有较少数氨基酸位点发生突变。

图8 茶树与其他植物TMF的多重序列比对Fig.8 Multiple sequence alignment of tea plant TMF with other plants
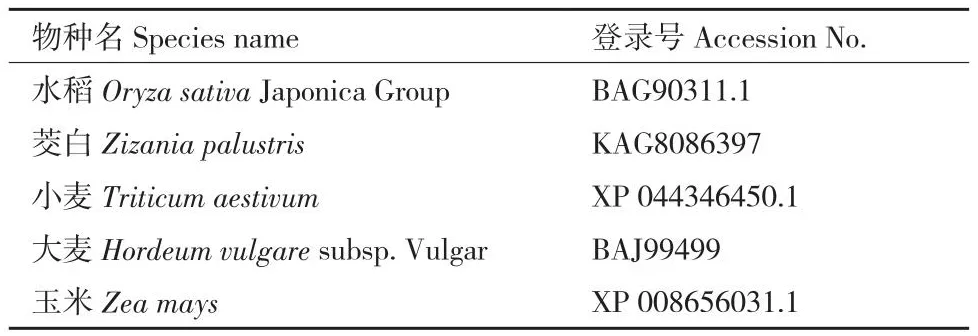
表3 不同物种TMF登录号Table 3 Accession numbers of different species of TMF
2.9 CsTMFs表达分析
从公共转录组数据库下载CsTMF1与CsTMF2在茶树中不同组织(分别为芽、花、果实、嫩叶、成熟叶、老叶、根和茎)表达数据,对CsTMF1与CsTMF2的组织表达模式进行分析(图9),CsTMF1与CsTMF2在茶树的各个组织中均有表达,其中,CsTMF1在花和茎中的表达量最高,果实和老叶中表达最少;CsTMF2在成熟叶和茎中的表达较高,芽和嫩叶中表达量最少。2个基因在成熟叶、老叶和根中的表达量均相当,CsTMF2总体表达量低于CsTMF1。

图9 CsTMF1与CsTMF2在茶树不同组织中的表达情况Fig.9 Expressions of CsTMF1 and CsTMF2 in different tissues of tea plant
采集冷处理和干旱处理后的舒茶早叶片进行qPCR试验,用来分析不同胁迫下CsTMFs的表达情况。数据分别以不同处理舒茶早0 h表达量为1进行计算(图10-A),CsTMF1在4℃处理3 h后表达量开始增高,并在第6小时达到最高值随后开始显著降低(P<0.05),在第12-36小时表达量较平稳,第48小时开始显著升高。CsTMF1在25℃处理后的趋势与4℃相似,均呈现先增高再降低的趋势,但25℃处理是在9 h达到最高值。CsTMF2在4℃处理后先显著降低(P<0.05),6 h有升高趋势但是没有达到显著差异(P>0.05),随即表达量降低,在第48小时开始显著升高。CsTMF2在25℃处理后则出现表达量先降低再升高再降低的趋势,在9 h达到最高值。
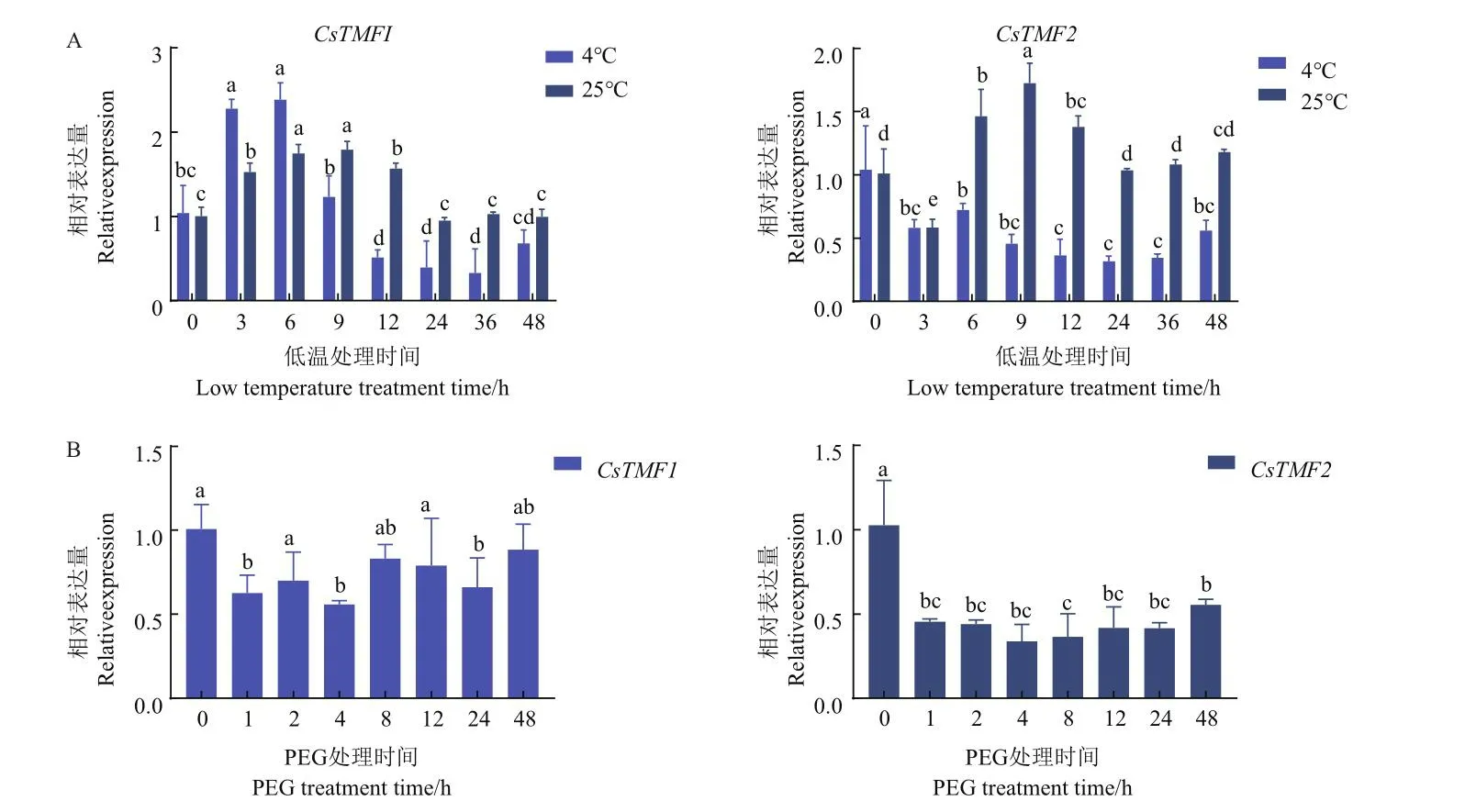
图10 CsTMFs在低温和干旱胁迫下的表达模式Fig.10 Expression pattern of CsTMFs gene under drought stress and low temperature
CsTMF1在PEG处理后1 h表达量先显著降低(P<0.05,图10-B),随后表达量出现不规律的波动。CsTMF2在PEG处理后呈现较长时间的显著降低,在48 h后有升高趋势但未达到显著差异,与0 h的表达量相比仍显著降低(P<0.05)。
3 讨论
前人研究表明,在人、小鼠、鱼、酵母、水稻等物种中,TMF为单拷贝[9],本研究对茶树TMF进行筛选与鉴定,结果表明,茶树中CsTMFs家族共有2个成员——CsTMF1与CsTMF2。结构域分析发现CsTMFs均具有典型的TMF-DNA-bd和TMFTATA-bd结构域,这可能是由于在2 800万年前发生过一次全基因组重复事件,导致许多与茶树抗逆性和次级代谢合成相关的转录因子家族发生了显著扩张[16],进一步说明茶树基因组较为庞大且杂合程度较高。本研究进一步克隆茶树CsTMFs的CDS区全长,证明CsTMFs在茶树中真实存在。对茶树CsTMFs与其他物种的TMF结构域进行对比发现,TMF-DNA-bd和TMF-TATA-bd结构域具有高度的相似性,说明该结构域在不同物种中高度保守,与前人的研究结果一致[13]。
大量研究表明,TMF参与基因表达调控、蛋白降解、细胞内的膜运输等,并在精子发育、肿瘤发生、细胞的代谢应激也发挥关键作用[9]。然而,TMF至今还未被纳入传统意义上的转录调控因子之中,表明该基因在植物中参与转录调控的分子机制尚不明确,亟待解决。通过STRING在线预测了OsTMFs蛋白潜在的互作关系,发现其与Ras相关蛋白Rab-6A、GTP结合蛋白Rab6相互作用,相关系数均为0.980。Rab家族不仅与细胞极性生长有关,还在植物的生物胁迫和非生物胁迫(干旱、低温和盐胁迫等)中起到重要作用[17],因此,推测CsTMFs可能与植物抗逆相关。
本研究发现CsTMF1在花和茎中的表达量最高;CsTMF2在成熟叶和茎中表达量高,在幼嫩部位表达量低,2个基因在相同组织中表达量的差异可能预示着两个基因的功能发生了分化。qPCR结果说明,CsTMF1在4℃和25℃处理时的表达量均为先上升后下降再上升,但是由于低温影响,使表达量峰值从9 h提前到6 h,表明CsTMF1的表达受到低温的诱导。CsTMF2的表达量在25℃处理时为先降低再升高再降低的趋势,在4℃处理时呈下降趋势,说明CsTMF2可能响应低温的影响,使CsTMF2的表达在本该升高的时间点降低,即低温抑制了CsTMF2的表达。2个CsTMF基因在PEG处理后的表达量变化情况也不同,CsTMF1整体呈波动状态,无明显趋势。CsTMF2呈现降低的趋势,表明CsTMF2的表达可能受到PEG的抑制,与茶树响应干旱胁迫有关。
研究表明,冷胁迫下OsTMF的表达水平上升,且OsTMF与TATA顺式元件的结合能力增强。细胞壁对于植物的生长发育有着至关重要的作用,主要表现在结构支持作用、抵御逆境作用[18],通常认为细胞壁增厚可能对细胞起保护作用。OsTMFOE水稻在2 d的冷胁迫后,次生细胞壁纤维素含量增加,次生细胞壁厚度增加,对寒冷的抵抗变弱。OsTMF-OE水稻在受寒冷刺激后,OsTMF特异性结合OsBURP16、水稻纤维素合酶亚基A(cellulose synthase A,CesA)OsCesA4和OsCesA9启动子中含TATA的区域的能力增强[10]。OsCESA4、OsCESA7和OsCESA9组成水稻次生壁纤维素复合酶[19],OsCesA4和OsCesA9启动后,纤维素含量增加,纤维素是细胞壁的主要成分,OsTMF-OE水稻的次生细胞壁增厚。在OsTMF的调控下OsBURP16启动,其编码一种前体果胶降解酶。OsBURP16过表达导致水稻细胞间黏附减少和耐寒性下降[20],导致细胞壁中果胶降解,细胞间黏附性变低,这可能是OsTMF-OE水稻抗寒性变弱的另一个原因。一般情况下,水稻通过增加果胶含量减少纤维素合成,以应对寒冷胁迫,因此,OsTMF对水稻耐寒性有负面影响。关于茶树在冷胁迫下CsTMFs与细胞壁组成成分变化的关系还在进一步探究中。
4 结论
在茶树全基因组中共鉴定出2个TMF家族基因,CDS区长度分别为2 934和2 904 bp,分别位于第2和第15染色体上,该家族基因具有组织表达特异性。CsTMFs响应茶树冷胁迫和干旱胁迫。