黄颡鱼养殖群体遗传多样性与遗传结构的微卫星分析
李燕平,覃川杰,吕云云,龚 全,王 均,颉 江,文正勇
(1.内江师范学院,长江上游鱼类资源保护与利用四川省重点实验室,四川内江 641112;2.四川省农业科学院水产研究所,成都 611730)
黄颡鱼(Pelteobagrusfulvidraco)隶属于鲶形目(Siluriformes)鲿科(Bagridae),广泛分布于长江、黄河、珠江、沅江及黑龙江等各水域[1,2],是我国重要的经济鱼类和主要养殖对象[3]。然而,因人工繁育随意留种可能出现的近亲繁殖、广泛交易、增殖放流、自然逃逸等原因有可能使种群的遗传多样性水平降低。库喜英等[4]以线粒体DNA为标记,对采自长江、珠江、辽河、闽江、富春江和韩江6个水系的21个采样点的黄颡鱼进行遗传结构分析,结果表明黄颡鱼的群体遗传多样性为中度多态,群体间遗传分化极小,已缺乏明显的地理结构。
微卫星DNA即短串联重复序列(short tandem repeat,STR)或简单重复序列(simple sequence repeat,SSR),广泛存在于真核生物基因组中。与传统分子标记相比,它具有突变速率快、呈共显性、稳定性好、易检测等优势,已被广泛应用于水生生物遗传多样性评估、种群遗传结构分析、物种分类、亲缘关系分析、标记辅助育种以及遗传图谱构建等[5-8]。李大宇等[9]利用微卫星标记对6个群体(吉林省大安市月亮湖野生黄颡鱼、四川野生黄颡鱼、黑龙江省哈尔滨市松花江段黄颡鱼、湖北省荆州市长湖野生黄颡鱼亲本以及子代和天津市换新国家级原良种场黄颡鱼1号)的黄颡鱼遗传多样性及亲缘关系进行解析。胡玉婷等[10]采用微卫星标记分析了长江、淮河两水系安徽区段黄颡鱼群体的遗传多样性。
目前,黄颡鱼遗传多样性及群体亲缘关系的研究主要集中在自然地理群体。随着长江十年禁渔计划的实施,天然水域亲本采集的难度变大。因此,了解和掌握人工养殖群体的黄颡鱼种群结构、群体遗传多样性,有助于为黄颡鱼良种选育、苗种繁育等提供理论依据。本实验基于8对微卫星标记对14个黄颡鱼养殖群体的遗传多样性和遗传结构进行评估,以期为黄颡鱼家系建立,提高黄颡鱼遗传育种潜力、缩短育种年限和加快新品种的选育提供数据支撑。
1 材料与方法
1.1 实验室动物
本实验所用黄颡鱼取自四川省14个养殖群体,通过咨询其亲本来源,确定其亲本群体来自于江苏溧阳(HJ)、四川邛崃(HQ)、广东佛山(LG)、安徽安庆(YB)、湖北仙桃(YH)、浙江德清(YZ)、湖南岳阳(DB)、湖北荆门(WH)、四川宜宾(XB)、湖北咸宁(XH)、江苏盐城(ZJ)、四川乐山(ZL)、浙江湖州(LZ)和广西南宁(RB),每个亲本群体的详细信息见表1。其中,除HJ,YH和XH分别采集到17,13和14尾样本外,其余11个群体每个群体的样本数均为15尾,共采集209尾样本。取部分尾鳍组织保存于95%乙醇中,置于-20℃冷藏备用。
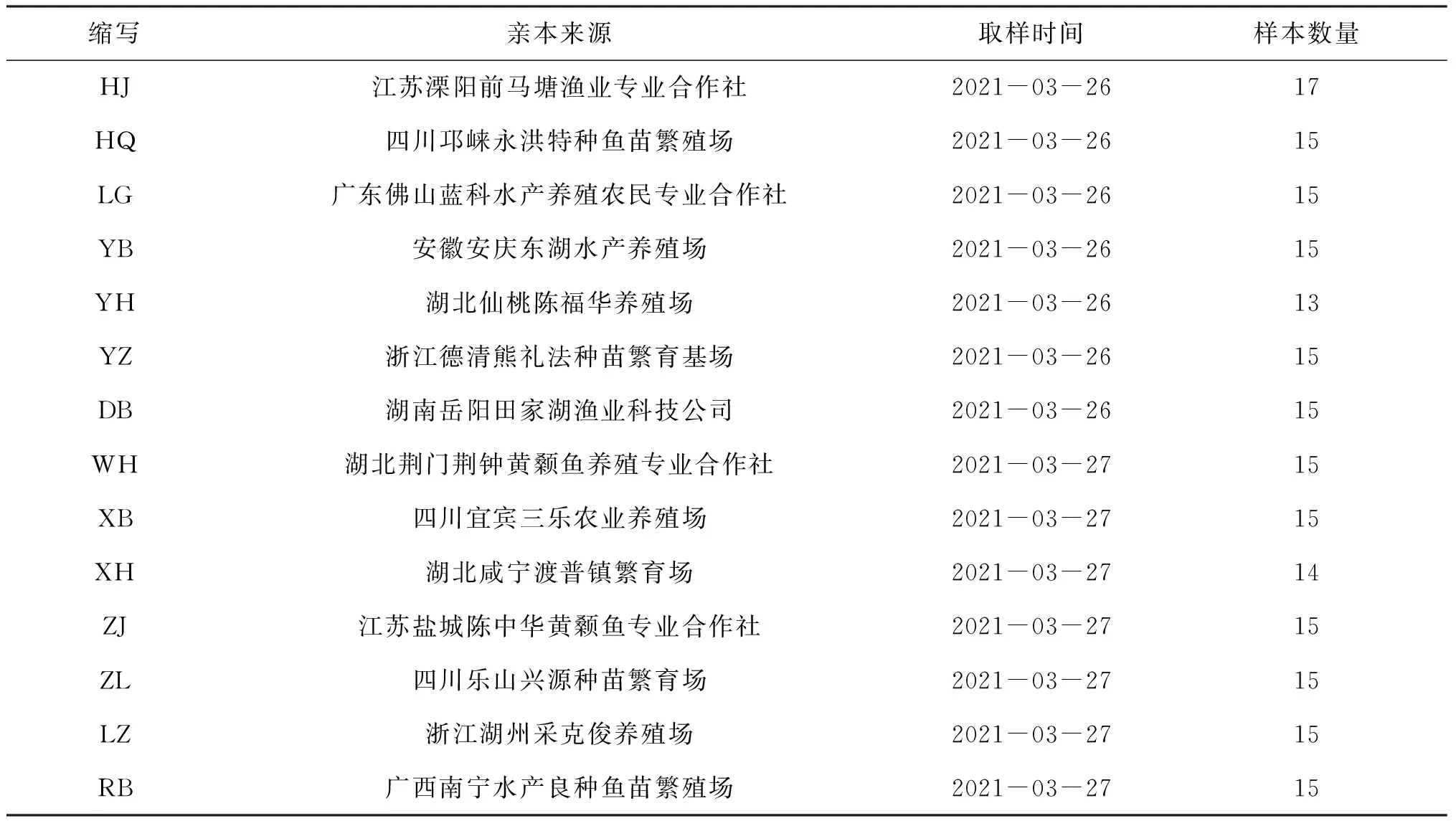
表1 14个养殖黄颡鱼群体的采样信息Tab.1 Sampling information of 14 cultured populations of P.fulvidraco
1.2 实验方法
1.2.1 基因组DNA提取和质量检测
对所有样本采用酚氯仿抽提法获得基因组DNA,具体DNA提取步骤参考文献[11]。对提取出的DNA,首先用1%琼脂糖凝胶电泳检测DNA的质量,并使用Nanodrop 2000(赛默飞,美国)进一步检测DNA的浓度和纯度,满足PCR扩增质量要求的DNA保存于-20 ℃备用。
1.2.2 多态性微卫星筛选、扩增和分型检测
本研究参考文献[9]随机挑选11对引物用于本研究的黄颡鱼养殖群体的多态性初筛。随机挑选的11对引物的具体信息见表2。经过在14个群体,每个群体挑选8个个体进行预实验,结果显示有3对引物(SS37,SS81和SS89)在本研究的样本中扩增失败或呈单态,最终确定8对引物用于本研究,并在这8对引物的正向5′端加上FAM荧光标记,方便后续进行毛细管电泳分型。本实验所用的引物及荧光染料由苏州金唯智生物科技有限公司合成。

表2 用于本研究14个黄颡鱼养殖群体的引物序列情况Tab.2 Microsatellite marker sequences used in 14 cultured populations of P.fulvidraco
PCR扩增体系为:20 μL反应体系包含2 μL 10×Pfu Buffer(含有Mg2+),10 μmol/L正反向引物各0.8 μL,0.8 μL dNTP(10 μmol/L),1.5 μL基因组DNA(20 ng/μL)和0.4 μL Taq酶,最终用去离子水补到20 μL。采用降落PCR扩增,具体反应程序:95 ℃预变性5 min;95 ℃变性30 s、61 ℃退火30 s、72 ℃延伸30 s,共10个循环(每个循环退火温度降低0.2 ℃);95变性30 s、58 ℃退火30 s、72 ℃延伸30 s,共32个循环(每个循环退火温度降低0.1 ℃);72 ℃延伸5 min;10 ℃保存。PCR扩增产物采用聚丙烯酰胺凝胶电泳(polyacrylamide gel electrophoresis,PAGE)进行微卫星位点多态性检测,在6.0%的非变性聚丙烯酰胺凝胶上进行。非变性聚丙烯酰胺凝胶制作的标准步骤参照《分子克隆实验指南》进行[12]。将1.5 μL PCR产物与6×上样缓冲液混合后依次加样于凝胶的加样孔内,于220 V恒定电压下电泳1.5~2.5 h。电泳结束后,用EB染色,并在紫外凝胶成像系统中扫描、保存。通过pBR322 DNA/MspI分子质量标准对比初步确定等位基因大小。将检测到目的条带的产物上样于ABI3730xl分析仪(苏州金唯智生物科技有限公司)进行毛细管电泳,检测产物片段大小。
1.3 数据分析
根据14个群体在8个微卫星位点的基因分型数据,首先使用CONVERT 1.3将其转化为STRUCTURE,POPGENE以及ARLEQUIN等软件能够识别的格式[13],再利用POPGENE 1.3.1[14]计算14个群体各个微卫星位点的等位基因数(Na)、观测杂合度(Ho)、期望杂合度(He)、有效等位基因数(Ne)、Hardy-Weinberg平衡遗传偏离指数和Nei′s标准遗传距离等参数;利用CERVUS 3.0.3计算多态信息含量(PIC)[15];使用ARLEQUIN 3.5计算两两群体之间的遗传分化指数(Fst)并进行分子方差分析(analasis of molecular variation,AMOVA)[16];使用MEGA 7软件基于Nei′s遗传距离构建UPGMA聚类树[17];最后利用STRUCTURE 2.3.4中的贝叶斯聚类[18],基于混合模型找到聚类值(K值),MCMC链长设置为20 000,每次运行舍弃的样本数设置为2 000,遗传分组K值设置为1~14,每次分析重复运行10次,将运行的结果提交到Structure Harvester确定最佳的K值[19],再用CLUMPP 1.1.2软件进行重复聚类分析[20],最后利用DISTRUCT 1.1软件将CLUMPP的结果进行可视化[21]。
2 结果与分析
2.1 14个黄颡鱼养殖群体的遗传多样性分析
8个位点在14个群体中的等位基因数、观测杂合度、期望杂合度、多态信息含量、有效等位基因数,以及各个群体的平均值见表3。14个黄颡鱼养殖群体中,平均观测杂合度在0.633~0.717之间,其中HQ和WH群体最低,最高的为XB和LZ群体;平均期望杂合度在0.358~0.749之间,最低群体为RB,最高群体为LG;平均多态信息含量在0.893~0.681之间,多态信息含量最低的为WH群体,最高的为YB群体,14个群体的平均多态信息含量均大于0.5,说明14个黄颡鱼养殖群体的遗传多样性均处于高度多态的水平;平均有效等位基因数在5.160~6.882之间,RB群体最低,DB群体最高(表3)。

表3 14个黄颡鱼养殖群体的遗传多样性信息Tab.3 Information on genetic diversity in four-teen populations of P.fulvidraco

续表3
2.2 黄颡鱼养殖群体遗传分化关系
对14个黄颡鱼养殖群体的分子方差分析(AMOVA)结果显示,3.69%的遗传变异来自群体间,3.93%来自群体内个体间,92.38%来自所有个体间,表明群体间的遗传变异主要来自所有个体间(表4)。

表4 基于8个微卫星标记的分子方差分析结果Tab.4 Result of AMOVA based on 8 microsatellite loci
14个黄颡鱼群体的遗传分化指数在-0.009~0.116之间,其中RB群体和WH群体的遗传分化最大(Fst=0.116),ZL群体和XB群体的遗传分化最小(Fst=-0.009)。根据WRIGHT[22]对遗传分化指数(Fst)的划分标准,当Fst<0.05时,表明群体间遗传分化较小;当0.05
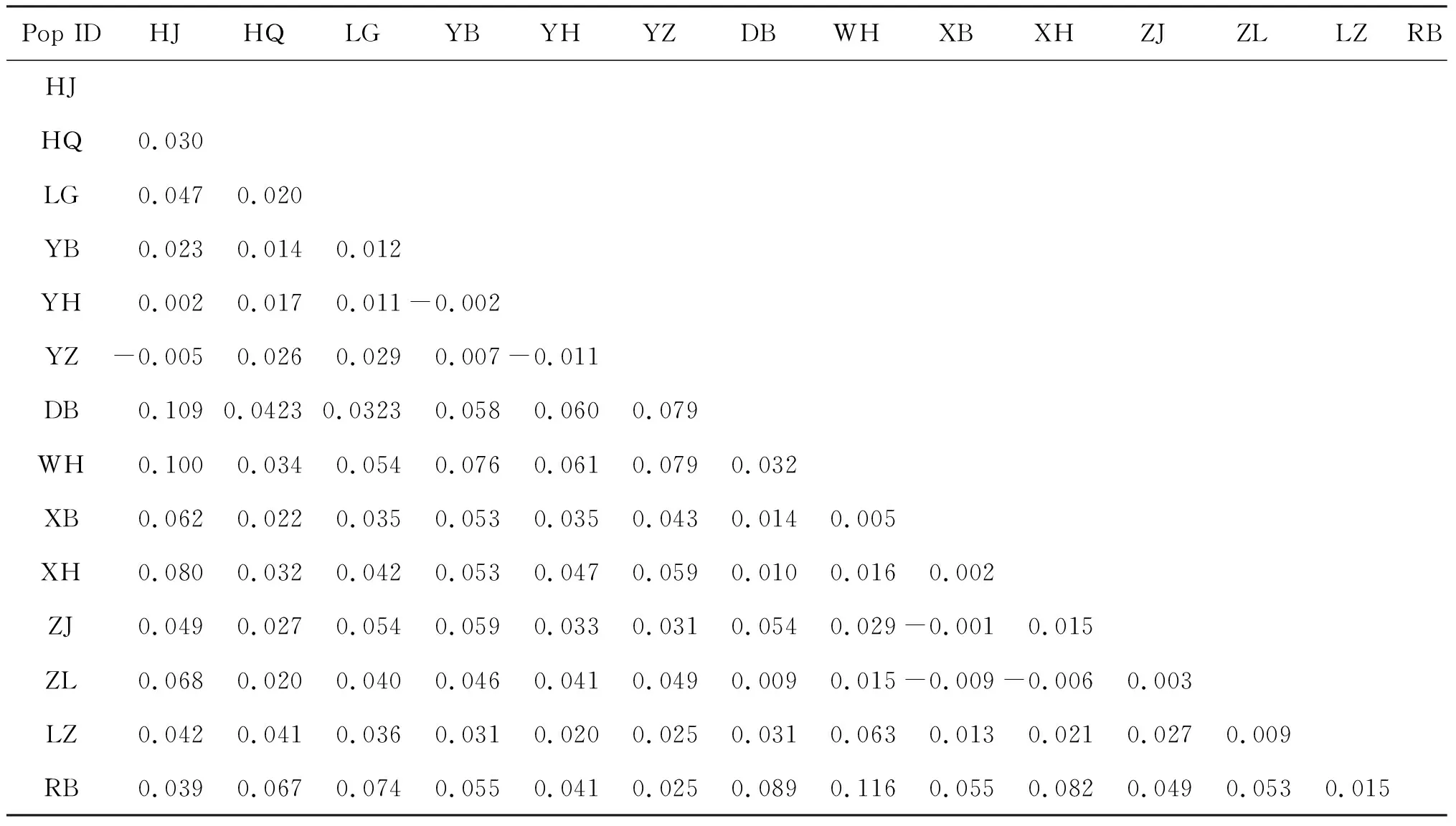
表5 14个黄颡鱼养殖群体的遗传分化指数(Fst)Tab.5 Pair-wise Fst values calculated from eight microsatellite loci among 14 populations of P.fulvidraco
8个微卫星位点的平均群体内近交系数(Fis)和总近交系数(Fit)分别为0.007 1和0.072 0(表6),说明黄颡鱼群体内存在一定程度的近交现象。各基因座的基因交流均值为3.573。

表6 黄颡鱼养殖群体8个微卫星位点的F-统计检验Tab.6 F-statistics for P.fulvidraco populations at eight microsatellite loci
2.3 遗传结构聚类分析
14个黄颡鱼养殖群体的遗传相似度和Nei′s遗传距离的结果表明,群体间的Nei′s遗传距离在0.055 0~0.431 4之间变化,遗传相似度在0.649 6~0.946 0之间(表7)。其中,HJ和DB群体的遗传距离最远,遗传相似度最低,而XB和ZL的遗传距离最近,遗传相似度最高。说明HJ和DB群体的遗传分化最大,亲缘关系最远,而XB和ZL群体的亲缘关系最近。
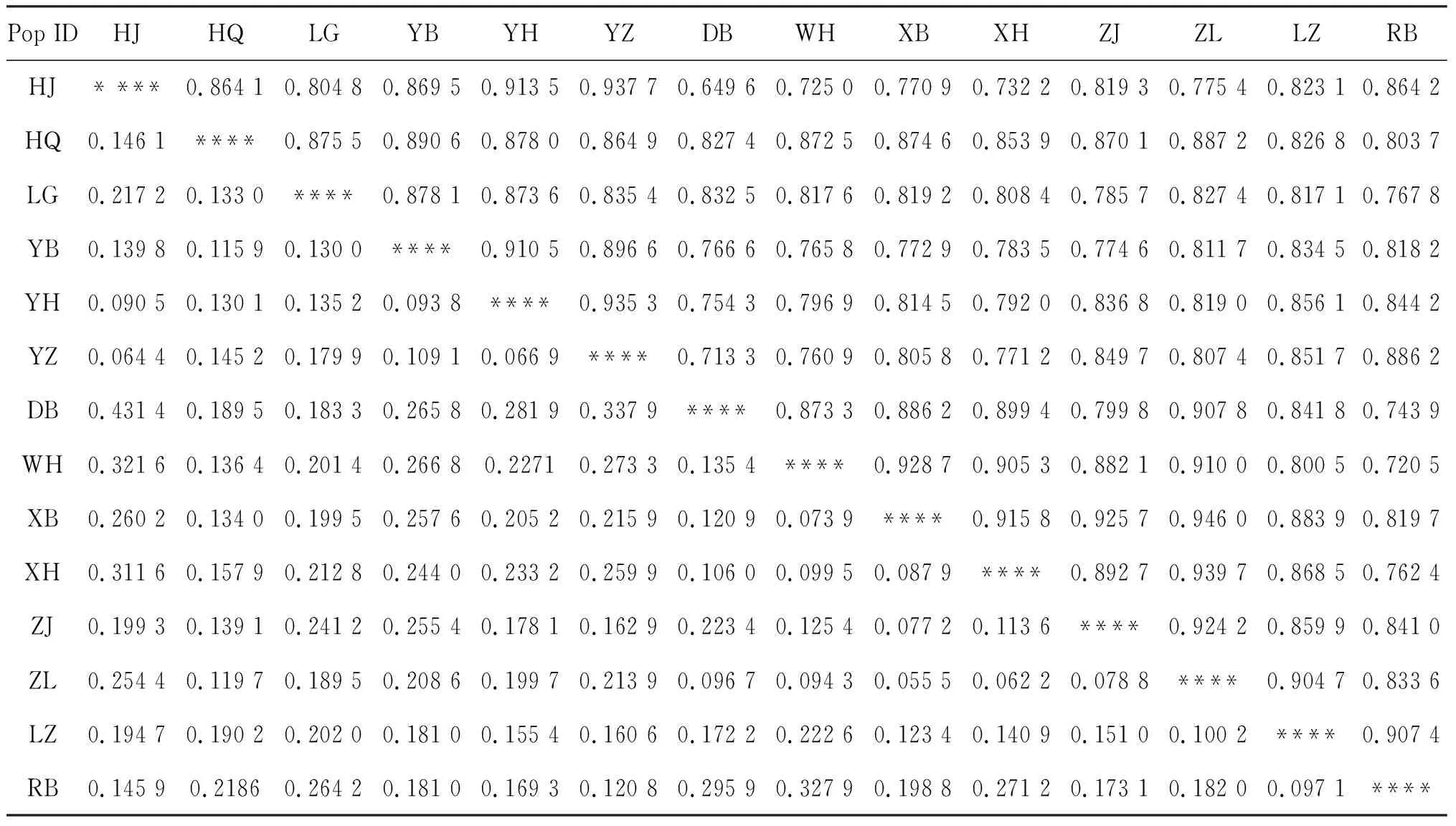
表7 14个黄颡鱼养殖群体的遗传相似性系数(右上角)和Nei′s遗传距离(左下角)Tab.7 Matrix of pair-wise genetic similarity coefficient(above diagonal)and Nei′s genetic distance(below diagonal)among 14 populations of P.fulvidraco
基于Nei′s遗传距离构建的UPGMA聚类树结果显示,HJ、YZ、YH、YB首先聚为一个分支,HQ和LG分别聚为一个分支,三个分支聚类后再与LZ和RB聚在一起,最后与余下的DB、ZJ、WH、XH、XB和ZL聚在一起,与遗传距离的结果一致(图1a)。在黄颡鱼Structure群体遗传结构分析中,Delta K随着亚群数K的增加而变动,K=5时,Delta K出现最大的拐点(图1b),表示K=5是最可能的模型,推测本研究的14个黄颡鱼养殖群体分为5个亚群最合适(图1c),与UPGMA聚类树的结果一致。
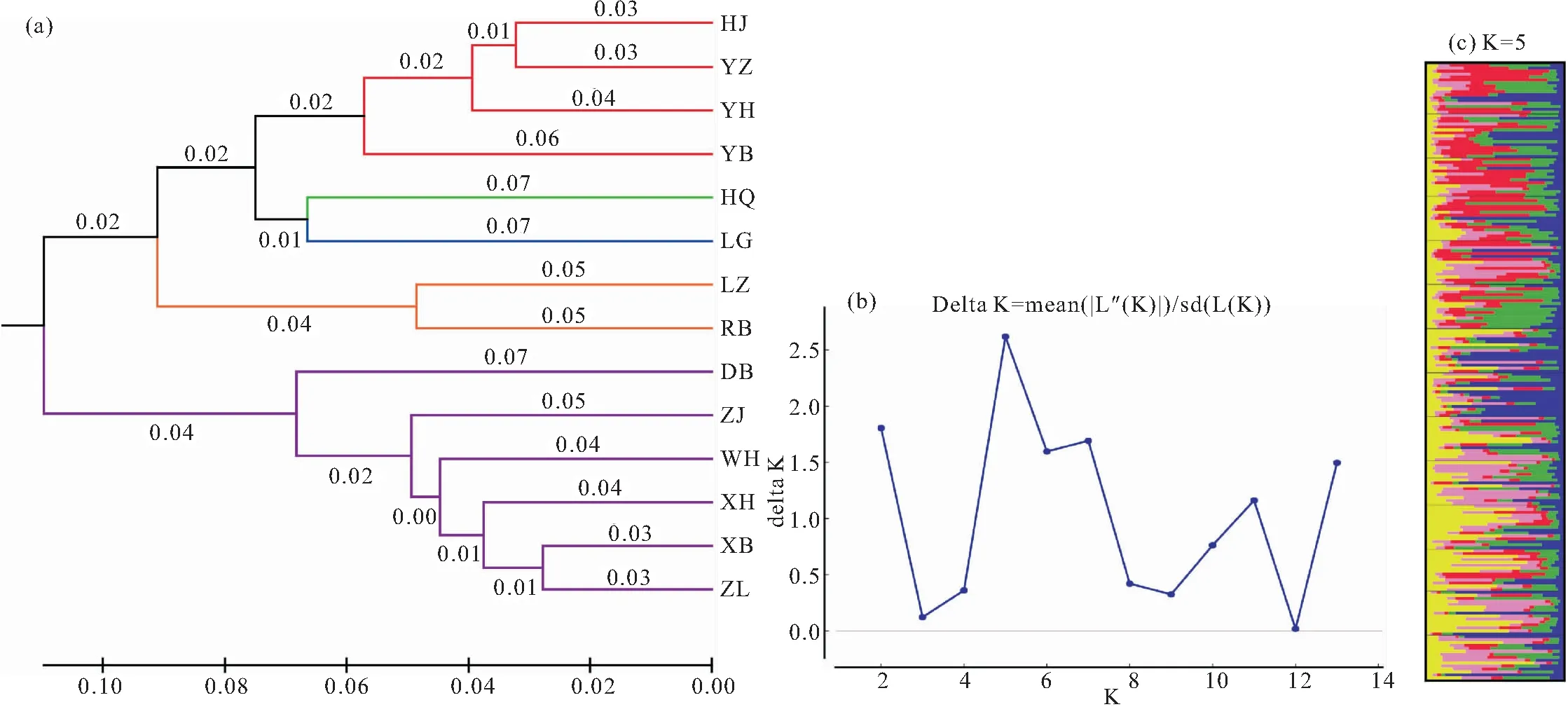
图1 黄颡鱼群体的遗传结构聚类分析Fig.1 Genetic clustering analysis among populations of P.fulvidraco(a)基于Nei′s遗传距离构建的UPGMA聚类图;(b)Delta K值的变化曲线;(c)K=5时的structure聚类结果,不同颜色代表不同的簇。
3 讨论
3.1 黄颡鱼养殖群体的遗传多样性水平
物种间亲缘关系越近,SSR引物的通用性越高。本研究选用11对前期通过磁珠富集法结合同位素杂交法在黄颡鱼野生群体中开发出的微卫星引物[9],检测黄颡鱼养殖群体中的遗传多样性,结果显示8对多态性引物扩增成功且呈多态,通用率为72.73%,表现出较为理想的扩增结果,说明野生黄颡鱼群体中开发出的微卫星位点可用于黄颡鱼养殖群体的遗传多样性和种群结构的评估、保护遗传学研究。
生物群体的遗传多样性是评价物种资源状况的一个重要依据。李大宇等[9]发现吉林、四川、黑龙江、湖北等省天然水系野生的6个黄颡鱼群体的多态性指标均适中。王婷婷等[23]报道了湖州菱湖养殖场(取自太湖支流大海漾群体的后代)、苏州澄湖、太湖和四川眉山养殖场的4个黄颡鱼群体的遗传多样性及亲缘关系,结果发现4个群体的多态性指标处于中等水平,群体间遗传分化不明显,4个群体的亲缘关系较近。目前有关黄颡鱼遗传多样的研究主要集中在不同地理分布群体,有关人工养殖群体种质资源评价的研究较少。黄颡鱼已是中国广泛养殖的小型名优经济类,具有繁育场多、交易广泛、逃逸和放流频繁等特点。因此,探讨人工养殖群体的遗传多样性是持续利用种质资源的前提。
多态信息含量PIC用来描述微卫星位点遗传变异程度,含量的高低与变异程度成正比。根据BOTSTEIN等[24]对PIC的划分标准,当PIC<0.25,该位点为低度多态;当0.25
基因杂合度也是衡量群体变异的最适参数之一,代表群体中杂合子在某个位点上的频率,体现群体的进化程度[27]。TAKEZAKI等[28]通过微卫星标记研究发现,当期望杂合度和观测杂合度大于0.3时,表明群体具有遗传多样性的基础。本研究14个群体的平均期望杂合度Ho在0.633~0.717之间,平均观测杂合度He在0.358~0.749,表明14个养殖黄颡鱼群体具有较高的遗传多样性,有较好的选育潜力。14个黄颡鱼养殖群体有12个群体(HJ、HQ、LG、YB、YH、YZ、DB、WH、XB、XH、ZL和LZ)的平均观测杂合度低于平均期望杂合度,推测可能是由于普遍存在近交情况而使得群体发生纯合,进而导致杂合度降低。其余2个群体(ZJ和RB)的观测杂合度大于期望杂合度,其中,ZJ群体观测杂合度超出期望杂合度的程度较小,说明该群体的遗传性较为稳定;相反,RB群体的观测杂合度超出期望杂合度的程度较大,可能是由于该群体掺杂了野生群体或其他群体中的放流个体,这些个体与RB群体杂交导致遗传平衡遭到破坏。建议在考虑RB群体繁殖选育过程中应该尽量扩大选育的范围,同时也要防止养殖个体的放流逃逸,避免具有同样群体遗传背景的个体之间交配,增加人工选育的难度。
3.2 黄颡鱼养殖群体的遗传分化
本研究14个黄颡鱼养殖群体的遗传分化指数在-0.009~0.116之间波动,其中在两两群体比较的组别中,有64组的Fst小于等于0.05,其余27组的Fst均在0.05~0.15之间。当Fst<0.05时,表明群体间遗传分化较弱;当0.05
本研究黄颡鱼养殖群体间的基因交流是引起其群体间遗传分化程度低的重要因素。本研究中群体间基因流的均值(Nm)为3.573。研究表明,当Nm大于1时,基因流引起群体间遗传分化大于来自于遗传漂变的影响;而当Nm小于1时,遗传漂变对群体间遗传分化的影响要大于基因流的作用[29]。本研究中,在对14个黄颡鱼养殖群体8对多态性微卫星分析结果中,只有一对引物SS9的基因流小于1,其余7对引物的基因流均大于1,表明黄颡鱼养殖群体间有明显的基因流存在,导致各群体间未有明显的遗传分化。
3.3 黄颡鱼养殖群体的遗传结构
本研究基于Nei′s遗传距离构建的UPGMA聚类树与Structure分析中的结果表现出较强的一致性,通过Structure软件对黄颡鱼群体进行种群遗传结构检测,采用ΔK推测K=5是最可能的模型,即14个黄颡鱼养殖群体可以分为5个亚群。此外,聚类的先后顺序可以反映出种群之间亲缘关系的远近。基于本研究的聚类结果可以看出,XB和ZL的遗传相似度最高,亲缘关系最近;而HJ和DB的遗传相似度最小,亲缘关系最远,这个结果与遗传距离的结果也是一致的,即XB和ZL的遗传距离最小,而HJ和DB的遗传距离最大。XB和ZL的亲缘关系最近的原因可能是由于这两个群体的亲本都来自四川,而HJ和DB两个群体的遗传相似度最低,亲缘关系最远的原因可能是由于这两个群体的亲本来源相差大造成的,HJ群体的亲本来自江苏、而DB群体的亲本来自四川。此外,尽管通过Structure分析把14个黄颡鱼群体分为5个亚群,但是各亚群之间基因交流频繁,这和14个黄颡鱼养殖群体处于中等偏低的遗传分化水平吻合。
综上所述,基于微卫星标记对14个黄颡鱼养殖群体的遗传结构分析结果表明,14个黄颡鱼群体之间存在中等偏低的遗传分化水平,且变异来源主要来自个体间。14个养殖群体的遗传多样性均处于高度多态的水平,遗传多样性丰富,可作为家系选育的基础群体。在今后的育种工作中,可以利用不同群体不同个体间的遗传距离以及聚类关系图进行辅助繁殖配组,指导家系建立和群体选育。可基于遗传距离的结果,把亲缘关系较远的群体建立繁育群组,最大限度地避免近亲交配,同时也保持群体的遗传多样性,这对提高黄颡鱼遗传育种潜力、缩短育种年限和加快新品种的选育具有重要的指导意义。

