多黏类芽孢杆菌的生防机制研究进展
贺丹 李鹏 赵珅 姜虹 田苗 赵婷婷 仁安 周彦丽 李明昊 任毅
摘要:多黏类芽孢杆菌(Paenibacillus polymyxa)是一种常见的植物根际促生细菌(PGPR),具有巨大的生物防治潜力,且在可持续农业中发挥的作用越来越重要。多黏类芽孢杆菌可以防治细菌、真菌、线虫和病毒,这是通过生产各种抗菌物质、生长必需物质的竞争以及通过引发植物的过敏防御反应来实现的。多黏类芽孢杆菌衍生的抗菌物质包括葡聚糖酶、几丁质酶、淀粉酶、纤维素酶、黏菌素、羊毛硫菌素、杀镰孢菌素、多肽菌素等,生长必需物质的竞争主要是铁载体,可通过诱导系统抗性 (ISR)引发植物的过敏防御反应。因此,多黏类芽孢杆菌是生产生物农药的重要菌种,由多黏类芽孢杆菌及其衍生物制备的微生物农药以其良好的环境友好性、生物防治机制的多样性和对土壤病害的良好防治效果,成为植物病害防治的重要策略之一。探究多黏类芽孢杆菌的生防机制具有重要的理论和应用价值,并对近年来多黏类芽孢杆菌通过产生抗菌物质、竞争作用以及诱导植物抗病性等生防机制的研究进展进行综述。
关键词:多黏类芽孢杆菌;生防机制;抗菌物质;竞争作用;诱导植物抗病性
中图分类号:S476;S182 文献标志码:A
文章编号:1002-1302(2023)08-0001-07
基金项目:黑龙江省自然科学基金(编号:LH2019C080、LH2019C079、LH2021C086、LH2021C087)。
作者简介:贺 丹(1990—),女,黑龙江哈尔滨人,硕士,助理研究员,从事农业微生物、农业生态学研究。E-mail:448077927@qq.com。
生物防治可定义为“利用自然生物及其产物来防治有害生物的技术,其中自然生物主要包括人类、作物、树木、动物和有益微生物”[1]。真菌细胞壁和昆虫角质层的降解提供了一种在不伤害人类和其他哺乳动物的情况下防治植物病原真菌和昆虫的方法。生防菌的拮抗作用归因于几丁质水解酶对几丁质的水解降解,而微生物或其分泌产物在控制植物病原真菌、线虫或害虫方面的实际应用方面提供了一个新的途径[2-8]。研究微生物组生物技术工具的快速发展,能够更精准地了解微生物生存的不同生态位(如土壤和植物组织)及多样性。当前大量研究集中在特定植物组织尤其是根际的微生物组上[9]。通过对多种作物和土壤进行研究,发现重要的微生物组通常由适合定植于不同植物根部表面和内部的细菌物种组成。通过对土壤微生物组进行研究,发现在植物附近富集的这些细菌属之一是类芽孢杆菌,其中多黏类芽孢杆菌(Paenibacillus polymyxa)以其促进植物生长的潜力而闻名,成为重要的、有潜力的生物防治和生物肥料剂。本文阐述了关于多黏类芽孢杆菌的已知信息及其对植物的生物防治机制,明确该生物体如何在可持续发展的农业中发挥重要作用。
1 多黏类芽孢杆菌
多黏类芽孢杆菌自1880年被发现以来,已有多个名称,开始它被命名为多黏梭菌,1889年因其杆状细胞而改名为多黏芽孢杆菌[10];1993年通过对芽孢杆菌16S rRNA序列种属的比较分析,将类芽孢杆菌重新分类,而类芽孢杆菌与芽孢杆菌属的密切关系体现在其名称上,是后者和拉丁语副词“paene”的缩写,表示“几乎”的意思。类芽孢杆菌属由165种组成,其中多黏类芽孢杆菌是类芽孢杆菌的模式菌种。多黏类芽孢杆菌是一种革兰氏阳性、兼性厌氧、嗜中性、产孢子、杆状细菌,具有周毛鞭毛[11]。它主要存在于土壤、根际和植物组织中,但也有部分是从海洋沉积物和发酵食品中分离出来的[12]。在美国国家生物技术信息中心(NCBI)中,有57个带注释的多黏类芽孢杆菌全基因组组装菌株保藏的总基因组长度中位数为5.8 Mb,蛋白质计数中位数为4 909,GC含量中位数为45.5% (https://www.ncbi.nlm.nih.gov/)。Zhou等对14株多黏类芽孢杆菌菌株的全基因组进行分析,共鉴定出9 345个基因,其中3 063个在所有研究菌株中共有,3 194个基因属于附属基因组,平均每个菌株有220个基因是特有的[13]。在这14个菌株的独特基因组中,5个最具有研究价值的功能之一是“次级代谢产物合成、运输和分解代谢”。在基因组中鉴定9个次级代谢物簇(非核糖体肽合成酶[NRPS]簇、Ⅰ型聚酮化合物合成酶[T1PKS]、细菌素、β内酯、羊毛肽、膦酸盐、铁载体和反式乙酰转移酶 [AT]-PKS),其中NRPS簇存在于所有菌株中,且在每个菌株中的含量也最高。次生代谢产物基因簇总数平均占基因组的12%,表明該菌种生物合成天然产物的潜力很大[13]。迄今为止,已发表了7个多黏类芽孢杆菌菌株的完整基因组,包括菌株SC2[14]、E681[15]、YC0136[16]、M-1[17]、SQR-21[18]、CR1[19]和YC0573[20]。多黏类芽孢杆菌E681是多黏类芽孢杆菌的模式菌株,它是从韩国冬大麦根际中分离出来的,具有促进植物生长和抑制植物病害的能力[21]。SC2是从中国贵州省辣椒植物的根际分离出来的,并被证明对植物病原真菌有抑制作用,如尖孢镰刀菌、灰霉病等植物病原菌,但这些细菌的种类尚未发表[22]。
2 多黏类芽孢杆菌产生的抗菌物质
2.1 肽类
多黏类芽孢杆菌能产生抗菌肽,一般分子量较小,在1~2 ku之间。当前研究较广泛的抗菌肽有多黏菌素(polymyxin)、黏菌素(colistin)、羊毛硫菌素(lantibiotics)、环杆菌素(circulin)、杀镰孢菌素(fusaricidins)、多肽菌素(polypeptins)、谷缬菌素(gatavalin)等[23]。多黏类芽孢杆菌所产生的抗菌肽主要分为2种,分别为核糖体合成的细菌素和非核糖体合成的肽。羊毛硫抗生素是细菌素中重要的一类,也称为Ⅰ类细菌素,含有非编码氨基酸羊毛硫氨酸。它们通常对革兰氏阳性菌具有活性,因为革兰氏阴性菌的外膜具有天然屏障。羊毛硫抗生素通常在细菌生长的晚期指数期或早期稳定期表达,并与它们广泛翻译后修饰所需的基因一起编码在一个簇中。在多黏类芽孢杆菌OSY-DF中,多黏类芽孢杆菌编码在一个簇中,该簇还包含假定的羊毛硫抗生素脱水酶、羊毛硫抗生素环化酶、乙酰化酶、肽酶和ATP转运蛋白,这些基因都具有输送至胞外的功能[24]。
也有一部分多黏类芽孢杆菌产生的许多抗菌肽是非核糖体合成的,独立于RNA。氨基酸残基由非核糖体肽合成酶(NRPS)拼接在一起,NRPS是一种多酶复合物,可以结合D-氨基酸和L-氨基酸的混合物。NRPS的每个模块都将1个或多个特定氨基酸合并到肽链中,所得肽在序列和结构上表现出极大的多样性,且对蛋白水解酶的抗性增强。非核糖体脂肽主要通过破坏靶细胞的膜起作用,且由于靶细胞很难重组它们的膜,因此抗病效果通常较慢[25]。1996年首次报道,在多黏类芽孢杆菌 KT-8 中的六肽环,除酰胺键(缩肽)外还包含1个或多个酯键,并连接有胍基化的β-羟基脂肪酸[26-27]。单个操纵子产生多种杀镰孢菌素(fusaricidins),不同之处在于它们在肽环的6个位置中有3个位置掺入了氨基酸,此处多样性是由于NRPS的底物特异性决定的,与多黏菌素合酶的模块化正好相反。
多黏菌素作用于细菌的细胞膜,能使病原菌细胞的胞内物质流失,进而失活。在促进植物生长的根际杆菌(PGPR)多黏类芽孢杆菌M-1的全基因组序列中检测到9个基因簇,这些基因簇具有抗菌作用的次级代谢产物具有非核糖体合成功能,其中有多黏菌素和杀镰孢菌素。通过进一步研究,发现多黏类芽孢杆菌M-1会抑制植物病原性梨火疫病菌Ea273和胡萝卜软腐欧文氏菌(Erwinia carotovora)(分别是火疫病和软腐病的病原体)的生长。通过MALDI-TOF质谱和反相高效液相色谱(RP-HPLC)检测到2种分子量,分别为1 190.9、1 176.9 u ,其抗菌化合物为多黏菌素P、多黏菌素P1和P2的2种成分。作用于2种欧文氏菌菌株的活性成分从TLC板中分离获得,并通过(PSD)-MALDI-TOF质谱法鉴定为多黏菌素P1和多黏菌素P2,这些发现被M-1染色体中检测到的多黏菌素(pmx)基因簇的域结构分析所证实,该基因簇显示对应于多黏菌素P的化学结构。用粗多黏菌素P或 M-1 培养上清液处理的细菌植物病原体细胞壁的相同形态变化,证实了多黏菌素P是菌株M-1对植物病原欧文氏菌产生的生物防治效果的主要成分[28]。
杀镰孢菌素是一种具有出色抗真菌活性的脂肽类抗生素。它是一类从多黏类芽孢杆菌中分离出来的、由6个氨基酸残基和1个2-胍基-3-羟基十五烷酸(GHPD)组成的环状多肽类抗生素,在1株防治植物枯萎病的多黏类芽孢杆菌HY96-2发酵液中分离得到一种抗真菌活性物质6B,被鉴定为杀镰孢菌素 A。杀镰孢菌素对真菌具有活性,其中包括许多重要的植物病原体和多种革兰氏阳性菌。杀镰孢菌素天然存在的结构和合成修饰都可以化学合成,从而提高稳定性并降低对人体细胞的非特异性细胞毒性[27]。Wang等认为,多黏类芽孢杆菌 WLY78对尖孢镰刀菌有明显抑制作用,为探究其机理,筛选WLY78的基因组进行研究,结果发现8个潜在的抗生素生物合成基因簇在杀镰孢菌素合成(FUS)基因簇参与抑制镰孢属。进一步突变分析表明,除fusTE外,fus簇内的7个基因fusG、fusF、fusE、fusD、fusC、fusB、fusA均参与抑制真菌的作用,其在控制黄瓜枯萎病中发挥了重要作用。此外,qRT-PCR表明杀镰孢菌素可以通过水杨酸 (SA) 信号诱导黄瓜枯萎病的系统抗性。多黏类芽孢杆菌 HK4能够对一系列真菌植物病原体具有抗菌功能,通过对HK4细胞沉淀粗提物的 LC-ESI-MS/MS,可以证实杀镰孢菌素作为主要抗真菌代谢物的存在,并证明HK4具备作为生物防治剂和植物生长促进剂的潜力[29]。Li发表了多黏类芽孢杆菌 KF-1 的基因组序列草图,并表明KF-1具有出色的抗菌活性。它编码杆菌肽、卡里曼泰星、杆菌霉素、伊图林、镰刀菌素、十三菌素和培吉肽的合酶以及果聚糖多糖的生物合成途径。此外,该基因组中涉及一种新的原噬菌体,其中包含编码内溶素的基因[30]。
2.2 蛋白质类
多黏类芽孢杆菌能产生多种具有抗菌活性的蛋白质类物质,这种抗菌蛋白的分子量从10 ku到37 ku不等。多黏类芽孢杆菌抗菌蛋白可以攻击真菌和卵菌竞争者的细胞壁。丝状真菌的细胞壁含有大量β-1,3-葡聚糖和几丁质,而卵菌的细胞壁主要由β-1,3-葡聚糖、β-1,6-葡聚糖和纤维素组成,两者蛋白质含量都高达11%。目前已发现多类芽孢杆菌可以产生葡聚糖酶、几丁质酶、纤维素酶和蛋白酶,这些酶与真核细胞壁的破坏有关[31]。糖聚合物几丁质是一种不分枝的结构多糖,由β-D-异聚体构型的1,4-连接N-乙酰氨基葡萄糖(GlcNAc)残基组成。
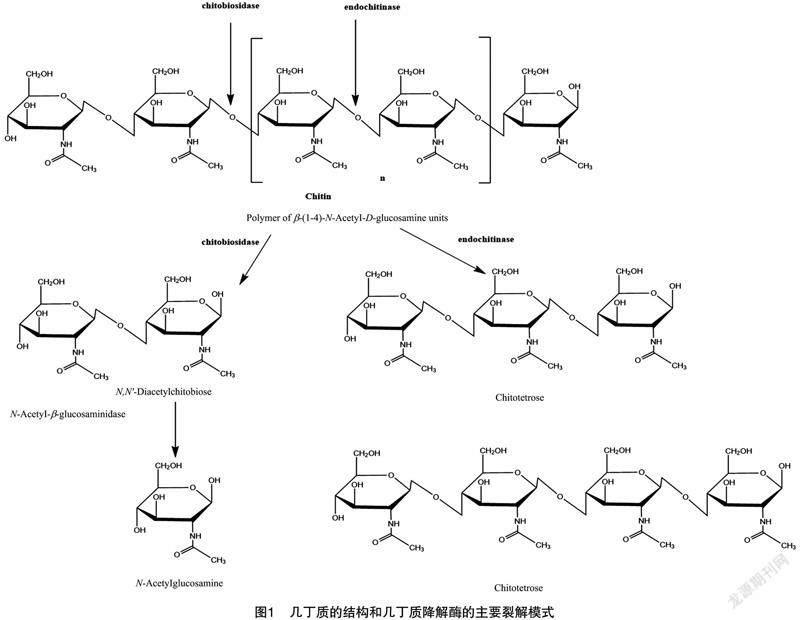
与纤维素不同,甲壳素可以同时作为氮和碳的来源(C ∶N=8 ∶1)。几丁质酶(EC 3.2.1.14)是普遍存在的几丁质裂解糖基水解酶,可水解几丁质N-乙酰氨基葡萄糖残基之间的β-1,4-糖苷键存在于多种生物体中,包括不含几丁质的生物体,如细菌、病毒、高等植物和动物中,在其中发挥着重要的生理和生态作用[32-36]。几丁质酶可分为内几丁质酶和外几丁质酶两大类。内切几丁质酶(EC 32.1.14)在内部位点随机切割几丁质,生成可溶性N-乙酰氨基葡萄糖(GlcNAc)低分子低聚物,如几丁质四糖、几丁质醇和二聚体二乙酰几丁质二糖。外几丁质酶分为2个亚类:几丁糖苷酶(EC 32.1.29),它催化从几丁质微纤丝的非还原端开始的二乙酰几丁质糖和1,4-β-N-乙酰氨基葡萄糖苷酶的渐进释放(EC 3.2.1.30),切割产生GIcNAc单体的内切几丁质酶和几丁糖苷酶的低聚物产物(图1)[35]。几丁质酶因其广泛的应用[37],主要是其在真菌原生质形成中的应用[38],可作为一种潜在的植物病原真菌生物防治剂[39-42],培养转基因真菌對携带几丁质酶转基因的植物/作物的抗性[43-46],以及生产低聚糖作为生物活性物质[47],而受到广泛关注。
Cho等从韩国人参(Panax ginseng C. A. Meyer)根内分离到1株多黏类芽孢杆菌GS01[48]。从该菌株中克隆了cel44C-man26A基因,表达和纯化的Cel44C基因对纤维素酶和地衣酶活性的最适pH值为7.0,对木聚糖酶和甘露聚糖酶活性的最适pH值为5.0。所有底物酶活性的最适温度为 50 ℃。菌株9X166表现出最高的拮抗活性,其产生β-1,3-葡聚糖酶的能力是抑制病原体生长的关键机制。从牛瘤胃中分离的多黏类芽孢杆菌ND25的基因组序列是木质纤维素植物生物质水解的潜在候选物。基因组序列草案生成了5.73 Mb数据,包含4 922个假定的蛋白质编码基因,其中140个被注释为糖苷水解酶。多黏类芽孢杆菌 ND25菌株包含多种木质纤维素分解成分,尤其是12种纤维素酶、23 种半纤维素酶和11种酯酶,表明其具有木质纤维素水解的潜力。随后的酶测定结果显示,菌株利用甘蔗渣作为唯一碳源分别产生0.49、0.24、044 U/mL内切葡聚糖酶、外切葡聚糖酶、β-葡糖苷酶,上述研究结果表明有效应用多黏类芽孢杆菌 ND25促进植物生物质利用。多黏类芽孢杆菌 NSY50从醋渣基质中分离,可以抑制黄瓜根际尖孢镰刀菌的生长,保护寄主植物免受病原体入侵。通过Illumina对16S rRNA基因和内部转录间隔区(ITS)区域(ITS1和ITS2)进行测序,分析细菌和真菌群落,结果表明,NSY50通过改变土壤理化性质(如pH值、C mic、R mic、总N和C org)可以有效降低枯萎病的发生率(56.4%)和酶尤其是脲酶和 β-葡萄糖苷酶的活性,相对于病原体处理条件,它们分别显著增加2.25、2.64倍。 更具体地说,NSY50的应用降低了镰刀菌的丰度并促进了潜在的有益菌群(包括芽孢杆菌属、放线菌属、链霉菌属、放线菌属、链孢菌属和假单胞菌属)。结果表明,NSY50的施用可以改善土壤特性,通过增加有益菌株和减少黄瓜根际病原体定植来改变微生物群落,并减少黄瓜枯萎病的发生,从而促进黄瓜生长[49]。
Kavitha等将多黏类芽孢杆菌VLB16的发酵液用40%硫酸铵沉淀分离得到一分子量为37 ku的抗菌蛋白,可作用于病原真菌稻纹枯病菌(Rhicoctnia solani)和稻瘟病(Magnaporthe oryzae)的菌丝细胞壁,引起细胞形态发生改变,抗菌蛋白对这2种菌表现出良好的抑制作用[50]。多黏类芽孢杆菌WY110分离得到一种P2抗菌蛋白,其具有广泛的抗菌性[51]。多黏类芽孢杆菌VLB16的發酵液中分离出一种分子量为37 ku的抗菌蛋白,可作用于多种植物病原真菌的细胞壁,使真菌细胞形态发生改变,从而抑制植物病原真菌的活性[52]。
3 竞争作用
竞争作用主要为营养和空间位点竞争,是存在于同一微生态环境中2种或2种以上微生物之间对其生长都必需的物质及条件进行竞争的现象。铁载体对铁的竞争是很多细菌生防菌剂抑制植物病原真菌的重要特征。铁载体主要由非核糖体肽合成酶(NRPS)合成,由基因簇编码。这些多酶复合物由各种模块组成,每个模块都将1个或多个特定氨基酸整合到肽主链中(另见“2.1”节)。3类铁载体根据其官能团进行分类,即儿茶酚酸盐、异羟肟酸盐和α-羟基碳酸盐[53]。paenibactin是由类芽孢杆菌(Paenibacillus elgii)B69产生的2,3-DHB-丙氨酸-苏氨酸的环状三聚内酯,在多黏类芽孢杆菌SQR-21的培养物中未检测到儿茶酚酸盐,而是在对数后期产生低浓度的异羟肟酸盐型铁载体[54]。铁载体合成基因簇仅存在于某些类芽孢杆菌属的某些菌株中,且被认为从最近发生的水平基因转移事件中获得[55-56]。
Zhou等研究了多黏类芽孢杆菌BFKC01通过刺激拟南芥以增强其对铁的吸收的机制,发现BFKC01利用转录激活缺铁诱导的转录因子1(FIT1),从而上调IRT1和FRO2的表达[57]。此外,BFKC01不仅可以增加MYB72的转录来诱导植物系统反应,还可以激活酚类化合物的生物合成途径。在接种BFKC01的植物根系分泌物中检测到丰富的酚类化合物,这有效地促进了碱性条件下Fe的迁移。此外,BFKC01还能分泌生长素,进一步改善根系,增强植物从土壤中获取铁的能力。
4 诱导植物抗病性
许多多黏类芽孢杆菌是与根相关的共生菌,当其以足够高的种群密度存在时,可以触发诱导系统抗性(ISR)。ISR是一种发生在植物组织中的潜在防御机制,不是立即激活防御状态,而是通过使植物对潜在威胁高度敏感,能够启动更快、更强的防御[58]。多黏类芽孢杆菌可以诱导植物产生针对病原菌、线虫和病毒的抗病机制。当植物识别来自多黏类芽孢杆菌的诱导物(如结构蛋白、酶、活性氧或挥发性有机化合物)时,ISR途径就开始了[59-60]。ISR可导致植物激素水杨酸的全身水平升高(SA依赖性反应)或导致SA非依赖性反应。后者可包括受植物激素茉莉酸调节的基因转录增加,或对茉莉酸或乙烯有反应的基因表达增强,然后在受到攻击时诱导这些基因。此外,不依赖于SA的ISR可以引发物理反应,如病原体侵染植物的一些部位,通过脱落酸的调节,该部位的细胞质沉积增强,会形成防止病原菌进一步攻击的结构屏障[58]。植物系统抗性被诱导后,便具有广谱抗性[60]。
多黏类芽孢杆菌 KNUC265分泌的细菌挥发物和可扩散代谢物作为诱导物,防治辣椒与烟草中的细菌病原体柑橘溃疡病菌(Xanthomonas axonopodis)和欧文氏菌(Erwinia carotovora)[61]。多黏类芽孢杆菌E681被证实,使用挥发性有机化合物诱导剂来保护拟南芥免受丁香假单胞菌的侵害[62]。多黏类芽孢杆菌菌株产生的挥发物和NRP也有助于诱导系统抗性(ISR)的发生,从而为植物随后的病原体侵染做好防御准备[63-67]。
挥发性有机化合物(VOC)可用于作物病害(特别是水果)的生物防治。它可以增强土壤微生物之间的相互作用,因为这些化合物通过土壤中充满空气的孔隙扩散到物理分离的生物体[68]。大量VOC由多黏类芽孢杆菌以及其他微生物产生。如发现多黏类芽孢杆菌 WR-2产生42种VOC,其中超过30种对尖孢镰刀菌(F.oxysporum)具有一定程度的抗真菌活性,有13种会完全抑制其生长。这些化合物包括苯、醛、酮和醇,但有些产量很低;苯并噻唑、苯甲醛、十一醛、十二醛、十六醛、2-十三烷酮和苯酚是主要的抗真菌化合物。菌株KM2501-1和Sb3-1可以通过分泌有毒挥发性化合物(VOC)保护宿主免受南方根结线虫和真菌长轮枝孢菌的侵害[69-70]。
参考文献:
[1]Monte E,Llobell A. Trichoderma in organic agriculture[C]//Proceedings V World Avocado Congress (Actas V Congreso Mundial del Aguacate),2003:725-733.
[2]Ajit N S,Verma R,Shanmugam V. Extracellular chitinases of fluorescent pseudomonads antifungal to Fusarium oxysporum f. sp. dianthi causing carnation wilt[J]. Current Microbiology,2006,52(4):310-316.
[3]Gortari M C,Hours R A. Fungal chitinases and their biological role in the antagonism onto nematode eggs:a review[J]. Mycological Progress,2008,7(4):221-238.
[4]Simmons C R,Litts J C,Huang N,et al. Structure of a rice β-glucanase gene regulated by ethylene,cytokinin,wounding,salicylic acid and fungal elicitors[J]. Plant Molecular Biology,1992,18(1):33-45.
[5]Neeraja C,Anil K,Purushotham P,et al. Biotechnological approaches to develop bacterial chitinases as a bioshield against fungal diseases of plants[J]. Critical Reviews in Biotechnology,2010,30(3):231-241.
[6]Radjacommare R,Venkatesan S,Samiyappan R. Biological control of phytopathogenic fungi of Vanilla through lytic action of Trichoderma species and Pseudomonas fluorescens[J]. Archives of Phytopathology and Plant Protection,2010,43(1):1-17.
[7]Wang S L,Shih I L,Liang T W,et al. Purification and characterization of two antifungal chitinases extracellularly produced by Bacillus amyloliquefaciens V656 in a shrimp and crab shell powder medium[J]. Journal of Agricultural and Food Chemistry,2002,50(8):2241-2248.
[8]Zhang Z,Yuen G Y. The role of chitinase production by Stenotrophomonas maltophilia strain C3 in biological control of Bipolaris sorokiniana[J]. Phytopathology,2000,90(4):384-389.
[9]Lebeis S L,Powell K R,Merlin D,et al. Interleukin-1 receptor signaling protects mice from lethal intestinal damage caused by the attaching and effacing pathogen Citrobacter rodentium[J]. Infection and Immunity,2009,77(2):604-614.
[10]Padda K P,Puri A,Zeng Q W,et al. Effect of GFP-tagging on nitrogen fixation and plant growth promotion of an endophytic diazotrophic strain of Paenibacillus polymyxa[J]. Botany,2017,95(9):933-942.
[11]Ash C,Priest F G,Collins M D. Molecular identification of rRNA group 3 bacilli (Ash,Farrow,Wallbanks and Collins) using a PCR probe test[J]. Antonie Van Leeuwenhoek,1993,64(3/4):253-260.
[12]Lal S,Tabacchioni S. Ecology and biotechnological potential of Paenibacillus polymyxa:a minireview[J]. Indian Journal of Microbiology,2009,49(1):2-10.
[13]Zhou L L,Zhang T,Tang S,et al. Pan-genome analysis of Paenibacillus polymyxa strains reveals the mechanism of plant growth promotion and biocontrol[J]. Antonie Van Leeuwenhoek,2020,113(11):1539-1558.
[14]Lohans C T,Huang Z D,van Belkum M J,et al. Structural characterization of the highly cyclized lantibiotic paenicidin A via a partial desulfurization/reduction strategy[J]. Journal of the American Chemical Society,2012,134(48):19540-19543.
[15]MacKelprang R,Grube A M,Lamendella R,et al. Microbial community structure and functional potential in cultivated and native tallgrass prairie soils of the Midwestern United States[J]. Frontiers in Microbiology,2018,9:1775.
[16]Liu J,Luo J G,Ye H,et al. Preparation,antioxidant and antitumor activities in vitro of different derivatives of levan from endophytic bacterium Paenibacillus polymyxa EJS-3[J]. Food and Chemical Toxicology,2012,50(3/4):767-772.
[17]Niu B,Vater J,Rueckert C,et al. Polymyxin P is the active principle in suppressing phytopathogenic Erwinia spp.by the biocontrol rhizobacterium Paenibacillus polymyxa M-1[J]. BMC Microbiology,2013,13:137.
[18]Li Y L,Chen S F. Fusaricidin produced by Paenibacillus polymyxa WLY78 induces systemic resistance against Fusarium wilt of cucumber[J]. International Journal of Molecular Sciences,2019,20(20):5240.
[19]Eastman A W,Heinrichs D E,Yuan Z C. Comparative and genetic analysis of the four sequenced Paenibacillus polymyxa genomes reveals a diverse metabolism and conservation of genes relevant to plant-growth promotion and competitiveness[J]. BMC Genomics,2014,15:851.
[20]Liu H,Wang C Q,Li Y H,et al. Complete genome sequence of Paenibacillus polymyxa YC0573,a plant growth-promoting rhizobacterium with antimicrobial activity[J]. Genome Announcements,2017,5(6):e01636-e01616.
[21]Kim J F,Jeong H,Park S Y,et al. Genome sequence of the polymyxin-producing plant-probiotic rhizobacterium Paenibacillus polymyxa E681[J]. J Bacteriol 2010,192(22):6103-6104.
[22]Guo L L,Karpac J,Tran S L,et al. PGRP-SC2 promotes gut immune homeostasis to limit commensal dysbiosis and extend lifespan[J]. Cell,2014,156(1/2):109-122.
[23]陳 浩. 多黏类芽孢杆菌X33发酵条件的优化及生物防治效果研究[D]. 哈尔滨:哈尔滨工业大学,2019
[24]王 波,王 幸,周兴根,等. 多黏类芽孢杆菌(Paenibacillus polymyxa)XZ-2发酵条件优化的研究[J]. 江西农业学报,2018,30(11):57-61.
[25]郑 维,权春善,赵 晶,等. 芽孢杆菌产环脂肽类化合物结构与生物合成机制研究进展[J]. 山东化工,2016,45(8):51-52,54.
[26]Mousa W K,Raizada M N. Biodiversity of genes encoding anti-microbial traits within plant associated microbes[J]. Frontiers in Plant Science,2015,6:231.
[27]Bionda N,Pitteloud J P,Cudic P. Cyclic lipodepsipeptides:a new class of antibacterial agents in the battle against resistant bacteria[J]. Future Medicinal Chemistry,2013,5(11):1311-1330.
[28]Niu B,Rueckert C,Blom J,et al. The genome of the plant growth-promoting rhizobacterium Paenibacillus polymyxa M-1 contains nine sites dedicated to nonribosomal synthesis of lipopeptides and polyketides[J]. Journal of Bacteriology,2011,193(20):5862-5863.
[29]Wang L Y,Zhang L H,Liu Z Z,et al. A minimal nitrogen fixation gene cluster from Paenibacillus sp. WLY78 enables expression of active nitrogenase in Escherichia coli[J]. PLoS Genetics,2013,9(10):e1003865.
[30]Li Y M,Li Q,Li Y M,et al. Draft genome sequence of Paenibacillus polymyxa KF-1,an excellent producer of microbicides[J]. Genome Announcements,2016,4(4):e00727-e00716.
[31]閆博巍,闫凤超,赵婷婷,等. 类芽孢杆菌源抗病基因在作物病害防治上的应用研究进展[J]. 现代化农业,2019(8):4-8.
[32]Bhattacharya D,Nagpure A,Gupta R K. Bacterial chitinases:properties and potential[J]. Critical Reviews in Biotechnology,2007,27(1):21-28.
[33]Boot R G,Blommaart E F C,Swart E,et al. Identification of a novel acidic mammalian chitinase distinct from chitotriosidase[J]. Journal of Biological Chemistry,2001,276(9):6770-6778.
[34]Kragh K M,Jacobsen S,Mikkelsen J D,et al. Tissue specificity and induction of class Ⅰ,Ⅱ and Ⅲ chitinases in barley (Hordeum vulgare)[J]. Physiologia Plantarum,1993,89(3):490-498.
[35]Shinshi H,Neuhas J M,Ryals J,et al. Structure of a tobacco endochitinase gene:evidence that different chitinase genes can arise by transposition of sequences encoding a cysteine-rich domain[J]. Plant Molecular Biology,1990,14(3):357-368.
[36]Cohen-Kupiec R,Chet I. The molecular biology of chitin digestion[J]. Current Opinion in Biotechnology,1998,9(3):270-277.
[37]Vaidya R,Roy S,Macmil S,et al. Purification and characterization of chitinase from Alcaligenes xylosoxydans[J]. Biotechnology Letters,2003,25(9):715-717.
[38]Kishimoto K,Nishizawa Y,Tabei Y,et al. Transgenic cucumber expressing an endogenous class Ⅲ chitinase gene has reduced symptoms from Botrytis cinerea[J]. Journal of General Plant Pathology,2004,70(6):314-320.
[39]Gupta R,Saxena R K,Chaturvedi P,et al. Chitinase production by Streptomyces viridificans:its potential in fungal cell wall Lysis[J]. Journal of Applied Bacteriology,1995,78(4):378-383.
[40]Jobin G,Couture G,Goyer C,et al. Streptomycete spores entrapped in chitosan beads as a novel biocontrol tool against common scab of potato[J]. Applied Microbiology and Biotechnology,2005,68(1):104-110.
[41]Vaidya R,Shah I,Vyas P,et al. Production of chitinase and its optimization from a novel isolate Alcaligenes xylosoxydans:potential in antifungal biocontrol[J]. World Journal of Microbiology and Biotechnology,2001,17:691-696.
[42]Yuan W,Crawford D. Characterization of Streptomyces lydicus WYEC108 as a potential biocontrol agent against fungal root and seed rots[J]. Applied and Environmental Microbiology,1995,61:3119-3128.
[43]Ojaghian S,Wang L,Xie G L. Enhanced resistance to white rot in Ipomoea batatas expressing a Trichoderma harzianum chitinase gene[J]. Journal of General Plant Pathology,2020,86(5):412-418.
[44]Distefano G,la Malfa S,Vitale A,et al. Defence-related gene expression in transgenic lemon plants producing an antimicrobial Trichoderma harzianum endochitinase during fungal infection[J]. Transgenic Research,2008,17(5):873-879.
[45]Shah J M,Raghupathy V,Veluthambi K. Enhanced sheath blight resistance in transgenic rice expressing an endochitinase gene from Trichoderma virens[J]. Biotechnology Letters,2009,31(2):239-244.
[46]Xu X J,Zhang L Q,Zhu Y Y,et al. Improving biocontrol effect of Pseudomonas fluorescens P5 on plant diseases by genetic modification with chitinase gene[J]. Chinese Journal of Agricultural Biotechnology,2005,2(1):23-27.
[47]Rattanakit N,Yano S,Plikomol A,et al. Purification of Aspergillus sp. S1-13 chitinases and their role in saccharification of chitin in mash of solid-state culture with shellfish waste[J]. Journal of Bioscience and Bioengineering,2007,103(6):535-541.
[48]Cho K M,Hong S Y,Lee S M,et al. A [WTBX][STBX]cel44C-man26A[WTBZ][STBZ] gene of endophytic Paenibacillus polymyxa GS01 has multi-glycosyl hydrolases in two catalytic domains[J]. Applied Microbiology and Biotechnology,2006,73(3):618-630.
[49]Du N S,Shi L,Yuan Y H,et al. Isolation of a potential biocontrol agent Paenibacillus polymyxa NSY50 from vinegar waste compost and its induction of host defense responses against Fusarium wilt of cucumber[J]. Microbiological Research,2017,202:1-10.
[50]Kavitha S,Senthilkumar S,Gnanamanickam S,et al. Isolation and partial characterization of antifungal protein from Bacillus polymyxa strain VLB16[J]. Process Biochemistry,2005,40(10):3236-3243.
[51]Heyndrickx M,Vandemeulebroecke K,Scheldeman P,et al. Paenibacillus (formerly Bacillus) gordonae (Pichinoty et al. 1986) Ash et al. 1994 is a later subjective synonym of Paenibacillus (formerly Bacillus) validus (Nakamura 1984) Ash et al. 1994:emended description of P.validus[J]. International Journal of Systematic Bacteriology,1995,45(4):661-669.
[52]Heyndrickx M,Vandemeulebroecke K,Hoste B,et al. Reclassification of Paenibacillus (formerly Bacillus) pulvifaciens (Nakamura 1984) Ash et al. 1994,a later subjective synonym of Paenibacillus (formerly Bacillus) larvae (White 1906) Ash et al. 1994,as a sub species of P.larvae,with emended descriptions of P.larvae as P.larvae subsp.larvae and P.larvae subsp. pulvifaciens[J]. International Journal of Systematic Bacteriology,1996,46(1):270-279.
[53]Hertlein G,Müller S,Garcia-Gonzalez E,et al. Production of the catechol type siderophore bacillibactin by the honey bee pathogen Paenibacillus larvae[J]. PLoS One,2014,9(9):e108272.
[54]Raza W,Shen Q R. Growth,Fe3+ reductase activity,and siderophore production by Paenibacillus polymyxa SQR-21 under differential iron conditions[J]. Current Microbiology,2010,61(5):390-395.
[55]Eastman A W,Heinrichs D E,Yuan Z C. Comparative and genetic analysis of the four sequenced Paenibacillus polymyxa genomes reveals a diverse metabolism and conservation of genes relevant to plant-growth promotion and competitiveness[J]. BMC Genomics,2014,15:851.
[56]Wen Y P,Wu X C,Teng Y,et al. Identification and analysis of the gene cluster involved in biosynthesis of paenibactin,a catecholate siderophore produced by Paenibacillus elgii B69[J]. Environmental Microbiology,2011,13(10):2726-2737.
[57]Zhou C,Guo J S,Zhu L,et al. Paenibacillus polymyxa BFKC01 enhances plant iron absorption via improved root systems and activated iron acquisition mechanisms[J]. Plant Physiology and Biochemistry,2016,105:162-173.
[58]Pieterse C M J,Zamioudis C,Berendsen R L,et al. Induced systemic resistance by beneficial microbes[J]. Annual Review of Phytopathology,2014,52:347-375.
[59]Farag M A,Zhang H M,Ryu C M. Dynamic chemical communication between plants and bacteria through airborne signals:induced resistance by bacterial volatiles[J]. Journal of Chemical Ecology,2013,39(7):1007-1018.
[60][JP2]楊得强,周春发,黄龙伟,等. 内生芽孢杆菌对植物生长发育及病害防治的研究进展[J]. 安徽农业科学,2020,48(4):11-14.
[61]胡 琼,任国平. 多黏类芽孢杆菌在植物生产中的应用及作用机制[J]. 北方园艺,2020(24):137-144.
[62]Lee B,Farag M A,Park H B,et al. Induced resistance by a long-chain bacterial volatile:elicitation of plant systemic defense by a C13 volatile produced by Paenibacillus polymyxa[J]. PLoS One,2012,7(11):e48744.
[63]Du N S,Shi L,Yuan Y H,et al. Isolation of a potential biocontrol agent Paenibacillus polymyxa NSY50 from vinegar waste compost and its induction of host defense responses against Fusarium wilt of cucumber[J]. Microbiological Research,2017,202:1-10.
[64]Luo Y C,Cheng Y J,Yi J C,et al. Complete genome sequence of industrial biocontrol strain Paenibacillus polymyxa HY96-2 and further analysis of its biocontrol mechanism[J]. Frontiers in Microbiology,2018,9:1520.
[65]Park K Y,Seo S Y,Oh B R,et al. 2,3-butanediol induces systemic acquired resistance in the plant immune response[J]. Journal of Plant Biology,2018,61(6):424-434.
[66]Jeong H,Choi S K,Ryu C M,et al. Chronicle of a soil bacterium:Paenibacillus polymyxa E681 as a tiny guardian of plant and human health[J]. Frontiers in Microbiology,2019,10:467.
[67]Cheng W L,Yang J Y,Nie Q Y,et al. Volatile organic compounds from Paenibacillus polymyxa KM2501-1 control Meloidogyne incognita by multiple strategies[J]. Scientific Reports,2017,7:16213.
[68]Morath S U,Hung R,Bennett J W. Fungal volatile organic compounds:a review with emphasis on their biotechnological potential[J]. Fungal Biology Reviews,2012,26(2/3):73-83.
[69]Garbeva P,Hordijk C,Gerards S,et al. Volatile-mediated interactions between phylogenetically different soil bacteria[J]. Frontiers in Microbiology,2014,5:289.
[70]Raza W,Yuan J,Ling N,et al. Production of volatile organic compounds by an antagonistic strain Paenibacillus polymyxa WR-2 in the presence of root exudates and organic fertilizer and their antifungal activity against Fusarium oxysporum f.sp. niveum[J]. Biological Control,2015,80:89-95.

