20种m6A甲基化调节基因与甲状腺乳头状癌的相关性分析
李祖飞,赵晓畅,何帅
(首都医科大学北京朝阳医院 耳鼻咽喉头颈外科,北京 100020)
甲状腺癌是一种常见的内分泌系统恶性肿瘤,约占所有恶性肿瘤的1%[1]。由于近年来甲状腺超声检查的普及,甲状腺癌的发病率在过去几十年中不断上升,从1990年的5人/10万人上升到2014年的15人/10万人[2]。其中女性发病率明显高于男性,在美国甲状腺癌已被列为女性最常见的第五种癌症[1-2]。甲状腺乳头状癌(papillary thyroid carcinoma, PTC)是最常见的甲状腺癌病理类型,PTC的预后相对好于其他类型的甲状腺恶性肿物,但由于一些患者存在隐性颈淋巴结转移(cervical lymph node metastasis, CLNM),这导致术后复发可能性较高,可能需行二次手术。与初次手术相比,二次手术难度增大,并发症风险增加[3-4]。此外,少部分患者常规治疗的效果不佳,尤其是累及气管/食管的晚期PTC患者预后较差。因此,有必要进一步研究并挖掘潜在的PTC疾病发生、发展与预后相关的分子标志物及生物治疗靶点。
N6-甲基腺苷(N6-methyladenosine,m6A)是一种RNA修饰,最早发现于20世纪70年代,已被证实与哺乳动物的多种生理功能有关[5]。m6A甲基化调节基因主要包括3种:①甲基转移酶,也称为“书写”蛋白,如METTL3、METTL4、WTAP等,负责将m6A标记装配到mRNA的特定位点;② 去甲基化酶,也称为“擦除”蛋白,如FTO等,负责去除书写复合物装配的m6A标记;③ 甲基结合蛋白,“读取”蛋白,如YT521-B同源性(YTH)的结构域家族,包含YTHDF1、YTHDF2、YTHDF3等,负责RNA转录物中m6A标记的读取[6]。
近年来,有研究发现m6A甲基化调节基因与各种癌症的发生和发展相关,包括乳腺癌、膀胱癌等[7-8]。m6A甲基化调节基因在PTC发生和发展中的作用尚不清楚。尽管Yu等[9]研究了m6A甲基化调节基因在甲状腺癌中的功能和预后价值,但他们只分析了13个m6A甲基化调节基因(METTL3、YTHDF1、VIRMA、YTHDC2、ALKBH5、YTHDF2、YTHDC1、ZC3H13、METTL14、FTO、WTAP、RBM15与HNRNPC),且研究对象是所有类型的甲状腺癌,而非单纯PTC这一种类型,而且并未研究m6A甲基化调节基因在PTC患者CLNM、临床分期、性别之间的相关性。Xu等[10]分析了m6A甲基化调节基因在分化型甲状腺癌中的预后作用,但同样只分析了13种m6A甲基化调节基因。随着对m6A甲基化的深入研究,研究人员发现更多的m6A甲基化调节基因[11-13]。除以上13种外,“书写”基因RBM15B[11]以及6种“读取”基因IGF2BP2、HNRNPA2B1、IGF2BP3、YTHDF3、IGF2BP1、RBMX也被认为与肿瘤的发生发展存在一定相关性[12-13]。因此,m6A甲基化调节基因在PTC中的具体作用应得到进一步研究与更新。
本研究的主要目的是通过从癌症基因组图谱(the cancer genome atlas, TCGA)数据库下载的患者数据,系统地探讨20种m6A甲基化调节基因在PTC中的作用。包括分析20种m6A甲基化调节基因在PTC组织和癌旁正常组织之间的差异表达,不同临床参数(包括淋巴结转移、性别、T分期和临床分期)与m6A甲基化调节基因之间的差异表达与相关性分析,对20种m6A甲基化调节基因进行聚类分析,探索不同聚类组与PTC预后之间的相关性;识别与预后相关的m6A甲基化调节基因,建立PTC预后模型,探索各预后标记基因与肿瘤免疫浸润之间的相关性。
1 资料与方法
1.1 数据收集
从TCGA数据库(https://portal.gdc.com)中获得395例PTC组织以及59例癌旁组织的基因表达谱数据,其中女性患者286例,男性患者109例。下载RNA测序表达谱以及各PTC患者相应的临床信息。
1.2 20种m6A甲基化调节基因筛选
本研究中共选取20种m6ARNA甲基化调节基因作为深入研究,包括2个“擦除”基因:FTO和ALKBH5;7种“书写”基因:METTL3、METTL14、RBM15、RBM15B、VIRMA、WTAP和ZC3H13;11种“读取”基因:IGF2BP2、YTHDC1、HNRNPA2B1、IGF2BP3、YTHDF3、YTHDC2、IGF2BP1、YTHDF1、HRNPC、RBMX、YTHDF2。
1.3 生物信息学综合分析
本研究中的生物信息学分析主要分为5个步骤。首先,比较各m6A甲基化调节基因在PTC组织和癌旁正常组织之间的差异表达,并绘制热图进行可视化分析,通过网络图展示各基因在PTC组织表达中的相关性。其次,进一步比较m6ARNA甲基化调节基因在PTC组织中表达与患者各临床参数的关系,包括性别、是否存在CLNM、T分期和临床分期。由于本次纳入的PTC患者数据中只有4例有远处转移,例数过少,因此未分析m6A甲基化调节基因的表达与PTC患者远处转移状态之间的相关性。第三,对20种m6A甲基化调节基因表达进行聚类分析,分析每个聚类的PTC患者其不同的生存状态。第四,进一步分析m6A甲基化调节基因表达在PTC患者中的预后作用,用Log-rank检验分析各组间的生存差异,用基于R语言的timeROC(V0.4)工具包分析各组m6A甲基化调节基因的预测准确性和风险评分。使用最小绝对收缩和选择算子(least absolute shrinkage and selection operator,LASSO)回归算法,使用Kaplan-Meier进行生存分析,使用对数秩检验和单变量Cox比例风险回归计算生存曲线的P值和95%置信区间(confidence interval, CI)的风险比(hazard ratio, HR)。最后,采用Spearman相关性分析探索3种预后相关m6A甲基化调节基因与PTC肿瘤免疫浸润之间的相关性。
1.4 统计学分析
所有统计分析方法均由R语言(4.0.3版)进行,使用ggplot2和heatmap软件包计算并绘制PTC组织和正常组织之间m6A甲基化调节基因表达的热图。Wilcox检验用于计算不同临床数据之间m6A调节基因的差异表达。使用Spearman相关分析定量变量之间的关系。Consensus Cluster Plus R包(v1.54.0)用于m6A甲基化调节基因的聚类分析,最大聚类数设置为6。 Heatmap工具包(v1.0.12)用于聚类热图绘制。采用Log-rank检验分析各组之间的生存差异,采用timeROC(v0.4)分析软件包分析各组m6A甲基化调节基因的预测准确性和风险评分。LASSO回归算法采用glmnet R软件包完成。生存分析采用Kaplan-Meier算法。以P<0.05为差异具有统计学意义。
2 结果
2.1 PTC与正常组织m6A甲基化调节基因的比较
在395例PTC组织和59例癌旁组织中,除RBMX(Z=-1.313,P=0.189)与YTHDF2(Z=-0.475,P=0.635)外,共鉴定出18种差异表达的m6A甲基化调节基因。其中15种表达上调,分别为:RBM15(Z=-9.869,P=0.000),YTHDC1(Z=-8.477,P=0.000),METTL14(Z=-8.208,P=0.000),FTO(Z=-7.391,P=0.000),HNRNPA2B1(Z=-6.895,P=0.000),IGF2BP3(Z=-6.791,P=0.000),ZC3H13(Z=-6.709,P=0.000),ALKBH5(Z=-6.460,P=0.000),WTAP(Z=-6.325,P=0.000),YTHDF3(Z=-5.011,P=0.000),YTHDC2(Z=-4.917,P=0.000),VIRMA(Z=-4.885,P=0.000),IGF2BP1(Z=-4.853,P=0.000),YTHDF1(Z=-4.739,P=0.000),METTL3(Z=-4.610,P=0.000);3种表达下调:IGF2BP2(Z=-11.008,P=0.000)、HNRNPC(Z=-4.556,P=0.000)和RBM15B(Z=-2.087,P=0.037)。图1A显示了20种m6A甲基化调节基因在PTC组织与癌旁组织中的差异,图1B示m6A甲基化调节基因在PTC组织中的表达热图,图1C和图1D分别示各m6A甲基化调节基因之间的相关性。
2.2 m6A甲基化调节基因表达与PTC患者临床资料的相关性
分别分析m6A甲基化调节基因的表达与PTC患者临床资料(包括PTC患者的性别、T分期、有无淋巴结转移、临床分期)之间的相关性。如图2所示,HNRNPC和IGF2BP2的表达与PTC的淋巴结转移呈显著正相关,ALKBH5与PTC淋巴结转移负相关。HNRNPC和IGF2BP2与T分期相关。YTHDF1与性别相关。METTL14、YTHDC1、YTHDF2与PTC临床分期相关,与早期(Ⅰ、Ⅱ期)相比,3者表达量在晚期(Ⅲ、Ⅳ期)PTC组织中有所下降。
2.3 基于m6A甲基化调节基因的聚类分析
为深入了解20种m6A甲基化调节基因在PTC中的亚组分型特点,本研究采用用一致性聚类法根据20种m6A甲基化调节基因在PTC组织中的表达进行聚类分组,该方法的原理为:采取重抽样方法抽数据,计算k值计以及不同聚类数目下的合理性[14]。结果显示:分为2组时,组间差异最显著(图3A、B)。图3C示不同组别各基因的表达热图,可见亚组1中的大多数基因表达上调,而亚组2中的大部分基因表达下调。图3D显示不同分组之间各PTC患者的生存曲线,可见亚组2的PTC患者总体生存率显著高于亚组1的PTC患者。
2.4 m6A甲基化调节基因在PTC预后中的作用
Cox单变量分析用于探讨m6A甲基化调节基因在PTC预后中的作用(图4),结果发现FTO、IGF2BP1和YTHDF3高表达的PTC患者生存率较差。这3个m6A甲基化调节基因被进一步用于构建生存预测模型,以预测PTC患者的预后。根据中位评分将PTC患者分为高风险和低风险亚组,低风险组预后较好(图4B,P<0.05)。最后通过LASSO回归算法构建PTC患者预后预测模型(图5),公式为:Riskscore=0.090 8×FTO+0.688 1×YTHDF3+0.629 5×IGF2BP1。该预后模型预测PTC患者3年生存率的ROC曲线下面积为0.753(95%CI0.615-0.891),预测PTC患者5年生存率的ROC曲线下面积为0.729(95%CI0.613-0.846),说明该预后预测模型可较准确预测PTC患者的预后。
2.5 3种预后相关m6A甲基化调节基因与免疫系统的相关性
我们进一步探讨了前述3种预后相关m6A甲基化调节基因与PTC组织肿瘤免疫浸润之间的相关性,通过Spearman相关分析发现YTHDF3与内皮细胞、巨噬细胞、NK细胞、CD4+T细胞相关;FTO与巨噬细胞、NK细胞、CD4+T细胞和CD8+T细胞相关。说明这3个基因不仅与PTC患者的预后相关,还与肿瘤免疫浸润相关(图6)。
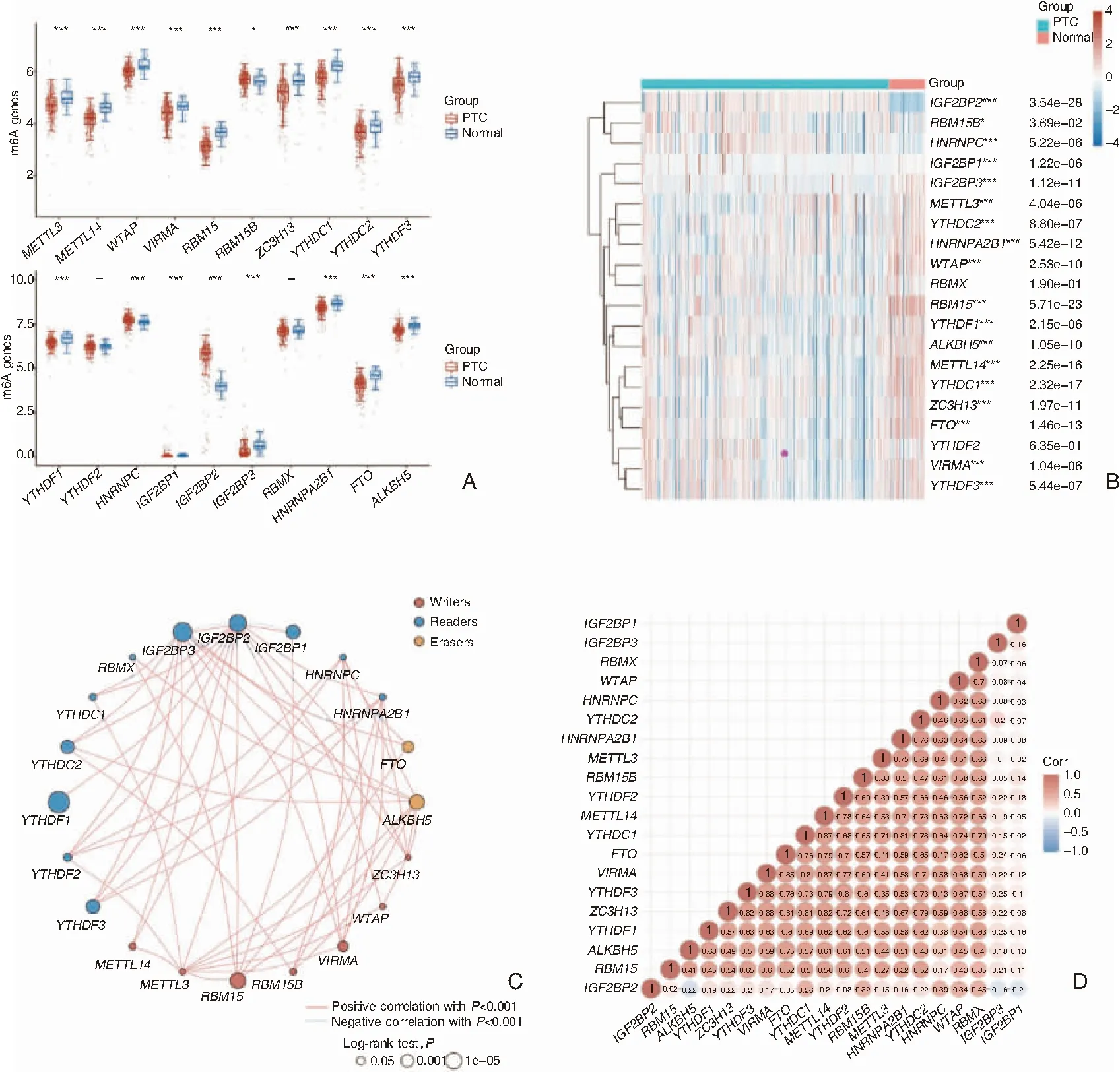
图1 20种m6A RNA甲基化调节基因在PTC组织中的表达 A:癌旁组织和PTC组织中各m6A甲基化调节基因的差异表达;B:热图展示PTC中差异表达的m6A甲基化调节基因; C、D:各基因在PTC表达之间的相关性 注:*P<0.05;***P<0.001。m6A(N6-甲基腺苷);PTC(甲状腺乳头状癌)。下同。
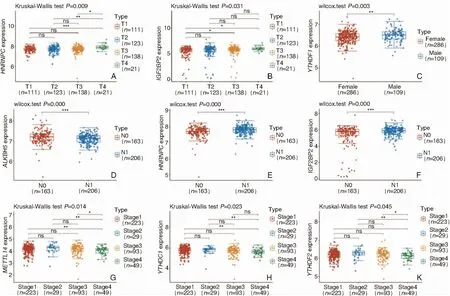
图2 m6A甲基化调节基因与PTC患者临床病理参数之间的关系 A、B:与T分期的相关性; C:与性别的相关性; D~F:与是否合并颈部淋巴结转移的相关性; G~K:与临床分期的相关性 注:stage1、stage2、stage3、stage4分别代表Ⅰ、Ⅱ、Ⅲ、Ⅳ期;TCGA数据库中T分期存在2例Tx分期不明数据,26例Nx淋巴结转移情况不明数据,临床分期1例不明数据,故均未纳入分析。
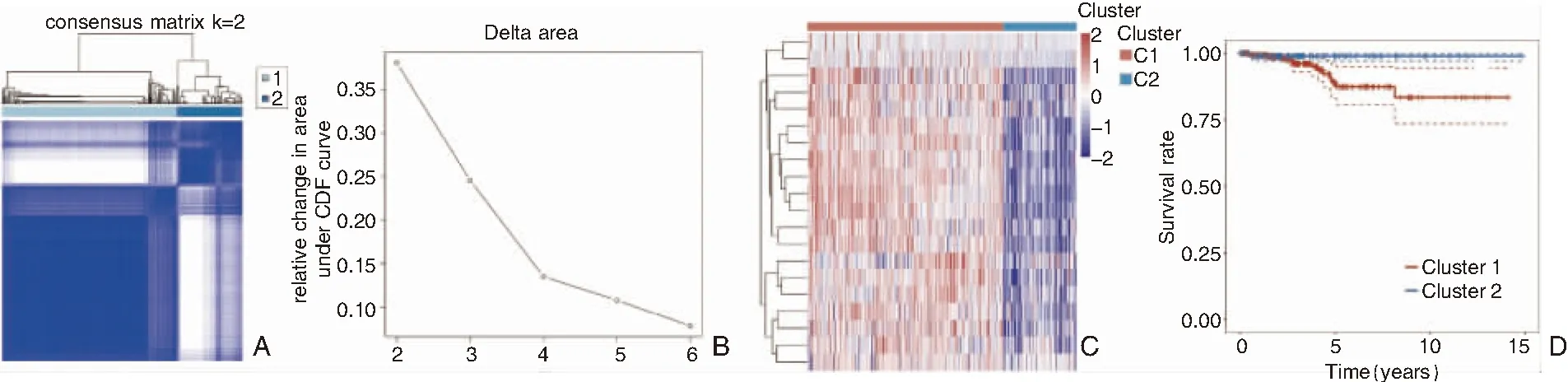
图3 20种m6A甲基化调节基因在PTC组织中表达的一致性聚类分析 A:当k为2时,一致性聚类结果矩阵; B:当k取值2~6时,CDF曲线下的相对面积变化,可见当k值为2时聚类效果最佳; C:热图显示不同组别m6A甲基化调节基因在各PTC患者中的表达模式。C1(亚组1);C2(亚组2); D:不同聚类亚组PTC患者的生存曲线 注:CDF(累积分布函数)。
3 讨论
本研究为首次对目前已知更多种类的(20种)m6A甲基化调节基因在PTC中的作用进行综合生物信息学分析的研究。本研究发现20种m6A甲基化调节基因中,有18种在PTC组织和癌旁组织之间差异表达。既往研究仅对13种m6A甲基化调节基因进行研究,并仅发现12种在PTC组织中差异表达[9-10],它们是METTL3、YTHDC2、HNRNPC、WTAP、YTHDF1、ALKBH5、METTL14、YTHD11、FTO、ZC3H13、RBM15、KIAA1429(也称为VIRMA)。本研究结果发现,除了上述12种m6A甲基化调节基因外,PTC组织和癌旁组织之间还存在6种差异表达的m6A甲基化调节基因:IGF2BP1、IGF2BP2、IGF2BP3、RBM15B、YTHDF3和HNRNPA2B1。这些发现弥补了以往研究在这方面的不足。
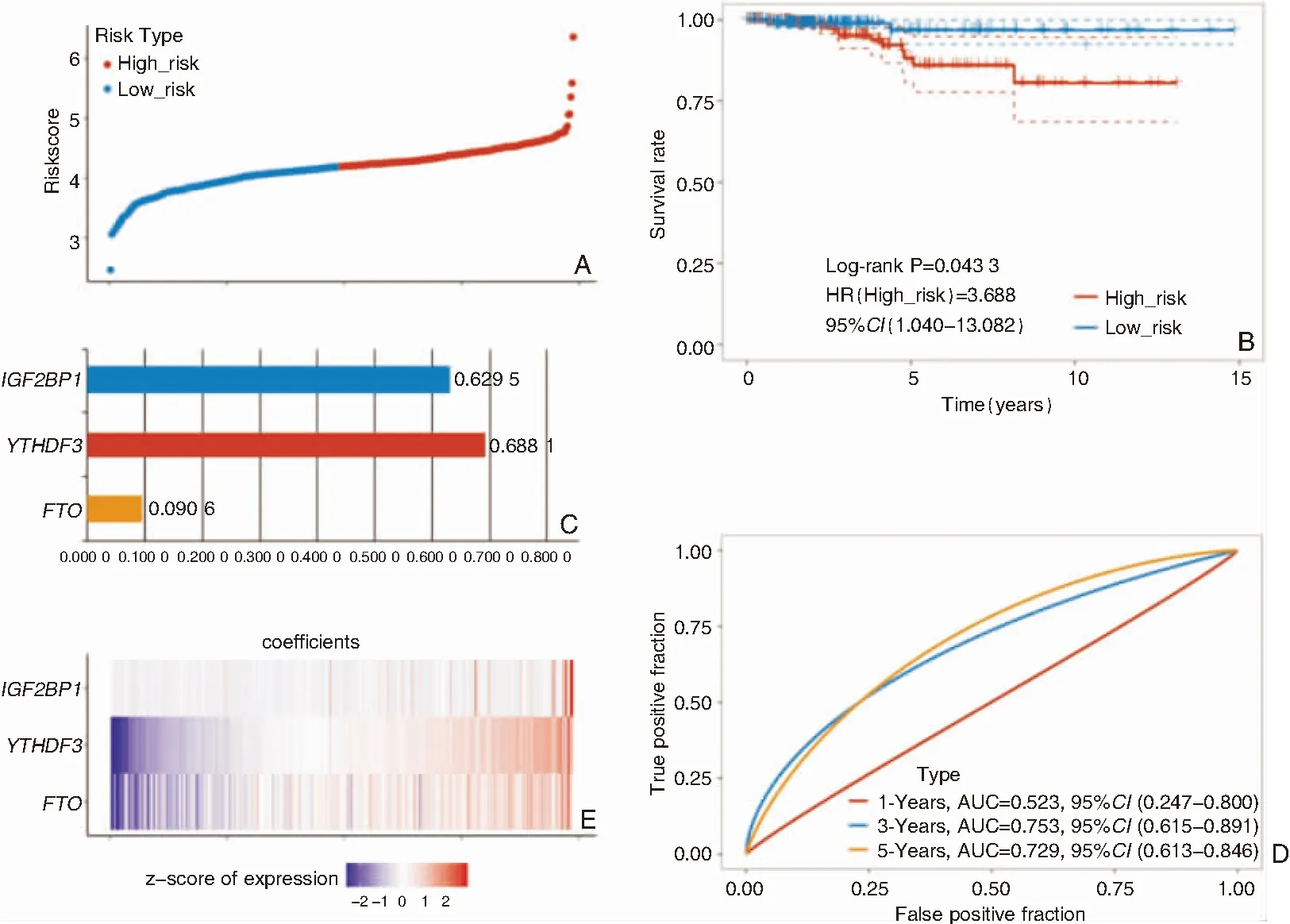
图5 基于LASSO回归算法建立的由3个预后相关的m6A甲基化调节基因构建的生存预测模型 A:预后指数的等级和组的分布; B:高风险组和低风险组患者的生存曲线; C:3种预后相关m6A甲基化调节基因在模型中的系数值; D:PTC预后预测模型的ROC曲线; E:FTO、YTHDF3与IGF2BP1的基因表达谱热图

图6 3种预后相关m6A甲基化调节基因与肿瘤免疫浸润之间的关系
此外本研究还发现3种m6A甲基化调节基因与PTC患者CLNM相关,其中ALKBH3与其呈负相关,而HNRNPC和HGF2BP2与CLNM呈正相关。CLNM是PTC术后局部复发的重要原因之一,据报道约30%~80%的PTC患者会出现CLNM[15-17]。因此,术前对患者进行CLNM转移预测非常重要。尽管术前超声检查可对PTC患者CLNM状态进行初步评估,但研究发现仅20%~31%的CLNM可通过术前超声检查检测,仅通过超声检查远不足以正确预测CLNM[18-19]。因此,探索更多的方法来预测PTC患者是否存在CLNM非常重要,本研究结果发现3种m6A甲基化调节基因(ALKBH3、HNRNPC、IGF2BP2)与PTC患者的CLNM状态相关,而这3种m6A甲基化调节基因可能是预测PTC患者是否存在CLNM的分子标记物。此外,本研究还发现HNRNPC和IGF2BP2不仅与CLNM相关,还与PTC患者的T分期相关,因此,值得通过更多实验进一步探讨这2个m6A甲基化调节基因在PTC中的作用。
本研究发现男性和女性PTC患者之间存在差异表达的m6A甲基化调节基因,男性患者的YTHDF1表达水平高于女性患者。既往研究提示男性PTC与女性相比,具有侵袭性更高,预后更差的特点,但其潜在机制尚不清楚[20]。本研究认为男性PTC患者中YTHDF1的表达高于女性患者可能是解释男性PTC恶性程度高于女性的原因,但一观点还需要进一步的实验对此进行验证。
本研究发现3种与预后相关的m6A甲基化调节基因,FTO、IGFBP1和YTHDF3高表达的PTC患者预后更差。FTO,也称为脂肪团和肥胖相关蛋白,其功能为m6A脱甲基酶。既往研究结果提示FTO是一种促癌基因,以m6A依赖性的方式在多种癌症中高度表达,如乳腺癌、肺癌、宫颈癌等,抑制FTO可以抑制肿瘤进展[21]。IGFBP1,也称为胰岛素样生长因子结合蛋白-1,是胰岛素样生长因子(IGF)系统的一员,已被证实与各种癌症的病理生理学相关[22]。YTHDF3是一种m6A“读取”蛋白,在多种肿瘤中也表达上调,并且与患者的不良预后有关[18]。本研究结果表明,3种m6A甲基化调节基因FTO、IGFBP1和YTHDF不仅在PTC组织中高表达,而且与PTC患者的不良预后相关。基于上述3种基因建立的PTC患者预后预测模型,其预测PTC患者5年及3年的生存率ROC曲线下面积均大于0.7,提示其可较为准确预测PTC患者预后。
最后,本研究对3种PTC预后相关的m6A甲基化调节基因与肿瘤免疫浸润之间的相关性进行分析,发现YTHDF3与内皮细胞、巨噬细胞、NK细胞和CD4+T细胞相关;FTO与巨噬细胞、NK细胞、CD4+T细胞和CD8+T细胞相关。癌症的一个共同特征是免疫逃逸,研究表明CD4+和CD8+T细胞的负调节可能有助于肿瘤的进展,这种现象在甲状腺癌患者中也可以观察到[23]。此外,巨噬细胞,特别是肿瘤相关巨噬细胞,是主要的免疫逃逸因子。3种预后相关的m6A甲基化调节基因中有2种与免疫细胞相关:YTHDF3与内皮细胞、巨噬细胞、NK细胞和T细胞CD4+相关;FTO与巨噬细胞、NK细胞、T细胞CD4+和T细胞CD8+相关。癌症的共同特征之一是免疫逃逸,研究表明CD4+和CD8+T细胞的负调节可能有助于肿瘤的进展,这种现象在PTC患者中也可以观察到[23]。此外,巨噬细胞,尤其是肿瘤相关巨噬细胞,是肿瘤微环境的主要成分,也在肿瘤恶性进展中发挥作用[24]。本研究结果表明,FTO和YTHDF3均与CD4+和CD8+T细胞以及巨噬细胞和NK细胞相关,这表明,其可能通过与免疫细胞相互作用的方式,在PTC患者的预后中发挥作用。
综上所述,本研究分析了20种m6A甲基化调节基因在PTC中的作用,发现m6A甲基化调节基因在PTC的发生和发展中起着重要作用,为PTC的深入研究提供了新的思路,具有较为重要的意义,未来需大量实验进行进一步研究验证。

