陆地棉粉花花蕾与黄花花蕾的比较转录组分析
刘林强 韩笑 杨兰 闫青地 胡伟 靳羽莹 于洁 杨召恩 李付广
摘要:【目的】挖掘影響棉花花色多态性的代谢物质和候选基因。【方法】以陆地棉黄色花品种中棉所24和粉色花品系中遗红为研究材料,取花瓣变色前后的花蕾进行花色苷含量测定和转录组测序分析。【结果】在开花前,中棉所24和中遗红的花蕾颜色已表现出差异,随着花蕾的发育,花色差异逐渐增大,其中天竺葵素-3-O-葡萄糖苷、矢车菊素-3-O-半乳糖苷、矢车菊素-3-O-葡萄糖苷和矢车菊素-3-O-(6''-O-丙二酰)-葡萄糖苷在中遗红花蕾中特异性积累。对中棉所24和中遗红花蕾中差异表达基因(differentially expressed gene, DEG)进行联合分析,共获得8 790个上调表达基因和8 521个下调表达基因,它们在液泡质子ATP酶(V型ATP酶)复合体、类黄酮生物合成过程等通路富集。进一步分析发现,花色苷合成相关的基因在2个材料中的表达水平并无明显差异,而调控花色苷合成、转运以及液泡酸碱平衡的基因Gh_A07G083500、bHLH基因Gh_D11G130400、GST基因Gh_A07G079800和V型ATP酶基因Gh_A09G123000、Gh_A08G012100和Gh_D09G115200在中遗红中高水平表达。【结论】本研究构建了粉花花色形成的调控通路,鉴定了与棉花花色相关的候选基因,为棉花育种提供基因资源。
关键词:陆地棉;花色苷;转录组;花蕾
Comparison of the transcriptomes between pink flower buds and yellow flower buds in upland cotton
Liu Linqiang, Han Xiao, Yang Lan, Yan Qingdi, Hu Wei, Jin Yuying, Yu Jie, Yang Zhaoen*, Li Fuguang*
(Institute of Cotton Research, Chinese Academy of Agricultural Sciences/State Key Laboratory of Cotton Biology, Anyang, Henan 455000, China)
Abstract: [Objective] This study aims to detect the metabolites and candidate genes which affect the polymorphsim of cotton flower color. [Method] Before and after petal color was changed, the flower buds of yellow flower cotton variety CCRI 24 and pink flower cotton line Zhongyihong were extracted to perform anthocyanin content detection and RNA-seq analysis. [Result] Before flowering, the color of CCRI 24 buds and Zhongyihong buds was different, and the difference was gradually increased with the development of buds. Among them, pelargonidin-3-O-glucoside, cyanidin-3-O-galactoside, cyanidin-3-O-glucoside (Kuromanin) and cyanidin-3-O-(6''-O-malonyl)-glucoside were specifically accumulated in Zhongyihong buds. A total of 8 790 up-regulated and 8 521 down-regulated genes were obtained by differentially expressed gene (DEG) analysis between CCRI 24 and Zhongyihong, which were enriched in pathways such as proton-transporting V-type ATPase complex and flavonoid biosynthesis. Further analysis showed that there was no significant difference in the expression of genes related to anthocyanin synthesis between the two materials, while Gh_A07G083500, bHLH gene Gh_D11G130400, GST gene Gh_A07G079800, and V-type ATPase genes Gh_A09G123000, Gh_A08G012100, and Gh_D09G115200 involved in regulating anthocyanin synthesis, transporting and vacuolar acid-base balance were highly expressed in CCRI 24. [Conclusion] This study demonstrated the gene expression pathway of pink flower formation, and identified several candidate genes related to the flower color of cotton, which provide genetic resources for cotton breeding.
Keywords: upland cotton (Gossypium hirsutum L.); anthocyanin; transcriptome; flower bud
花是植物重要的生殖器官,起着繁衍后代的作用。花瓣具有不同颜色,能够吸引昆虫鸟兽帮忙传递花粉完成受精过程[1]。此外,花色还可以保护植物免受紫外线辐射、病原体入侵和草食动物摄食[2]。
类胡萝卜素、甜菜碱和类黄酮的积累导致植物形成不同的颜色,其中花色苷类黄酮的积累是花色形成的主要原因[3]。花色苷广泛分布在植物器官中,在位于内质网上一系列酶的催化作用下从苯丙烷类化合物衍生而来[4]。编码花色苷合成的基因在许多植物中被广泛分离,其可以分为两类,一类是苯丙氨酸代谢相关基因,包括苯丙氨酸裂解酶(phenylalanine ammonia-lyase, PAL)、肉桂酸-4-羟化酶(cinnamate-4-hydroxylase, C4H)、4-香豆酸辅酶A连接酶(4-coumarate coenzyme A ligase, 4CL)、查耳酮合酶(chalcone synthase, CHS);另一类是黄酮类代谢相关基因,包括查尔酮异构酶(chalcone isomerase, CHI)、黄烷酮3-羟化酶(flavanone 3-hydroxylase, F3H)、类黄酮 3'-羟化酶(flavonoid 3'-hydroxylase, F3'H)、二氢黄酮醇4-还原酶(dihydroflavonol 4-reductase, DFR)、花青素合酶(anthocyanidin synthase, ANS)和UDP-类黄酮糖基转移酶(UDP-glycose flavonoid glycosyltransferase, UFGT)。花色苷合成后在谷胱甘肽S-转移酶(glutathione-S-transferase, GST)的协助下,由ATP结合盒转运蛋白(ATP-binding cassette transporter, ABC)将花色苷从内质网转运到液泡中储存起来[5-6]。
研究表明,花色苷生物合成途径的调控涉及多个转录因子,包括R2R3型MYB、WD40和bHLH(基本螺旋-环-螺旋,basic helix-loop-
helix)蛋白[7],这些蛋白质共同形成三元复合物MYB-bHLH-WD40,该三元复合物与花色苷合成相关结构基因的启动子结合,增强相关基因的表达[8]。在这些调节因子中,R2R3-MYB是引起花色改变的关键转录因子[9-10]。例如:ZmC1是植物中首个报道的MYB类转录因子,可调控玉米花色苷的合成[11]。随后的系列研究发现,MYB转录因子在多种植物中调控花色苷代谢过程,例如拟南芥中的AtMYB75(AT1G56650)、AtMYB90(AT1G66390)、AtMYB113(AT1G66370)和AtMYB114(AT1G66380)[12-15],水稻中的OsC1(Os06g10350)[16-17],葡萄中的VvMYBA1(VIT_202s0033g00380)和VvMYBA2(VIT_02s0033g00390)以及苹果中的MdMYB3(MDP-0000187872)、MdMYB10(MDP0000259614)和MdMYB110a(MD17G1261000)[18-22],都属于激活型转录因子,对花色苷代谢途径具有正向调控作用。此外,抑制型MYB转录因子也参与花色苷的代谢调控,例如拟南芥AtMYBL2、矮牽牛PhMYB27和PhMYBx等与激活型MYB转录因子产生拮抗作用,避免花色苷的过度积累[23-24]。
棉属(Gossypium spp.)包含50多个种,花色具有极高的多样性,有白色、黄色、粉色和红色等。陆地棉花色以黄色为主,开花后随着花色苷在花瓣中的积累,花色发生改变。花色苷生物合成相关基因的上调表达刺激花色苷的合成和积累,最终导致棉花的花色发生改变[25]。本研究从陆地棉核心资源材料中筛选获得1个粉花材料——中遗红,通过对花蕾中花色苷含量测定和转录组分析,比较粉花的中遗红与黄花的中棉所24在花蕾变色前后花色苷类色素含量,鉴定2种材料在相同发育时期和同一材料在不同发育时期的差异表达基因(differentially expressed gene, DEG),对DEG进行功能富集分析,并重点对花色苷合成、调控及转运相关的基因进行分析,筛选关键候选基因,为深入研究棉花花色的遗传机制提供基因资源和材料资源。
1 材料与方法
1.1 试验材料
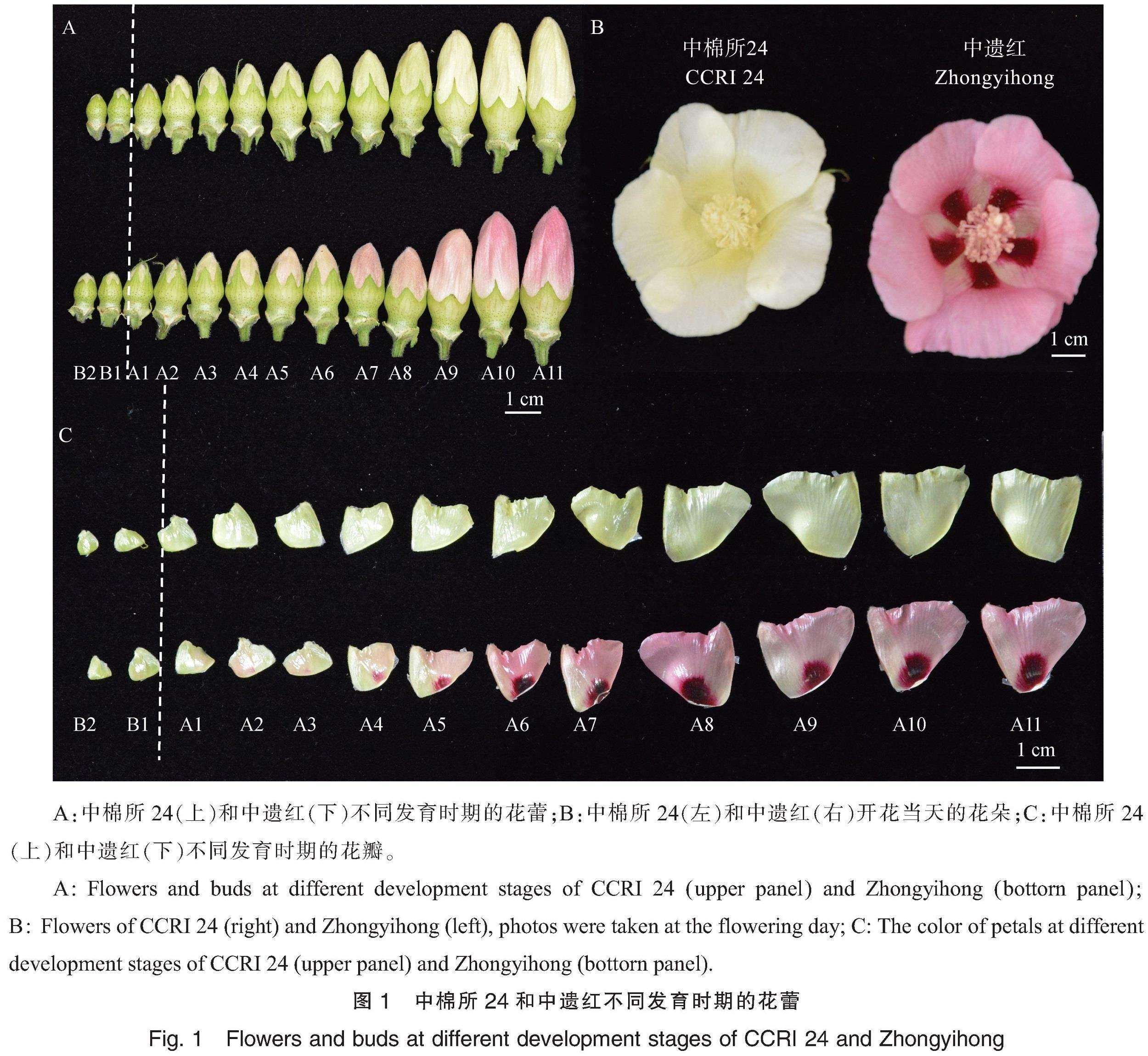
本研究以陆地棉黄花品种中棉所24和粉花品系中遗红为试验材料,种植于中国农业科学院棉花研究所三亚大茅南繁基地(109°65′E, 18°34′N)。每个材料种植6行,行长5 m,行距80 cm、株距20 cm。2019年10月27日种植,次年1月中旬观察花色变化。依据花蕾颜色变化和大小,分为变色前B1、B2和变色后A1、A2、A3、A4、A5、A6、A7、A8、A9、A10和A11共13个发育时期(图1)。上午10:00,分别选取变色前B1时期和红色趋于稳定的A9时期的花蕾,5个单株的花蕾混为1个样品,取样后用锡箔纸包住,立即放入液氮中速冻,并置于-80 ℃超低温冰箱保存,用于转录组测序和花色苷类代谢物分析。每个处理设3个生物学重复。以W-B1和W-A9分别表示中棉所24的B1和A9时期的花蕾,以P-B1和P-A9分别表示中遗红B1和A9时期的花蕾。取B1时期的花蕾以及A9时期的花蕾、根、茎、叶,检测基因表达情况,每个材料设3个生物学重复。
1.2 花色苷含量的测定
将花色变化前B1时期和变化后A9时期的花蕾进行冷冻干燥和研磨。称取100 mg粉末溶解于1.2 mL的70%(体积分数)甲醇提取液中,溶解后的样品于4 ℃冰箱过夜,期间涡旋震荡6次以提高提取率。浸提后于12 000 r·m-1离心10 min,经0.22 μm微孔滤膜过滤,得到滤液,利用超高效液相色谱和串联质谱进行花色苷类色素含量测定[28]。
1.3 总RNA提取及转录组测序
利用天根生化科技(北京)有限公司的RNA提取试剂盒(货号:DP441)提取样品的总RNA。对获得的总RNA进行质量检测,检测合格的样品利用Oligo(dT)富集mRNA,并通过 Fragmentation 缓冲剂使其短片段化,以短片段mRNA合成双链cDNA,纯化后进行末端修复、加碱基A、加测序接头,进行PCR扩增,最后使用因美纳(Illumina)NovaSeq 6000测序平台进行测序。
1.4 mRNA-seq数据分析
为了保证后续信息分析的可靠性,剔除原始序列中的接头和低质量的读长(reads)序列,获得高质量的干净序列(clean reads)。使用Hisat2[26]软件将干净序列比对到陆地棉TM-1的参考基因组上(G.hirsutum_TM-1_ICR, https://grand.cricaas.com.cn/),利用Stringtie软件进行基因表达量的分析,以TPM(transcripts per kilobase of exon model per million mapped reads,每千个碱基的转录每百万映射读取的转录本)对表达量进行均一化处理,用Feature count进行不同基因座上序列读长数目(reads count)统计。
1.5 差异基因的筛选
利用R语言包DESeq2对中遗红和中棉所24的表达基因进行差异分析[27]。使用Benjamini Hochberg方法校正P值,以校正后的P值小于0.05和log2(FC)(fold change, 差异表达倍数)绝对值大于等于1为标准筛选差异表达基因。
1.6 GO富集分析和KEGG代谢通路分析
使用perl脚本提取TM-1基因组的基因注释,获得DEG的功能注释。使用BioConduct的GO.seq包,利用超几何检验进行DEG的功能富集分析和KEGG代谢通路分析。
1.7 候选基因表达分析
利用Primer3 plus设计引物(表1),参照GenStar公司的2×RealStar Green Fast Mixture试剂盒(货号:A301-01)说明书,进行实时定量聚合酶链式反应(quantitative real-time polymerase chain reaction, qRT-PCR)检测。扩增体系为20 μL(1 μL cDNA,上下游引物各0.5 μL,10 μL 2× RealStar Green Fast Mixture,8.0 μL ddH2O)。反应程序为:95 ℃预变性2 min,95 ℃变性15 s,60 ℃退火15 s,72 ℃延伸10 s,40个循环。以棉花Gh_D03G042200(GhHistone)为内参基因,每个取样时间点设3次生物学重复,每个样品设置3次技术重复。使用2-ΔΔCt方法进行表达量的计算。
2 结果与分析
2.1 棉花花蕾不同发育时期的着色情况
对中棉所24和中遗红不同发育时期的花蕾进行调查,并根据花色变化和花蕾大小将中遗红和中棉所24的花色差异分为变色前(B1和B2)和变色后(A1~A11)两个阶段13个时期(图1A和1C)。在开花前(图1A和1C)和开花当天(图1B),中棉所24的花蕾和花瓣均呈现黄色,而中遗红的花蕾和花瓣顏色在发育过程中呈动态变化,红色逐渐加深。在B1和B2时期,中遗红的花蕾和花瓣颜色与中棉所24并无明显差别;而中遗红花蕾和花瓣颜色自A1开始呈现出浅粉色,并且随着发育进程推进从A1~A8时期颜色逐渐加深,A9~A11时期花蕾颜色趋于稳定。
花色苷是棉花花蕾显色的主要原因,花瓣中的花色苷主要可以分为矢车菊素、飞燕草素、锦葵花素、天竺葵素、矮牵牛素和芍药花素等6大类,其中矢车菊素和天竺葵素在酸性液泡中呈现红色是花瓣显色的主要原因[29]。分别对中遗红和中棉所24花色变化前的B1时期、快速着色期A9时期的花蕾进行花色苷类类色素含量检测,共检测到11种花色苷色素(表2),在B1时期,与中棉所24花蕾相比,中遗红花蕾中矢车菊素-3-O-(6''-O-丙二酰)-葡萄糖苷含量明显增高,其它10种花色苷色素的含量无显著差异;在A9时期,矢车菊素-3-O-葡萄糖苷、矢车菊素-3-O-半乳糖苷、天竺葵素-3-O-葡萄糖苷和矢车菊素-3-O-(6''-O-丙二酰)-葡萄糖苷4种花色苷类色素在中遗红花蕾中的含量明显高于中棉所24花蕾中的含量,表明这些色素的积累可能是粉花花色形成的主要原因。
2.2 测序数据质量分析
经过严格的质量控制后,RNA-seq共获得3.1亿对双末端序列,共计91.55 Gb的序列数据,GC含量为43.63%~43.99%,Q20碱基均大于97.5%,Q30碱基均大于93.74%(表3),说明测序的错误率较低,数据质量符合后期数据分析的要求。
2.3 基因表达量分析
分析不同样品生物学重复之间的皮尔逊相关系数(图2),除了P-B1-2和P-B1-3的相关系数较低为0.86外,其他同一样品的不同生物学重复之间具有很高相关性,相关系数均大于等于0.90;同一发育时期的不同样品间也具有较高的相关性,相关系数均大于等于0.78。以上结果表明,试验测序样品的生物学重复性好、数据可靠。此外,同一发育时期不同花色的花蕾转录组间的相关系数(0.78≤r≤0.99)大于同一材料不同发育时期的花蕾转录组间的相关系数(0.28≤r≤0.54),说明棉花花蕾生长发育对基因表达的影响远大于花色对基因表达的影响。
为了验证转录组中基因表达水平的结果,选取6个DEG进行qRT-PCR分析。由图3可知,qRT-PCR检测的6个基因的表达量的变化趋势与其在转录组的趋势基本一致,两者相关性很高(r为0.75~1.00),表明转录组数据较为可靠。
2.4 差异表达基因的筛选
在4个测序样品间共检测出17 311个DEG,包括8 790个上调基因和8 521个下调的基因。在花色变化前2个材料间(P-B1/W-B1)有768个DEG,在花色变化后(P-A9/W-A9)2个材料间仅有99个DEG。同一材料的花蕾不同发育时期间进行比较,P-A9/P-B1组和W-A9/W-B1组的DEG数量分别为14 776和12 149个(表4)。
为了全面评估DEG对花色的影响,筛选在P-A9/W-A9组和P-A9/P-B1组中同时差异表达的基因共55个,包含20个上调DEG和35个下调DEG(图4)。进一步分析发现,调控花色苷生物合成的MYB转录因子的编码基因Gh_A07G083500和参与将花色苷由细胞质转运到液泡中的谷胱甘肽-S-转移酶的编码基因Gh_A07G079800均上调表达。对这2个基因的表达模式进行分析发现,Gh_A07G083500和Gh_A07G079800在中遗红的根和叶中的相对表达量都低于其在中棉所24中的表达水平,但在中遗红花蕾中的相对表达量显著高于其在中棉所24花蕾中的相对表达量(图5)。
2.5 差异表达基因的功能富集
为了进一步分析花色发育中DEG的功能,取粉花与黄花花色变化前后以及粉花2个发育时期的DEG的集合,并对所得的基因集合减去黄花2个发育时期的DEG,获得可能与花色发育相关的DEG,包括2 215个上调表达基因和2 947个下调表达基因。
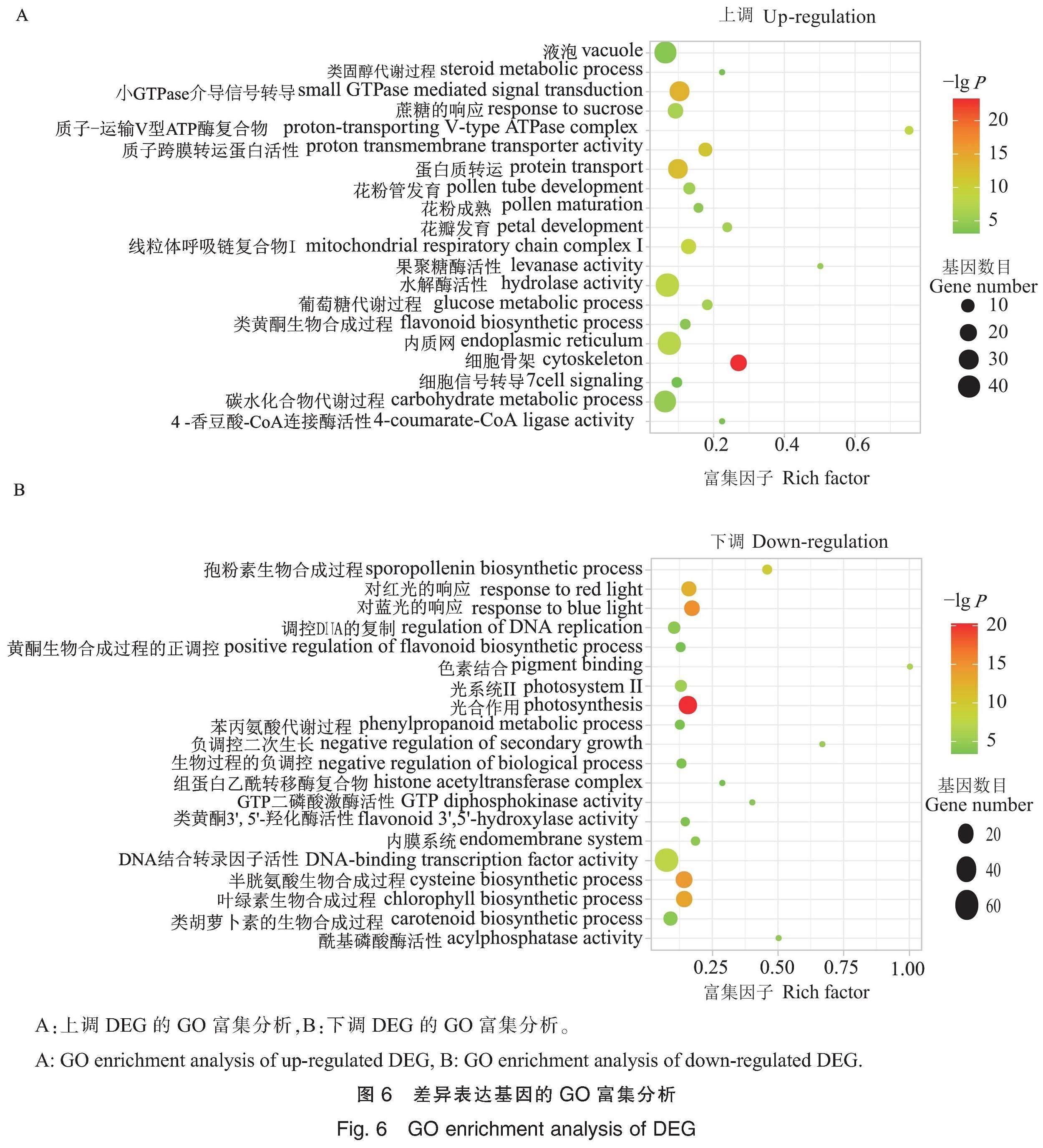
对这些DEG进行GO富集分析,发现它们主要富集在细胞骨架、光合作用、质子-运输V型ATP酶(液泡质子ATP酶,V型ATP酶)复合物的组装、类黄酮生物合成过程、4 -香豆酸-CoA连接酶活性、黄酮生物合成过程的正调控、类黄酮-3',5'-羟化酶活性和苯丙氨酸代谢等GO条目(图6)。其中,V型ATP酶复合物的组装通路包括3个参与液泡内质子转运、调节液泡pH的V型ATP酶基因Gh_A09G123000、Gh_A08G012100和Gh_D09G115200;编码调控黄酮类生物合成的bHLH转录因子Gh_D11G130400。研究表明,V型ATP酶和bHLH转录因子主要与液泡pH和类黄酮代谢调节相关,影响植物器官呈色[31-32]。
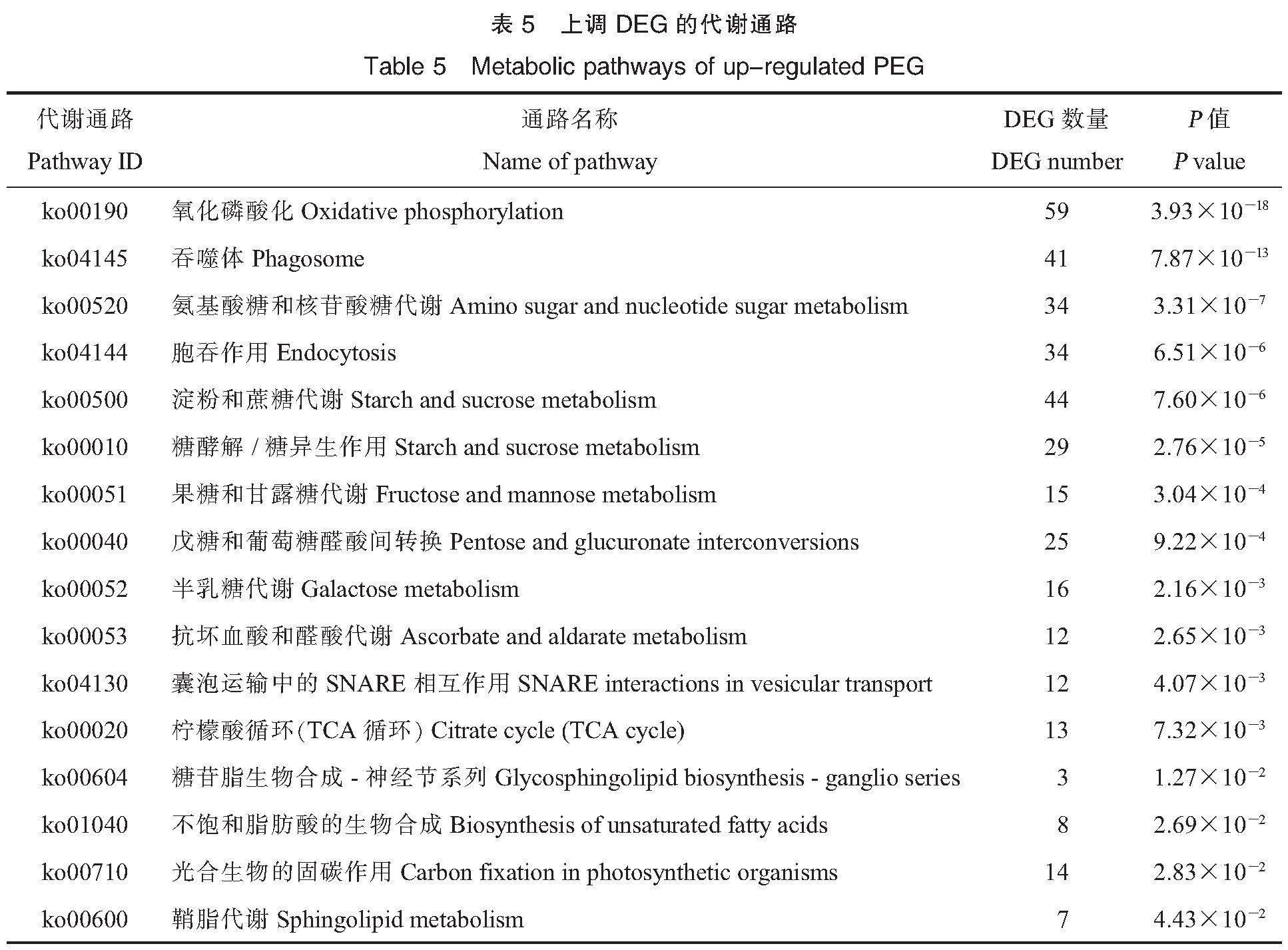
对DEG进行KEGG分析,上调DEG富集到氧化磷酸化、吞噬体、氨基酸糖和核苷酸糖代谢、胞吞作用、淀粉和蔗糖代谢、糖酵解/糖异生等16条代谢通路(表5),下调DEG富集到光合作用、卟啉与叶绿素代谢、抗坏血酸和醛酸代谢、淀粉和蔗糖代谢、二萜类生物合成等13条代谢通路(表6)。然而上述代谢通路在花色苷合成和花色形成中的功能仍不清楚,需要进一步的研究。
2.6 花青素代谢通路中基因表达情况
对花色苷合成通路中的基因的表达量进行分析(图7),发现在黄花和粉花2个材料中花色苷合成途径中关键酶基因的表达量无明显差异,说明花色苷合成不是花色差异的原因。进一步对花色苷转运相关基因进行分析(图7),发现仅有花色苷转运相关的谷胱甘肽-S-转移酶基因Gh_A07G079800在粉花A9时期明显上调表达,与2.4中qRT-PCR结果一致,推测该基因参与的花色苷转运与粉花的花色形成密切相关。
3 讨论
花色苷在植物中广泛存在,苯丙氨酸是花色苷生物合成的前体物质[33],苯丙氨酸代谢通路相关基因表达模式的差异导致花色苷种类的多样性。本研究通过对粉花材料中遗红与黄花材料中棉所24的花蕾进行转录组测序并分析,结果表明,苯丙氨酸合成相关基因的表达量在2个材料间没有明显差异(图7),而调控花色苷转运的MYB基因Gh_A07G083500、bHLH基因Gh_D11G130400、谷胱甘肽-S-转移酶基因Gh_A07G079800和3个编码V型ATP酶的基因Gh_A08G012100、Gh_D09G115200和Gh_A09G123000在中遗红中高水平表达,是粉花形成的重要候选基因。
植物中多种转录因子在花色苷的生物合成中起重要作用[34],MYB-bHLH-WD40复合物调控花色苷的合成与转运[35-36]。本研究筛选的Gh_A07G083500基因与拟南芥中的AtPAP1为同源基因。在拟南芥中过表达AtPAP1,导致花色苷在其根、茎、叶和花中积累[12]。AtPAP1在其他物种中的同源基因过表達也会导致花色苷含量增多,DCmyb113在胡萝卜中过表达,导致整个植株花色苷剧增,从而呈现深红色[37]。Li等[38]在红色棉花T586定位了R1红色基因GhMYB113,在绿色棉花中过表达该基因会产生红色棉花植株,而GhMYB113转录因子与Gh_A07G083500为同源基因。Gh_D11G130400也是1种重要的转录因子,属于MYC家族,具有螺旋-环-螺旋(helix-loop-helix, HLH)结构域,氨基酸序列比对结果显示,Gh_D11G130400与拟南芥AtbHLH /TT8最为相似,后者调节花色苷的生物合成,由此我们推测Gh_A07G083500和Gh_D11G130400与中遗红的花瓣颜色形成相关。
研究表明,花色苷必须沉积在酸性的液泡中才能显示出鲜艳的颜色[39-40]。因此将花色苷运输到液泡对植物花瓣器官的显色至关重要。Francisco等[5]用生物化学的方法在葡萄中验证了ABC蛋白参与花色苷的运输,该转运过程必须依赖于谷胱甘肽转移酶,拟南芥中AtGSTF(TT19)作为载体将花色苷从胞质转移到液泡膜表面[41]。此外,从荔枝中分离出的花色苷转运体LcGST4能够回补拟南芥tt19突变体,并且该转运体受到LcMYB1转录因子的调控[42]。本研究通过转录组学分析,筛选到1个与拟南芥AtTT19 同源的基因Gh_A07G079800,在中遗红粉色花蕾中高水平表达,可能与花色苷的代谢途径相关,其具体生物学功能还有待进一步验证。
液泡的pH會改变花色苷的吸收光谱,从而改变组织和器官的颜色。在液泡膜上,不同类型的V型ATP酶控制着液泡的pH[31]。花色苷在酸性条件下(pH<3)呈稳定的红色,在弱酸性条件下(3<pH<6)时呈蓝色且稳定性显著下降[43]。Hu等[31]在苹果中用生化的方法证明了MdMYB1/10转录因子直接调节2个编码V型ATP酶的基因MdVHA-B1和MdVHA-B2的表达;MdVHA-B1在苹果的愈伤组织、果实、花瓣以及烟草中过表达,可以引起液泡酸化和花色苷积累。通过转录组分析,我们筛选到了3个编码V型ATP酶的基因Gh_A08G012100、Gh_D09G115200和Gh_A09G123000,它们在粉色花蕾中高水平表达,使液泡中H+含量增加,pH值降低,这可能是导致花瓣颜色变化的原因之一。
综上所述,我们构建了棉花花蕾中花色苷的调控和转运的模型(图8)。MYB转录因子和bHLH转录因子形成复合物参与调控GST,将花色苷从内质网经液泡膜上的ABC转运蛋白运到液泡中;同时该复合物也提高了Gh_A08G012100、Gh_D09G115200和Gh_A09G123000编码产物(H+-ATP酶)的活性,使大量H+运输到细胞液泡中,导致液泡中pH降低,在两者的共同作用下,使花瓣呈现出不同的颜色。
4 结论
以陆地棉黄花品种中棉所24和粉花品系中遗红开花前后(B1和A9时期)的花蕾为材料,进行代谢物分析,共鉴定出11种花色苷,其中4种在粉花A9时期花蕾中明显富集。进一步对2个材料B1时期和A9时期的花蕾进行转录组测序分析,发现花色苷合成通路的基因在2个材料中表达并无明显差异,而调控类黄酮类物质合成的Gh_A07G083500和Gh_D11G130400、花色苷转运Gh_A07G079800以及液泡酸碱平衡相关的Gh_A08G012100、Gh_D09G115200和Gh_D09G123000)等相关基因差异表达。本研究构建了棉花粉花形成的表达调控通路,筛选出调控棉花粉色花形成的候选基因,为研究棉花花色的遗传和分子机制研究提供参考。
参考文献:
[1] Stintzing F C, Carle R. Functional properties of anthocyanins and betalains in plants, food, and in human nutrition[J/OL]. Trends in Food Science and Technology, 2004, 15(1): 19-38[2020-12-04]. https://doi.org/10.1016/j.tifs.2003.07.004.
[2] Mouradov A, Spangenberg G. Flavonoids: a metabolic network mediating plants adaptation to their real estate[J/OL]. Frontiers in Plant Science, 2014, 5(10): 620[2020-12-04]. https://doi.org/10.3389/fpls.2014.00620.
[3] Tanaka Y, Sasaki N, Ohmiya A. Biosynthesis of plant pigments: anthocyanins, betalains and carotenoids[J/OL]. The Plant Journal, 2008, 54(4): 733-749[2020-12-04]. https://doi.org/10.1111/j.1365-313X.2008.03447.x.
[4] Belwal T, Singh G, Jeandet P, et al. Anthocyanins, multi-functional natural products of industrial relevance: recent biotechnological advances[J/OL]. Biotechnology Advances, 2020, 43(1): 107600[2020-12-04]. https://doi.org/10.1016/j.biotechadv.2020.107600.
[5] Francisco R M, Regalado A, Ageorges A, et al. ABCC1, an ATP binding cassette protein from grape berry, transports anthocyanidin 3-O-glucosides[J/OL]. The Plant Cell, 2013, 25(5): 1840-1854[2020-12-04]. https://doi.org/10.1105/tpc.112.102152.
[6] Alfenito M R, Souer E, Goodman C D, et al. Functional complementation of anthocyanin sequestration in the vacuole by widely divergent glutathione S-transferases[J/OL]. The Plant Cell, 1998, 10(7): 1135-1149[2020-12-04]. https://doi.org/10.1105/tpc.10.7.1135.
[7] Xu W, Lepiniec L, Dubos C. New insights toward the transcriptional engineering of proanthocyanidin biosynthesis[J/OL]. Plant Signaling and Behavior, 2014, 9(4): e28736[2020-12-04]. https://doi.org/10.4161/psb.28736.
[8] Xu W, Grain D, Bobet S, et al. Complexity and robustness of the flavonoid transcriptional regulatory network revealed by comprehensive analyses of MYB-bHLH-WDR complexes and their targets in Arabidopsis seed[J/OL]. New Phytologist, 2014, 202(1): 132-144[2020-12-04]. https://doi.org/10.1111/nph.12620.
[9] Lai Y S, Li H X, Yamagishi M. A review of target gene specificity of flavonoid R2R3-MYB transcription factors and a discussion of factors contributing to the target gene selectivity[J/OL]. Frontiers in Biology, 2013, 8(6): 577-598[2020-12-04]. https://doi.org/10.1007/s11515-013-1281-z.
[10] Dubos C, Stracke R, Grotewold E, et al. MYB transcription factors in Arabidopsis[J/OL]. Trends in Plant Science, 2010, 15(10): 573-581[2020-12-04]. https://doi.org/10.1016/j.tplants.2010.06.005.
[11] Paz-Ares J, Ghosal D, Wienand U, et al. The regulatory c1 locus of Zea mays encodes a protein with homology to myb proto-oncogene products and with structural similarities to transcriptional activators[J/OL]. The Embo Journal, 1987, 6(12): 3553-3558[2020-12-04]. https://doi.org/10.1002/j.1460-2075.1987.tb02684.x.
[12] Borevitz J O, Xia Y J, Blount J, et al. Activation tagging identifies a conserved MYB regulator of phenylpropanoid biosynthesis[J/OL]. The Plant Cell, 2000, 12(12): 2383-2393[2020-12-04]. https://doi.org/10.1105/tpc.12.12.2383.
[13] Zimmermann I M, Heim M A, Weisshaar B, et al. Comprehensive identification of Arabidopsis thaliana MYB transcription factors interacting with R/B-like bHLH proteins[J/OL]. The Plant Journal, 2004, 40(1): 22-34[2020-12-04]. https://doi.org/10.1111/j.1365-313X.2004.02183.x.
[14] Gonzalez A, Zhao M, Leavitt J M, et al. Regulation of the anthocyanin biosynthetic pathway by the TTG1/bHLH/Myb transcriptional complex in Arabidopsis seedlings[J/OL]. The Plant Journal, 2008, 53(5): 814-827[2020-12-04]. https://doi.org/10.1111/j.1365-313X.2007.03373.x.
[15] Stracke R, Werber M, Weisshaar B. The R2R3-MYB gene family in Arabidopsis thaliana[J/OL]. Current Opinion in Plant Biology, 2001, 4(5): 447-456[2020-12-04]. https://doi.org/10.1016/s1369-5266(00)00199-0.
[16] Saitoh K, Onishi K, Mikami I, et al. Allelic diversification at the C(OsC1) locus of wild and cultivated rice: nucleotide changes associated with phenotypes[J/OL]. Genetics, 2004, 168(2): 997-1007[2020-12-04]. https://doi.org/10.1534/genetics.103.018390.
[17] Sun X, Zhang Z, Chen C, et al. The C-S-A gene system regulates hull pigmentation and reveals evolution of anthocyanin biosynthesis pathway in rice[J/OL]. Journal of Experimental Botany, 2018, 69(7): 1485-1498[2020-12-04]. https://doi.org/10.1093/jxb/ery001.
[18] Chagne D, Kui L W, Espley R V, et al. An ancient duplication of apple MYB transcription factors is responsible for novel red fruit-flesh phenotypes[J/OL]. Plant Physiology, 2013, 161(1): 225-239[2020-12-04]. https://doi.org/10.1104/pp.112.206771.
[19] Vimolmangkang S, Han Y, Wei G, et al. An apple MYB transcription factor, MdMYB3, is involved in regulation of anthocyanin biosynthesis and flower development[J/OL]. BMC Plant Biology, 2013, 13(1): 176[2020-12-04]. https://doi.org/10.1186/1471-2229-13-176.
[20] Azuma A, Kobayashi S, Mitani N, et al. Genomic and genetic analysis of Myb-related genes that regulate anthocyanin biosynthesis in grape berry skin[J/OL]. Theoretical and Applied Genetics, 2008, 117(6): 1009-1019[2020-12-04]. https://doi.org/10.1007/s00122-008-0840-1.
[21] Espley R V, Hellens R P, Putterill J, et al. Red colouration in apple fruit is due to the activity of the MYB transcription factor, MdMYB10[J/OL]. The Plant Journal, 2007, 49(3): 414-427[2020-12-04]. https://doi.org/10.1111/j.1365-313X.2006.02964.x.
[22] Li Z T, Dhekney S A, Gray D J. Use of the VvMybA1 gene for non-destructive quantification of promoter activity via color histogram analysis in grapevine (Vitis vinifera) and tobacco[J/OL]. Transgenic Research, 2011, 20(5): 1087-1097[2020-12-04]. https://doi.org/10.1007/s11248-010-9482-6.
[23] Albert N W, Davies K M, Lewis D H, et al. A conserved network of transcriptional activators and repressors regulates anthocyanin pigmentation in Eudicots[J/OL]. The Plant Cell, 2014, 26(3): 962-980[2020-12-04]. https://doi.org/10.1105/tpc.113.122069.
[24] Matsui K, Umemura Y, Ohme-Takagi M. AtMYBL2, a protein with a single MYB domain, acts as a negative regulator of anthocyanin biosynthesis in Arabidopsis[J/OL]. The Plant Journal, 2008, 55(6): 954-967[2020-12-04]. https://doi.org/10.1111/j.1365-313X.2008.03565.x.
[25] Tan J, Wang M, Tu L, et al. The flavonoid pathway regulates the petal colors of cotton flower[J/OL]. PLoS One, 2013, 8(8): e72364[2020-12-04]. https://doi.org/10.1371/journal.pone.0072364.
[26] Pertea M, Kim D, Pertea G M, et al. Transcript-level expression analysis of RNA-seq experiments with HISAT, stringtie and ballgown[J/OL]. Nature Protocols, 2016, 11(9): 1650-1667[2020-12-04]. https://doi.org/10.1038/nprot.2016.095.
[27] Robinson M D, Mccarthy D J, Smyth G K. edgeR: a bioconductor package for differential expression analysis of digital gene expression data[J/OL]. Bioinformatics, 2010, 26(1): 139-140[2020-12-04]. https://doi.org/10.1093/bioinformatics/btp616.
[28] Wang Y T, Li S P, Zhu Z Q, et al. Transcriptome and chemical analyses revealed the mechanism of flower color formation in Rosa rugosa[J/OL]. Frontiers in Plant Science, 2022, 3(1): 231[2023-08-29]. https://doi.org/10.3389/fpls.2022.1021521.
[29] Freyre R, Uzdevenes C, Gu L, et al. Genetics and anthocyanin analysis of flower color in mexican petunia[J/OL]. Journal of the American Society for Horticultural Science, 2015, 140(1): 45-49[2020-12-04]. https://doi.org/10.21273/JASHS.140.1.45.
[30] Zhang Q, Zhang J, Gong M, et al. Transcriptome analysis of the gene expression profiles associated with fungal keratitis in mice based on RNA-Seq[J/OL]. Investigative Ophthalmology and Visual Science, 2020, 61(6): 32[2020-12-04]. https://doi.org/ 10.1167/iovs.61.6.32.
[31] Hu D G, Sun C H, Ma Q J, et al. MdMYB1 regulates anthocyanin and malate accumulation by directly facilitating their transport into vacuoles in apples[J/OL]. Plant Physiology, 2016, 170(3): 1315-1330[2020-12-04]. https://doi.org/10.1104/pp.15.01333.
[32] Xie X B, Li S, Zhang R F, et al. The bHLH transcription factor MdbHLH3 promotes anthocyanin accumulation and fruit colouration in response to low temperature in apples[J/OL]. Plant Cell Environment, 2012, 35(11):1884-1897[2020-12-04]. https://doi.org/10.1111/j.1365-3040.2012.02523.x.
[33] 龐红霞, 祝长青, 覃建兵. 植物花青素生物合成相关基因研究进展[J]. 种子, 2010, 29(3): 60-64. Pang Hongxia, Zhu Changqing, Qin Jianbing. Advancement of plant genes related with anthocyanins synthetic biology[J]. Seed, 2010, 29(3): 60-64.
[34] Falcone M L, Rius S P, Casati P. Flavonoids: biosynthesis, biological functions, and biotechnological applications[J/OL]. Frontiers in Plant Science, 2012, 3(1): 222[2020-12-04]. https://doi.org/10.3389/fpls.2012.00222.
[35] Wu Y, Guo J, Zhou Q, et al. De novo transcriptome analysis revealed genes involved in flavonoid biosynthesis, transport and regulation in Ginkgo biloba[J/OL]. Industrial Crops and Products, 2018, 124: 226-235[2020-12-04]. https://doi.org/10.1016/j.indcrop.2018.07.060.
[36] Allan A C, Hellens R P, Laing W A. MYB transcription factors that colour our fruit[J/OL]. Trends in Plant Science, 2008, 13(3): 99-102[2020-12-04]. https://doi.org/10.1016/j.tplants.2007.11.012.
[37] Xu Z S, Yang Q Q, Feng K, et al. DcMYB113, a root-specific R2R3-MYB, conditions anthocyanin biosynthesis and modification in carrot[J/OL]. Plant Biotechnology Journal, 2020, 18(7): 1585-1597[2020-12-04]. https://doi.org/10.1111/pbi.13325.
[38] Li X, Ouyang X, Zhang Z, et al. Over-expression of the red plant gene R1 enhances anthocyanin production and resistance to bollworm and spider mite in cotton[J/OL]. Molecular Genetics and Genomics, 2019, 294(2): 469-478[2020-12-04]. https://doi.org/10.1007/s00438-018-1525-3.
[39] Winkel B. Flavonoid biosynthesis. a colorful model for genetics, biochemistry, cell biology, and biotechnology[J/OL]. Plant Physiology, 2001, 126(2): 485-493[2020-12-04]. https://doi.org/10.1104/pp.126.2.485.
[40] Yamazaki M, Yamagishi E, Gong Z Z, et al. Two flavonoid glucosyltransferases from Petunia hybrida: molecular cloning, biochemical properties and developmentally regulated expression[J/OL]. Plant Molecular Biology, 2002, 48(4): 401-411[2020-12-04]. https://doi.org/10.1023/a:1014043214943.
[41] Sun Y, Li H, Huang J R. Arabidopsis TT19 functions as a carrier to transport anthocyanin from the cytosol to tonoplasts[J/OL]. Molecular Plant, 2012, 5(2): 387-400[2020-12-04]. https://doi.org/10.1093/mp/ssr110.
[42] Hu B, Zhao J, Lai B, et al. LcGST4 is an anthocyanin-related glutathione S-transferase gene in Litchi chinensis Sonn.[J/OL]. Plant Cell Reports, 2016, 35: 831-843[2023-9-11]. https://doi.org/10.1007/s00299-015-1924-4.
[43] Levi M, Scarminio L, Poppi R, et al. Three-way chemometric method study and UV-vis absorbance for the study of simultaneous degradation of anthocyanins in flowers of the Hibiscus rosa-sinensys species[J/OL]. Talanta, 2004, 62(2): 299-305[2020-12-04]. https://doi.org/10.1016/j.talanta.2003.07.015.

