小麦硝酸盐转运蛋白基因 TaNRT1.1的鉴定及其等位变异分析
王沙沙,黄绍敏,张珂珂,宋 晓,3
(1.河南省农业科学院小麦研究所/河南省小麦生物学重点实验室,河南郑州 450002;2.河南省农业科学院植物营养与资源环境研究所,河南郑州 450002;3.河南省农业生态环境重点实验室,河南郑州 450002)
相比于拟南芥和水稻,小麦硝酸盐转运蛋白方面的研究主要集中在小麦NRT1基因家族成员的鉴定方面[6,24-26],而对于每个家族成员潜在的分子机制尚不清楚。因此,本研究拟克隆小麦硝酸盐转运蛋白基因TaNRT1.1,并通过生物信息学分析、不同组织qRT-PCR分析及多态性筛选等方法对其进行研究,为解析小麦硝酸盐转运蛋白基因的生物学功能提供参考,并为进一步开发其功能标记及小麦分子标记辅助育种奠定基础。
1 材料与方法
1.1 试验材料
小麦品种许科168、郑麦113、中育1211、周麦27、漯麦18和郑品麦8号来自黄淮麦区,具有不同的遗传背景且氮利用效率差异明显。取普通小麦中国春露地栽培自然生长的幼苗根、茎、叶,用于分析TaNRT1.1三个同源基因在小麦不同组织材料中的表达模式。取小麦品种周麦27、许科168、郑麦113苗期的根,用于分析TaNRT1.1基因不同等位变异在小麦根中的表达模式。所有小麦品种均由河南省农业科学院小麦研究所小麦营养与品质研究室提供。
1.2 DNA、总RNA的提取及cDNA合成
参照Chen等[27]的SLS(十二烷基肌氨酸钠)方法提取小麦种子DNA,并略有改进。利用Trizol法提取中国春幼苗的根、茎、叶总RNA。用PrimeScriptTMRT reagent Kit with gDNA Eraser试剂盒(TaKaRa,大连)说明书将RNA反转录为cDNA,-20 ℃保存。
1.3 小麦 TaNRT1.1基因的克隆
根据水稻OsNRT1.1B基因(LOC_Os08g12780)序列,通过中国春参考基因组2.0以及Ensemble Plants进行同源Blast,获得小麦硝酸盐转运蛋白基因TaNRT1.1(TaNRT1.1-1A、TaNRT1.1-1B、TaNRT1-1D)及其cDNA序列。通过矮抗58数据库(未公布)和已克隆的TaNRT1.1三个同源基因,获得这三个同源基因的启动子序列。
分别设计TaNRT1.1-1A、TaNRT1.1-1B、TaNRT1.1-1D基因的特异性引物TaNRT1.1-P1、TaNRT1.1-P2、TaNRT1.1-P3(表1),以中国春cDNA为模板进行PCR扩增。PCR反应体系25 μL,包括cDNA 100 ng,上、下引物(10 pmol·μL-2)各0.5 μL,10×Taqbuffer (Mg2+plus)2.5 μL,dNTP (2.5 mmol·L-1)0.5 μL、TaqDNA聚合酶1.25 U,灭菌ddH2O补足至25 μL(以上所有试剂均购自于北京天根生化科技有限公司)。PCR扩增程序:94 ℃预变性5 min;94 ℃变性30 s,55~60 ℃(各引物的具体温度见表1)退火30 s,72 ℃延伸2 min,共35个循环;72 ℃延伸7 min。用含溴化乙锭(EB)的1.5%琼脂糖凝胶电泳对PCR产物进行分离和检测。之后利用SanPrep柱式DNA胶回收试剂盒(生工,上海)进行片段回收,回收的目的片段连接至pMD18-T克隆载体上,并转化至大肠杆菌DH5α感受态细胞中。用单克隆菌落PCR检测筛选阳性克隆,然后送至上海生工工程有限公司进行测序。用Chromos 2.4.3 (http://technelysium.com.au/?page_id=13)和FinchTV 1.5.0(http://www.geospiza.com/ Products/finchtv.shtml)软件对测序结果进行可靠性检验和分析。最终选择测序正确的单克隆(TaNRT1.1-1A-pMD18-T、TaNRT1.1-1B-pMD18-T和TaNRT1.1-1D-pMD18-T)进行摇菌,并提取质粒,-20 ℃保存。
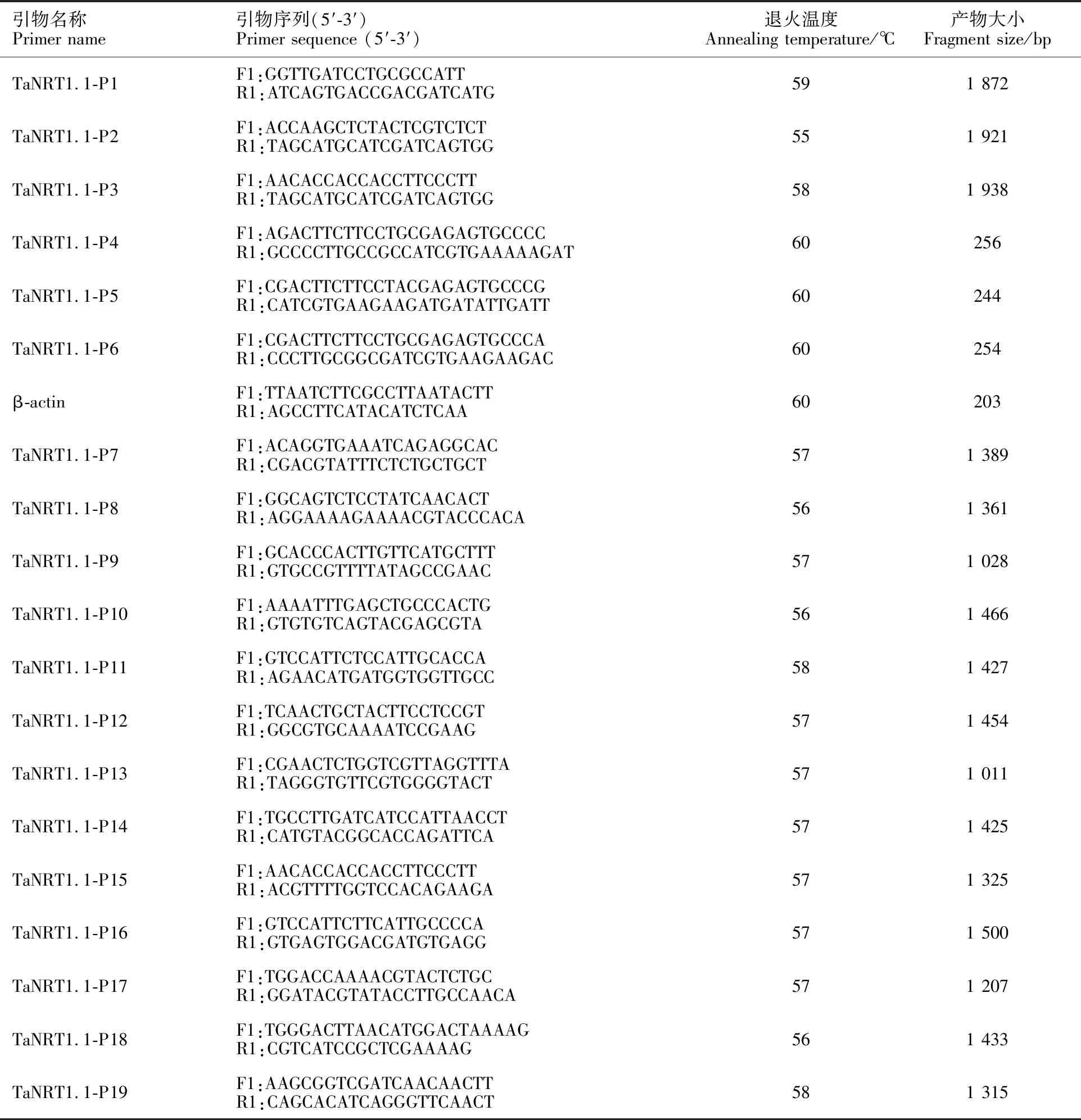
表1 本研究所有的引物Table 1 Primers used in this study
1.4 小麦 TaNRT1.1基因的顺式作用元件及其编码蛋白的生物信息学分析
用SMART软件(http://smart.embl-heidelberg.de/)及NCBI-CDD (https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)预测TaNRT1.1编码蛋白的信号肽及结构域;用ProParam (http://web.expasy.org/protparam/) 分析TaNRT1.1编码蛋白的氨基酸数目、相对分子量、等电点等理化性质;用TMHMM 2.0在线软件 (http://www.cbs.dtu.dk/services/TMHMM-2.0/)预测TaNRT1.1编码蛋白的跨膜区;用SPOMA (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_opma.html)和SWISS-MODEL(http://swissmodel.expasy.org/workspace/)在线分析软件预测TaNRT1.1编码蛋白的二级结构和三级结构;用PSORT II Prediction(http://psort.hgc.jp/form2.html)在线软件进行亚细胞定位;用Plantcare在线软件(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)预测TaNRT1.1基因启动子的顺式作用元件。
1.5 小麦 TaNRT1.1基因的表达模式及其等位变异分析
设计3个TaNRT1.1基因的荧光定量PCR特异性引物TaNRT1.1-P4、TaNRT1.1-P5、TaNRT1.1-P6以及小麦内参基因β-actin的特异性引物。分别以合成的中国春幼苗根、茎、叶cDNA为模板,应用iQ5荧光定量扩增仪(伯乐,美国)进行扩增。PCR反应体系为20 μL,包括上、下游引物(10 pmol·μL-1)各0.4 μL、2×SYBR Premix Ex Taq II(TakaRa,大连)10 μL、cDNA模板2 μL、灭菌ddH2O补足至20 μL。PCR反应程序:95 ℃ 2 min;95 ℃ 10 s,60 ℃ 20 s,72 ℃ 15 s,40个循环;72 ℃ 10 min。每个样品3次重复。采用2-△△CT方法分析TaNRT1.1基因在各组织中的表达情况。
另外,根据已克隆的TaNRT1.1基因序列,设计13对特异性引物(TaNRT1.1-P7~TaNRT1.1-P19)用于扩增其同源基因DNA序列及启动子序列,具体引物见表1。其中,特异性引物TaNRT1.1-P10用于小麦TaNRT1.1基因的多态性筛选,所用小麦材料为1.1节提到的6个不同氮利用效率小麦品种。以合成的周麦27、许科168、郑麦113苗期根cDNA为模板,用TaNRT1.1-1A基因的特异性引物TaNRT1.1-P4及小麦内参基因β-actin引物(表1)用于分析TaNRT1.1-A不同等位变异在小麦根中的表达情况。
2 结果与分析
2.1 小麦 TaNRT1.1基因的克隆结果
按照1.3节中的方法,获得水稻OsNRT1.1B基因在小麦中的三个同源基因,分别命名为TaNRT1.1-1A(TraesCS1A02G210900.1)、TaNRT1.1-1B(TraesCS1B02G224900.1)和TaNRT1.1-1D(TraesCS1D02G214200.1),其DNA序列全长分别为2 170、2 498和2 272 bp。序列分析发现,这三个基因均由3个外显子和2个内含子组成(图1),其编码序列长度均为 1 791 bp,可编码596个氨基酸。以中国春cDNA为模板进行PCR扩增,将目的片段回收后,将其连接到pMD18-T克隆载体上,经测序验证,最终获得小麦TaNRT1.1-1A、TaNRT1.1-1B和TaNRT1.1-1D的cDNA序列。
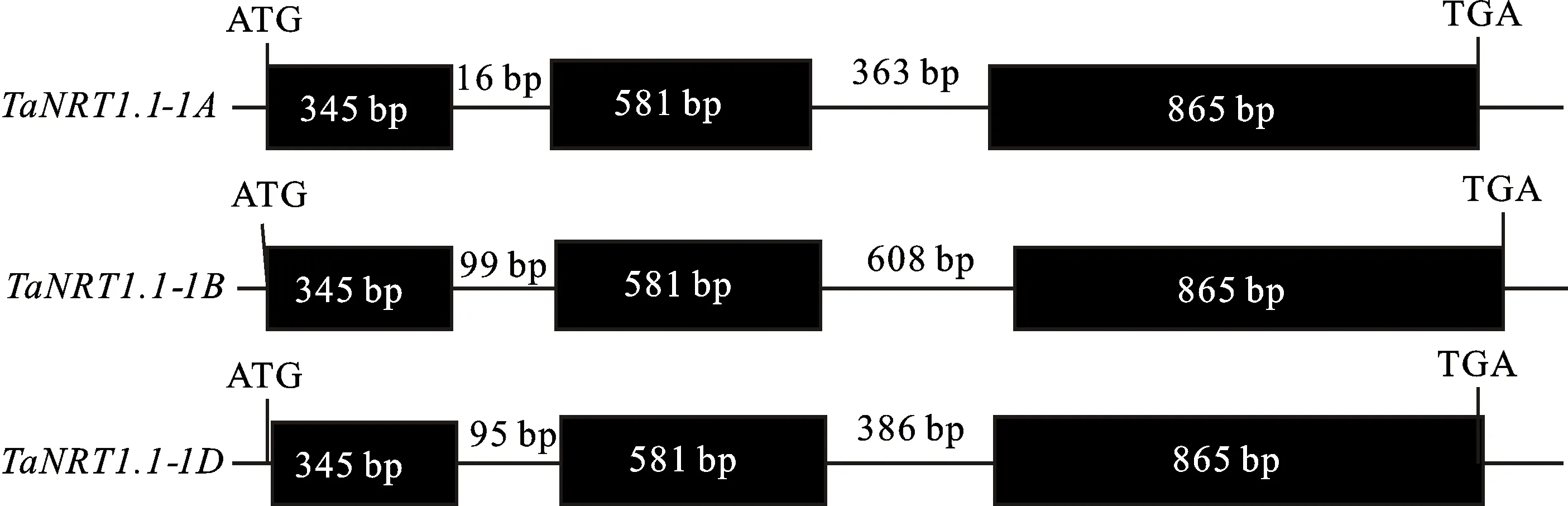
黑色方框代表 TaNRT1.1基因的外显子序列;黑色方框之间的连线为内含子序列。Black rectangular boxes represent the exons of TaNRT1.1gene;Lines between two black rectangular boxes represent the introns of TaNRT1.1gene.图1 小麦 TaNRT1.1基因结构示意图Fig.1 Schematic graph of the structures of TaNRT1.1gene in wheat
随后,根据矮抗58数据库(未公布)和已克隆的TaNRT1.1三个同源基因,并结合中国春的测序结果,成功获得长度均为3 000 bp的三个同源基因的启动子序列,分别是lcl|chr1A:373 762 258~373 765 258、lcl|chr1B:403 537 134~403 540 134和lcl|chr1D:299 571 952~299 574 952。
2.2 小麦 TaNRT1.1基因编码蛋白的生物信 息学
用SMART在线软件预测表明,小麦TaNRT1.1基因编码的蛋白包含PTR2(peptide transport family 2)家族的两个结构域,分别位于33~301 aa 和345~548 aa之间 (图2A)。生物信息学分析表明,TaNRT1.1基因编码的蛋白分子量为63.9 kD,理论等电点(pI)为7.55,为疏水性蛋白。用SOPMA软件预测表明,蛋白二级结构由4种形式构成(图2B),其中α-螺旋占比49.66%、延伸链占比12.58%、β转角占比3.69%、无规则卷曲占比34.06%。用SMART和TMHMM 2.0软件预测表明,该蛋白有跨膜结构区,为膜蛋白,无信号肽(图2C)。利用SWISS-MODEL进一步进行三级结构预测,结果表明,TaNRT1.1基因编码的蛋白含有丰富的α螺旋和无规则卷曲,与二级结构预测结果一致(图2D)。PSORT II Prediction软件预测结果显示,该蛋白主要定位在质膜上。
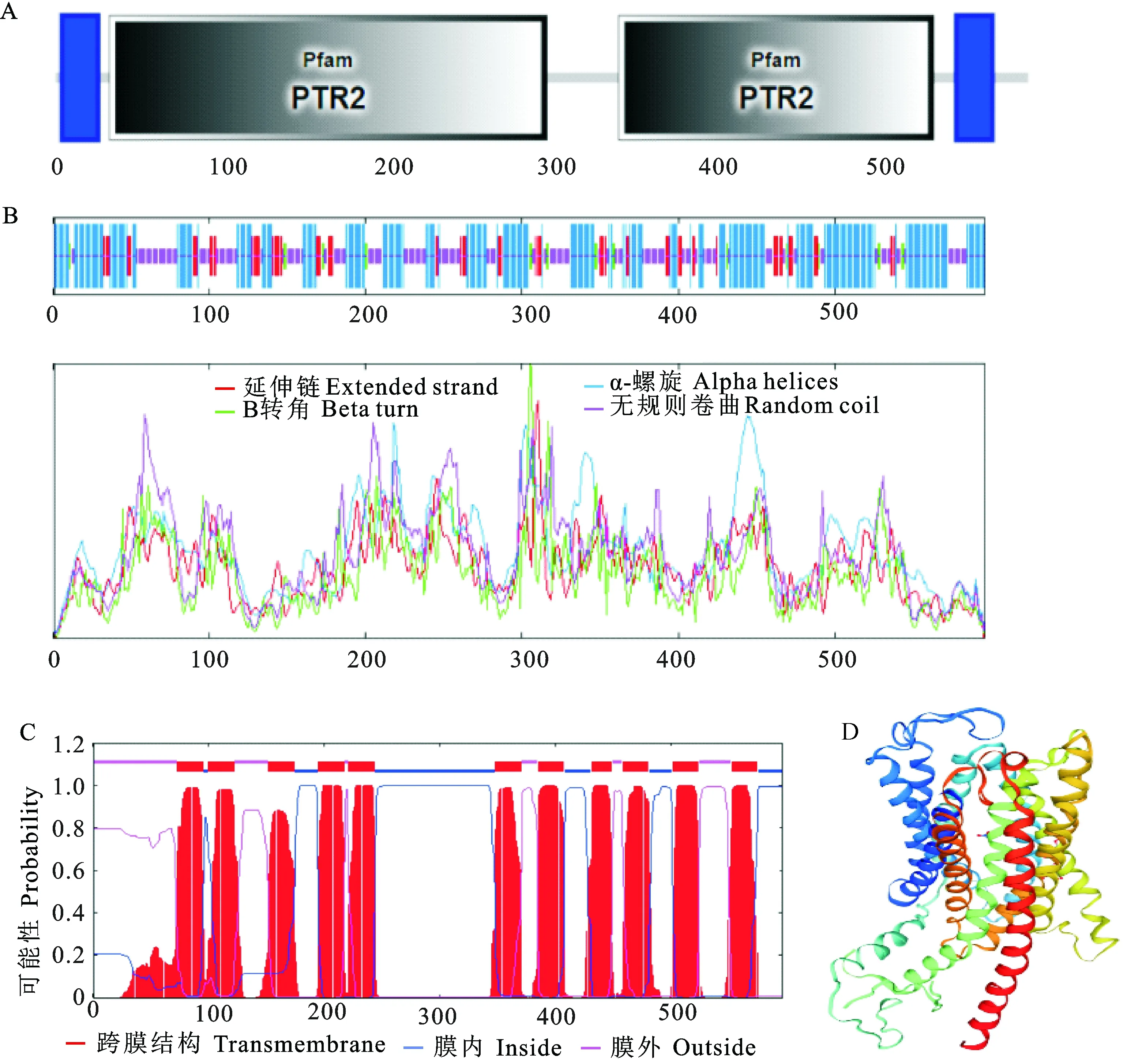
A:结构域预测;B:二级结构预测;C:跨膜结构预测;D:空间结构预测。A:Domain prediction;B:Secondary structure prediction;C:Transmembrane structure prediction;D:Spatial structure prediction.图2 小麦TaNRT1.1蛋白的生物信息学分析Fig.2 Bioinformatics analysis of TaNRT1.1 protein in wheat
2.3 小麦 TaNRT1.1基因启动子的顺式作用元件
为进一步研究小麦TaNRT1.1基因的调控机制,分别截取其起始密码子上游2 000 bp的基因组序列,利用Plantcare在线软件预测小麦TaNRT1.1基因启动子区域的顺式作用元件。结果(表2)发现,小麦TaNRT1.1基因启动子除含有一些基本元件(如TATA-box、CAAT-box)外,还富含光响应元件(如Box 4、GA-motif、GT1- motif等)、逆境响应元件(如CGTCA-motif、ARE、TCA-element、ABRE、MBS等)以及与生长发育有关的顺式作用元件(如HD-Zip 1、RY-element、CAT-box、Circadian等)。
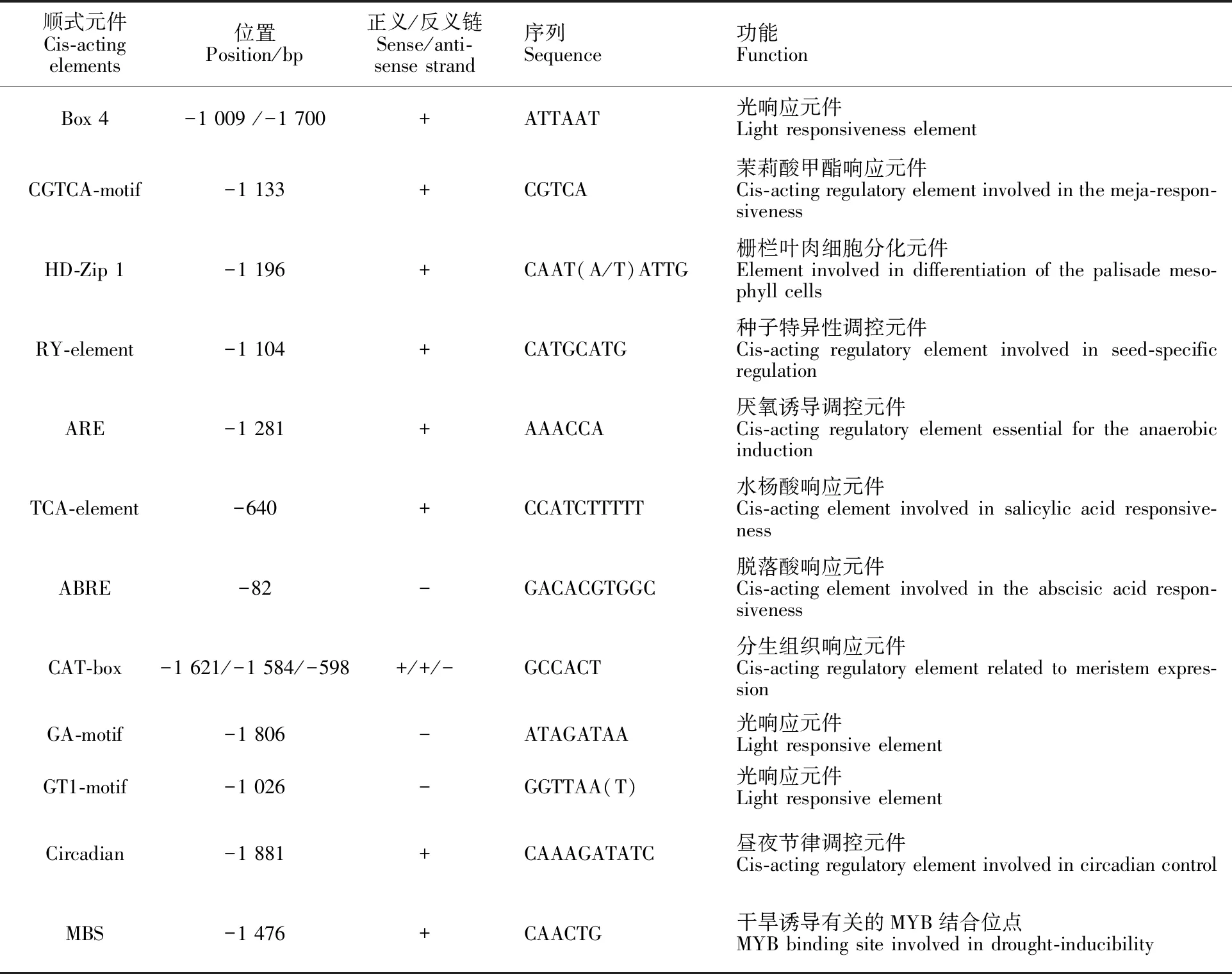
表2 小麦 TaNRT1.1基因启动子区域具有特殊功能的顺式作用元件Table 2 Cis-acting elements with special function of the promoter sequence of TaNRT1.1gene in wheat
2.4 小麦 TaNRT1.1基因的表达模式
对中国春小麦不同组织材料进行qRT-PCR检测,结果(图3)表明,TaNRT1.1-1A、TaNRT1.1-1B和TaNRT1.1-1D三个同源基因在根、茎、叶等组织中均有表达。其中,TaNRT1.1-1A和TaNRT1.1-1B基因均在根中表达量最高,叶中表达量次之,茎中表达量最低;而TaNRT1.1-1D基因在小麦茎中表达量最高,叶中表达量次之,根中表达量最低。
2.5 小麦 TaNRT1.1基因等位变异的检测结果
根据宋 晓等[28]的研究结果,挑选6个氮利用效率差异明显的小麦品种,利用13对特异性引物TaNRT1.1-P7~TaNRT1.1-P19对TaNRT1.1-1A、TaNRT1.1-1B和TaNRT1.1-1D三个基因DNA序列及启动子序列进行PCR扩增并测序,发现在TaNRT1.1-1A基因启动子上游1 120 bp的位置有一个8 bp(TGCATGCA)的插入位点(图4),测序结果证实了该位点的可靠性。而TaNRT1.1-1B和TaNRT1.1-1D基因DNA序列及启动子序列中均未发现多态性。结合前期对TaNRT1.1基因启动子顺式作用元件预测的结果,发现该位点位于光响应元件Box 4和GT1-motif附近。因此,按照Mcintosh等[29]对基因的命名方法,将TaNRT1.1-1A基因启动子上游 1 120 bp处按有无8 bp碱基插入分别命名为TaNRT1.1-1A-a和TaNRT1.1-1A-b。进一步研究发现,TaNRT1.1-1A-a基因型主要存在于氮低效型小麦品种中,而TaNRT1.1-1A-b基因型主要存在于氮高效型小麦品种中(表3)。因此,推测TaNRT1.1基因可能与小麦氮利用效率相关。
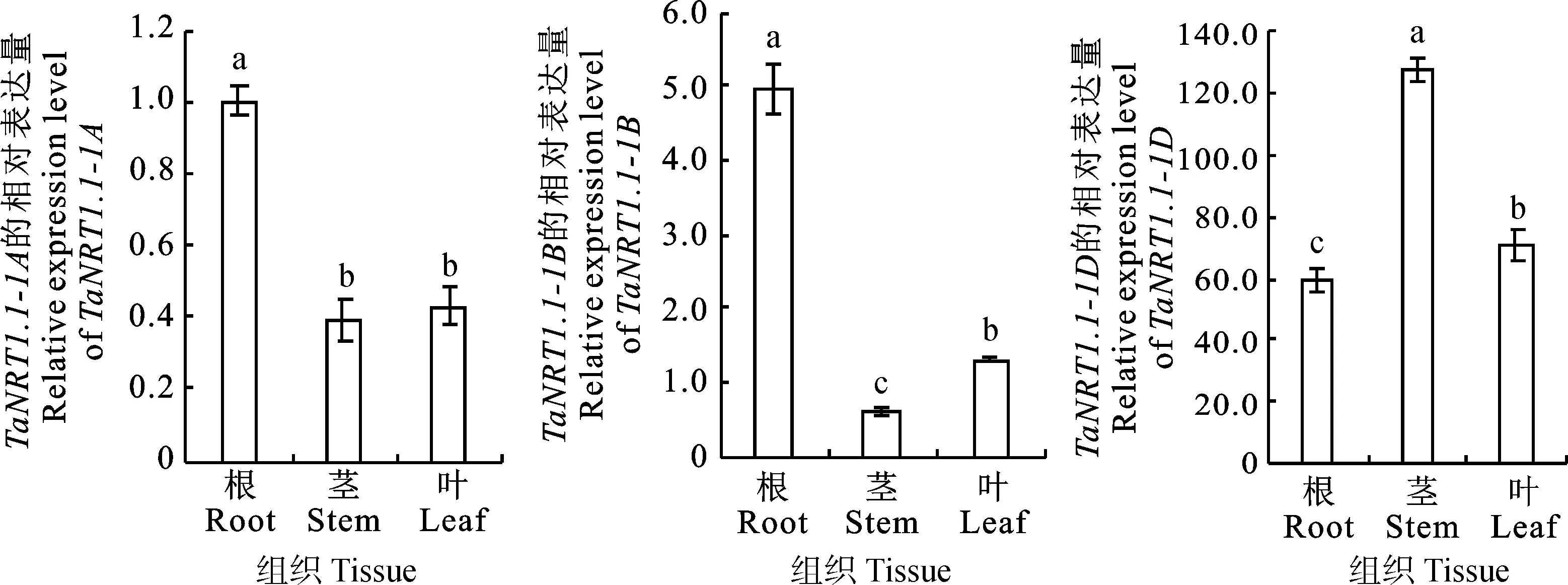
图柱上不同字母表示差异显著(P<0.05)。图5同。Different letters above columns mean significant difference(P<0.05)。The same in Fig.5图3 小麦 TaNRT1.1三个同源基因在不同组织中的相对表达量Fig.3 Relative expression levels of TaNRT1.1homoeologs in different tissues of wheat
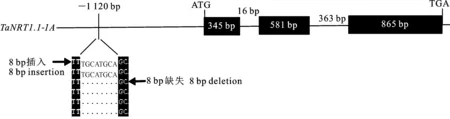
图4 TaNRT1.1基因的多态性分析Fig.4 Polymorphism analysis of TaNRT1.1gene
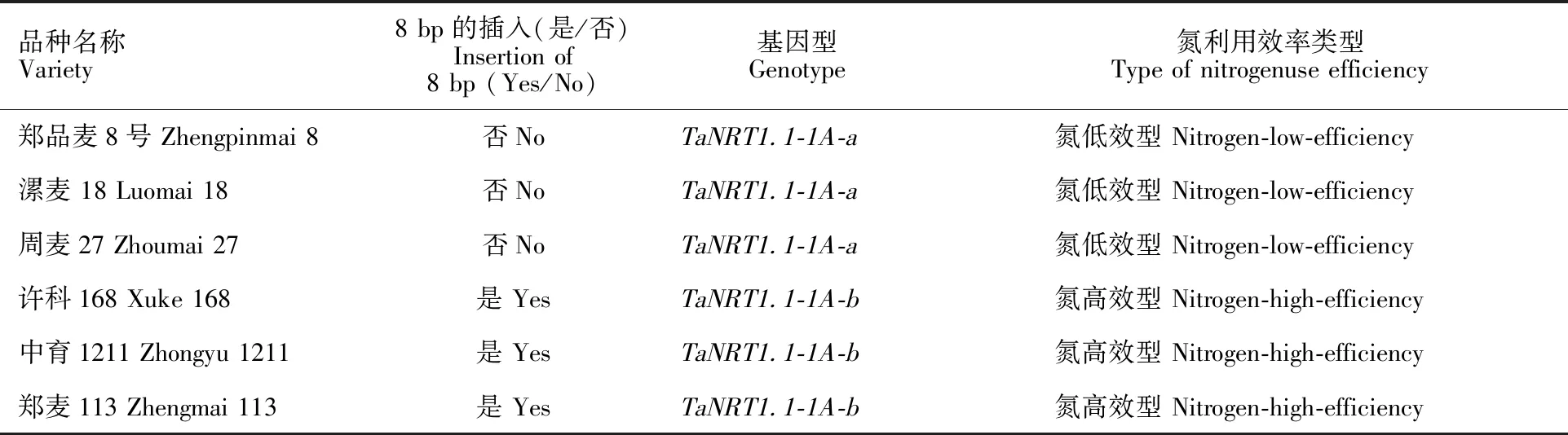
表3 TaNRT1.1基因等位变异对6个小麦品种氮利用效率的影响Table 3 Effect of TaNRT1.1allelic variations on nitrogen use efficiency of the six wheat varieties
2.6 TaNRT1.1-1A基因两种等位变异的表达模式
分别以1个氮低效型代表性小麦品种周麦27(基因型为TaNRT1.1-1A-a)和2个氮高效型代表性小麦品种许科168、郑麦113(基因型为TaNRT1.1-1A-b)苗期的根cDNA为模板,对TaNRT1.1基因进行表达模式分析。实时荧光定量PCR结果(图5)显示,TaNRT1.1-1A基因在氮高效小麦品种苗期根中的相对表达量显著高于在氮低效小麦品种中的相对表达量。推测TaNRT1.1基因可能正调控小麦氮利用效率。
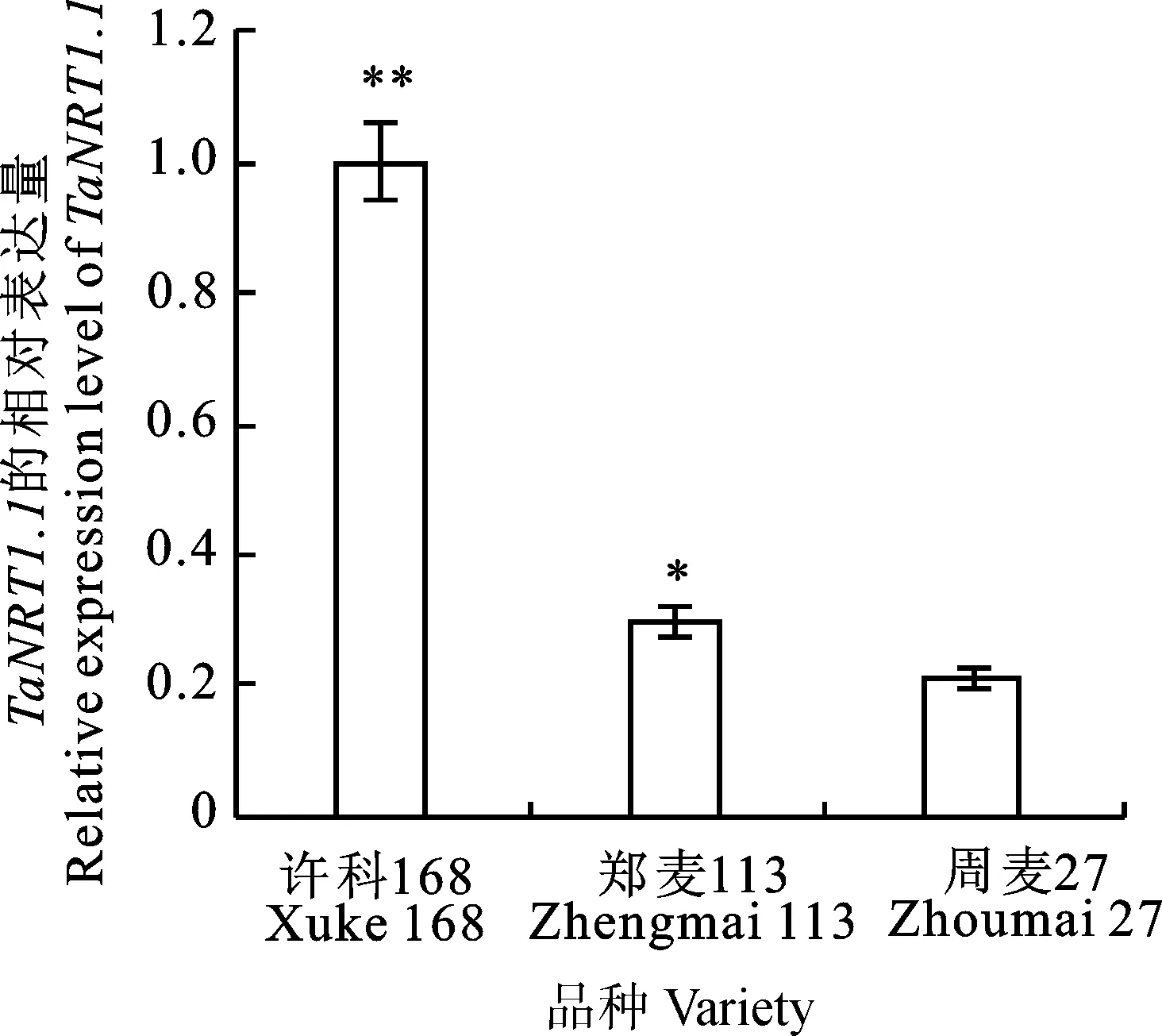
图5 许科168、郑麦113和周麦27苗期根中TaNRT1.1基因等位变异的相对表达量Fig.5 Relative expression levels of TaNRT1.1 alleles in the seedling root of Xuke 168,Zhengmai 113 and Zhoumai 27
3 讨 论
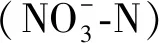
在此基础之上,本研究首先同源克隆了水稻OsNRT1.1B基因在小麦中的三个同源基因TaNRT1.1-1A、TaNRT1.1-1B和TaNRT1.1-1D,并通过生物信息学分析,发现这三个同源基因所编码的蛋白均为疏水蛋白,含有丰富的α-螺旋和无规则卷曲,其亚细胞定位于质膜上。此研究结果与郭志强等[26]的研究结果一致。另外,通过对小麦不同组织中三个同源基因TaNRT1.1-1A、TaNRT1.1-1B和TaNRT1.1-1D进行qRT-PCR分析,结果显示,TaNRT1.1-1A和TaNRT1.1-1B基因在不同组织中表现出相似的表达模式,即根中表达量最高,叶、茎中次之。TaNRT1.1-1D基因的表达模式则不同,茎中表达量最高,叶、根中较低。因此,推测TaNRT1.1-1A和TaNRT1.1-1B基因在植物根从土壤中吸收硝酸盐过程中发挥了重要作用,而TaNRT1.1-1D基因在硝酸盐转运方面发挥了重要作用。
本研究发现,TaNRT1.1-1A基因启动子上游1 120 bp处存在新的8 bp插入位点。该8 bp的插入与TaNRT1.1基因的高表达、氮利用效率密切相关,且其位于光响应元件Box 4和GT1-motif附近,因此,可为研究TaNRT1.1基因启动子区域光反应元件所发挥的作用提供参考。此外,由于本研究中用于筛选TaNRT1.1基因多态性的材料较少,未来仍需进一步探究TaNRT1.1-1A基因启动子上游1 120 bp处8 bp的插入对小麦氮利用效率的影响。
- 麦类作物学报的其它文章
- 国审高产节水小麦新品种—冀麦665
- 优质抗病粮用大麦新品种—龙紫裸1号

