基于KG-GCNASL方法的人类癌症合成致死预测研究
朱晓敏, 刘 爽
(大连民族大学 计算机科学与工程学院,辽宁 大连 116650)
1 问题提出
合成致死指对于细胞中的两个基因,其中任何一个单独突变或不发挥作用时,都不会导致细胞死亡,两者同时突变或者不能表达时,会导致细胞死亡[1]。在合成致死基因对中,一个基因突变不会影响细胞的生存能力,两个基因同时突变则会导致细胞死亡;通过抑制致癌突变基因的合成致死伙伴基因,可杀死致癌基因发生突变的癌细胞,且不损害正常细胞。SL作为一种选择性杀死癌细胞新的靶向策略,为癌症治疗带来了新机遇;也为发现新的药物靶标和潜在的癌症药物联合策略提供了可能性。
SL预测是链接预测在生物医学领域一个很重要的应用。随着人们生活压力越来越大和快餐式的生活方式越来越频繁,癌症已经成为了危害人类健康的主要杀手之一,其主要原因是细胞生长不受控制导致过度增殖而引起的。传统化学疗法通过药物靶向快速分裂细胞从而杀死癌细胞,当患者使用这些药物时,会快速损害正常细胞的分裂,对不能迅速分裂的正常细胞也有毒性,因此限制了抗癌药物的有效性。
知识图谱是由语义网络发展而来的[2],当知识图谱被应用到各个领域后发现存在一些问题需要被解决,如链接预测问题。链接预测是将知识图谱中实体和关系的内容映射到连续向量空间中,对实体或关系进行预测,包涵(h,r,?),(?,r,t),(h,?,t)三种任务[3],癌症合成致死预测是知识图谱链接预测在生物医疗领域非常重要的研究。
1.1 现有癌症合成致死预测方法与面临的挑战
合成致死是抗癌药物研发的全新思路,目前用于癌症合成致死预测的方法主要包括以下三种。第一种是基于代谢网络模型进行基因敲除模拟[4],如图1。通过整合基因组、转录组、蛋白组和热力学数据实现基于各种约束的模型构建,在基因靶点识别、系统代谢工程研究等多方面取得进一步发展和理论突破;但缺点是严重依赖于代谢网络模型、领域知识和基因组数据等,不能充分利用已知合成致死对象有价值的信息。
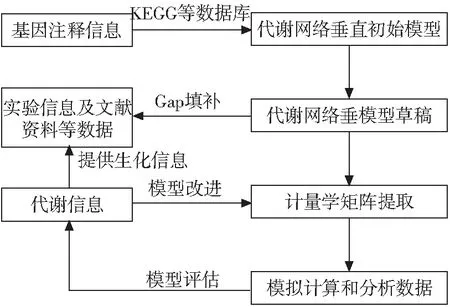
图1 基于代谢网络的预测方法
第二种是基于知识数据挖掘即面向知识的方法[5]进行SL预测,如图2。主要是利用特定领域的知识进行特征工程,通过相关方法从海量数据中抽取出潜在且有价值的知识规则,其缺点是SL预测的湿实验筛选存在着成本高、成批效应和脱靶等问题,不能充分利用有价值的信息。
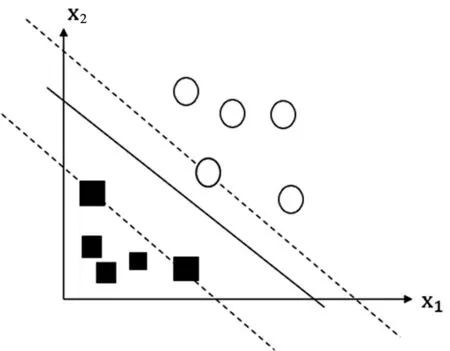
图2 支持向量机模型的预测方法
第三种方法应用机器学习的算法进行癌症合成致死预测,如图3所示:其特征基于领域知识和启发式函数设计的[6],如支持向量机等注入基因组和蛋白质组数据来促进癌症合成致死预测;基于图网络的方法[7]对输入特征等信息进行编码,但缺点在于需手工提取特征,会遗漏特征。
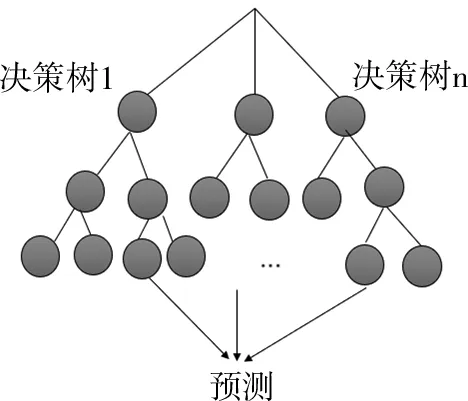
图3 基于决策树的预测方法
综上可知,现有方法大多倾向于假设合成致死对象是相互独立的,并未考虑到潜在的共享生物机制。一些方法虽结合基因组和蛋白质组数据来帮助癌症合成致死预测,但涉及手工特征工程,严重依赖医药学、肿瘤学等相关领域知识。
1.2 癌症合成致死预测的研究意义
综上,其现有方法的局限和意义启发着应该寻找一个新改进方法或模型去更多地进行癌症合成致死预测等相关研究。本文将进行如下工作:基于知识图谱链接预测与图神经网络及注意力机制Attention方法使得实体特征向量融合所有邻域实体特征及相应的关系特征,更好地捕捉给定多跳邻域中的信息和关联特征,从而达到更好的效果来解决相关问题。可知知识图谱与图卷积网络及注意力机制等相结合的方法进行人类癌症致死预测研究对医疗领域与生物信息领域的研究具有重要意义,尤其是癌症治疗方面。
2 相关方法与模型介绍
2.1 癌症合成致死预测相关方法介绍
2.1.1 基于知识图谱图卷积神经网络模型介绍
KG-GCNASL方法将知识图谱与图卷积网络结合引入癌症合成致死关系预测中,基于图卷积神经网络模型通过结合知识和数据更好地解决生物医药领域的复杂问题[8],图卷积网络模型如图4。新预测的合成致死基因可帮助生物学家更快筛选到新抗癌药物靶点[9],实现AI技术加速新药研发进程。通过知识图谱来揭示SL背后的生物学机理,使深度学习模型具有更好的可解释性,加速癌症药物靶点发现,促进AI制药技术发展。
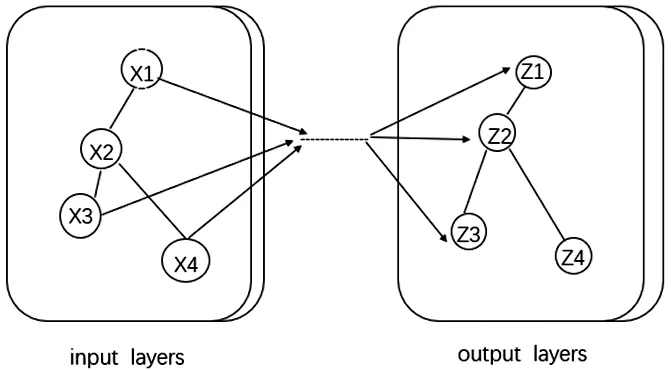
图4 图卷积网络模型
2.1.2 注意力机制模型介绍
通过引入注意力机制模型来跟踪不同基因间发生癌症合成致死的可能性,从而实现可解释性;除此还解决了语义向量无法关注到表示序列的重要信息问题[10]。当获取词向量被逐个送入图卷积网络模型后会产生一系列的编码端隐藏状态参与到注意力系数的计算。每轮训练中,解码端输出状态也将参与注意力系数的计算,然后使用注意力权重将原子集成到分子表示中。解码器状态与隐藏状态经过加权求和后得到最终的概率分布。此方法可以在任何给定实体的邻域中同时捕获实体和关系特征;还在模型中封装关系聚类和多跳关系,从而捕捉给定药物多跳邻域中的信息和关联特征,为基于注意力模型的有效性提供见解,其注意力机制模型如图5。
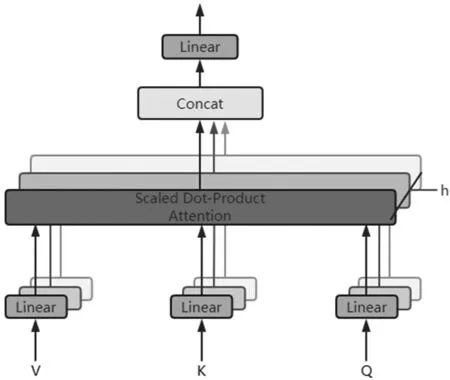
图5 注意力机制模型图
2.2 癌症合成致死预测模型介绍
本文提出KG-GCNASL合成致死预测方法,将预测问题定义为图上链接预测问题,扩展到图结构上神经网络方法进行非线性节点嵌入学习,并重构新邻接矩阵或重构新图上链接,以得到基因间合成致死关系,整体模型框架如图6。
模型将知识图谱合并到图卷积神经网络中,通过直接在图中引入潜在因素作为节点缓解独立性问题;知识图谱中注入各种可能与合成致死相关的生物过程、疾病等因素来解决独立性问题。KG-GCNASL主要由三部分组成:首先从每个基因的原始知识图谱中推导出一个基因特异性子图;其次在基因特异性子图上进行MP,自动将基因与可能识别合成致死对象过程中起决定性作用的因素关联起来,加入注意力机制以捕获给定实体多跳邻域中的实体和关系特征,使得模型能够对不同邻居节点指定不同权值,避免采集的有效邻居节点信息量过大带来的噪声影响从而影响预测的结果;最后,定义了一个以监督方式重构基因-基因相似度的译码器实现癌症合成致死预测。此模型与目前先进的合成致死预测方法进行了比较,在ROC曲线下面积(AUC)、precision-recall曲线下面积(AUPR)和F1值等方面优于目前流行的baseline方法,证明了该模型的有效性。
(1)图谱的生成:SynLeth KG中包含11个实体、24种关系,如(gene, regulates, gene)、(gene, interactions, gene)等。 11种实体中有7种与基因直接相关,即途径、细胞成分、疾病、化合物等。知识图谱生成如图7。首先在SynLethKG数据库中筛选出需要的信息,给定一个癌症合成致死相关基因;然后使用Bio2RDF工具构建链接数据网络,基于传输定义从不同格式数据源中获取数据后创建与RDF数据格式兼容的链接数据;最后,使用RDF将数据集处理成三元组形式用于知识图谱构建,从构建好的KG中构建一个加权子图,再识别出相关的节点和决定边感重。
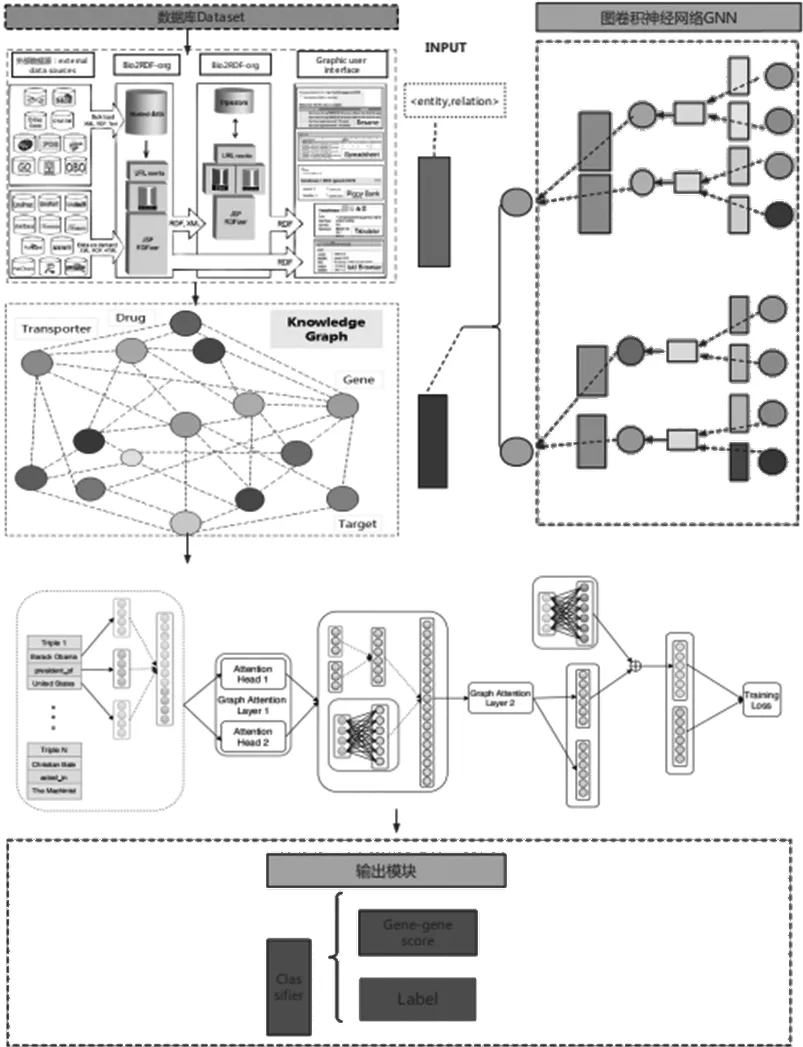
图6 模型框架图
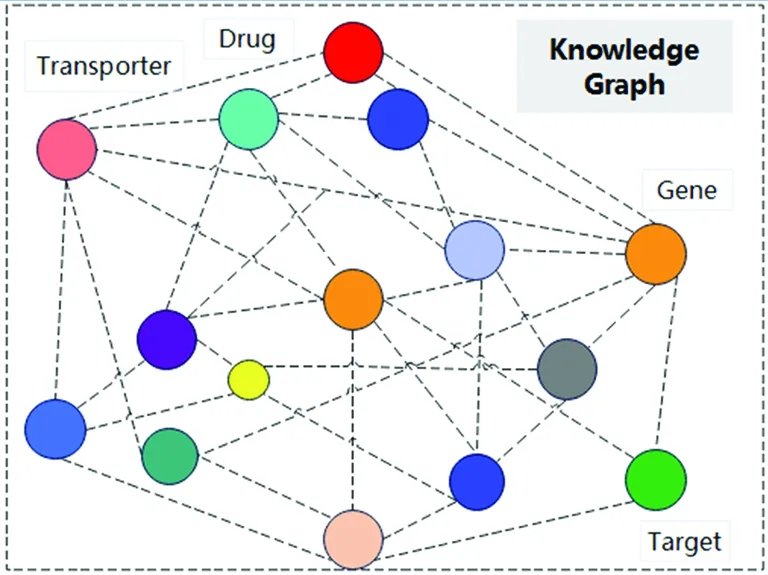
图7 知识图谱生成图
(2)对实体进行邻居采样:为每个实体抽取固定数量的邻居表征局部结构引入参数H(CNN感知域)重复H跳,节点可被重复采样。然后将信息聚合起来,作为下个网络的输入[11]。由于每一个基因实体的邻域分布情况是不一样的,先对实体进行邻域采样:H=1时只考虑与当前节点直接相连的邻居节点,H=2时考虑二阶相连的节点情况,能够学习到更多邻域实体信息。每个实体抽取固定数量k个邻居来表征其局部结构,并重复该过程H跳(H >=1)。边上权重代表关系重要性,则边权重在子图计算方式:
(1)
式中an表示基因,ra,a′表示关联的embedding。
(3)聚合邻域信息:在构建的知识图谱中,和基因直接相连的节点定义为Nneigh(a)。由于每个药物节点邻域的分布不同,在采样完成后,通过聚合方法将实体自身嵌入表示和邻域信息嵌入表示聚合起来,最终得到当前实体的嵌入表示。
①对子图中每个节点进行信息聚合与更新,在对每个节点计算加权平均和,公式如下所示:
(2)
式中a′表示子图中一个实体,Za表示子图中实体集合,wama,a′表示基因关系间的重要性权重。
Q是使用softmax函数进行normalize后的基因关联分数,公式如下所示:
(3)
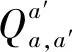
②得到中心节点的邻居的表达后,再对其进行信息的聚合与更新,公式如下所示:
A[h+1]=Ø(Q(a[g]+Az(a))+g)。
(4)
式中:Q表示线性transform层权重;g表示线性transform层偏置;Ø表示激活函数,A表示实体表示;h+1表示更新后的实体表示;a[g]表示线性变化后的权重;Az(a) 表示计算后的加权平均和。
③在得到两个基因的表达之后,它们之间的反应概率通过下列公式计算:
sm,n=Ø(f(am,an)。
(5)
其中,f()表示基因表达公式,am,表示基因。
(4)注意力机制Attention层:将上层输出作为注意力机制模型的输入,从而有效捕获局部邻居及全局邻居的注意力权重,用来学习节点的局部和全局表示。利用多层感知器将原始特征、局部和全局表示进行聚合,从而得到特定的特征表示并对其进行整合。对于一个节点,在图中与其直接相连的节点定义为局部邻居。使用下面公式计算注意力打分:
④对于一个节点,在图中与其直接相连的节点定义为它的局部邻居。考虑到不同的邻居重要性不同,设计注意力机制来学习节点表示:
(6)
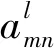
⑤将注意力打分进行归一化,公式如下:
(7)
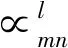
⑥同时基于局部邻居信息聚合节点vi的表示,公式如下所示:
(8)
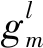
⑦由于注意力系数的不稳定性,单个节点的注意力机制可能会引入噪声。公式如下所示:
(9)
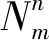
(5)总loss和优化:模型设计了两种loss,基本loss和L2 loss,进行cross-entropy计算:
J=min(sm,n,0)-sm,n*sm,n+log(b+exp(-|sm,n|)。
(10)
式中:sm,n是预测值;sm,n是真实值;b是常数。
⑧‖Γ‖代表对实体embedding,关联embedding及聚合权重的L2正则:
(11)
(3)还加入了L2正则loss,公式如下所示:
minW,K,bι=minW,K,b∑m,nj+α‖Γ‖。
(12)
式中:K表示可训练权重矩阵;bι表示基因关系评分权值;∑m,nJ表示关联embedding及聚合权重后的正则;α表示平衡超参数。
3 实验部分
SynLethDB是一个合成致死基因对的综合数据库,包含11个实体及24种关系。为使正、负样本平衡,随机选取未知对作为负对,使正、负SL pair数量相等,包含10 004个基因之间的72 804对基因,去除孤立节点后,最终包含了54 012个节点和2 231 921条边,SL数据集见表1。

表1 数据集介绍
实验部分分别用AUC、AURP、F1值三个指标进行分析,与多种baseline方法比较,包括ML、GRSMF、HOPE、DeepWalk、Node2vec、LINE、GCN、GAT等,实验结果对比见表2。
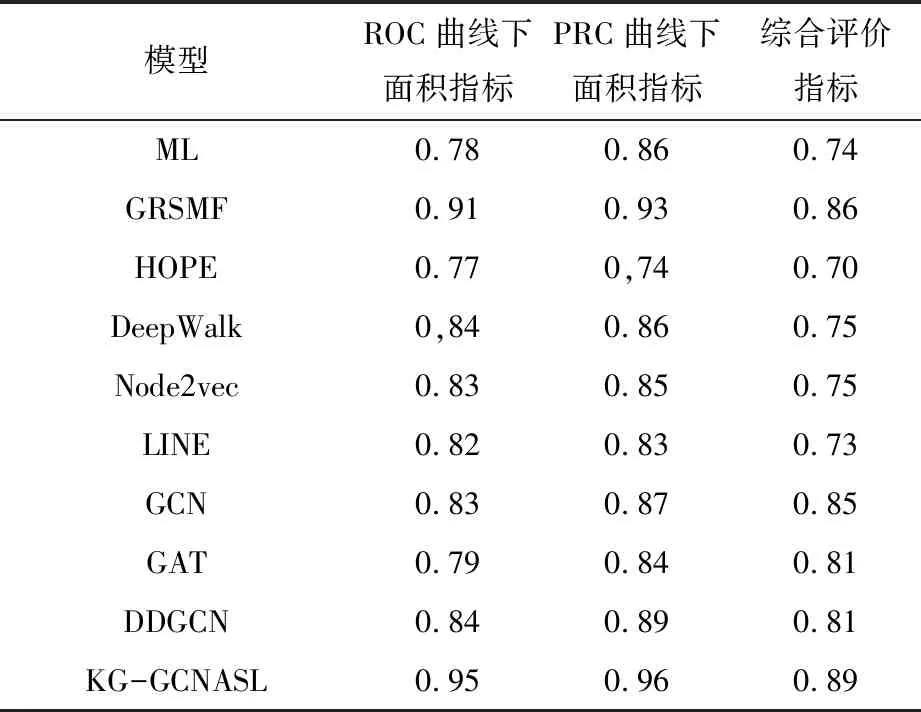
表2 各模型实验结果
KG-GCNASL优于表中所有baseline方法,与第二优模型GRSMF相比,KG-GCNASL在AUC、AUPR和F1上的性能分别提高了4%、3%和3%,证明了模型的有效性。因为KG-GCNASL模型可从合成致死对象的相似性中学习,丰富SL预测的基因嵌入,表明从包含GO信息的KG中学习基因表征和其它基因特征可进一步提高SL预测。
另外对本实验中一些关键超参数进行了敏感性分析:括邻居采样大小k和实体嵌入维数d。首先,通过改变邻居k的样本数观察模型性能,不同邻居采样大小对模型的的敏感度分析如图8。可知,该模型在相邻采样尺寸k=64时AUC、F1和AUPR效果最好。当k值越高时邻居采样越多,采样信息变得冗余,k为128时模型性能略有下降。
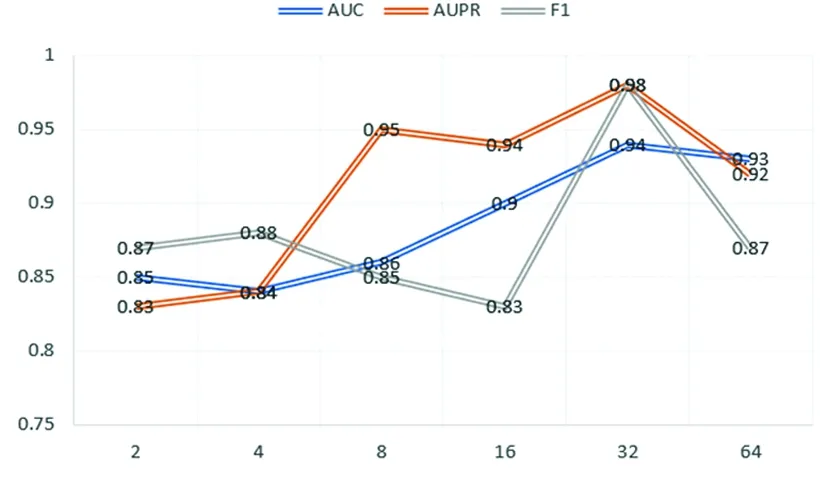
图8 邻居采样大小k的敏感分析
其次,实验还分析了实体嵌入维度d对模型敏感度的影响,如图9。当模型嵌入维数d为256时已经有了很好的性能。太大的嵌入维度会给内存和计算带来负担。最终,实验中设置模型的邻居采样大小为64,嵌入维数为256。
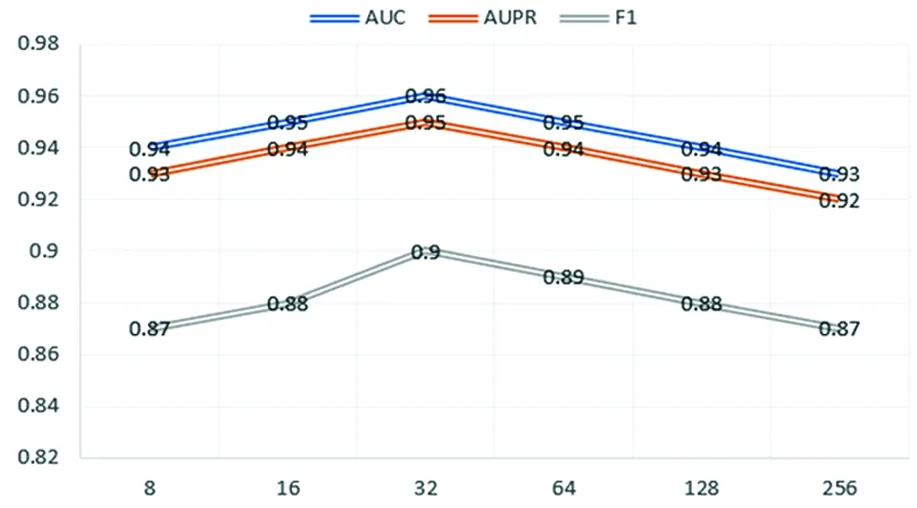
图9 实体嵌入维度d的敏感分析
通过对SL领域知识的了解与分析,对该领域图谱实体类别进行设计:11种实体有7种与基因直接相关,即途径、分子功能、疾病等。每类实体类别中包含多个实体,每个实体中包含相应属性信息用于刻画该实体的内在特征,定义关系来刻画实体和实体或属性间的联系,其癌症合成致死预测研究生成的图谱如图10。
图10 生成的知识图谱
4 结 语
合成致死是一种很有前途的基因相互作用类型,在靶向抗癌治疗中起着关键作用。本文提出KG-GCNASL方法实现癌症合成致死预测,将知识图消息传递纳入到图卷积神经网络与注意力机制模型预测中:利用包括基因、疾病等在内11种实体和24种SL关系进行构建,通过对KG进行信息传递解决独立性问题,模型虽取得了良好的预测性能但仍有一些局限,希望研究自动特征提取预训练策略,基于更新的版本的SynLethDB验证预测的SL对。此模型在AUC、AUPR和F1指标上优于所有最先进baseline方法,并且证明了将知识图谱纳入GCN中对SL预测的显著影响。

