茶树品种‘金牡丹’自然杂交后代遗传鉴定
杨军, 孔祥瑞, 郑国华, 邱陈华, 王让剑
茶树品种‘金牡丹’自然杂交后代遗传鉴定
杨军, 孔祥瑞, 郑国华, 邱陈华, 王让剑*
(福建省农业科学院茶叶研究所/国家茶树改良中心福建分中心/农业部福建茶树及乌龙茶加工科学观测实验站,福州 350000)
为了对‘金牡丹’茶树自然杂交后代进行遗传鉴定,利用EST-SSR毛细管电泳荧光标记技术对65个金牡丹自然杂交后代进行研究。结果表明,28对SSR标记共扩增出192个等位片段,平均等位基因数(Na)、平均观测杂合度(Ho)及遗传多态信息量(PIC)分别为6.86、0.540、0.532。单亲基因型已知时的累积排除概率为0.999,说明选择的28对SSR标记位点具有高度的多态性和较高的排除概率,适用于遗传分析和个体的亲子鉴定。15个‘金牡丹’自然杂交后代的遗传鉴定结果表明,MD44、JMD45、JMD47、JMD32为早生绿茶类型;JMD51、JMD53为闽北肉桂乌龙茶类型;MD2、JMD56为闽南‘铁观音’乌龙茶类型;JMD24、JMD26、JMD29、JMD55、JMD59、JMD27、JMD61为‘黄棪’乌龙茶类型。
茶树;自然杂交;EST-SSR;遗传鉴定; 模拟分析
茶树是山茶科(Theaceae)山茶属()茶组(sect.)的多年生常绿木本植物[1],且为常异花授粉植物,具有高度杂合性,杂交结实率低[2],自然杂交收获种子比人工杂交收获种子相对容易,但缺乏对其父本信息了解,对自然杂交后代父本的模拟鉴定有助于提高茶树育种者对茶树自然杂交后代的选择。EST-SSR具有共显性、多态性高、重复性好等特点[3–8],被应用在不同生物上进行模拟亲本鉴定。用于亲本模拟分析的软件主要有CERVUS、COLONY和PAPA,李博等[9]对比了这3个软件的准确率,认为CERVUS亲本模拟分析的准确率可达81.2%,鉴定效果较好。不同作物都有大量采用CERVUS软件进行亲本模拟分析的报道,艾畅等[10]通过马尾松()自由授粉子代模拟父本分析,建立遗传多样性与同父本组成之间的联系,为种子园集约生产种子提供理论依据;张冬梅等[11]利用SSR对油松()种子进行父本分析, 结合油松在园内的分布,明确种子园内花粉传播的最大距离和花粉活力;冯源恒等[12]对广西马尾松第2代育种群体进行父本分析,表明个体间的近交程度较低,为开展马尾松高世代杂交育种奠定了基础;王建忠等[13]从桉树()候选父本中鉴定出D2、D4、D11、D19、D20等7个子代的父本,探究了桉树自交不亲和与近交衰退机制,并准确进行子代父本鉴定和杂交亲本重建; 邵文豪等[14]认为高多态性和高累积排除概率的SSR位点适用于对油橄榄()品种子代进行父本分析,‘城固32’具有一定的自交亲和性,这为品种建园时授粉品种的选择和配置提供重要依据;何勇凤等[15]准确找出了圆口铜鱼子1代()的父母本,为圆口铜鱼的家系管理、种群遗传管理和增殖放流效果评估提供了科学依据;余书平等[16]对开化县茶树种质资源进行父本分析,了解茶树种质资源的遗传背景。同时,利用CERVUS软件对子代模拟亲本鉴定在黄鳍棘鲷()[17]、鳙()[18]、羊()[19]、黄喉拟水龟()[20]、许氏平鲉()[21]和唇䱻()[22]等生物上有较多的报道。总的来说,亲本模拟分析主要用于分析亲本的遗传方式(花粉转播距离、自交不亲性)、后代群体遗传差异评估(遗传多样性、种群遗传管理、遗传背景、近交程度)、辅助杂交后代选择等方面。
茶树品种‘金牡丹’为“九五”国家科技攻关的一级优异茶树种质资源[23],适制性广,制优率高,制乌龙茶香气馥郁芬芳,滋味醇厚甘爽,“韵味”显, 制红、绿茶花香显、味醇厚。本试验以42个福建茶树良种为候选父本对‘金牡丹’自然杂交后代进行分子鉴定,并调查其节间长度、叶片长度、叶片宽度等叶片性状,旨在分析‘金牡丹’自然杂交后代亲缘关系与遗传差异,为茶树品种选育提供参考。
1 材料和方法
1.1 材料
2015年11月收取福建省茶树种质资源圃内‘金牡丹’成熟茶果,2016年1月播种。2016年11月选取生长正常的实生苗,与福建省茶树种质资源圃邻近种植,位于福建省农业科学院茶叶研究所二号山(27°12′58″ N,119°34′31″ E)。种植规格为行距1.5 m,株距0.3 m,按顺序编号JMD1~JMD65。2019年分别采集65个‘金牡丹’自然杂交后代和42个福建省茶树品种(福建省茶树种质资源圃内)的嫩叶,用液氮冷冻处理,于-70 ℃冰箱中保存备用(表1)。
1.2 方法
基因组DNA的提取 采用改进的CTAB法[24]提取茶树基因组DNA。采用1.0%的琼脂糖凝胶电泳检测,用756-MC型紫外分光光度计测定OD260和OD280,检测茶树基因组DNA的纯度。
引物合成 参照Ma等[25]的引物序列,序列由上海Sangon公司合成,引物信息见表2。
PCR扩增和产物鉴定 PCR反应体系为: 双蒸水18.8L,2.5L 10×Buffer (Mg2+),0.5L dNTP (10 mmol/L),正、反向引物(10mol/L)各0.5L,聚合酶0.2L (0.5 U),模板DNA 1L。PCR反应于美国ABI-9600型扩增仪上进行,反应程序为: 94 ℃预变性4 min;然后94 ℃变性45 s,不同温度下退火60 s,72 ℃延伸75 s,共35次循环;最后72 ℃延伸10 min。反应产物于4 ℃保存。统一在反向引物(R)的5′段标记荧光(FAM/TAMRA), 由上海百力格生物科技有限公司合成。扩增产物0.5L,GeneScan™ 500 ROX™ 0.5L,HiDi 9L,混匀后使用ABI公司3730XL进行毛细管电泳。
叶片性状测量 测量17个‘金牡丹’自然杂交后代(父本模拟达到显著性)与40个福建良种的主要叶片性状,主要有第1~3叶芽稍的长度、第1~3节间长度、叶齿数、叶脉对数、第1~4叶片长度与宽度,每个性状调查重复5次。
1.3 数据处理
采用ABI公司的GeneMapper 4.0软件,选择GeneScan™ 500 ROX™作为分子量标准,对每条扩增条带记录大小。使用Cervus 3.0[26]软件计算多态信息含量、期望杂合度、等位基因频率与非父排除概率等参数。利用SAS9.3进行叶片性状聚类。运用NTSYSpc 2.1对参试材料进行遗传距离计算,利用MEGA7进行UPGMA聚类分析,并利用网站https://itol.embl.de/进行图片优化。
1.4 亲子鉴定模拟分析
使用Cervus 3.0软件进行亲子鉴定模拟分析,分别运行Simulation程序与Parentage Analysis程序中的paternity子程序,模拟10 000次亲子鉴定,candidate fathers的设置为42,候选亲本检测率为80%, 位点检测率为90%, 基因型判别错误率为0.01, 计算候选亲本基因型似然对数比(LOD)值和95%置信水平,根据LOD值判定亲子关系。对福建省农业科学院茶叶所审(鉴)定品种、65个‘金牡丹’自然杂交后代分别进行双亲模拟分析与单亲模拟分析。
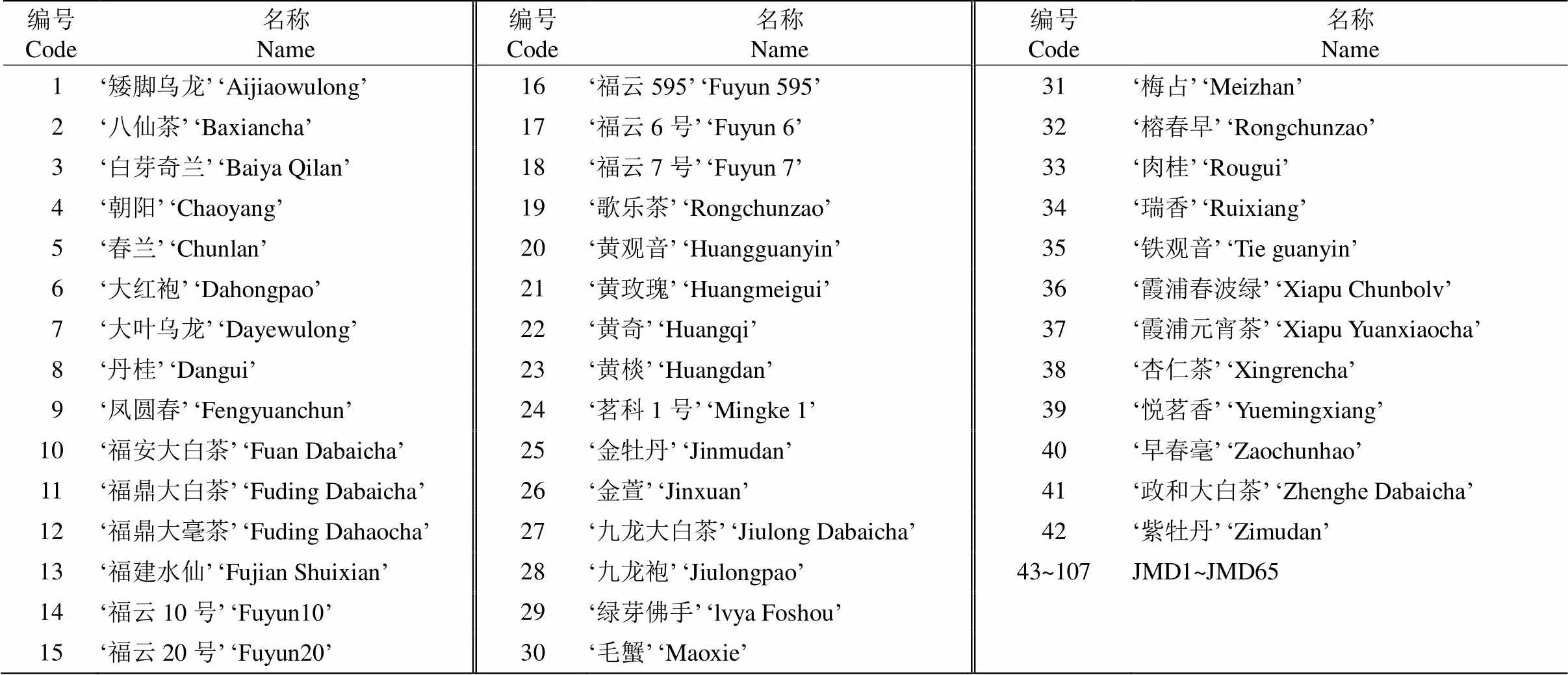
表1 供试材料
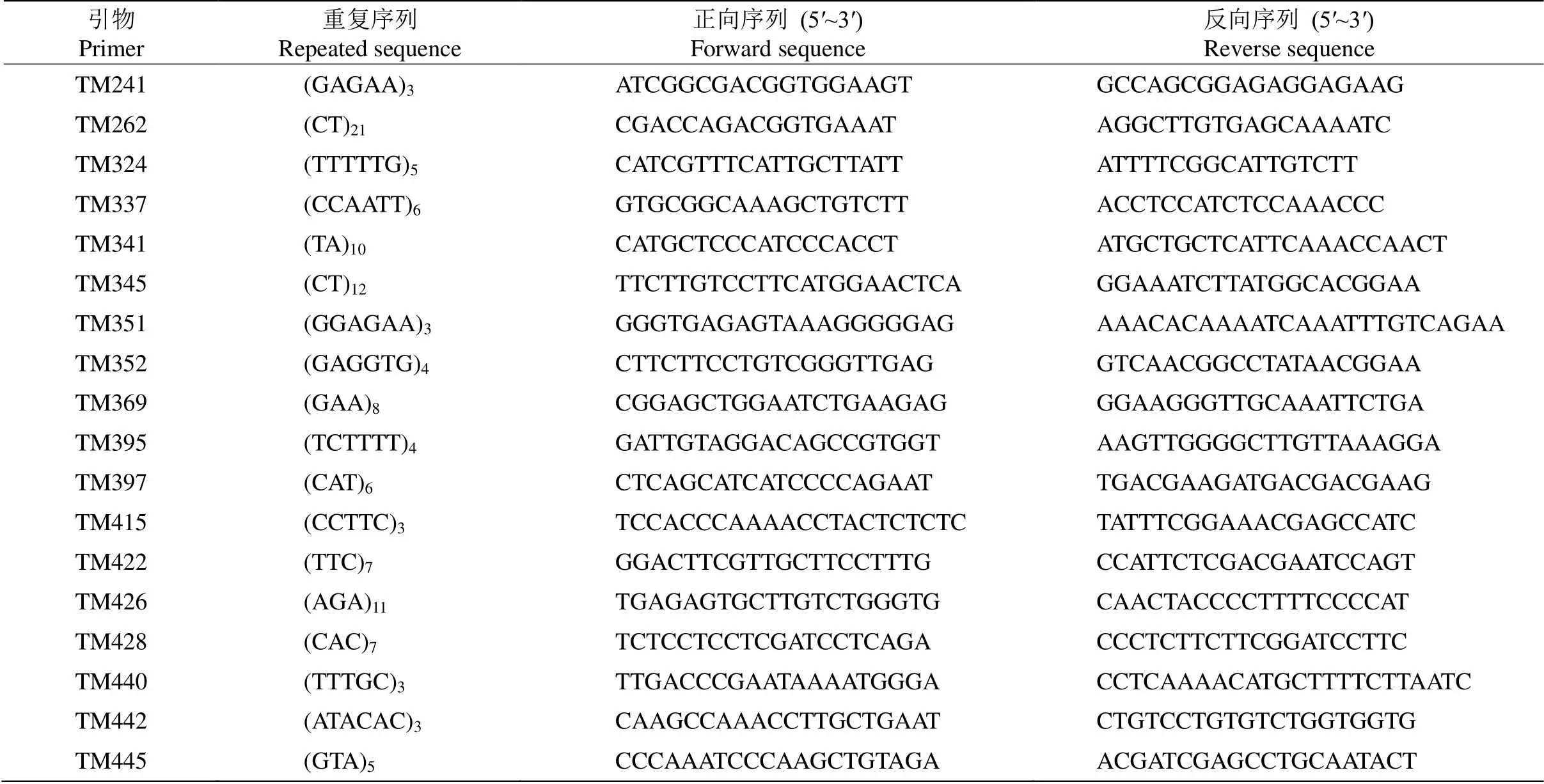
表2 SSR引物信息

续表(Continued)
2 结果和分析
2.1 遗传多样性分析
对65个‘金牡丹’自然杂交后代与42个模拟亲本进行扩增,28个SSR位点的平均等位基因数6.86个(表3),其中,TM422位点上的等位基因数目最多(14个),TM499和TM589最少(3个),均满足利用SSR标记进行父本分析所需等位基因数要求。28个SSR位点的平均观测杂合度和期望杂合度分别为0.540和0.583,变异范围分别为0.247~0.794和0.240~0.805。Ho与HE均值相近,表明茶树品种受选择及近交等因素的影响较小,平均杂合度较高, 等位基因在群体中分布较均匀。
2.2 多态信息含量及排除概率
28个SSR位点的平均多态信息含量(PIC)为0.532, 其中,TM499位点最低(0.221),TM262位点最高(0.778)。28个SSR位点具有丰富的多态性, 能够为研究提供足够的多态信息。
当双亲基因型皆未知时,单个SSR位点的非父排除概率(NE-1P)为0.551~0.972,均值0.793;当1个亲本基因型已知时,单个SSR位点的非父排除概率(NE-2P)为0.375~0.882,均值0.645;当双亲基因型已知时,单个SSR位点的非父排除概率(NE-PP)为0.187~0.793,均值0.485 (表4)。
依据PIC由高至低次累积计算28个SSR位点的排除概率,随着SSR位点数增加,累积排除概率增大(图1)。对于单亲已知类NE-2P,基于前6个SSR位点累积排除概率0.975,基于前13个SSR位点累积排除概率0.999,而基于全部28个SSR位点累积排除概率接近1。因此,28对引物适合用于亲本鉴定分析。
2.3 亲子模拟鉴定分析
从表4可见,18个福建省农业科学院茶叶所审(鉴)定品种的亲子鉴定可分为2个类型,类型I真实亲本不在实验模拟亲本中,‘悦茗香’(母本为‘赤叶观音’)、‘早春毫’(母本为‘迎春’)、‘朝阳’(母本为‘崇庆枇杷茶’)未能找到显著性模拟亲本;类型II真实亲本在实验模拟亲本中,‘福云6号’、‘福云7号’、‘福云10号’、‘福云20号’和‘福云595’的真实亲本之一为‘福鼎大白茶’,另一真实亲本(‘云南大叶种’)未在42个模拟亲本中;‘黄奇’、‘春兰’、‘瑞香’、‘丹桂’、‘紫牡丹’的真实亲本之一分别为‘黄棪’、‘铁观音’、‘黄棪’、‘肉桂’、‘铁观音’,单一亲本鉴定模拟结果与真实亲本相一致;‘茗科1号’、‘金牡丹’真实亲本为母本(‘铁观音’)、父本(‘黄棪’),双亲亲本模拟鉴定结果与真实亲本相一致;‘黄玫瑰’、‘黄观音’的真实亲本与模拟鉴定结果不完全一致,能准确模拟到其中之一亲本为‘黄棪’;‘九龙袍’模拟亲本之一为‘肉桂’,且达到显著性水平,‘九龙袍’真实父本未知,推测‘肉桂’为‘九龙袍’真实父本。类型II中的12个品种能找到真实亲本,准确率80%,因此,模拟亲本结果较为准确。
65个‘金牡丹’自然杂交后代中的25个子代的LOD值均大于0,且置信度均达到95%,说明25个‘金牡丹’自然杂交后代均能准确地找到显著性模拟父本。后代中模拟父本为‘茗科1号’的有14个, 占56%,其次模拟父本为乌龙茶品种的有‘丹桂’、‘黄棪’和‘黄奇’,模拟父本为绿茶品种的有‘福鼎大白茶’、‘福云6号’和‘福云7号’ (表5)。
2.4 ‘金牡丹’自然杂交后代亲缘关系分析
为进一步解读‘金牡丹’自然杂交后代的遗传信息,将模拟父本结果标注在107个参试材料遗传距离聚类图上(图2)。‘金牡丹’自然杂交后代的显著性模拟父本(天蓝色)为‘黄棪’、‘茗科1号’、‘丹桂’、‘福鼎大白茶’与‘福云6号’等,为福建省主要种植乌龙茶和绿茶品种,模拟父本来源广泛,遗传多样性丰富。‘金牡丹’为‘铁观音’ (母本)与‘黄棪’ (父本)的人工杂交后代,大多数‘金牡丹’自然杂交后代受双亲的影响,分别与祖母、祖父、母本、模拟父本聚类在一起,形成5个类群。5个类群分别为‘铁观音’ (祖母)与‘金牡丹’自然杂交后代类群(草绿色)、‘黄棪’ (祖父)与‘金牡丹’自然杂交后代类群(深蓝色)、‘金牡丹’(母本)与‘金牡丹’自然杂交后代类群(红色)、‘茗科1号’ (模拟父本)与‘金牡丹’自然杂交后代类群(金黄色)、‘丹桂’ (模拟父本)与‘金牡丹’自然杂交后代类群(浅紫色);Jmd51、Jmd53模拟父本为‘丹桂’,与‘丹桂’ (模拟父本)形成一个类群。少部分‘金牡丹’自然杂交后代未与亲本聚类在一起, Jmd44、Jmd45模拟父本为‘福云6号’,遗传聚类分别未与‘金牡丹’ (母本)、‘福云6号’ (父本)聚类在一起,推测Jmd44、Jmd45遗传信息受双亲共同作用影响。另外, JMD9、JMD3未能模拟到显著性父本,从遗传距离看分别与‘白芽奇兰’、‘杏仁茶’遗传距离较近,推测JMD9、JMD3的真实父本与‘白芽奇兰’、‘杏仁茶’亲缘关系较近,且遗传信息偏向未知父本。
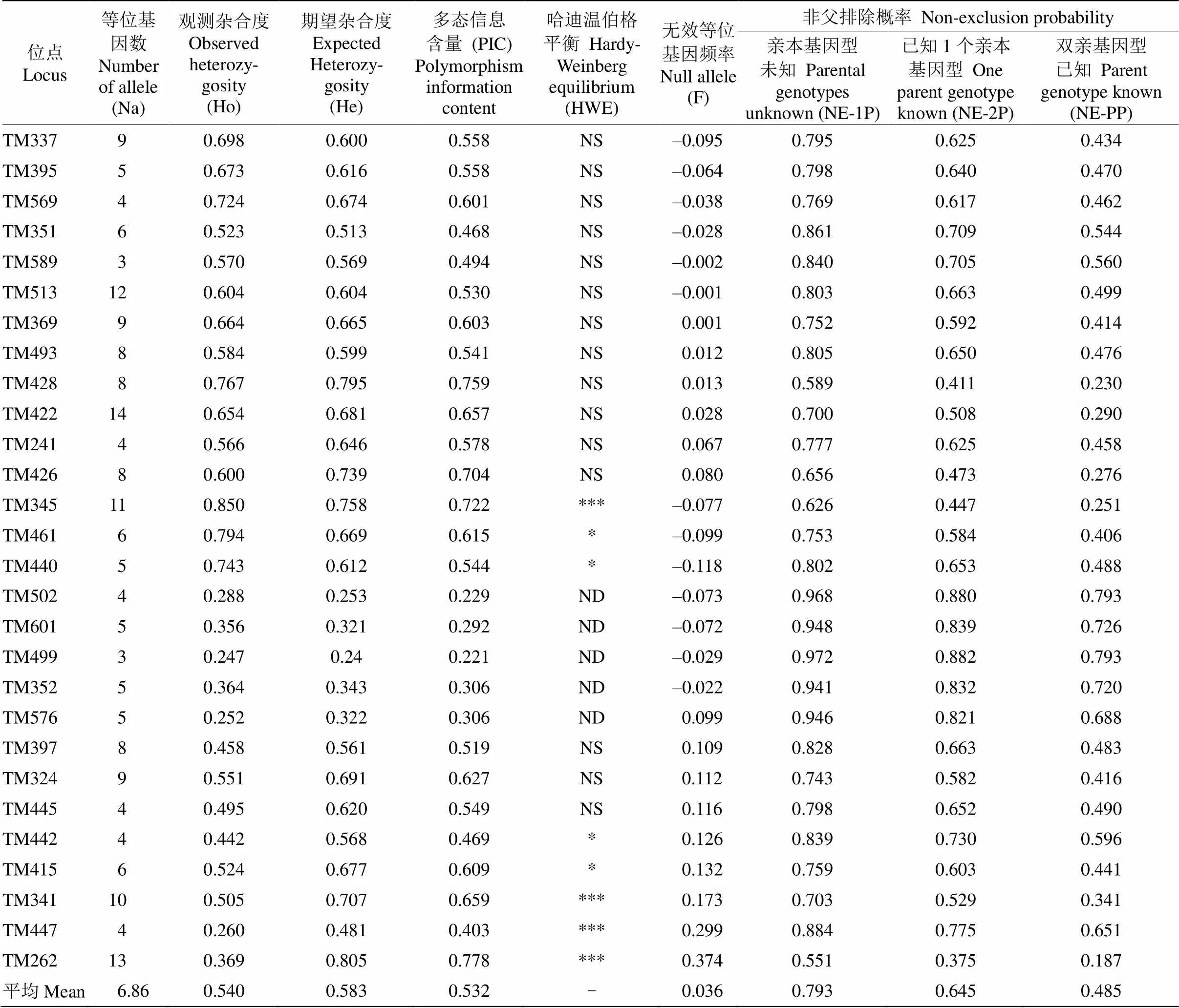
表3 28对SSR引物遗传多样性与鉴定排除率
NS:>0.05; *:<0.05; ***:<0.001; ND: 未检验。
NS:>0.05; *:<0.05; ***:<0.001; ND: Not detected.
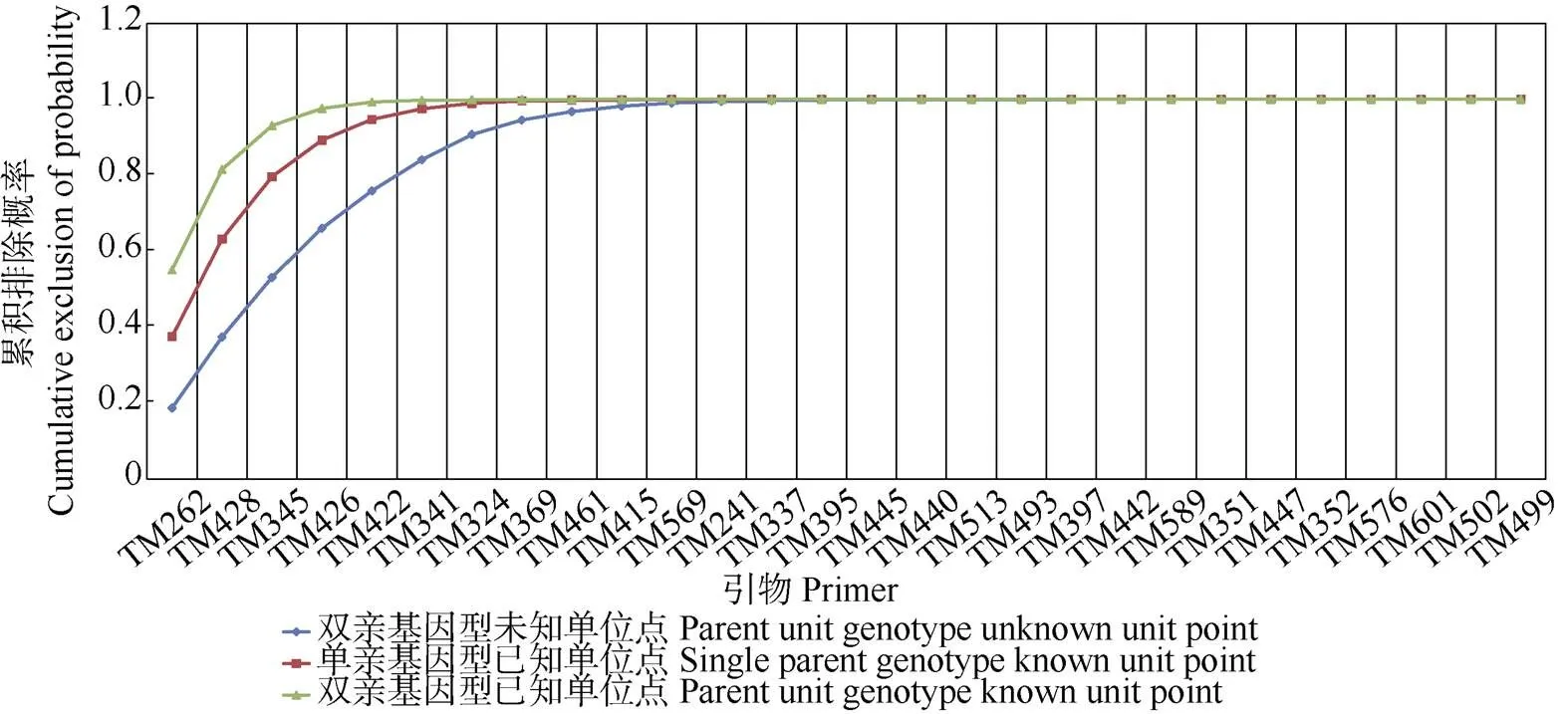
图1 28个SSR位点的累积排除概率
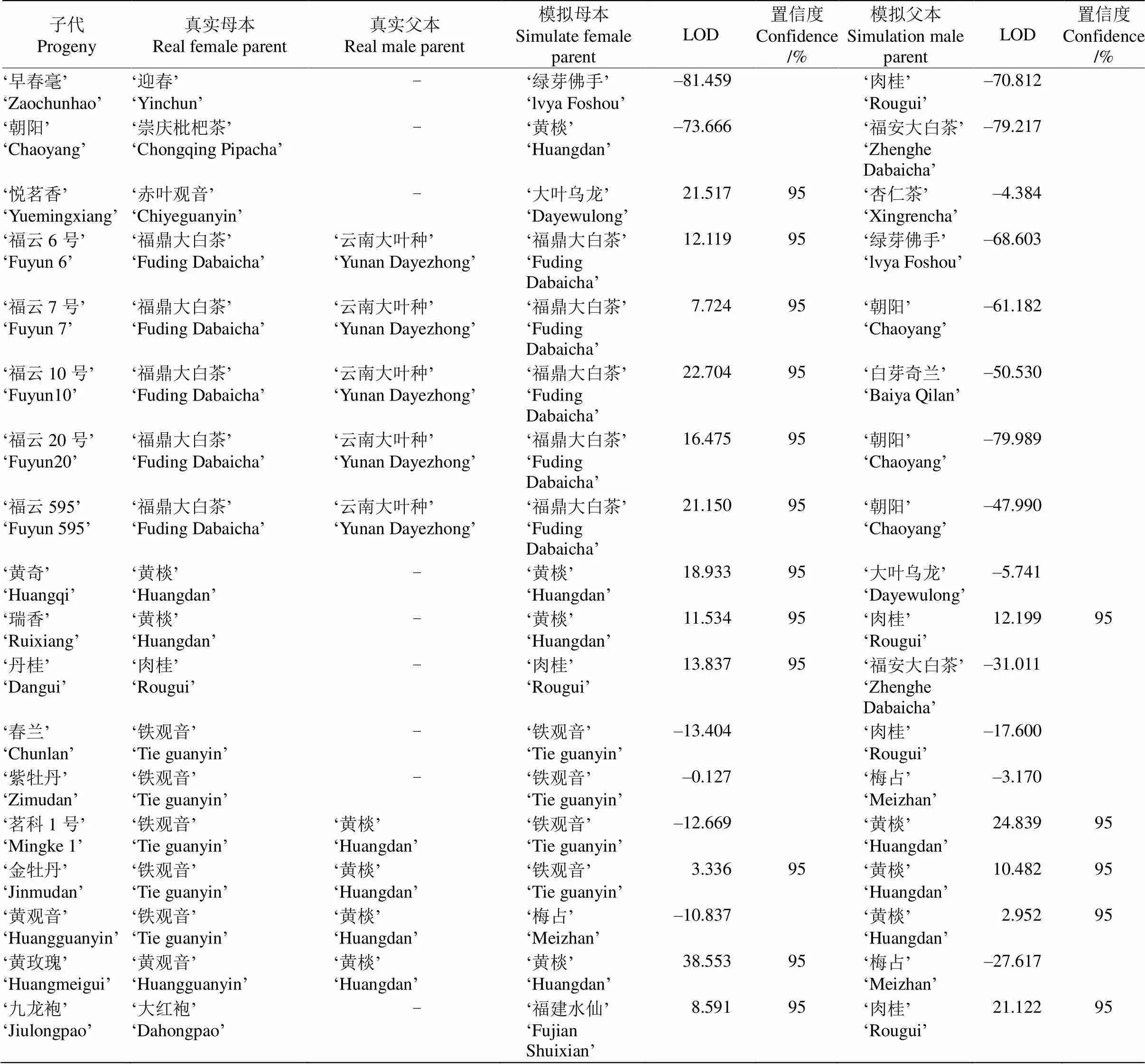
表4 18个茶树品种的亲子鉴定结果
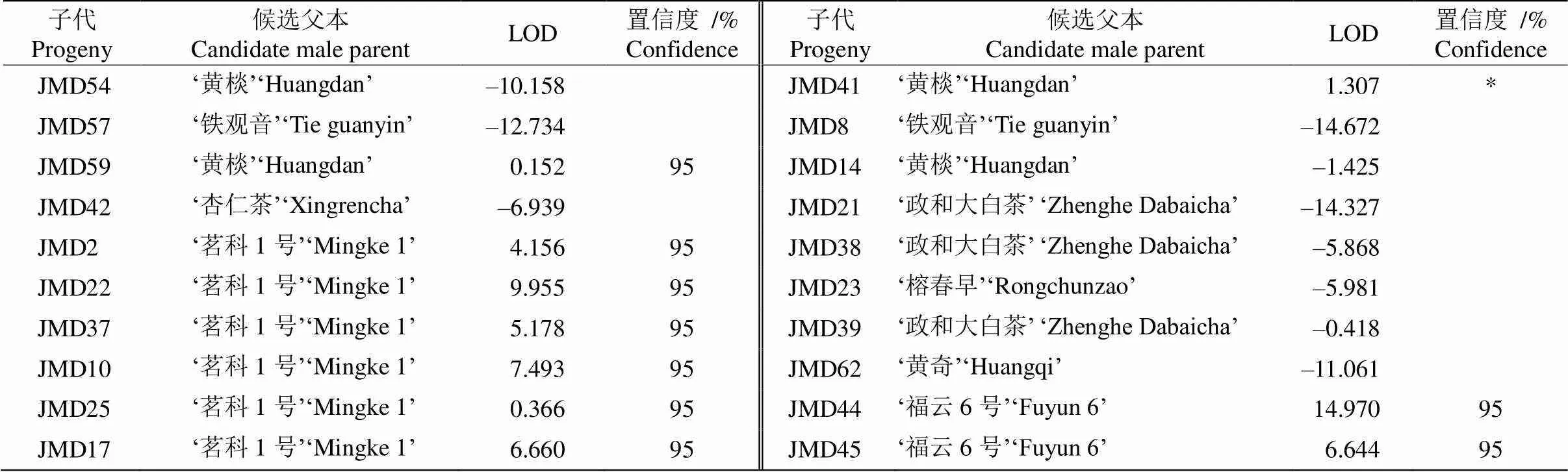
表5 65个茶树品种的父本
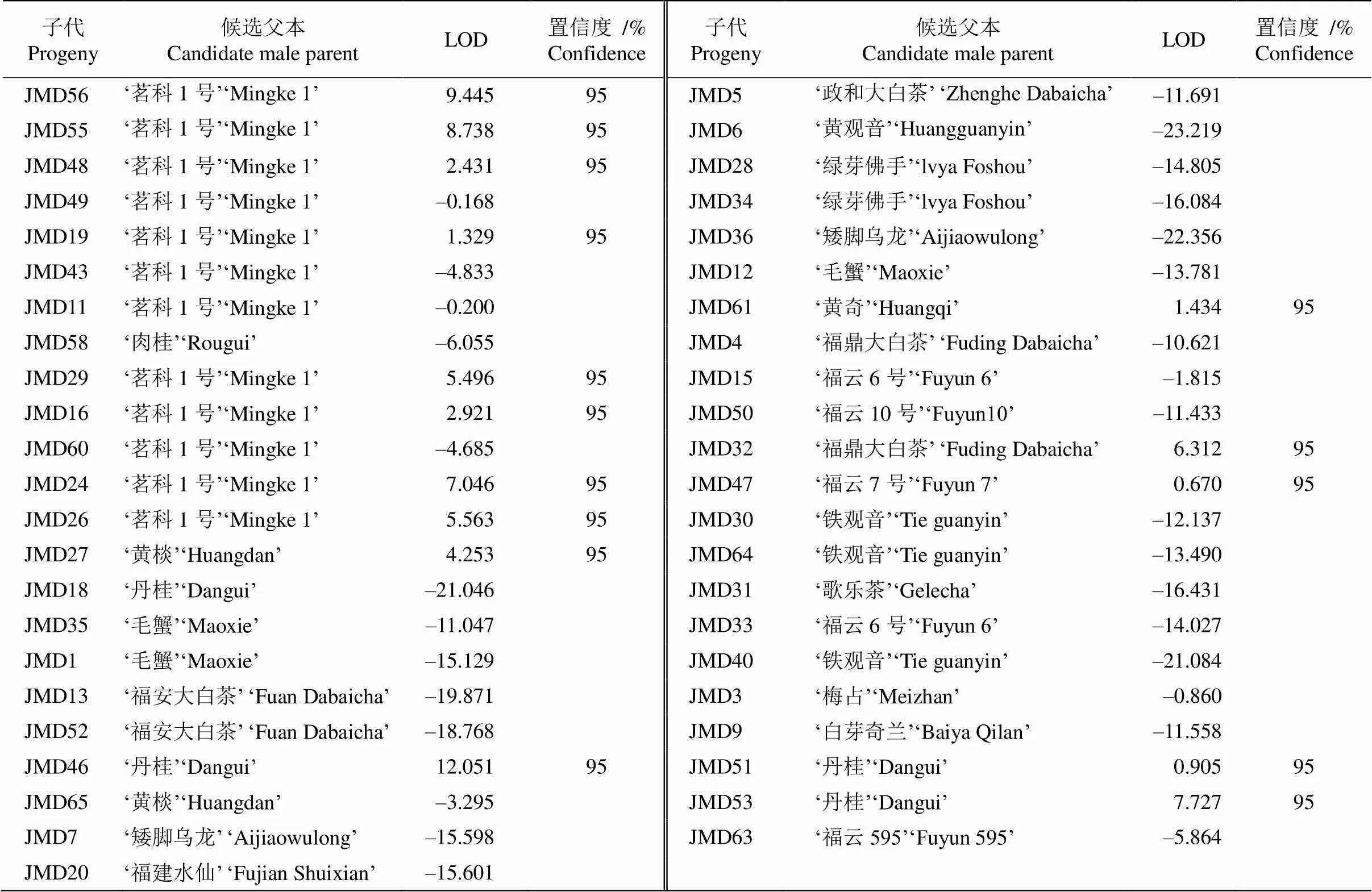
续表(Continued)
2.5 ‘金牡丹’自然杂交后代遗传多样性分析
将107份试验材料按来源与是否找到显著性父本进行分类,分为4个类群进行遗传多样性分析。从表6看出,参试材料的Shannon信息指数为0.828~ 1.245,类群的遗传多样性为:类群D>所有材料>类群C>类群B>类群A,说明65个‘金牡丹’自然杂交后代降低参试材料整体的遗传多样性水平,且遗传多样性略低于福建省茶树品种。Ho和He分别为0.497~0.587和0.481~0.625。其中,类群C的Ho与He相当,属于基因型的分布就越接近于平衡状态,说明65个‘金牡丹’自然杂交后代群体基因型相对稳定;类群A的Ho大于He,说明存在杂合子过剩,推测类群A基因型杂合性较高;类群D的Ho小于He,说明存在杂合子缺失现象,福建省茶树品种间遗传交流较少。
2.6 ‘金牡丹’自然杂交后代叶片性状辅助鉴定
基于叶片长宽、叶齿数、叶脉对数等性状,利用SAS软件对父本模拟显著性的‘金牡丹’自然杂交后代与福建主要茶树品种进行叶片性状聚类(图3)。模拟父本为‘福云6号’、‘福云7号’的‘金牡丹’自然杂交后代JMD44、JMD45、JMD47都聚类在类群I中,且类群I中主要为‘福云6号’、‘福云7号’、‘福云10号’、‘福云20号’等绿茶品种,推测JMD44、JMD45、JMD47叶片主要性状偏向于模拟父本,可优先进行绿茶、白茶加工品质鉴定。
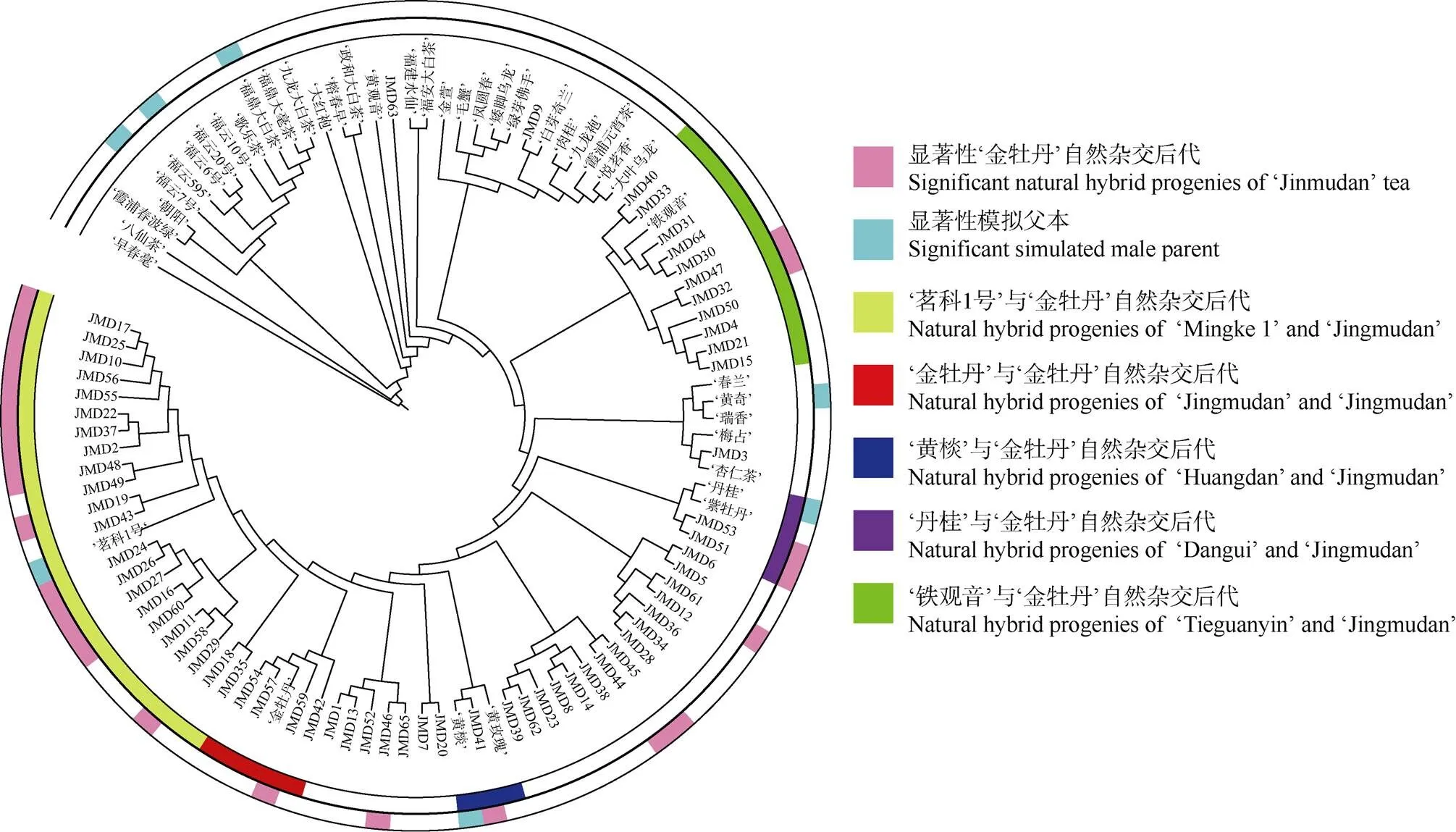
图2 遗传距离聚类图
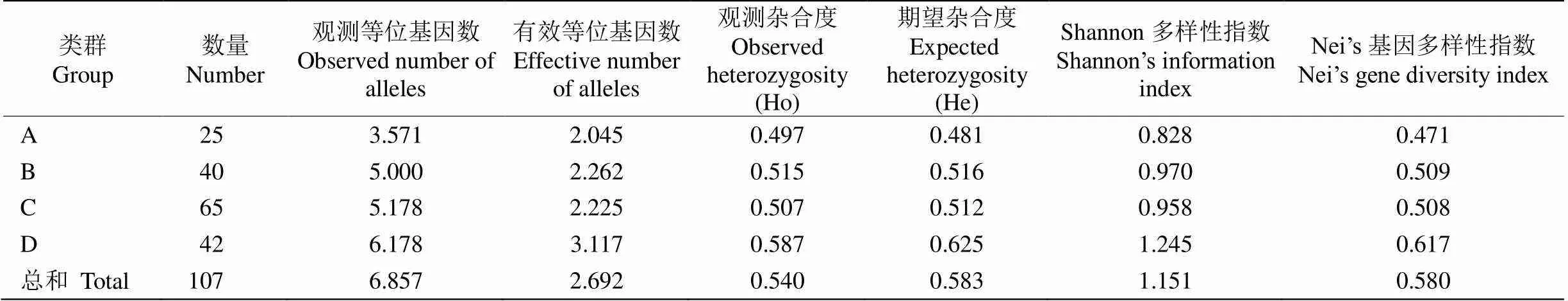
表6 4个群体的遗传多样性
类群III内JMD51、JMD53模拟父本为‘丹桂’,叶片性状与‘丹桂’的母本‘肉桂’聚类在一起,叶片性状更偏向‘肉桂’(祖母)。JMD61模拟父本为‘黄奇’, 叶片性状更偏向‘黄棪’(祖母)。JMD59模拟父本为‘黄棪’,叶片性状与‘黄棪’聚类在一起,叶片性状偏向于模拟父本‘黄棪’。MD24、JMD26、JMD29、JMD55与JMD27、JMD41的模拟父本分别为‘茗科1号’与‘黄棪’,叶片性状与‘黄棪’聚类在一起,叶片性状更偏向于‘黄棪’(祖父、模拟父本),可优先进行乌龙茶加工品质鉴定。
类群IV中JMD56、JMD2模拟父本为‘茗科1号’,叶片性状与‘茗科1号’的母本‘铁观音’聚类在一起,叶片性状更偏向‘铁观音’(祖母),可优先进行乌龙茶加工品质鉴定。
3 结论和讨论
3.1 影响模拟父本筛选的因素
本试验选择的候选父本为42个福建省鉴定审(鉴)定品种,引物28对,在95%置信水平下为25个自然杂交后代找到模拟父本,显著性模拟父本的概率为38.4%,与其他植物的模拟父本概率(37.67%[10]、34.5%[11]、36.2%[14])相近。影响模拟父本筛选的因素较多,耿瑞静等[27]认为无效等位基因的存在是影响亲子鉴定结果准确性的首要因素;祝招玲等[28]研究表明无效等位基因是影响亲权鉴定准确性的基础因素;朱克诚等[17]认为无效等位基因与等位基因的缺失导致低质量的DNA序列是影响准确性的主要原因。基因分型错误也会导致子代与亲本的错配,一方面是实验操作包括PCR扩增、电泳等诸多因素影响, 使得条带扩增不清导致分型错误, 另一方面是数据处理过程中的人为分型错误[29]; 亲子关系鉴定试验中应尽可能降低标记基因型在扩增和判定时的错误率,有些微卫星基因座在扩增时可能产生无效基因,导致某些等位基因无法出现,本可能为杂合子的个体可能出现为纯合子,这会影响鉴定的准确性[30];进行排除法亲本分析时, 群体中候选亲本数量不宜大于100,否则很难为大多数子代确定亲本来源[31]。本试验认为,无效等位基因是不可避免的,只能尽可能的降低;候选父本的数量不是越多越好,而是模拟父本代表性越强越好,在理论上,选择相同母本与亲缘关系相近的品种作为父本,杂交后代可能为基因型一致的或极其相似的, 因此,增加亲缘关系相近的品种作为模拟父本数量会影响到软件鉴定的结果;同时构建核心种质与确定核心引物有利于提高模拟亲本鉴定结果准确性。
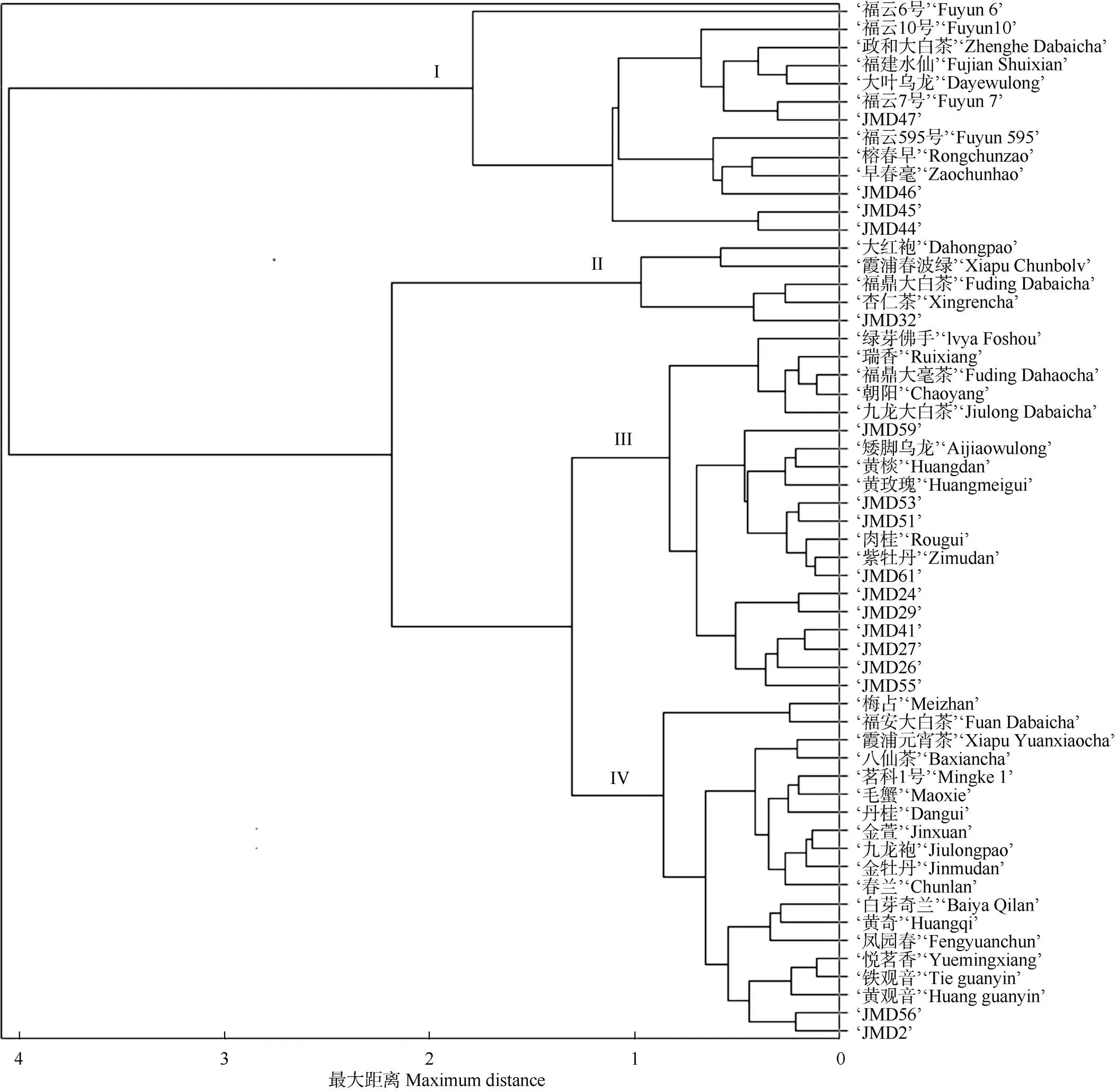
图3 叶片性状最大距离法聚类图
3.2 ‘金牡丹’自然杂交后代鉴定与辅助筛选
本研究中父本模拟分析达到显著性的‘金牡丹’自然杂交后代一共有25个,其中8个模拟父本为‘茗科1号’的自然杂交后代种植4 a后,树势衰弱或自然死亡,不能满足叶片性状调查的需要,可能由于‘茗科1号’与‘金牡丹’杂交组合属于近亲杂交,部分杂交后代生长势弱。对父本模拟达到显著性的‘金牡丹’自然杂交后代需进一步分析。
模拟父本相同的‘金牡丹’自然杂交后代,亲缘关系聚类可能有差异。JMD59、JMD27、JMD41模拟父本同为‘黄棪’,叶片性状与‘黄棪’相似,但亲缘关系聚类显示有所不同,JMD59与‘金牡丹’(母本)亲缘关系更接近,JMD41与‘黄棪’(模拟父本)亲缘关系更接近,JMD27与‘茗科1号’亲缘关系更接近。JMD51、JMD53与JMD46的模拟父本相同,JMD51、JMD53与‘丹桂’(模拟父本)亲缘关系更接近,叶片性状更接近‘肉桂’(祖母);JMD46与‘金牡丹’(母本)亲缘关系更接近,叶片性状更接近福云系列绿茶品种,叶片性状遗传可能来自于‘丹桂’(模拟父本)的未知父本。
模拟父本相同的试验材料,叶片性状聚类可能有差异。JMD24、JMD26、JMD29、JMD55与MD2、JMD56模拟父本同为‘茗科1号’,JMD24、JMD26、JMD29、JMD55的叶片性状聚类与‘黄棪’(祖父)在一起,后代可能与‘黄棪’(祖父)品质性状相似; MD2、JMD56的叶片性状聚类与‘铁观音’(祖母)在一起,后代可能与‘铁观音’(祖母)品质性状相似。
因此, ‘金牡丹’自然杂交后代在相同模拟父本的情况下,叶片性状可以分别与父本、母本、祖父、祖母相似。进行初步辅助选种,选种目标为早生优质绿茶品系的可以选择JMD32、MD44、JMD45、JMD46和JMD47进行鉴定;选种目标为闽北高香岩茶乌龙茶品系可以选择JMD51和JMD53进行鉴定;选种目标为闽南‘铁观音’乌龙茶品系可以选择MD2和JMD56进行鉴定;选种目标为与‘黄棪’相似的早生高香乌龙茶类型可以选择JMD24、JMD26、JMD27、JMD29、JMD41、JMD55和JMD59。
[1] YU F L. Discussion on the originating place and the originating center of tea plant [J]. J Tea Sci, 1986, 6(1): 1–8. [虞富莲. 论茶树原产地和起源中心 [J]. 茶叶科学, 1986, 6(1): 1–8. doi: 10.13305/j.cnki.jts. 1986.01.001.]
[2] QIAN Y H, PANG D D, WEI K, et al. Research progress on fertility of tea plant () [J]. Tea Commun, 2020, 47(2): 185–191. [钱因红, 庞丹丹, 韦康, 等. 茶树育性相关的研究进展 [J]. 茶叶通讯, 2020, 47(2): 185–191. doi: 10.3969/j.issn.1009-525X.2020.02.002.]
[3] WANG S L, MA C L, HUANG D J, et al. Analysis of genetic diversity and construction of DNA fingerprints of chlorophyll-deficient tea cultivars by SSR markers [J]. J Tea Sci, 2018, 38(1): 58–68. [王松琳, 马春雷, 黄丹娟, 等. 基于SSR标记的白化和黄化茶树品种遗传多样性分析及指纹图谱构建 [J]. 茶叶科学, 2018, 38(1): 58–68. doi: 10.3969/j.issn.1000-369X.2018.01.006.]
[4] JIANG X H, FANG K X, CHEN D, et al. Genetic diversity and relationship of two historical famousgroups in Guangdong by capillary electrophoresis detection with fluorescent EST-SSR marker [J]. Chin J Trop Crops, 2018, 39(1): 46–54. [姜晓辉, 方开星, 陈栋, 等. 基于EST-SSR毛细管电泳荧光标记技术分析广东2个历史名茶群体遗传多样性 [J]. 热带作物学报, 2018, 39(1): 46–54.]
[5] MAO J, JIANG H J, LI C X, et al. Genetic diversity analysis of tea plant in Baiyingshan Mountain of Yunnan [J]. J Tea Sci, 2018, 38(1): 69–77. [毛娟, 江鸿键, 李崇兴, 等. 云南白莺山地区茶树遗传多样性研究 [J]. 茶叶科学, 2018, 38(1): 69–77. doi: 10.3969/j.issn.1000- 369X.2018.01.007.]
[6] JIANG X H, LI H J, LI C X, et al. Genetic diversity and population structure of tea germplasm from Baiying Mountain in Yunnan [J]. Chin Agric Sci Bull, 2019, 35(14): 68–76. [姜晓辉, 李红建, 李崇兴, 等. 云南白莺山茶树种质资源遗传多样性及群体结构分析 [J]. 中国农学通报, 2019, 35(14): 68–76.]
[7] ZHOU B, WANG L B, XU X T, et al. Analysis of genetic diversity and genetic relationship of Leibo wild tea resources [J]. J Yunnan Agric Univ (Nat Sci), 2020, 35(1): 122–129. [周斌, 王留彬, 徐新涛, 等. 雷波野生茶树遗传多样性及亲缘关系分析 [J]. 云南农业大学学报(自然科学), 2020, 35(1): 122–129. doi: 10.12101/j.issn.1004-390X(n). 201904037.]
[8] ZHANG H Y, YANG T, LIU R, et al. Assessment of genetic diversity by using EST-SSR markers in[J]. Acta Agron Sin, 2020, 46(3): 330–340. [张红岩, 杨涛, 刘荣, 等. 利用EST-SSR标记评价羽扇豆属(L.)遗传多样性 [J]. 作物学报, 2020, 46(3): 330–340. doi: 10.3724/SP.J.1006.2020.94077.]
[9] LI B, PAN W T, LI K Q, et al. A comparison among three softwares of parentage analysis (CERVUS, COLONY and PAPA): A case study on parentage analysis of control pollination progenies in[J]. Mol Plant Breed, 2013, 11(2): 241–248. [李博, 潘文婷, 李康琴, 等. 三种常用亲本分析软件效率的比较——以鹅掌楸属树种控制授粉子代亲本分析为例 [J]. 分子植物育种, 2013, 11(2): 241–248.]
[10] AI C, XU L A, LAI H L, et al. Genetic diversity and paternity analysis of a seed orchard in[J]. Sci Silv Sin, 2006, 42(11): 146–150. [艾畅, 徐立安, 赖焕林, 等. 马尾松种子园的遗传多样性与父本分析 [J]. 林业科学, 2006, 42(11): 146–150. doi: 10.3321/j. issn:1001-7488.2006.11.026.]
[11] ZHANG D M, SUN P G, SHEN X H, et al. Paternity analysis of open- and control-pollinated seeds collected from a seed orchard of[J]. Chin J Plant Ecol, 2009, 33(2): 302–310. [张冬梅, 孙佩光, 沈熙环, 等. 油松种子园自由授粉与控制授粉种子父本分析 [J]. 植物生态学报, 2009, 33(2): 302–310. doi: 10.3773/j.issn. 1005-264x.2009.02.007.]
[12] FENG Y H, LI H G, YANG Z Q, et al. Construction of second generation breeding population ofin Guangxi [J]. Sci Silv Sin, 2017, 53(1): 54–61. [冯源恒, 李火根, 杨章旗, 等. 广西马尾松第2代育种群体的组建 [J]. 林业科学, 2017, 53(1): 54–61. doi: 10.11707/j.1001-7488.20170107.]
[13] WANG J Z, LAN J, LU Z X, et al. Study on using SSR markers for paternal identification in eucalyptus [J]. Eucalypt Sci Technol, 2019, 36(2): 1–8. [王建忠, 兰俊, 陆珍先, 等. 基于SSR分子标记的桉树父本鉴定技术研究 [J]. 桉树科技, 2019, 36(2): 1–8. doi: 10.3969/j. issn.1674-3172.2019.02.001.]
[14] SHAO W H, WANG Z S, ZHANG J G. Paternity analysis of main olive cultivars progenies based on SSR markers [J]. For Res, 2020, 33(3): 22–30. [邵文豪, 王兆山, 张建国. 基于SSR标记的油橄榄主要栽培品种子代父本分析 [J]. 林业科学研究, 2020, 33(3): 22–30. doi: 10. 13275/j.cnki.lykxyj.2020.03.003.]
[15] HE Y F, ZHU Y J, WU X B, et al. Parentage analysis of coreius guichenoti using microsatellites [J]. Acta Hydrobiol Sin, 2019, 43(6): 1216–1223. [何勇凤, 朱永久, 吴兴兵, 等. 基于微卫星标记的圆口铜鱼亲子鉴定技术 [J]. 水生生物学报, 2019, 43(6): 1216–1223. doi: 10.7541/2019.144.]
[16] YU S P, XU L Y, WU R M, et al. Genetic and phylogenetic analysis for resources offrom Kaihua County in Zhejiang Province [J]. J Tea Sci, 2020, 40(3): 341–351. [余书平, 徐礼羿, 吴荣梅, 等. 浙江开化县茶树种质资源的遗传多样性及亲缘关系分析 [J]. 茶叶科学, 2020, 40(3): 341–351. doi: 10.3969/j.issn.1000-369X. 2020.03.005.]
[17] ZHU K C, SONG L, LIU B S, et al. Establishment of parentage determination in yellowfin seabream () [J]. J Fish China, 2020, 44(3): 351–357. [朱克诚, 宋岭, 刘宝锁, 等. 黄鳍棘鲷家系亲缘关系鉴定 [J]. 水产学报, 2020, 44(3): 351–357. doi: 10. 11964/jfc.20181011480.]
[18] FENG X T, ZHANG G N, XUE X P, et al. Current germplasm situation of bighead carp () candidate parent and parent from hatchery in the lower reaches of Changjiang River based on SSR markers [J]. J Fish Sci China, 2020, 27(5): 589–597. [冯晓婷, 张桂宁, 薛向平, 等. 基于SSR标记的长江下游原良种场鳙亲本和后备亲本种质资源现状分析 [J]. 中国水产科学, 2020, 27(5): 589–597. doi: 10.3724/SP.J.1118.2020.19266.]
[19] LI B, REN T T, QIU M Y, et al. Paternity identification of Texel× Kazakh sheep based on microsatellite multiple PCR technology [J]. China Anim Husb Vet Med, 2020, 47(7): 2171–2180. [李彬, 任亭亭, 邱梅玉, 等. 基于微卫星多重PCR技术的特克塞尔×哈萨克杂交羊亲子鉴定 [J]. 中国畜牧兽医, 2020, 47(7): 2171–2180. doi: 10. 16431/j.cnki.1671-7236.2020.07.022.]
[20] WEN P, ZHAO J, LI W, et al. The parentage assignment ofusing multiplex PCR of microsatellites [J]. Acta Hydrobiol Sin, 2015, 39(6): 1134–1141. [文萍, 赵建, 李伟, 等. 基于微卫星多重PCR技术的黄喉拟水龟亲子鉴定 [J]. 水生生物学报, 2015, 39(6): 1134–1141. doi: 10.7541/ 2015.149.]
[21] HAN H Z, LIU Y, WANG T T, et al. Development of multiplex PCR systems of microsatellite and evaluation of parental contribution to offsprings in black rockfish () [J]. Chin Fish Qual Stand, 2018, 8(4): 48–57. [韩慧宗, 刘阳, 王腾腾, 等. 许氏平鲉微卫星多重PCR体系构建及亲本对子代贡献率 [J]. 中国渔业质量与标准, 2018, 8(4): 48–57. doi: 10.3969/j.issn.2095-1833.2018.04.006.]
[22] QUE Y F, XU X, XU N, et al. Parentage analysis ofbased on microsatellite markers [J]. J Dalian Ocean Univ, 2019, 34(5): 643–648. [阙延福, 胥贤, 徐念, 等. 基于微卫星标记的唇䱻亲子鉴定技术研究 [J]. 大连海洋大学学报, 2019, 34(5): 643–648. doi: 10. 16535/j.cnki.dlhyxb.2018-258.]
[23] YANG Y J, LIANG Y R. Clonal Tea Varieties of China [M]. Shanghai: Shanghai Science and Technology Press, 2014: 111. [杨亚军, 梁月荣. 中国无性系茶树品种志 [M]. 上海: 上海科学技术出版社, 2014: 111.]
[24] JIN J Q, CUI H R, GONG X C, et al. Studies on tea plants () germplasms using EST-SSR marker [J]. Hereditas, 2007, 29(1): 103–108. [金基强, 崔海瑞, 龚晓春, 等. 用EST-SSR标记对茶树种质资源的研究 [J]. 遗传, 2007, 29(1): 103–108. doi: 10.3321/j. issn:0253-9772.2007.01.019.]
[25] MA J Q, YAO M Z, MA C L, et al. Construction of a SSR-based genetic map and identification of QTLs for catechins content in tea plant () [J]. PLoS One, 2014, 9(3): e93131. doi: 10. 1371/journal.pone.0093131.
[26] KALINOWSKI S T, TAPER M L, MARSHALL T C. Revising how the computer program cervus accommodates genotyping error increases success in paternity assignment [J]. Mol Ecol, 2007, 16(5): 1099–1106. doi: 10.1111/j.1365-294X.2007.03089.x.
[27] GENG R J, XIE M H, WANG W M, et al. Microsatellite paternity of[J]. Freshwater Fish, 2018, 48(4): 9–15. [耿瑞静, 谢明辉, 王卫民, 等. 团头鲂的微卫星亲子鉴定 [J]. 淡水渔业, 2018, 48(4): 9–15. doi: 10.3969/j.issn.1000-6907.2018.04.002.]
[28] ZHU Z L, LIU Z S, GAO H, et al. Parentage verification and mating system ofin Helan Mountains based on Faecal DNA [J]. Acta Ecol Sin, 2019, 39(22): 8639–8647. [祝招玲, 刘振生, 高惠, 等. 基于粪便DNA的贺兰山岩羊亲权鉴定和婚配制研究 [J]. 生态学报, 2019, 39(22): 8639–8647. doi: 10.5846/stxb201806261410.]
[29] GU Y, LI C, LU C Y, et al. Microsatellite markers for parentage identification in Jian carp (var.) [J]. Hereditas, 2012, 34(11): 1447–1455. [顾颖, 李超, 鲁翠云, 等. 建鲤(var.)微卫星DNA亲权鉴定 [J]. 遗传, 2012, 34(11): 1447–1455. doi: 10.3724/SP.J.1005.2012.01447.]
[30] WANG M, ZHANG X H, CUI R, et al. Parentage testing of Dezhou donkey with microsatellite marker [J]. China Anim Husb Vet Med, 2019, 46(7): 2003–2011. [王敏, 张新浩, 崔冉, 等. 利用微卫星标记鉴定德州驴亲子关系 [J]. 中国畜牧兽医, 2019, 46(7): 2003–2011. doi: 10.16431/j.cnki.1671-7236.2019.07.015.]
[31] HE T H, GE S. Mating system, paternity analysis and gene flow in plant populations [J]. Acta Phytoecol Sin, 2001, 25(2): 144–154. [何田华, 葛颂. 植物种群交配系统、亲本分析以及基因流动研究 [J]. 植物生态学报, 2001, 25(2): 144–154.]
Genetic Identification of the Natural Hybrid Progenies of ‘Jinmudan’ Tea Varieties
YANG Jun, KONG Xiangrui, ZHENG Guohua, QIU Chenhua, WANG Rangjian*
(Tea Research Institute, Fujian Academy of Agricultural Sciences/Fujian Branch, National Center for Tea Improvement/Scientific Observing and Experimental Station of Tea Tree and Oolong Tea Processes in Fujian, Ministry of Agriculture,Fuzhou 350000, China)
In order to analyze the genetic identification of the natural hybrid progenies of tea, EST-SSR capillary electrophoresis fluorescence maker technique was used to analyze 65 the natural hybrid progenies of ‘Jinmudan’ tea. The results showed that a total of 192 polymorphic alleles were detected by 28 SSR markers, the average number of alleles (Na), observed heterozygosity (Ho) and polymorphism information content (PIC) was 0.58, 0.58 and 0.98, respectively. When the parental genotype was known, the combined exclusion probability was all reached 0.999, indicating that the 28 selected SSR marker sites had high polymorphism and high probability of exclusion, which were suitable for genetic analysis and paternity identification of individuals. The results of genetic identification showed that the 15 natural hybrid progenies of ‘Jinmudan’ tea could be divided into 4 types, JMD44, JMD45, JMD47 and JMD32 were early green tea, JMD51 and JMD53 were ‘Rougui’ oolong tea in northern Fujian, MD2 and JMD56 were ‘Tieguanyin’ oolong tea in southern Fujian, JMD24, JMD26, JMD29, JMD55, JMD59 and JMD27 were ‘Huangdan’ oolong tea.
Tea tree; Natural hybrid; EST-SSR; Genetic identification; Simulation analysis
10.11926/jtsb.4557
2021-11-02
2022-02-07
福建省现代农业(茶叶)产业技术体系项目(闽农综2019[144号]);公益类科研院所专项(2021R1029008)资助
This work was supported by the Project for Modern Agriculture (Tea) Industry Technology System in Fujian (Grant No. 2019[144]), and the Special Project for Public Welfare Research Institutes in Fujian (Grant No. 2021R1029008).
杨军(1981年生),男,硕士,助理研究员,主要从事茶树种质资源与遗传育种研究。E-mail: 99258768@qq.com
E-mail: wangrj@faas.cn

