绵羊组织特异性长链非编码RNA的鉴定与功能分析
赵 琛,刘晓懿,王喜宏
( 西北农林科技大学 动物科技学院,陕西 杨凌 712100)
长链非编码RNA(Long non-coding RNA, lncRNA)是指长度超过200个核苷酸,且不具备蛋白编码能力的RNA[1]。lncRNA在染色质调控、转录调控和转录后调控中具有重要作用[2],主要通过招募染色质重塑复合物,介导染色体间的相互作用,作为转录因子“向导”或“诱饵”,充当miRNA的“海绵”等方式发挥抑制或激活作用[3]。近年来高通量测序方法的迅速发展使得对家畜lncRNA的研究增加,Bakhtiarizadeh等[4]分析绵羊8种组织的RNA-seq数据并报告了第一个基因间lncRNA(lincRNA)数据集(n=325)。Bush等[5]根据绵羊3.1版本参考基因组(Oar_v3.1)在11个绵羊组织中鉴定出11646个新的lncRNA。但是与其他哺乳动物(例如人,小鼠,牛和猪)相比,绵羊基因组中注释的lncRNA很少。NONCODE数据库是一个比较全面的ncRNA相关注释的数据库,包含人类,小鼠,大鼠,牛,鸡和猪等17个物种的ncRNA信息[6],但是该数据库中缺少绵羊的lncRNA数据集。本研究以绵羊为研究对象,以高通量测序为手段,筛选并鉴定新型的lncRNA及组织性lncRNA,旨在发现对家畜重要经济性状有调控作用的lncRNA,丰富家畜lncRNA数据库,为lncRNA在育种工作中的应用提供借鉴和参考。
1 材料与方法
1.1 试验数据
绵羊(Ovisaries)RNA-seq测序数据包括特克赛尔羊♂×哈萨克羊♀杂交绵羊F1代个体、哈萨克羊、特克塞尔羊♂×苏格兰黑脸羊♀的杂交F1代个体的192个组织样本。由肌肉(半腱肌、半膜肌、背最长肌)、皮肤(腹侧皮肤、体侧皮肤)、脂肪(尾脂、皮下脂肪)、心脏、肺、肝脏、肾脏、小肠、脾脏构成,包含新生、出生后1周、8周、3个月、5个月、6个月与成年的数据。原始数据下载自NCBI SRA数据库,BioProject号为PRJNA485657[7]。
1.2 序列比对与转录本组装
使用Trimmomatic(版本0.36)[8]对每个样本的转录组测序原始数据进行质控,具体参数如下:LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:40。去除接头序列和过滤低质量的读段后,通过STAR软件(版本2.5.1)[9]将样本读段比对到绵羊参考基因组(Oar_v4.0)[10]。利用StringTie软件(版本1.3.4)[11]将比对结果进行样本的组装,并通过String Tie --merge将多个样本组装合并,得到绵羊的转录本集合。
1.3 lncRNA的鉴定
lncRNA的鉴定步骤:(1)去除长度小于200 bp,外显子数目小于2的转录本[12];(2)筛去蛋白编码基因;(3)利用CPC2软件[13],CNCI软件[14]预测候选lncRNA的编码能力,去除评分不合格的转录本;(4)使用blastx与UniProt数据库(版本2016_04)[15]和Pfam数据库(版本29)[16]中的已知蛋白质进行比对,认为E值≤10-3,比对长度≥10个氨基酸和一致性≥95%的转录本为“潜在的编码基因”,将此类转录本去除[17]。保留筛选下来的转录本作为绵羊候选lncRNA转录本。统计lncRNA与编码基因的外显子数目、GC含量、表达量等特征。
1.4 表达量评估
采用StringTie软件(版本1.3.4)[11]计算lncRNA在每一个组织中的表达量,使用每千碱基外显子百万片段数(fragments per kilobase of exons per million fragments mapped,FPKM)来评估表达水平,并保留至少在任意一个组织中有20%以上的样本FPKM表达量>1的转录本[7],作为绵羊lncRNA的组织表达谱。
1.5 组织特异性分析
选取每种组织的表达量的平均值代表该组织,对候选lncRNA转录本计算τ组织特异性指数。τ指数是基因特异或广泛表达的指标,取值范围为0-1,其中1表示仅在一个组织中特异表达,而0表示所有组织的广泛表达[18]。给定基因的τ指数可以用以下公式计算:
式中n是被研究组织的数量,xi是给定组织的表达谱。基于τ计算组织特异性分数,认为τ≥0.8且在所有组织中表达量排名前两位的为具有组织特异性的lncRNA,分别计算肌肉、脂肪、皮肤特异性的lncRNA。
1.6 靶基因功能预测
lncRNA的顺式作用主要通过影响邻近蛋白编码基因的表达发挥调控功能[19]。lncRNA的反式作用主要通过识别与候选lncRNA共表达的mRNA,调控远距离mRNA的转录激活与表达[20]。(1)顺式作用预测:皮尔森相关系数是用来反映两个变量线性相关程度的统计量。提取位于lncRNA上游10 kb,下游100 kb内的编码基因作为靶基因[21]。(2)反式作用预测:采用皮尔森相关系数法分析样本间lncRNA与蛋白编码基因的相关性,取相关性较高的蛋白编码基因作为靶基因(r≥0.90或r≤-0.90, Pvalue<0.01)[22]。
1.7 功能富集分析
使用R软件中的ClusterProfiler软件包[23]对基因进行KEGG通路富集分析,设置adjust.p<0.05作为显著性阈值。使用Metascape[24]对候选靶基因进行基因本体论(gene ontology, GO)富集分析,设置Pvalue<0.05作为显著性阈值。
1.8 lncRNA-mRNA互作网络的构建
利用STRING数据库(版本11.0)[22]分别对肌肉特异性lncRNA、皮肤特异性lncRNA、脂肪特异性lncRNA反式作用的靶基因数据集进行蛋白质相互作用(Protein-Protein Interaction, PPI)网络预测。整合PPI网络与共表达的lncRNA-mRNA来构建最终的互作关系。利用Cytoscape软件(版本3.7)[25]中的ClusterONE插件(版本1.0)[26]检测网络互作中的显著模块。设置Pvalue≤0.05,表达模块中最少的基因数量为3。使用Cytoscape软件[25]对lncRNA-mRNA互作网络进行可视化。
1.9 数量性状位点(QTL)分析
绵羊QTL数据(Oar_v4.0)来自AnimalQTLdb[27]。通过bedtools intersect[28]比较QTL和lncRNA在全基因组中的位置,预测lncRNA的功能。起始位置和结束位置都在QTL区域内的lncRNA被认为成功注释在QTL中[29]。
2 结果与分析
2.1 lncRNA的预测结果与组织特异性分析
利用软件CNCI、CPC2与蛋白质数据库Pfam和UniProt分别预测出94 721条、75 580条、58 136条和94 987条lncRNA,四者的交集为36 340条lncRNA,认为其为高可信度的绵羊lncRNA(图1A)。对lncRNA和蛋白编码基因的基本特征进行比较分析,lncRNA的GC含量为47.17%,低于编码基因的GC含量48.79%(图1B);lncRNA外显子平均数目为3.45个,低于蛋白编码基因的12.8个;但外显子个数在2~10区间中lncRNA为35 423个,多于编码基因的23 466个(图1C);lncRNA和蛋白编码基因的平均表达值分别为0.38和4.49,lncRNA的平均表达量低于蛋白编码基因(图1D)。

图1 绵羊lncRNA与蛋白编码基因的基本特征比较
基于RNA-Seq数据的主成分分析结果表明(图2),相似类型的组织样本更倾向于聚集在一起,如半腱肌、半膜肌、背最长肌三种肌肉组织聚集,体侧皮肤、腹侧皮肤两种皮肤组织聚集,尾脂和皮下脂肪两种脂肪组织聚集,呈现出明显的组织特异性。试验共获得肌肉特异性lncRNA 213个,皮肤特异性lncRNA 467个,脂肪特异性lncRNA 295个。
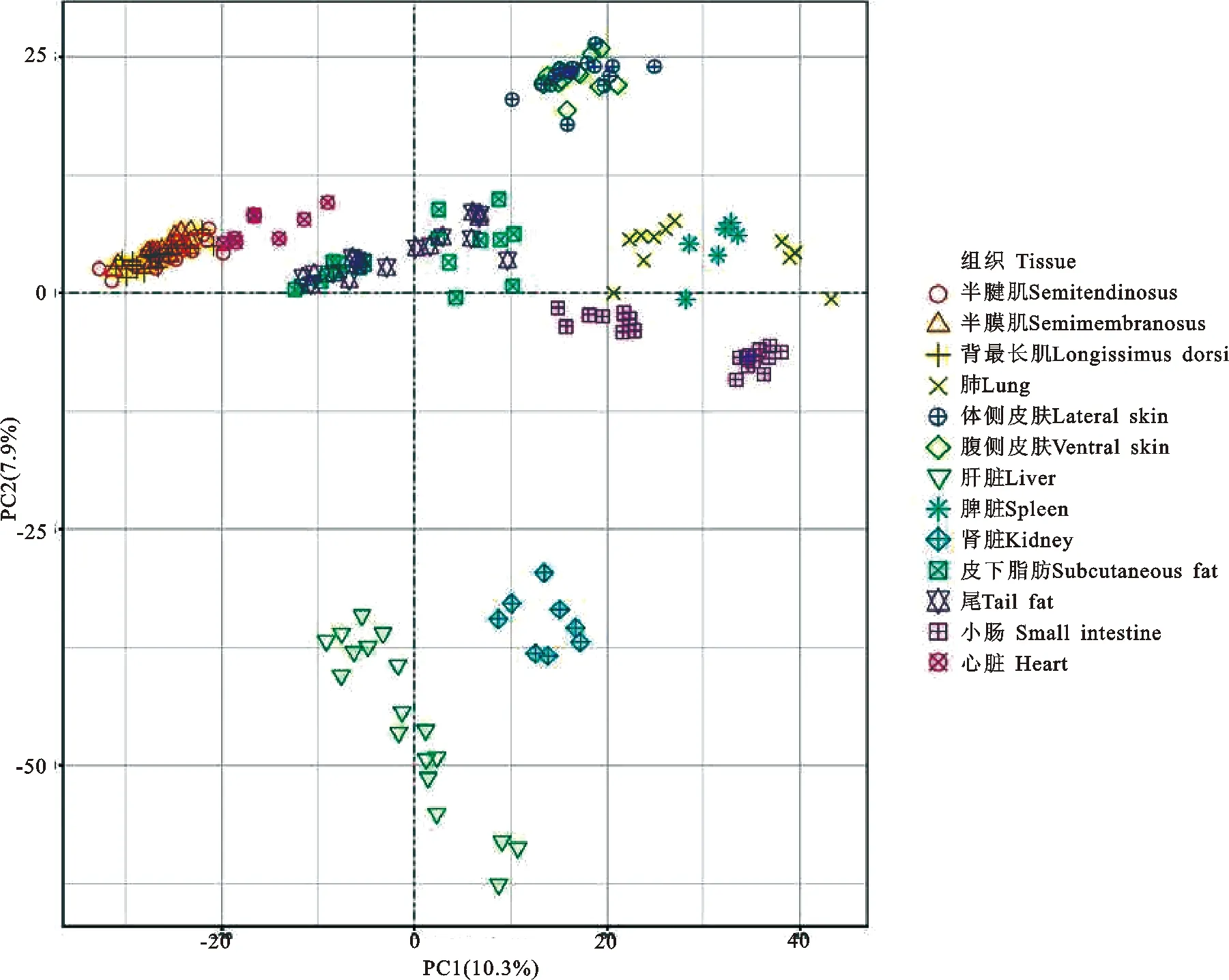
图2 组织样本的主成分分析
2.2 肌肉特异性lncRNA
基于绵羊的213个肌肉特异性lncRNA预测到了255个上下游基因。为了评估上下游基因的调控能力,从AnimalTFDB3.0数据库[30]中提取了绵羊转录因子集合,将其与顺式作用靶基因进行比较,发现有14个靶基因为绵羊转录因子。对肌肉特异性lncRNA上下游靶基因进行GO分析,发现显著富集在肌肉系统过程(19,GO:0003012)(P<0.05)和肌肉结构发育(21,GO:0007517)(P<0.05)。Six1为绵羊转录因子,是MSTRG.71513.3(7:69708802-69711134)的邻近基因,对骨骼肌发育和肌纤维类型转化具有调控作用[31]。肌肉特异性lncRNA共预测出63个反式作用的靶基因,GO富集注释结果显示,可注释到7个功能条目,显著富集是肌肉系统发育(16,GO:0003012)(P<0.05),肌肉器官发育(11,GO:0007517)(P<0.05),钙介导信号(7,GO:0019722)(P<0.05),肌动蛋白丝运动的调节(3,GO:1903115)(P<0.05)。括号内包含注释到该条目的基因数目与GO编号(图3A)。

图3 肌肉与皮肤特异性lncRNA反式作用靶基因的GO富集分析
2.3 皮肤特异性lncRNA
基于绵羊的467个皮肤特异性lncRNA预测到了1006个上下游基因,有44个为绵羊转录因子。对靶基因进行GO分析,共富集到11个功能条目,其中最显著富集的为皮肤发育(28,GO:0043588)(P<0.05),包括表皮生长因子家族基因AREG,骨形态发生蛋白受体1A型(BMPR1A),角蛋白基因(KRT),细胞间粘附分子(ICM)等。AREG位于MSTRG.68599.1(6:88881341-88903389)邻近,KRT(KRT9,KRT15,KRT17)位于MSTRG.12764.8(11:41165824-41274496)邻近,ICM(ICAM1,ICAM4,ICAM5)位于MSTRG.62954.1(5:12366881-12385388)邻近。皮肤特异性lncRNA共预测出334个反式作用的靶基因。GO富集注释结果显示(图3B),lncRNA反式作用的靶基因可注释到32个功能条目,显著富集的前7位分别是角质化(28,GO:0070268)(P<0.05),蜕皮周期(14,GO:0042303)(P<0.05),牙齿发生(11,GO:0042476)(P<0.05),表皮发育调节(10,GO:0045682)(P<0.05),长链脂肪酰基辅酶A代谢(7,GO:0035336)(P<0.05),蛋白交联(7,GO:0018149)(P<0.05),皮肤屏障的建立(6,GO:0061436)(P<0.05)。进一步选取更显著的lncRNA-mRNA对(r≥0.95或r≤-0.95)进行共表达分析,发现可分为4个表达模块,黄色圆圈表示ClusterONE[26]识别的显著表达模块,红色圆圈表示组织特异性lncRNA,蓝色圆圈表示lncRNA反式作用靶基因。若仅关注包含lncRNA的表达模块,最显著的为MSTRG.39053.2(2:228846018-228866160)所在模块(P<0.05)(图4),共10个基因,包含TGM1、IL36RN、PLA2G4E、THEM5、CERS3、PNPLA1、ALOX12B、KRT28、DSG3编码基因,对该模块富集分析发现这些基因显著富集在角质化(4,GO:0070268)(P<0.05)。
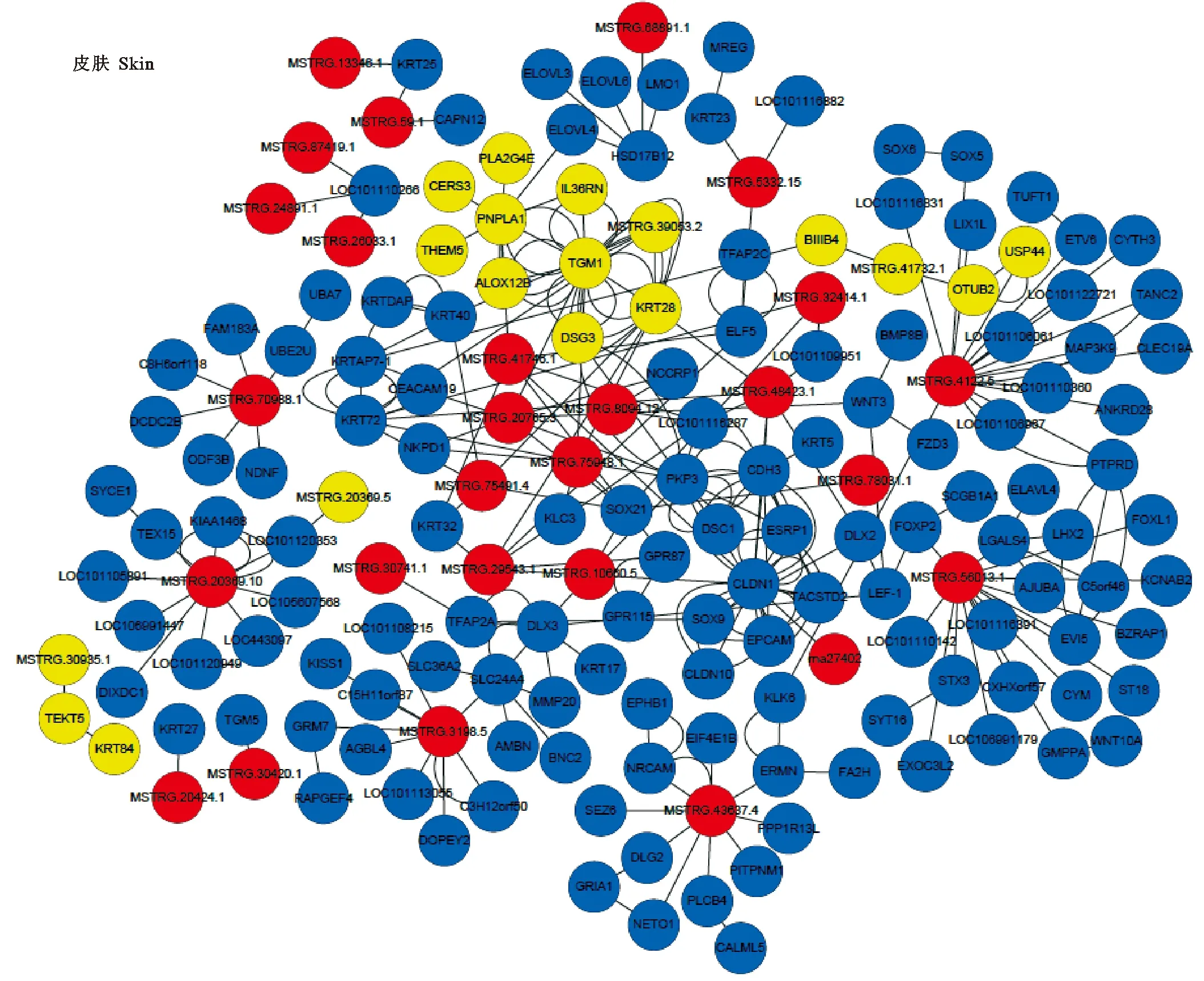
图4 皮肤lncRNA-mRNA共表达网络图
2.4 脂肪特异性lncRNA
基于绵羊的295个脂肪特异性lncRNA预测到了787个上下游基因,有43个为绵羊转录因子。对上下游基因进行GO分析,显著富集到脂质定位(22,GO:0010876)(P<0.05)。转录因子HOXC基因HOXC11、HOXC12、HOXC13位于LNC12062(LOC105611004,3:132242058-132246141)邻近。脂肪特异性lncRNA共预测出79个反式作用的靶基因。KEGG数据库注释结果显示,注释到8条通路(P<0.05),注释基因数最多的前5位分别为甘油脂代谢(5)(P<0.05)、脂肪酸伸长率(3)(P<0.05)、不饱和脂肪酸的生物合成(3)(P<0.05)、缬氨酸,亮氨酸和异亮氨酸的降解(3)(P<0.05)、脂肪的消化吸收(3)(P<0.05)。GO富集注释结果显示,注释到11个功能条目,显著富集的是脂质代谢(14,R-HSA-556833)(P<0.05),DAG和TAG的酰基链重塑(14,R-HSA-556833)(P<0.05),跨化学突触的传递(7,R-HSA-112315)(P<0.05),胆汁盐和有机酸,金属离子和胺化合物的转运(4,R-HSA-425366)(P<0.05)。lncRNA-mRNA共表达分析发现了1个显著表达模块(图5),包含8个基因,其中包含2个预测的lncRNA——MSTRG.57079.1(3:166886406-167025566)、MSTRG.68891.1(6:100780317-100820136)。对该模块进行GO富集分析发现主要富集在酰基辅酶A代谢过程(4,GO:0006637)(P<0.05),KEGG通路最显著富集于甘油三酯的生物合成(P<0.05),这两个过程都与脂肪合成相关。括号内的数字表示注释到该条目的基因数。
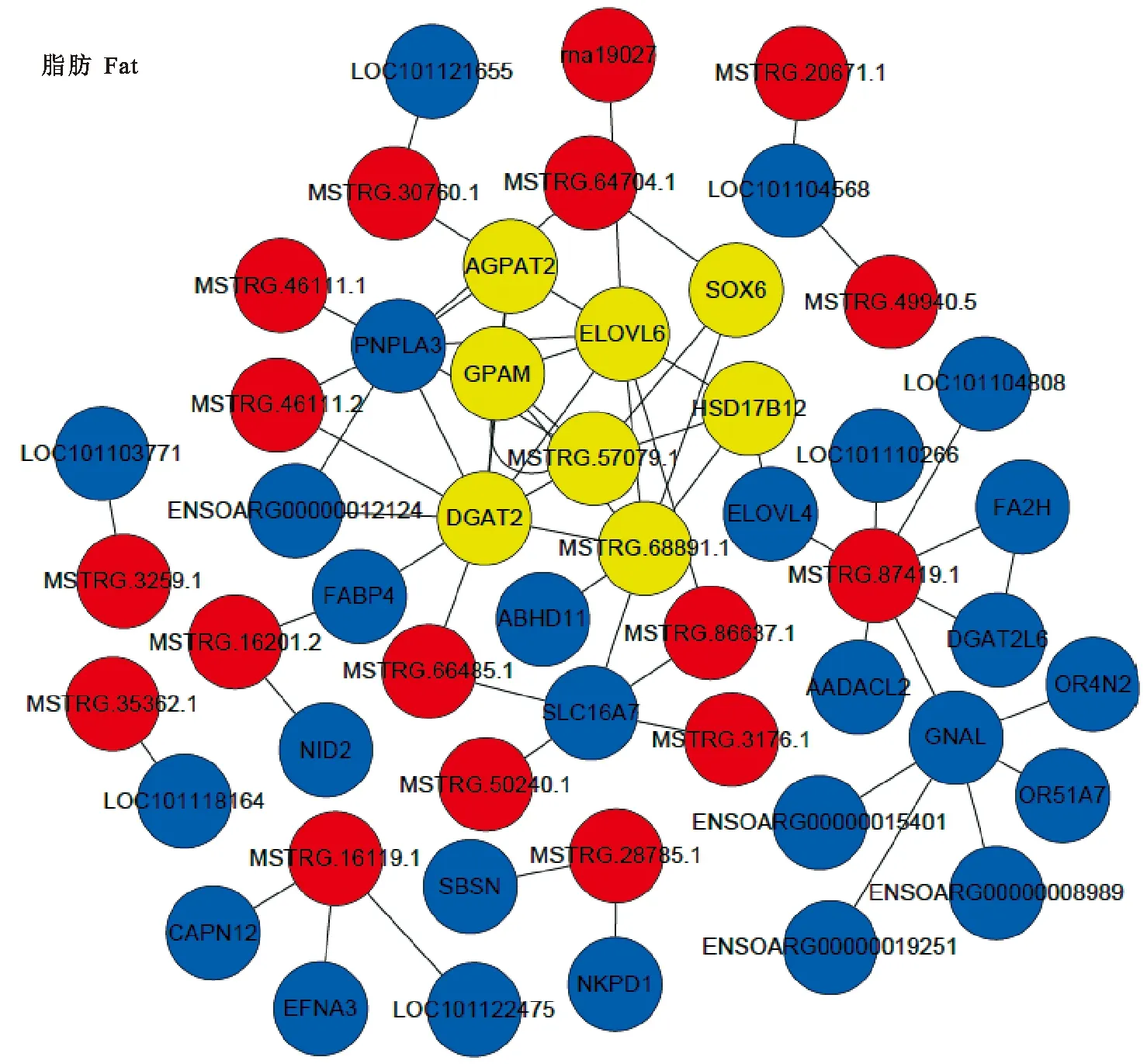
图5 脂肪lncRNA-mRNA共表达网络图
2.5 QTL分析
绵羊QTL数据库[27]包含肌肉相关QTL 681个,脂肪相关QTL 231个。对组织特异性lncRNA与绵羊QTL数据库的重合关系进行检索,共发现14个lncRNA可能作为绵羊肌肉与脂肪相关性状的候选lncRNA。存在6个肌肉特异性lncRNA与肌肉相关QTL区域重合,其中有3个位于胴体肌肉重量QTL区域,1个位于肌肉密度QTL区域,2个位于体重QTL区域(表1)。11个脂肪特异性lncRNA与脂肪相关QTL区域重合,其中有5个位于尾脂沉积区域,6个位于全部脂肪区域(表1)。MSTRG.77930.1(9:93629571-93634360)与尾脂沉积QTL重合,其顺式作用基因之一为与脂质合成相关的DEPTOR,可通过抑制胰岛素信号mTORC1介导的反馈抑制作用来促脂肪合成[32]。

表1 位于QTL区域的组织特异性lncRNA
3 讨 论
本研究共鉴定36 340个绵羊lncRNA,发现肌肉特异性lncRNA 213个,皮肤特异性lncRNA 467个,脂肪特异性lncRNA 295个,扩展了绵羊基因组lncRNA集合,为进一步分析提供了良好的基础。与编码基因相比,lncRNA的GC含量、表达丰度较低,但平均长度更长,并且组织特异性高。这些基本特征与前人报道的哺乳动物lncRNA相似[33]。
肌肉质量是决定畜禽经济价值的重要因素之一,肌肉发育和生长过程受遗传、环境和营养的影响。lncRNA已被证实通过顺式或反式作用调节肌肉生长发育[34],很可能是重要的潜在的肌肉生长调节因子。Cai等[35]发现位于鸡Six1基因上游432 bp的lncRNA-Six1通过编码一种微肽激活Six1基因,发挥顺式调节作用,促进细胞增殖并参与肌肉生长。Mueller等[36]发现小鼠成肌转录因子MyoD基因的转录起始位点上游5 kb处编码的lncRNA MUNC,可以促进MyoD在骨骼肌生成。MSTRG.46395.2的靶基因是肌球蛋白1(MYOM1),位于MYOM1转录下游区域。MYOM1在横纹肌的细胞分化中编码一种高度特异性的蛋白质标记,并可能在肌原纤维的组装或维持中起作用[37]。本研究发现MSTRG.46395.2在肌肉组织中特异表达,可能表明MSTRG.46395.2与MYOM之间可能存在调控关系,但是需要进一步研究以阐明确切的作用机理。Six1是参与骨骼肌发育的重要转录因子[38],肌肉特异性lncRNA MSTRG.71513.3位于其下游7 kb。有研究表明Six1不仅可以调节滩羊的肌肉纤维类型,而且还可以调节肉的嫩度,可以作为选择肉嫩度的候选基因[39]。MSTRG.71513.3在肌肉组织中特异表达,推测MSTRG.71513.3与Six1之间可能存在调控关系,调控肌肉发育。但是确切的作用机理需要进一步研究。
毛囊发育和周期性生长过程中受多种信号通路调控,主要有WNT通路、转化生长因子(TGF)家族、丝裂原活化蛋白激酶(MAPK)家族、Notch信号通路、骨形成蛋白(BMP)信号通路等[40]。有研究发现绵羊胎儿皮肤初级毛囊中下调的lncRNA与角蛋白(KRT14和KRT15)、WNT信号通路(WNT16和SFRP1)等通路基因相互作用,进而影响表皮和羊毛发育[41]。本研究发现,皮肤特异性lncRNA的上下游基因与反式作用共表达基因主要与角质化、毛囊发育相关。Notch信号通路和毛囊的形态发生具有直接的关系,可能参与了毛囊发育的早期环节,并在毛干分化过程中起重要作用[40]。DLL4(Notch配体)位于MSTRG.70607.8(7:33364804-33478104)邻近,猜测该皮肤特异性lncRNA可能对毛囊发育具有调控作用。角蛋白是一种中间纤维形成蛋白,是复杂上皮细胞的主要成分[42]。有研究表明具有无效的KRT17基因的小鼠在出生后的第一周内无法形成完整的皮毛[43],MSTRG.12764.8邻近基因包括多个KRT基因KRT9、KRT15、KRT17,猜测其对表皮角质化及毛囊发育有重要作用,但具体作用机制待进一步分析。
lncRNA在脂质代谢中的调节功能已经在不同生物体中得到了证实,包括人类[44],小鼠[45],猪[46],牛[47]等。有研究表明,皮下脂肪组织中HOXC簇的表达上调[48]。还有研究发现,HOXC11,HOXC12,HOXC13等发育基因与绵羊尾部脂肪沉积有关[49]。本研究发现LOC105611004分别位于HOXC11,HOXC12,HOXC13附近,猜测该lncRNA可能对绵羊尾脂沉积有调控作用。还发现MSTRG.77930.1顺式调控的靶基因是DEPTOR。DEPTOR通过激活脂肪细胞中的过氧化物酶体增殖物激活受体γ自主促进脂肪生成,是脂肪形成的新调节剂[50]。lncRNA-mRNA共表达分析发现了2个lncRNA(MSTRG.57079.1和MSTRG.68891.1)出现在与脂肪相关的表达模块中,与酰基辅酶A代谢和甘油三酯的生物合成功能相关,有研究表明骨骼肌脂肪储存的增加通常伴随脂质代谢产物(例如甘油二酯和长链酰基辅酶A)的逐步积累[51],推测这两个lncRNA可能对脂肪代谢具有调控作用。
4 结 论
本研究通过分析绵羊不同转录组的RNA-seq数据,获得了新的绵羊lncRNA数据集,并筛选出了具有肌肉、皮肤、脂肪组织特异性的lncRNA。基于顺式作用和反式作用两个方面,对lncRNA靶基因的功能进行分析,发现其可能在组织代谢、发育等多个功能中发挥调控作用,推测肌肉特异性lncRNA可能与肌肉生长功能相关,脂肪特异性lncRNA可能与脂肪合成相关,皮肤特异性lncRNA可能与表皮形成和毛囊发育相关。同时通过QTL筛选,获得了与绵羊肌肉、皮肤与脂肪性状相关的候选长链非编码RNA。本研究为探究lncRNA在绵羊重要经济性状的功能提供了新见解。

