基于影像组学的T1WI增强模型预测高级别脑胶质瘤TP53基因突变的研究
李明 郑金晶 董海波 王超超 卫雨果
脑胶质瘤是颅内最常见的原发恶性脑肿瘤,按WHO分类将Ⅲ级胶质瘤和胶质母细胞瘤称为高级别胶质瘤。2021年版《WHO中枢神经系统肿瘤分类》进一步明确中枢神经系统肿瘤以分子分型作为核心依据,基因型结合表观型的分型方法进行分类[1]。TP53基因是一种重要基因分子生物学标志物,分为野生型和突变型两种表达状态;其是一种抑癌基因,其突变或缺失是导致胶质瘤发生的原因之一,与患者预后和化疗敏感性有密切联系[2-3]。近年来,影像组学成为研究的热点,目前国内对TP53基因组学方面的研究较少,对增强MR序列的研究尚未见报道。本文拟通过构建高级别脑胶质瘤T1WI增强基因组学模型,预测TP53基因的表达状态,对指导临床精准医疗和肿瘤治疗疗效的评估提供依据[4]。
1 资料与方法
1.1 临床资料 回顾性分析2015年3月至2021年10月经宁波市医疗中心李惠利医院外科手术证实的高级别脑胶质瘤患者。纳入标准:①在本院神经外科病理证实为高级别脑胶质瘤患者;②术前均在GE 3.0 T MR行T1WI增强检查;③能够对增强序列脑肿瘤实质强化部分进行有效的分析,图像质量符合标准磁共振扫描数据。排除标准:①有颅脑手术史;②P53免疫组化结果缺失;③接受过放化疗及类固醇治疗。共纳入68例符合标准的高级别脑胶质瘤患者,其中WHO Ⅲ级12例(7例间变型星形细胞瘤、5例间变型少突胶质细胞瘤),WHO Ⅳ级56例胶质母细胞瘤;其中TP53突变型46例(男22例,女24例,年龄21~87岁,WHO Ⅲ级6例,WHO Ⅳ级40例),TP53野生型22例(男15例,女7例,年龄26~80岁,WHO Ⅲ级6例,WHO Ⅳ级16例)。按照7∶3分为训练集和测试集,其中训练集48例(TP53基因突变型33例,野生型15例);测试集20例(TP53基因突变型13例,野生型7例)。
1.2 仪器与方法 采用美国GE Discovery7503.0T MR超导型磁共振仪、8通道头颅相控阵线圈检查,仰卧位固定患者头部。(1)常规MR平扫序列:T1WI序列:TE24 ms,TR1,850 ms,TI780 ms;T2WI序列:TE 105 ms,TR 6,656 ms;矩阵288×224,FOV240 mm×240 mm,层间距1 mm,层厚4 mm,扫描层24层。(2)T1WI增强MR序列:注入对比剂钆喷酸葡胺(GE药业)注射液0.2 mL/kg后,横断面T1WI序列扫描,参数同平扫T1WI序列。(3)DWI序列:TR 4,500 ms,TE为最小值,b值设定1,000 s/mm2,矩阵160×160,FOV 240 mm×240 mm,层间距1.5 mm,层厚5 mm,扫描层数24层。
1.3 图像处理与特征提取 将收集到的所有患者颅脑T1WI增强图像,通过医院PACS系统以DICOM格式导出,然后导入GE A.K V3.3.0分析软件(Analysis Kit),对图像进行标准化预处理,包括图像的重采样和灰度级规范化,必要的图像滤波和配准处理等;然后将预处理后的图像导入ITK-SNAP软件,由2名具有5年以上工作经验的神经影像医师在双盲的情况下,分别沿肿瘤实质最大强化轮廓缘手动勾画感兴趣区ROI,包括肿瘤实质、囊变及坏死区,不包括病灶周围水肿区。勾画完成后由另一名高年资医师比对二者勾画ROI是否准确,选择其中最佳ROI入选,如对二者结果均有异议,三人讨论后适当调整ROI(见图1),最后将各层ROI合成3D-VOI。将合成的VOI导入A.K软件并提取特征,共得到396个符合IBSI标准的影像组学特征[5],包括灰度共生矩阵特征(GLCM)、灰度游程长度矩阵特征(RLM)、形态学特征(Formfactor)及直方图特征(Histogram)等特征。采用R语言Spearman相关性分析去除相关系数r绝对值>0.9的其它强相关特征,再使用最小绝对收缩和选择算法(leastabsoluteshrinkage and selection operator,LASSO)筛选出用于模型构建的影像组学特征参数(见图2)。LASSO回归是通过生成惩罚函数对回归模型中的变量系数进行压缩,起到防止过度拟合,解决严重共线性的作用,常被用于变量数据的筛选压缩[6];有研究表明LASSO回归分析适用于样本量较少而提取的组学特征较多的情况[7]。

图1 采用ITK-SNAP软件手动勾画病灶最大强化层面感兴趣区(ROI)

图2 应用Lasso回归十折交叉验证方法筛选最优特征集过程
1.4 统计学方法 采用SPSS 22.0统计软件。符合正态分布的计量资料以(±s)表示;用t检验,偏态分布计量资料以M(Q1,Q3)表示,用U检验。计数资料资料用χ2检验。采用Spearman相关性分析去冗余,Lasso回归筛选获取最终特征参数,采用Logistic回归组建预测模型,并构建Hosmer-Lemeshow校准曲线对模型校准。采用ROC曲线下面积AUC值对模型的效能进行评估,0.5<AUC≤0.7提示诊断效能较低,0.7<AUC≤0.9诊断效能中等,AUC>0.9诊断效能较高。P<0.05为差异有统计学意义。
2 结果
2.1 两组一般资料比较 见表1。

表1 TP53基因突变组与TP53野生组脑胶质瘤患者一般资料比较
2.2 特征参数 从T1WI增强图像上提取的396个特征经过去冗余筛选,最终获得9个影像组学特征,分别是1个形态学特征Form Factor Parameters(Compactness2);3个灰度共生矩阵GLCMParameters(GLCMEntropy_angle0_offset7,HaralickCorrelation_angle0_offset7,Correlation_angle45_offset7);3个灰度游程长度矩阵RLMParameters(ShortRunEmphasis_angle45_offset7,LongRunHighGreyLevelEmphasis_AllDirection_offset1_SD,LongRunEmphasis_AllDirection_offset4_SD);1个纹理参数Texture Parameters(ClusterProminence_angle0_offset4)和1个直方图特征Histogram(Quantile0.75)。
2.3 建立模型与效能评价 利用获得的9个特征,采用逻辑回归分析建立影像基因组学模型;用ROC曲线下面积AUC值评估影像组学模型对TP53基因突变型和野生型高级别脑胶质瘤的鉴别效能。在训练集中,在最佳截断值(0.787)下模型的AUC值为0.883、灵敏度为0.697、特异度为0.933;测试集模型的AUC值为0.802、灵敏度为0.769、特异度为0.857。见图3~4。

图3 训练集ROC曲线图
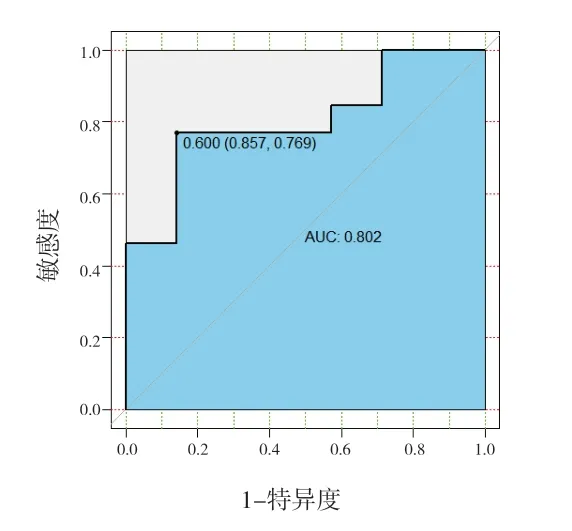
图4 测试集ROC曲线图
2.4 模型校准度评估 通过建立预测模型可计算得到训练集预测TP53基因突变的概率。为比较预测概率与实际概率吻合程度,进一步行预测模型的校准曲线分析[8]。该模型的Hosmer-Lemeshow校准曲线见图5,所示的校准线与理想模型的对角直线稍有偏差,但较为接近且趋势吻合(P=0.928),提示预测模型构建良好。

图5 为训练集模型Hosmer-Lemeshow校准曲线
3 讨论
脑胶质瘤具有很强的基因异质性,病理的获取需要有创的外科操作,由于肿瘤的基因型、蛋白表达模式等存在时间、空间分布异质性,其结果常无法全面反映肿瘤的性质,也是临床开展精准医疗的重要障碍,尤其是中晚期肿瘤和术后复发或治疗后进展的肿瘤,通过有创性手段获得组织标本更加困难。因此,术前发现与脑胶质瘤基因相关的预测因子,建立有效的预测模型,具有重要的临床意义。TP53基因是一种抑癌基因,其编码产物为P53蛋白,该蛋白可通过转录调节下游靶基因而发挥调节细胞周期、诱导细胞凋亡、抑制血管生成、调节能量代谢的作用,进而阻止DNA损伤和有丝分裂后异常染色体细胞的存活,抑制肿瘤形成、生长[9]。P53蛋白可通过免疫组织化学方法进行标记,利用免疫组化检测对TP53基因表达类型的获取,具有突变简单易行,特异度较高的优势[10]。
TP53基因的突变,使胶质瘤的生长抑制调节作用减弱,导致其生长过程出现变化。HASSE等[11]通过分析增强T1WI图像上胶质母细胞瘤的表面形态、肿瘤体积和坏死体积与Ki67和TP53基因的相关性,认为肿瘤形态学特征可以预测胶质母细胞瘤Ki67和TP53基因的表达状态。BERNABÉU等[12]通过利用MR光谱分析评估高级别胶质瘤的浸润生长状态,认为TP53基因的突变状态与脑胶质瘤的浸润袭性有关,TP53基因突变型脑胶质瘤的生长具有更高的侵袭性。LI等[13]发现TP53突变型与野生型胶质瘤内微血管密度具有差异性,可以导致T2WI加权图像上的信号差异,亦可表现在增强序列强化程度的不同。
本研究中,用于构建模型的9个影像组学特征中,有1个直方图特征、7个纹理特征(3个GLCM、3个RLM特征和1个Texture Parameters),1个形态特征。其中,直方图特征可以描述图像中单个像素灰度频率分布情况;纹理特征可以对像素灰度值空间分布情况进行描述,反映相邻像素的交互作用,如本研究提取到的GLCM纹理特征—熵,其是图像强度随机性的度量,表示图像压缩所需的图像信息量,度量了在传输信号中信息或信息的损失,也度量了图像信息,熵的值越大,表示图像的纹理信息越复杂。因此,可以通过直方图特征和纹理特征来描述胶质瘤的增强程度及信号间的差异,进而解释肿瘤间的异质性,从而可以有效预测胶质瘤TP53基因表达状态[14]。本研究对入组患者临床资料进行统计分析,结果显示TP53基因野生型与突变型两组患者性别、年龄与病理分级等差异均无统计学应用。故本研究仅采用TIWI增强图像筛选到的特征参数,进行影像组学基因预测模型的构建。
本研究采用在T1WI增强图像上勾画感兴趣区,能够较准确勾画靶区,可以直观观察病灶形态和浸润情况,对病灶T1WI增强图像提取到的特征,建立影像基因组学二分类模型,结果显示,训练集AUC值为0.883,校准曲线重合度较好;测试集AUC值为0.802,表明该模型对术前鉴别预测胶质瘤TP53基因突变与否具有较好的预测效能。
但本研究仍存在一定的局限性。第一,该研究为单中心回顾性分析研究,病例收集时间跨度大,部分样本因资料不全而排除,在样本选择上可能存在一定的偏倚:第二,纳入研究的样本量较少,模型性能可能存在不稳定性,模型的泛化能力有待验证:第三,因低级别胶质瘤多数强化不明显,故本研究仅对高级别胶质瘤进行了单一分析,对低级别胶质瘤的TP53基因表达未做探索。
综上,基于术前T1WI增强MRI建立的影像组学模型可以对高级别胶质瘤的TP53基因表达状态进行预测,从而对肿瘤治疗和预后效果提供参考。

