珠江广州段浮游真核微生物分布及其法医学意义
苏 秦,余仲昊,徐曲毅,吴伟斌,李 欢,杜蔚安,赵 建,陈 玲,刘 宏,刘 超,,*
(1.广州市刑事科学技术研究所,公安部法医病理学重点实验室,广州 510442;2.南方医科大学法医学院,广州 510515)
溺水是常见的非正常死因之一,而水中尸体的发现地通常并不是其实际溺水点[1-3]。确定溺水点对于寻找尸源、分析案情和侦破案件至关重要[4]。目前溺水地点的推断主要是通过分析溺死尸体的器官组织中是否含有嫌疑地点水样中的特异性标记物来进行[5]。这些标记物通常需要表现出在溺液中普遍存在且特异分布的特征。目前,硅藻和异物颗粒应用最为广泛。
迄今, 微波消解—真空抽滤—扫描电镜硅藻检验法为识别硅藻形态的标准方法[6],但形态学鉴定需要专业分类学的支持,且很难获得完整的丰度信息。此外,当水域中缺乏特异性硅藻、种类无分布差异或者某些硅藻因体积较大难以进入溺水者的脏器中时,硅藻检测对于溺水地点的推断便有局限性[7]。而异物颗粒通常与化工厂、钢铁厂、军工厂等重工业的排污口有关,在排放不稳定或缺乏工业排放的情况下,该方法的应用亦受到限制。随着二代测序技术的广泛应用,通过18S rDNA测序从分子层面获取包含硅藻在内的浮游真核微生物的种群和丰度信息,有助于建立特定水域中浮游真核微生物种群的背景数据库,进而为溺水地点的推断提供基础数据[8]。其中,18S rDNA V4—V5区段应用最多,且其数据库信息也最全、分类效果最好。
本文通过18S rDNA测序,对珠江水域(广州段)进行浮游真核微生物群落的调查,以解析包括硅藻在内的浮游真核微生物在同一水域不同地点的分布特征,同时为建立水中浮游真核微生物种群的背景数据库积累基础数据,为水中尸体的法医遗传学分析提供参考。
1 材料和方法
1.1 水样采集
在珠江广州段水域随机等距离选取4个采样点(相邻取水点相距25~30 km,从上游的佛山三江源景观点附近处至下游的近入海口处依此为W1、W2、W3、W4),进行水样采集,采样时间为2021年7月。在距岸边1 m、水下0.3 m处收集水样,每个取样点取3份水样(每份500 mL)。为避免不同采水点的微生物通过取水器产生交叉污染,每次取水前,用取样点处的水样将取水器冲洗3次,取样结束后再用纯净水冲洗3次。
1.2 微生物富集
采用HL-6型多联真空抽滤仪(珠海黑马医学仪器有限公司)处理水样,将水样中的浮游微生物富集至0.22 μm孔径的滤膜上,再将滤膜封装于无菌离心管中。液氮速冻,-80 °C冰箱中保存。
1.3 DNA提取和PCR扩增
根据 E.Z.N.A®Soil DNA Kit(Omega Bio-tek, 美国)说明书抽提水样浮游微生物群落的总DNA,采用TAReukFWD1F(CCAGCASCYGC- GGTAATTCC)和TAReukREV3R (ACTTTCGTTCT- TGATYRA)引物对18S rDNA的V4—V5区段进行扩增,程序如下:95 °C预变性3 min,27个循环(95 °C变性30 s,55 °C退火30 s,72 °C延伸30 s),72 °C稳定延伸10 min,4 °C保存。PCR反应体系为20 μL,每个样本重复3次。
1.4 建库与测序
将同一样本的PCR产物混合后以2%琼脂糖凝胶回收,利用AxyPrep DNA Gel Extraction Kit (Axygen Biosciences,Union City, CA, USA)纯化回收产物,并用Quantus™ Fluorometer (Promega, USA)对回 收产物检测定量。使用NEXTf lexTMRapid DNA-Seq Kit(Bioo Scientif ic, USA)建 库,再 以Illumina Miseq PE300平台测序(上海美吉生物医药科技有限公司)。
1.5 数据分析
使用fastp(https://github.com/OpenGene/fastp, version 0.20.0)软件对原始测序数据进行质控,再经FLASH(http://www.cbcb.umd.edu/software/f lash,version 1.2.7)软件拼接序列,最后利用QIIME2软件进行后续的18S扩增子分析,其中使用Deblur去噪并生成特征表,以Silva138数据库(https://www.arbsilva.de/)进行注释分析,使用R语言进行韦恩图分析和作图。
2 结果
2.1 Alpha多样性分析
18S rDNA的测序分析结果显示珠江流域(广州段)水样中观测到的分类操作单元(operational taxonomic unit,OTU)数(按照97%的相似性阈值)平均为483.73±39.06个。其中,W1的OTU数目最多(638.33±1.67个),而W4数目最少(298.33±10.73个)(图1A)。W1的香农指数(Shannon index)最高,W2和W3次之,W4最低(图1B)。这些结果表明,随着取水点离入海口越近,被检测到的浮游真核微生物种类和物种分布的均匀度均降低。

图1 四个取水点的α多样性指数(A:观测到的OTU数目;B:Shannon指数。不同字母表示存在统计学差异(P<0.05))Fig.1 Alpha-diversity i ndexes derived from four sampling locations (A: observed OTUs; B: Shannon index, with statistical differences(P<0.05) being indicated as letters (a, b, c))
2.2 Beta多样性分析
采用PCoA方法对数据降维后进行Beta多样性分析,确定不同取水点的微生物物种的异质性。分别采用非加权的Unifrac距离和Jaccard距离以及加权的Unifrac距离和Bray Curtis距离进行计算。其中,非加权的方法仅考虑浮游微生物成员在群落中存在与否,而不考虑其丰度高低;加权的方法需考虑浮游微生物成员的丰度情况。结果显示,取水点W4在各种算法中均与另外三个取水点存在显著差异,这与Alpha多样性的结果一致(图2)。虽然取水点W1、W2和W3在各种算法中也呈现出各自组内一致性且与其他取水点存在差异的趋势,但是W2在非加权算法中与W3更为接近(图2A,2C),而在加权算法中与W1更为接近(图2B,2D)。这些结果表明,W4的物种构成与另外三组存在显著差异,而W2的物种构成种类与W3接近,但W2的优势物种分布与W1更接近。
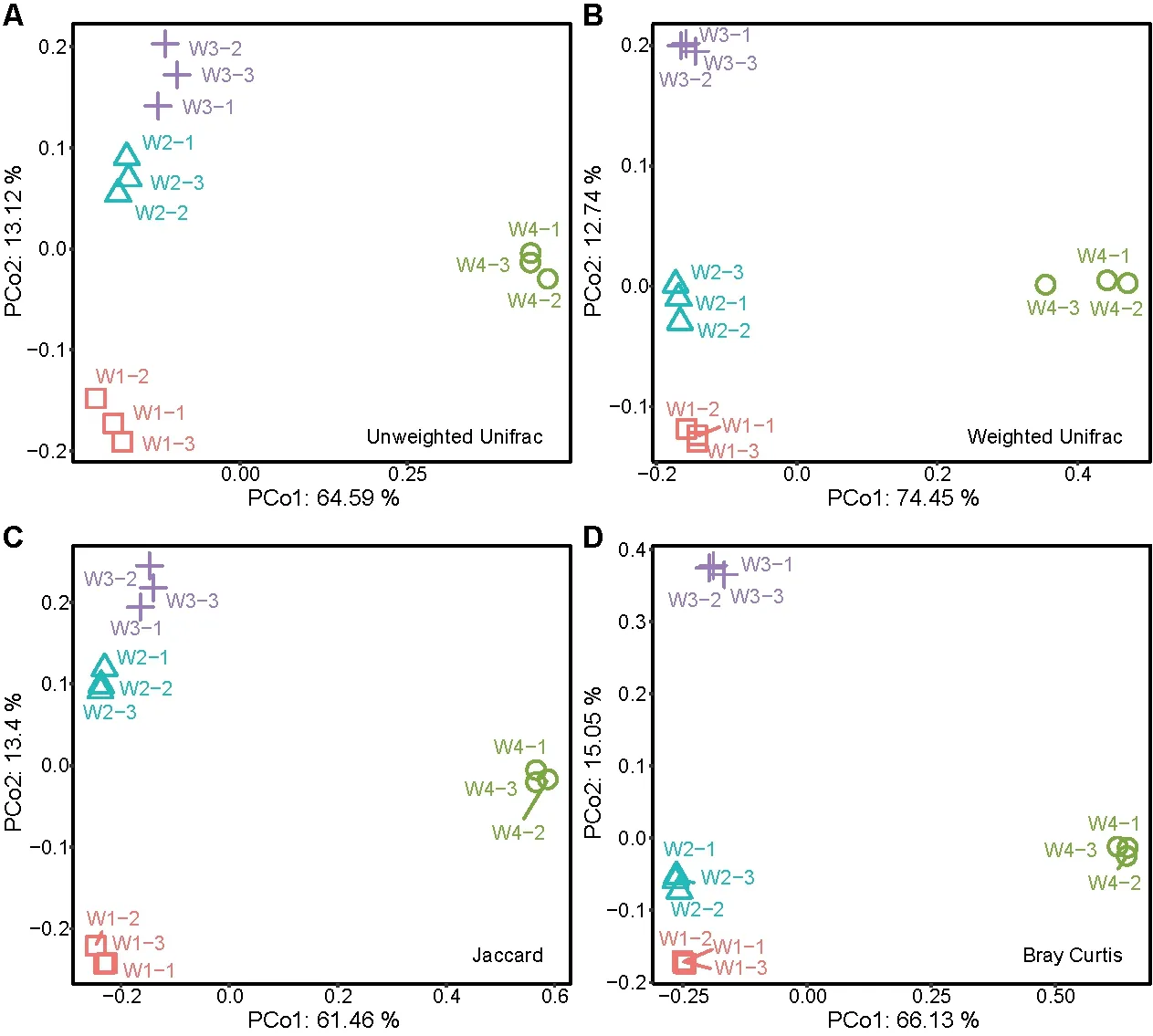
图2 四个取水点的β多样性指数Fig.2 Beta-diversity indexes derived from four sampling locations
2.3 取水点的群落组成
对各取水点的样本进行门水平分类群注释,按12个样本的丰度排序,每个取水点的3个样本取均值展示(图3)。

图3 门水平上的相对含量分布图Fig.3 The relative abundance distribution of microplanktons at the phylum level
从序列特征推断的优势微生物分类群依次为硅藻门、纤毛虫门、绿藻门、丝足虫门、隐藻门、皮胆虫门、囊泡虫门、褐藻门、壶菌门、甲藻门、隐真菌门和轮虫动物门等。硅藻门为排序第一的微生物类群,但在不同组别中比例并不相同,从取水点W3的最高比例46.33%到取水点W4的最低比例10.28%,而W1和W2的比例分别为24.25%和21.22%。对取水点W4的门水平群落组成分析的结果显示,里面含有较大比例的皮胆虫门类属,而这类微生物在其他三个取水点比例极低。结合Beta多样性分析的结果,不同取水点的微生物群落组成存在显著差异,应可为不同地点溺死尸体的鉴别提供可行性。
2.4 硅藻的含量差异
由于硅藻门在门水平为排序第一的优势微生物类群,且其对溺死案例的死因诊断具有重要意义,因此对各取水点的硅藻作属水平的含量统计。结果如表1所示,辐环藻属为取水点W1、W2和W3的优势硅藻属,但在W4的含量低,仅占0.69%±0.39%。而骨条藻属和Bacillariophyceae属在取水点W4的含量比其他三个取水点高,分别为39.88%±2.65%和15.88%±0.56%。此外,取水点W4还有三个特有硅藻属,分别为圆筛藻属、细柱藻属和泥生藻属。取水点W1中有一个特有硅藻属(脆杆藻属),而W2和W3中并不存在特有硅藻属。经分析,所有硅藻属的含量在4个取水点中的含量均存在统计学差异。
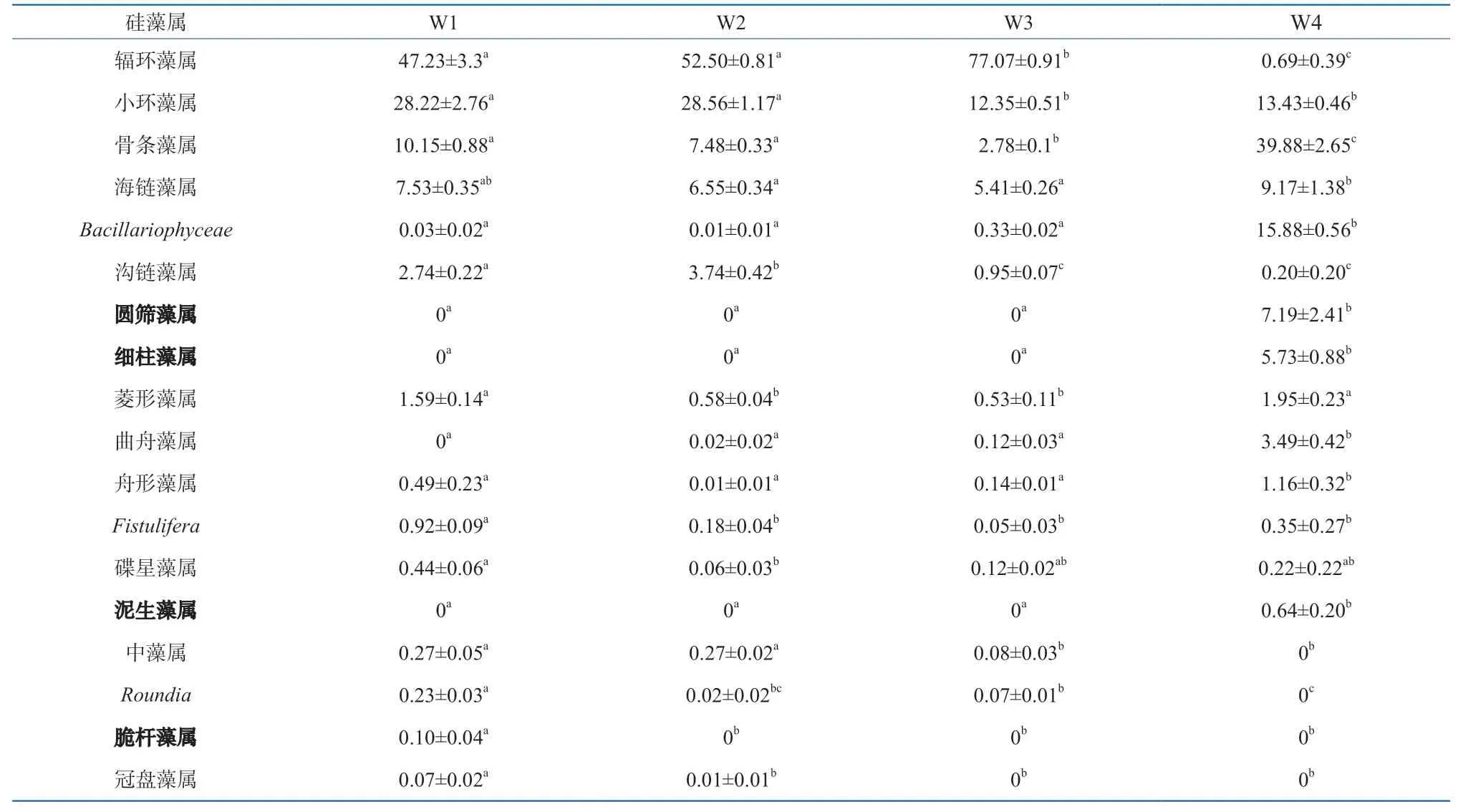
表1 硅藻属的相对含量分布Table 1 Relative content distribution of diatoms at the genus level %
2.5 特有物种分析
由于在取水点W2和W3中没有发现特有的硅藻属,因此我们将范围扩大到所有浮游真核微生物物种上。通过韦恩图发现,W4中的特有物种最多(98种),其次为W1(27种),而W2和W3的特有物种最少,分别为7种和6种(图4),这与硅藻属分析的结果类似。这些结果表明当分类水平细化到种水平时,各取水点相应的特异物种种类增多。

图4 特有浮游真核微生物物种在种水平上的韦恩图分析Fig.4 The analysis of venn diagram about endemic eukaryotic plankton species at the species level
3 讨论
3.1 微生物群落组成信息用于溺水地点推断的可行性
真核生物18s rDNA序列信息可用于物种鉴定,因该序列具有多个可变区,在不同物种间变化大,从而被广泛应用于分类学研究[9]。18s rDNA测序可通过提供更广泛的分类覆盖范围来增强对特定水域浮游真核微生物群落的生物学调查,其与形态学方法相比,对于某些群体而言,分辨率更高[10]。法医常通过溺死尸体组织与溺液中浮游微生物(主要是硅藻)种类的一致性分析来推断溺水地点[11],有时也依据溺死尸体随身衣物、鞋袜等检材上的微生物群落作推断[12]。要进行这些分析推断,特定水域的微生物组成背景信息至关重要。
本研究选取的4个取水点的位置分布于珠江广州段的上游至下游,根据α多样性指数发现各取水点内的OTU数及Shannon指数存在一定的差异,OTU数表明上游具有更多的浮游微生物物种数,且Shannon指数表明上游的浮游微生物物种丰度更为均衡。β多样性分析进一步表明各取水点的微生物组成差异显著,尤其是取水点W4与其他取水点的差异最为明显。这可能是因为W4的位置位于珠江的下游,靠近入海口,常年经受海水倒灌,从而形成了与淡水差异显著的微生物组成。W1位于佛山三江源景观点附近,常年接受西南涌、白泥水和流溪河的淡水灌入。W2位于广州闹市区广州塔附近,其前段水域接受了后航道的流道输入。W3位于广州大学城附近,其前段水域接受了大石水道的流道输入。这些水域结构的差异可能是造成各取水点微生物组成差异的主要原因之一,而这也为其用于溺水地点的推断提供了可能性。
3.2 特异微生物种类信息用于溺水地点推断的可行性
在法医工作实践中,溺死尸体中吸入的溺液量及溺液种类差异大,进入肺脏、血液及其他内脏器官的微生物数目差异大[13]。当从检材中分离到的微生物数量较少时,不能满足二代测序对检测样品DNA含量的要求。此外,若样本中混有人或动物DNA与微生物DNA竞争扩增,则还有可能干扰微生物18s rDNA可变区的扩增和检测,这使得要获取溺液中的微生物群落及组成就更加困难。此时,可通过特异微生物种类信息来推断溺水地点,如 在本研究的数据中发现硅藻中的脆杆藻属为W1取水点处特有,若在珠江流域广州段中发现的溺死尸体肺部等脏器中检测到脆杆藻属,则可快速确定溺水地点的大致范围,可为相关案件的侦破赢得时间。然而这需要建立不同水域的数据库,获取特异微生物的种类,并设计特异的扩增引物。本研究结果表明,珠江在不同的取水点有各自特异的微生物,这为后续的特异微生物扩增提供了可行性。此外,在取水点W2和W3中并未找到特异的硅藻属,这似乎显示利用常用的硅藻形态学观察推断溺水地点存在局限性。而细化到微生物物种水平分析中,尽管特异性物种数量偏少,但至少提供了特异性微生物种类用于推断溺水地点的可能性。
3.3 18S rDNA测序用于溺水地点推断的优劣势
目前常用硅藻形态学检测来进行溺死诊断,而其样本处理过程易使硅藻种类丢失和干扰硅藻实际丰度的测量,因此较难获取完整的硅藻种类及其丰度信息。此外,本研究发现有些采水点缺乏特异的硅藻属,使得硅藻形态学检测较难用于溺水地点推断。18S rDNA测序可在不损害样本信息的前提下,便捷、快速、高通量地获取器官组织和溺液中包含硅藻在内的浮游真核微生物在种水平上的丰度构成比例,得到更丰富的物种信息,再进一步与嫌疑溺水地点样本进行匹配比对,可使得溺水地点推断具有可行性。此外,18S rDNA可鉴定出更多的物种,其中的硅藻物种可与形态学检测相互验证。当然,目前缺乏检测水域的18S rDNA数据库,所以仍有大量的基础性工作有待进行。
4 总结
本研究通过对珠江广州段水域随机等距离选取4个采水点进行18S rDNA二代测序,检测浮游真核微生物群落并分析特异微生物物种,为推断潜在的溺水地点提供了一种新方法。在未来的法医实践中,可尝试通过二代测序建立不同水域的微生物数据库,使用生物信息学方法辅助溺水地点的推断。

