一种新型提高HDR效率的CRISPR/Cas9-Gal4BD供体适配基因编辑系统
张潇筠,徐坤,沈俊岑,穆璐,钱泓润,崔婕妤,马宝霞,陈知龙,张智英,魏泽辉
一种新型提高HDR效率的CRISPR/Cas9-Gal4BD供体适配基因编辑系统
张潇筠,徐坤,沈俊岑,穆璐,钱泓润,崔婕妤,马宝霞,陈知龙,张智英,魏泽辉
西北农林科技大学动物科技学院,杨凌 712100
近年来,CRISPR基因编辑及衍生技术迅速发展,在生命科学、生物医学研究以及动植物育种领域得到了广泛应用。基于DNA双链断裂(double-stranded break, DSB)同源指导修复(homology-directed repair, HDR)机制的基因敲入和点编辑是基因编辑的重要策略,但效率偏低亟待提高。本文提出了驱动供体DNA富集至DSB处以提高HDR效率的新策略,并设计了一套CRISPR/Cas9-Gal4BD 供体适配基因编辑系统(donor adapting system, DAS)。该系统主要利用Gal4 DNA结合域(Gal4 binding domain, Gal4BD)作为配体蛋白与Cas9融合表达,将Gal4BD结合序列(Gal4 binding sequence, Gal4BS)作为受体序列与双链DNA (double-stranded DNA, dsDNA)供体结合,以期提高HDR效率。使用HEK293T-HDR.GFP报告细胞系的初步研究结果表明当dsDNA供体同源臂在一定长度(100~60 bp)时该系统能够提高HDR效率2~4倍。进一步的优化研究表明,融合端口和融合使用连接子(linker)的选择会影响Cas9表达效果及活性,而GGS5作为Cas9-Gal4BD融合的连接子则影响较小。同时,本研究还发现Gal4BS-dsDNA供体的差异化设计也会影响HDR效率,将Gal4BS添加到dsDNA供体5′-端的效果最佳。综上所述,本研究利用CRISPR/Cas9-Gal4BD DAS在和位点上实现了HDR编辑效率的提高,为进一步利用该系统进行动物分子设计育种研究提供了参考和借鉴。
CRISPR/Cas9;基因编辑;供体DNA;供体适配;同源指导修复
CRISPR/Cas9基因编辑被称为能够“改变世界”的技术,以其为基础的分子设计育种能够实现动植物性状的快速改良,被广泛应用到了动植物育种研究中[1~3]。CRISPR/Cas9系统通过单链引导RNA (single guide RNA, sgRNA)在特定的靶位点诱导DNA双链断裂(double-stranded break, DSB),进而利用细胞自身的DSB修复机制实现基因编辑[4,5]。细胞主要通过非同源末端连接(non-homologous end joining, NHEJ)和同源重组(homologous recombination, HR)两种修复机制进行DSB的修复。其中,HR包括在基因组编辑中广泛应用的同源指导修复(homology- directed repair, HDR)和相对小众的单链退火(single- stranded annealing, SSA)[6]。
在哺乳动物细胞中,DSB绝大部分通过NHEJ机制进行修复,相关修复蛋白直接将DNA断裂末端拉近,通过连接酶进行重新连接。在此过程中通常会导致断裂末端核苷酸的丢失或插入,进而引起基因功能的丧失。HR修复发生的概率相对较低,当DNA双链断裂后,MRN复合物(包括MRE11、Rad50、Nbs1三种蛋白质)结合到DSBs,引起核酸内切酶(CtIP)对DSBs末端进行切除,导致长3′单链DNA片段被复制蛋白A (replication protein A, RPA)包裹。随后,Rad51取代了RPA与ssDNA结合,形成核蛋白突触前丝,促进寻找同源供体。待异源双链DNA结构形成后,Rad51分解,同时伴随着DNA合成和最后的连接步骤。
在使用CRISPR/Cas9进行相关研究时,基于NHEJ修复的基因编辑通常被用于目标基因的移码敲除,而HDR机制常被用于基因精确编辑(精确的点突变、小片段的插入或缺失等)、置换和敲入等。但是,HDR机制依赖于供体DNA的重组效应,普遍存在着效率低下的缺点[7]。近年来,研究者们开发了一系列的提高HDR效率的策略,包括抑制NHEJ通路、增强HDR机制、优化供体形式和控制打靶时效等(表1)。
Ruff等[8]首次提出了将供体DNA靶向募集到DSB附近以提高HDR效率的策略。该研究直接将I-I核酸酶作为配体蛋白,筛选出具有强结合活性的受体DNA元件(I-I适配子)。通过在单链DNA (single-stranded DNA, ssDNA)短供体(小于100 nt)一端引入适配子序列,成功在酵母和人类细胞中将HDR效率提高了32倍和16倍。受此策略的启发,多个类似的CRISPR/Cas9基因编辑衍生系统相继被开发[9~16]。为了便于描述,本文提出了供体适配基因编辑系统(donor adapting system, DAS)的概念,特指驱动供体DNA富集至DSB处以提高HDR效率的CRISPR/Cas9基因编辑衍生系统。
Gal4转录因子是半乳糖诱导基因表达的正调节因子,由DNA结合域(binding domain, BD)和激活域(activating domain, AD)两个功能域组成。两个结构域可以独立表达并行使功能,已被成功应用于商业化的酵母双杂交系统(yeast two-hybrid system, Y2H)。Gal4BD位于Gal4 N-末端,是一个属于Zn(2)-C6家族的锌指结构,能够特异性识别Gal1启动子中的上游激活序列(upstream activation sequence, UAS)[43]。野生型UAS由几个具有高度同源性的结合序列(binding sequence, BS)组成。研究人员发现单个结合序列是约为20 bp的保守序列,两端均有保守的GC碱基[44]。
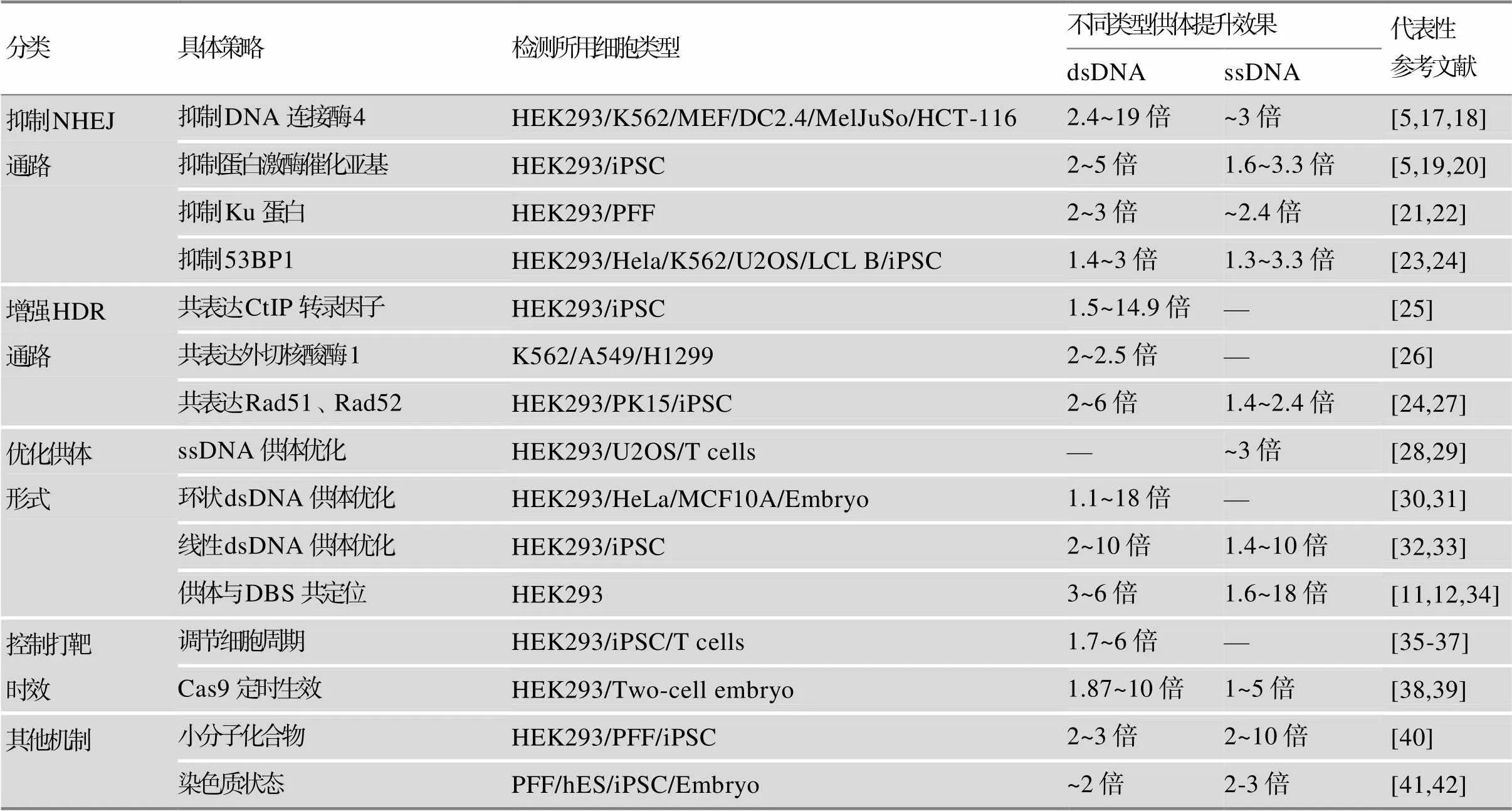
表1 不同HDR效率提高策略的比较
本研究基于课题组在CRISPR/Cas9和Y2H方面的研究基础提出了一种新型的CRISPR/Cas9-Gal4BD DAS基因编辑系统,通过将Cas9蛋白与酵母源的Gal4 BD[45]融合表达以提高HDR效率。
1 材料与方法
1.1 载体构建
采用pLL3.7作为载体骨架,分别用U6和CMV启动子起始CRISPR/Cas9系统sgRNA和Cas9的表达。通过PCR,从Y2H载体pGBKT7 (美国Clontech公司)上扩增获得Gal4BD及C-端28 aa链接的DNA表达序列,克隆至pLL3.7-U6/sgRNA-CMV-Cas9中基因的上游(N-端),构建获得N-ter Gal4BD Cas9融合蛋白表达载体;将课题组研究常用的针对VEGF基因相关靶位点(CTCGGCCACCACAGGGAAGCPAM)的sgRNA克隆至该载体中U6启动子下游,构建sgVEGF/N-ter Gal4BD Cas9表达载体,初步建立CRISPR/Cas9-Gal4BD DAS (图1A)。
为了进一步优化该系统,采用两类不同的柔性linker,即不同长度的GGSn (n=1/3/5/7,为GGS氨基酸串联重复的个数)和全长及截短的XTEN (XTEN1- SGSETPGTSESATPES和XTEN2-SESATPES),将基因克隆至基因的下游(C-端),进而构建获得相应的C-ter Gal4BD Cas9融合蛋白表达载体,同样采用sgVEGF作为后续功能验证实验的sgRNA构建相应的CRISPR/Cas9-Gal4BD DAS表达载体。
本研究中所使用的pSSA.GFP.VEGF和pHDR.GFP.VEGF荧光报告载体分别为课题组前期研究所构建[27,46]。两个报告载体中的报告基因均被插入的VEGF靶序列打断,经过CRISPR/Cas9系统打靶造成DSB后,分别通过SSA和HDR机制进行修复,进而可以通过荧光细胞流式计数评估Cas9活性和HDR效率。
1.2 细胞培养和转染
采用HEK293T细胞进行相关细胞实验检测。细胞培养条件均为:90% DMEM培养基,10% FBS,100 μg/mL的青链霉素,5% CO2(均为体积分数),温度37℃。采用上海YEASEN公司的Hieff Trans™脂质体核酸转染试剂根据说明书步骤进行转染实验:转染前1天将细胞接种于12孔板中,在细胞密度达到80%~90%后进行转染,每孔转染2 μg的质粒,每组最少设置3个平行转染孔。
1.3 报告细胞系的构建及检测
课题组前期通过转座系统将HDR.GFP报告基因表达盒随机整合至HEK293T基因组中构建了相应的HEK293T-HDR.GFP报告细胞系[46]。本研究利用该报告细胞系初步验证CRISPR/Gal4BD- Cas9 DAS的可行性,通过PCR在不同长度的GFP dsDNA供体5′-端添加长度为20 bp的Gal4BD结合序列(binding sequence, BS; 5′-TCCGGAGGACTGTCCTCCGG-3′) (图1B)。对照组供体添加同样长度的无关序列(irrelevant sequence, IS; 5′-TTCAGACGAGATAGTCTGAG-3′)。将上述构建的sgVEGF/ N-ter Gal4BD Cas9表达载体与经5′-端改造的GFP dsDNA供体以质量比1∶1转染HEK293T-HDR.GFP报告细胞系,转染48 h后使用BD FACS Aria III流式细胞仪对GFP阳性(GFP+)细胞进行计数以评估HDR效率。
1.4 SSA报告实验检测Cas9活性
采用SSA报告实验比较不同Cas9-Gal4BD融合表达方案对Cas9活性的影响。其中,pSSA.GFP.VEGF报告载体中的报告基因被CRISPR/Cas9系统打靶后无需供体DNA即可通过SSA机制直接修复(图2A)。以1∶1的质量比或摩尔比,将sgVEGF/Cas9表达载体与pSSA.GFP.VEGF报告载体共转染HEK293T细胞进行预实验,初步结果表明1∶1的质量比效果最佳。进而以质量比1∶1将不同融合表达方案(图2, B和D)的sgVEGF/Cas9-Gal4BD表达载体与pSSA.GFP.VEGF报告载体共转染HEK293T细胞,转染48 h后,使用美国BD公司FACS Aria III流式细胞仪对GFP阳性(GFP+)细胞进行计数以评估Cas9活性。同时收集每个转染组的细胞提取总蛋白,使用抗-Gal4BD (美国Abbkine公司,ABP57232)和抗-Cas9 (英国Abcam公司,ab191468)抗体分别进行免疫印迹(Western blot, WB)实验检测不同Cas9- Gal4BD融合蛋白的表达情况。
1.5 HDR报告实验验证HDR效率
为了获得最佳的BS-dsDNA供体设计,使用HDR报告实验对不同BS-dsDNA供体介导的HDR效率进行评估。其中,pHDR.GFP.VEGF报告载体中报告基因被CRISPR/Cas9系统打靶后仅能通过供体DNA依赖的HDR机制实现修复(图3A)。以1∶1∶1的质量比或摩尔比,将sgVEGF/Cas9表达载体、pHDR.GFP.VEGF报告载体和长度为700 bp的GFP dsDNA供体共转染HEK293T细胞进行预实验,初步结果表明1∶1∶1的质量比效果最佳。根据相关报道,Gal4BD结合序列的一般结构是5′- CGG-N11-CCG-3'[44,47,48],进一步选择长度为17 bp的短BS序列[49]进行BS-dsDNA供体的设计(图3,B和D)。以质量比1∶1∶1将sgVEGF/Cas9-GGS5- Gal4BD载体、pHDR.GFP.VEGF报告载体和通过PCR扩增获得的不同BS-dsDNA供体共转染HEK293T细胞。转染48 h后,使用美国BD公司FACS Aria III流式细胞仪对GFP阳性(GFP+)细胞进行计数以评估HDR效率。
1.6 基因组编辑
使用优化后Cas9-Gal4BD和BS-dsDNA组成的CRISPR/Cas9-Gal4BD DAS对HEK293T细胞三个基因组位点(、、)进行点编辑。将sgVEGF/Cas9-GGS5-Gal4BD载体中的sgVEGF替换为sgAAVS1、sgEMX1和sgNUDT5,构建相应的CRISPR/Cas9-Gal4BD DAS载体。以课题组前期构建的质粒供体[50]为模板,通过PCR引入5′-BS获得相应的BS-dsDNA供体。供体左右同源臂长度均设计为1000 bp左右,且序列中sgRNA靶点PAM均突变为限制性内切酶(RE)位点(图4A),以便通过限制性片段长度多态性(restriction fragment length polymorphism, RFLP)分析评估HDR编辑效率。
以质量比1∶1∶1将CRISPR/Cas9-Gal4BD DAS载体、相应的BS-dsDNA供体和HDR通用型报告筛选载体(HDR-universal surrogate reporter, HDR- USR)共转染HEK293T细胞。HDR-USR为课题组前期开发的HDR编辑阳性细胞辅助筛选报告载体[50]。转染后24 h,用含有嘌呤霉素的培养基筛选3天,更换正常培养基继续培养2天,收集各组细胞,提取基因组DNA,PCR扩增目的基因片段并进行限制性内切酶消化实验及RFLP分析。其中,一条PCR检测引物设计在dsDNA供体模版之外,PCR产物及酶切后片段大小(bp)为:, 1274=1087+187;, 2147=1087+1060;, 2186=1105+1081。通过ImageJ软件对消化和未消化的DNA条带进行灰度对比分析,进而评估目标基因位点的HDR编辑效率。
1.7 数据统计与分析
所有实验均至少设置三个平行重复或独立重复,数据以“平均值±SD”表示,使用检验进行显著性检验。*:<0.05为差异显著,**:<0.01为差异极显著。
2 结果与分析
2.1 报告细胞系初步验证CRISPR/Cas9-Gal4BD DAS功能
本研究最初将Gal4BD与Cas9 N-端进行融合,初步建立了CRISPR/Cas9-Gal4BD DAS 基因编辑系统(图1A),并尝试利用课题组前期研究构建的HEK293T-HDR.GFP报告细胞系进行功能验证。该报告细胞系中整合的报告基因修复原理如图1B所示。其中,Gal4BD结合序列BS可以通过PCR技术直接添加到dsDNA供体的5'-端,IS为无关对照序列。本实验采用了长度分别为215 bp、100 bp和60 bp的GFP dsDNA供体,初步检测结果表明,当供体DNA在一定长度内时,HDR编辑效率可提高2~4倍(图1C)。但CRISPR/Cas9-Gal4BD DAS能否有效提高长dsDNA供体的HDR效率尚有待商榷。
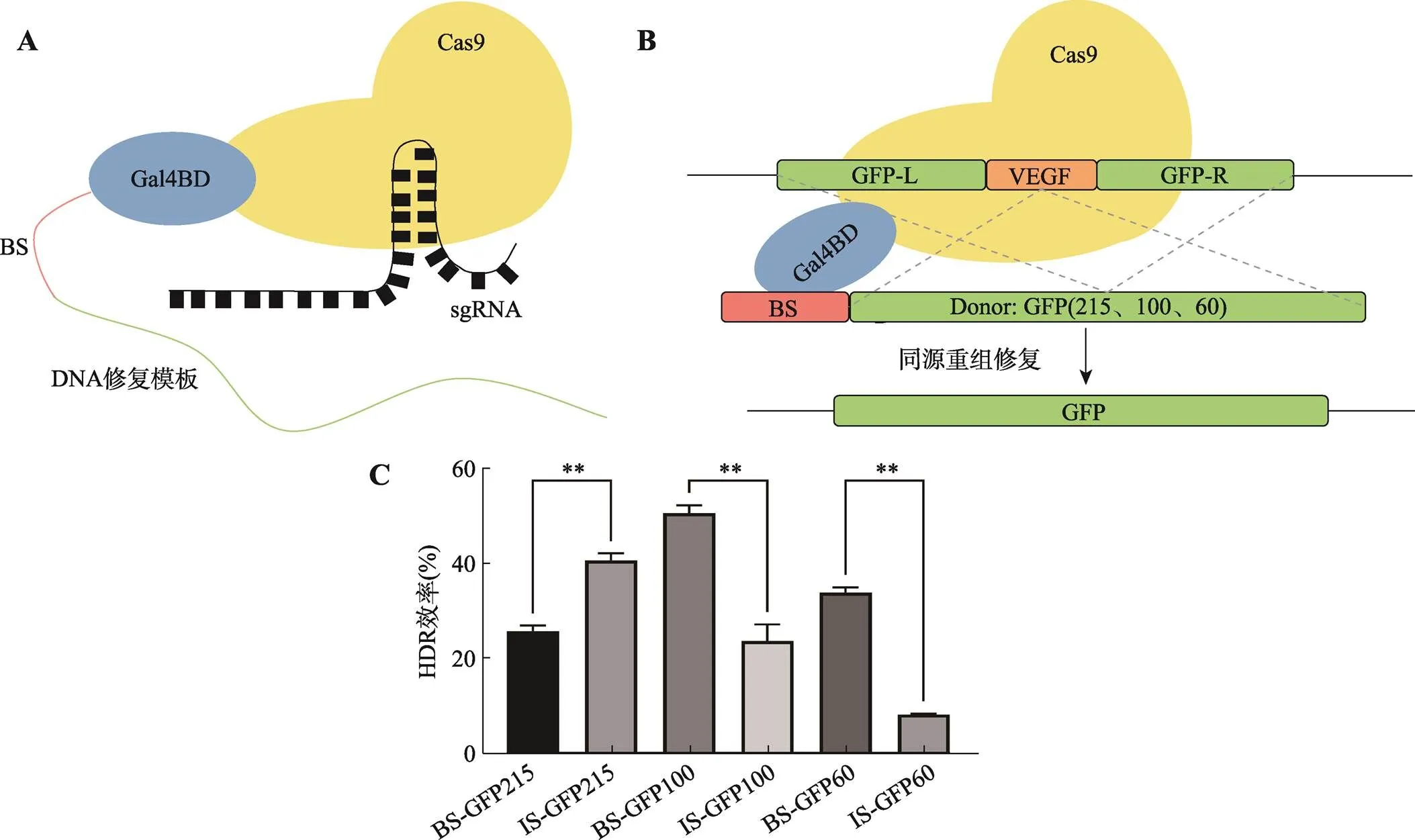
图1 CRISPR/Cas9-Gal4BD DAS及HEK293T-HDR.GFP报告细胞系验证结果
A:Cas9-Gal4BD融合蛋白驱动BS-dsDNA供体示意图;B:CRISPR/Cas9-Gal4BD DAS介导报告细胞系基因HDR示意图;C:CRISPR/Gal4BD-Cas9 DAS介导的不同GFP dsDNA供体HDR效率比较。BS/IS-GFP215/100/60分别代表5’-端连接BS或IS、长度为215/100/60 bp的GFP dsDNA供体;BS为Gal4BD结合序列,IS为无关对照序列。数据以平均值±SD表示,=3,**:<0.01。
2.2 SSA报告实验优化Cas9-Gal4BD融合方式
鉴于Gal4BD与Cas9 N-端融合(N-ter Gal4BD)可能会严重影响Cas9的活性,进一步将Gal4BD与Cas9 C-端进行融合(C-ter Gal4BD),并采用了多个不同的linker,构建了优化的CRISPR/Cas9-Gal4BD DAS。利用课题组前期开发的SSA报告系统(图2A)进行Cas9活性的验证,结果表明C-ter Gal4BD保留了与野生型(WT)Cas9相近的活性,而N-ter Gal4BD活性显著降低(图2,B和C);不同linker连接的C-ter Gal4BD对Cas9活性造成了不同程度的影响(图2,D和E)。进一步采用抗-Gal4BD和抗-Cas9抗体对Cas9-Gal4BD融合蛋白的表达情况进行WB检测,结果表明除了GGS5和XTEN1,其余linker连接的C-ter Gal4BD均对Cas9表达造成了显著影响(图2,F和G)。结合SSA报告实验结果及Cas9、Gal4BD蛋白表达检测情况,推测采用GGS5 (5个GGS重复) linker的C-ter Gal4BD融合表达策略是相对较优的选择。
2.3 HDR报告实验优化BS-dsDNA供体设计
作为CRISPR/Cas9-Gal4BD DAS重要组成部分,BS-dsDNA供体的设计也是提高HDR效率的关键。为了探讨最佳的BS-dsDNA供体设计,采用课题组前期开发的HDR报告系统(图3A)进行不同BS-dsDNA供体的HDR效率验证。鉴于Gal4 BS的一般结构为5′-CGG-N11-CCG-3′,改用长度为17 bp的短BS序列,并采用不含启动子的全长序列(约700bp)作为供体模板。首先,将不同数量的BS添加到dsDNA供体的5′-端,HDR报告实验结果表明增加BS并不能提高HDR效率,反而单个BS (1×BS)可能是最好的选择(图3,B和C)。其次,将BS序列添加到供体模板的5′-或/和3′-端(图3D),结果表明将BS添加到供体DNA 5′-端的效果较好(图3E)。
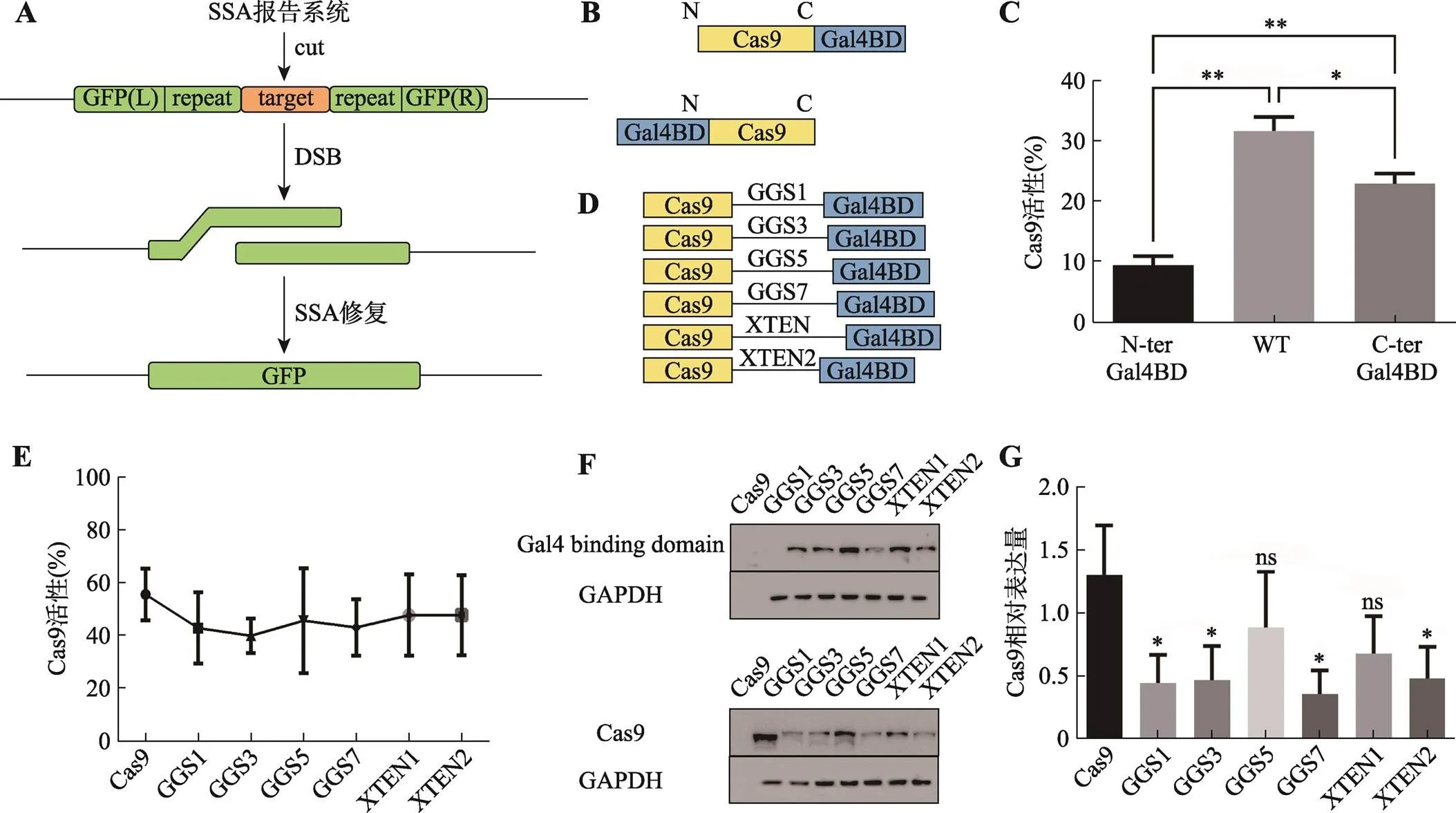
图2 SSA报告实验验证不同Cas9-Gal4BD融合蛋白活性的结果
A:pSSA.GFP报告载体修复原理示意图;B,D:Cas9 N-端和C-端融合Gal4BD (N-ter Gal4BD, C-ter Gal4BD)及linker示意图;C:N-ter Gal4BD和C-ter Gal4BD对Cas9活性影响的检测结果;E:不同linker链接的C-ter Gal4BD对Cas9活性影响的检测结果;F:使用抗-Gal4BD (上)和抗-Cas9 (下)抗体的WB检测结果;G:Cas9蛋白表达水平WB检测结果的灰度分析。数据以平均值±SD表示,n=3~5,*:<0.05;**:<0.01。
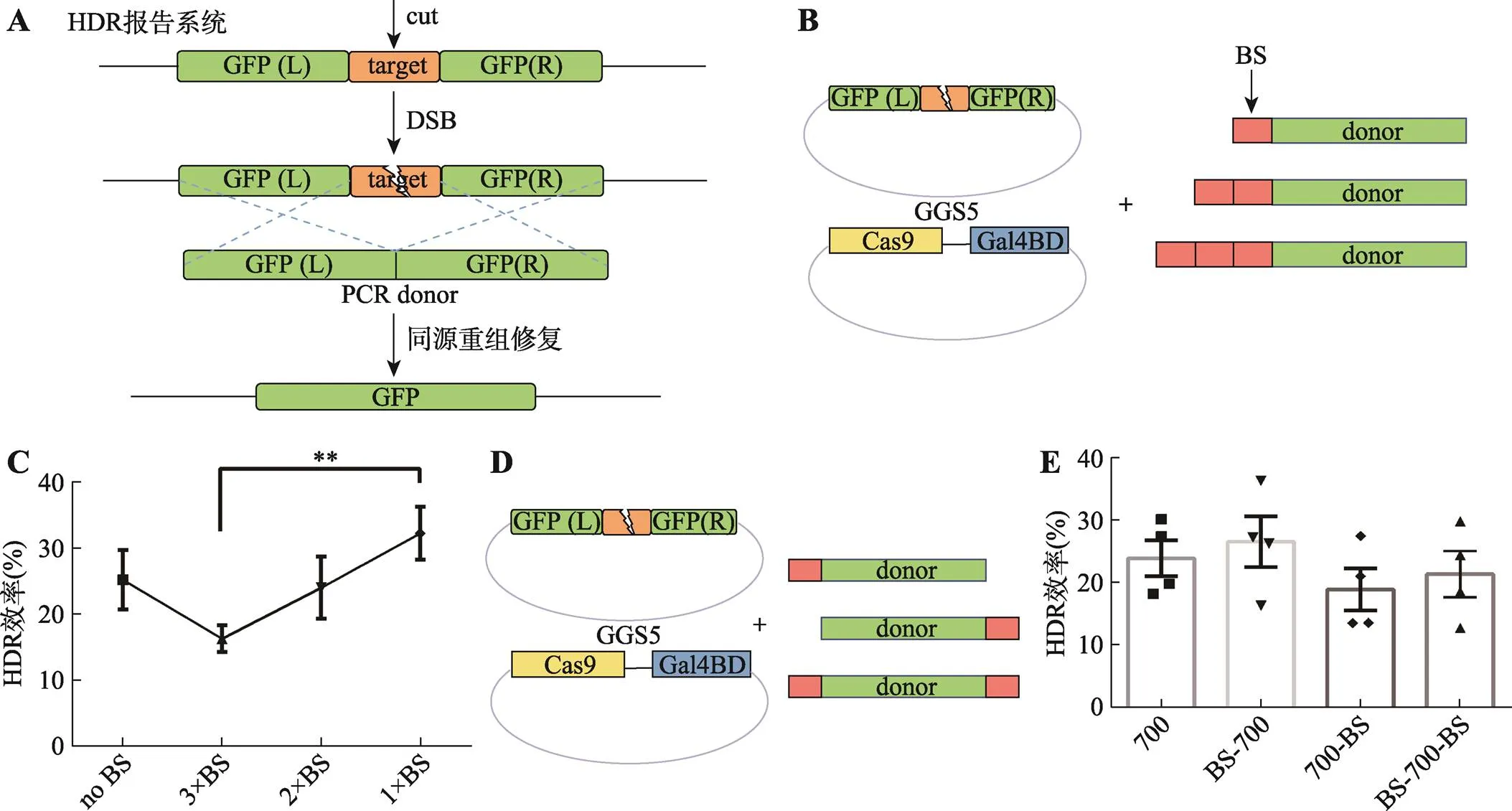
图3 HDR报告实验验证不同BS-dsDNA供体HDR效率的结果
A:pHDR.GFP报告载体修复原理示意图;B:5′-端添加不同数目BS的dsDNA供体及HDR报告实验设计示意图;C:5′-端添加不同数目BS的dsDNA供体HDR效率检测结果;D:5′-端或/和3′-端添加BS的dsDNA供体及HDR报告实验设计示意图;E:5′-端或/和3′-端添加BS的dsDNA供体HDR效率检测结果。数据以平均值±SD表示,=3~5,**:<0.01。
2.4 CRISPR/Cas9-Gal4BD DAS介导的基因组编辑
使用优化后Cas9-Gal4BD和BS-dsDNA组成的CRISPR/Cas9-Gal4BD DAS对HEK293T细胞的三个基因组位点(、、)进行点编辑。将BS-dsDNA供体中sgRNA靶序列的PAMs突变为RE位点防止供体DNA被打靶,同时方便后续通过RFLP实验进行HDR效率检测(图4A)。结果表明和位点的HDR编辑效率显著提高了约20%,但是基因位点则与对照组相比无显著差异(图4,B和C)。据此推测,HDR编辑效率的提高具有位点依赖性,可能与基因的结构和活跃情况有关。
3 讨论
为了提高供体DNA的HDR效率,本研究开发了新型的CRISPR/Cas9-Gal4BD供体适配基因编辑系统。优化结果表明Cas9-Gal4BD C-端融合效果较好,这可能是因为RuvC-1位于Cas9 N-端且N-端的完整性对Cas9的核酸酶活性至关重要[11]。为了降低Gal4BD融合对Cas9活性的影响,本研究采用了两类不同的柔性linker,即不同长度的GGSn和全长及截短的XTEN。虽然最终确认了相对较优的GGS5和XTEN1,但不同linker链接的Cas9-Gal4BD融合均对Cas9的表达及活性均造成了一定程度的影响。这可能与Cas9蛋白较大、空间结构折叠容易受到影响有关。在后续相关研究中可以尝试比较更多类型及不同长度的linker以降低融合蛋白对Cas9活性的影响。在BS-dsDNA供体的优化过程中,本研究发现单个BS序列添加在dsDNA 5′-端的效果较好,推测dsDNA供体的3′-游离端有助于同源重组,非同源BS的额外添加反而不利于重组,但该假设仍有待进一步探讨。
此外,课题组也注意到了预实验中HKE293T- HDR.GFP报告细胞系的验证结果与后续优化过程中的HDR报告载体实验及基因组编辑结果并不呼应,这可能与报告细胞系的质量、供体长度改变、融合linker优化以及后续检测手段不同等因素有关。在报告细胞系验证实验中,dsDNA供体长度为100 bp和60 bp时的HDR效率有所提高。推测修复模版较短时,可能存在效率较HDR高的微同源末端链接(microhomology-mediated end joining, MMEJ)修复,故提升效果明显。但CRISPR/Cas9-Gal4BD DAS能否有效提高长dsDNA供体的HDR效率尚有待商榷。由于HKE293T-HDR.GFP报告细胞系是一个转基因细胞混池,基因型背景不清晰,存在着一定的不确定因素,课题组后续放弃了使用该细胞系进行优化研究。另鉴于较短的dsDNA供体不能介导基因敲入,其应用前景有限,且与ssDNA供体相比HDR效率相对较低,后续优化研究及内源基因编辑均采用了长dsDNA供体,以期应用于基因大片段敲入研究。在进一步的内源基因编辑实验中,尽管利用CRISPR/Cas9-Gal4BD DAS在和位点实现了编辑效率的提高,但仍需在更多类型的细胞中针对更多基因组位点进行适用性验证。另外,通过药物瞬时筛选富集基因点编辑细胞后进行RFLP分析的检验方法具有一定的局限性,受细胞筛选、基因组提取、PCR及酶切等过程的影响,存在着一定的干扰因素。鉴于CRISPR/Cas9-Gal4BD DAS所采用的dsDNA供体主要优势在于基因大片段敲入,后续研究可采用荧光或抗性基因敲入的策略并优化下游检测手段,更严谨地论证该系统的可行性。
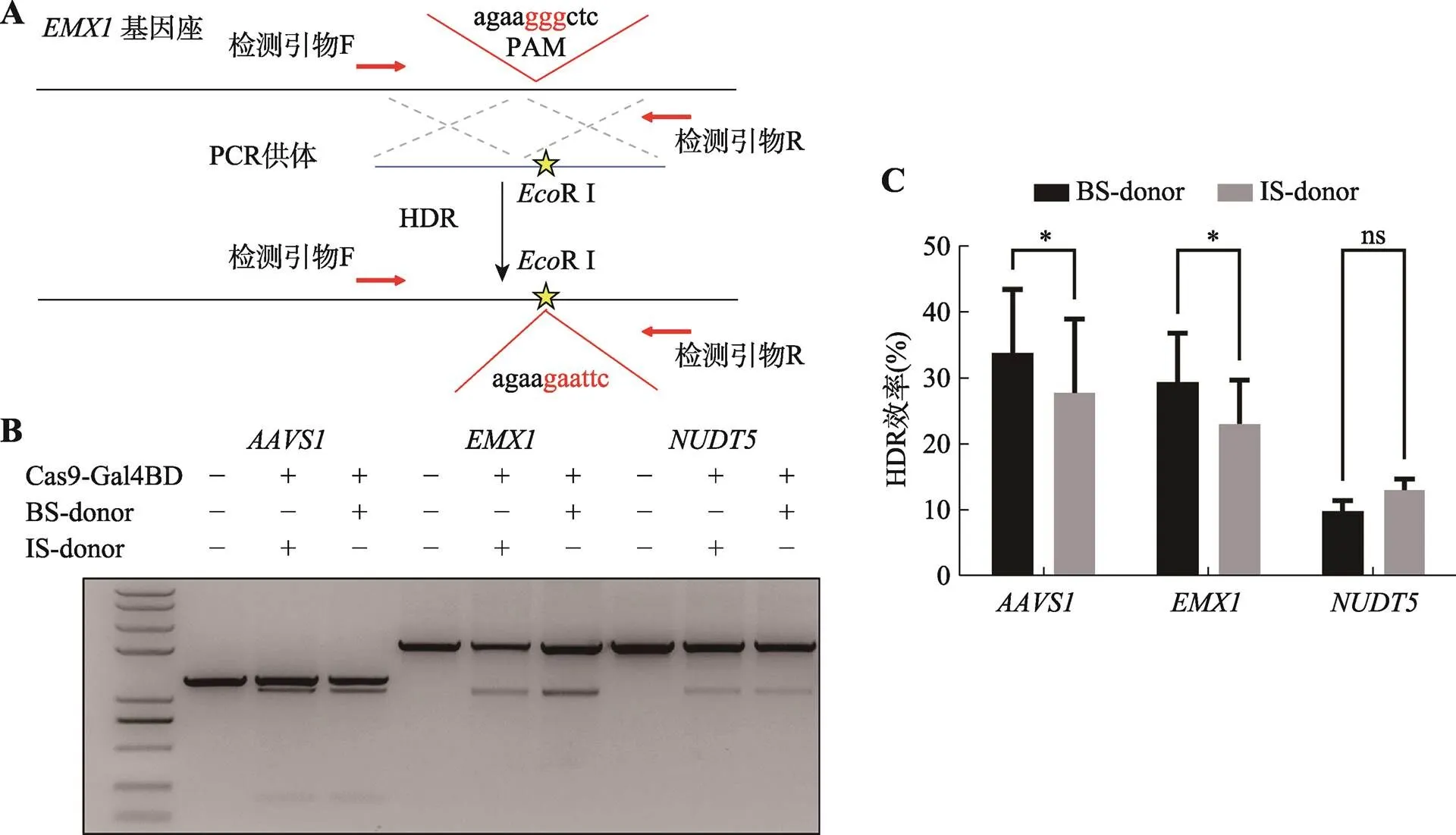
图4 CRISPR/Cas9-Gal4BD DAS介导的基因精确编辑
A:以基因座为例的基因编辑设计及检测示意图,其中一条PCR检测引物设计在dsDNA供体模版之外;B:RFLP实验检测HDR编辑效率的琼脂糖凝胶电泳结果,PCR产物及酶切后片段大小(bp)为:,1274=1087+187;,2147=1087+1060;,2186=1105+1081;C:基于RFLP实验DNA条带灰度分析的HDR编辑效率检测结果。数据以平均值±SD表示,=3,*:<0.05。
在本研究开展前后,多种以相似设计理念改进的CRISPR/Cas9 DAS被相继报道(表2) 。其中,Cas9-SNAP/O6-BG-ssDNA系统以SNAP-Tag作为适配配体,以O6-苄基鸟嘌呤(BG)作为“受体”修饰ssDNA供体[9]。Cas9-Avidin/Biotin-dsDNA系统[10]将与Cas9蛋白融合的亲和素作为适配配体,同时将生物素作为适配“受体”与PCR获得的dsDNA供体进行化学偶联。Cas9-Avidin/Biotin-ssDNA系统[11]与Cas9-Avidin/Biotin-dsDNA系统类似,但采用了长ssDNA供体(~1000 nt)。另一个sgRNA-S1m.Avidin/ Biotin-ssDNA系统[12]采用了新颖的sgRNA导向策略,该系统将亲和素特异性结合序列(S1mRNA)与sgRNA融合,作为驱动链霉亲和素/生物素-ssDNA供体的接头部分。该系统增加了供体和sgRNA设计的复杂程度,且ssDNA供体需要与生物素–链霉亲和素偶联,理论上并不实用。ssDNA供体与dsDNA供体介导的DNA修复机制有所不同,前者具有重组效率高、操作简单方便等优点,但是长ssDNA供体制备相对困难、价格昂贵、不稳定且容易降解。因此,基因编辑研究中多使用长度小于120 nt的短ssDNA供体进行点编辑,而大片段基因敲入研究仍受限于相对偏低的dsDNA供体重组效率。
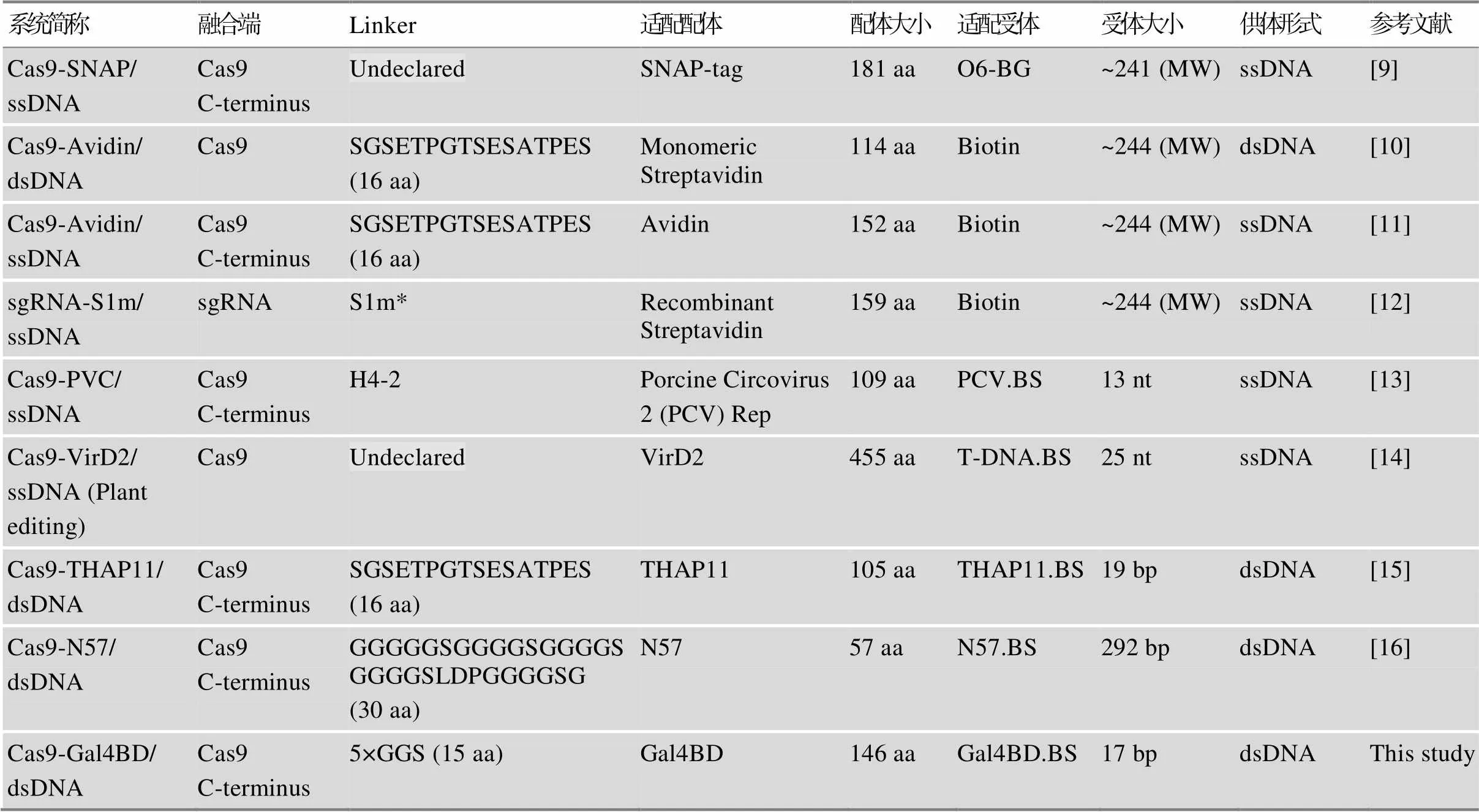
表2 目前已报道的CRISPR/Cas9 DAS系统
Linker,一段短肽链用于连接Cas9蛋白和适配器。S1m*,一段与sgRNA连接的用于连接链霉素亲和素的RNA序列。MW,分子量。
与本研究思路类似,在最新的报道中不同的dsDNA结合蛋白也被用于开发新型CRISPR/Cas9 DAS。对于这些DAS,通常可以通过PCR便捷地获得BS-dsDNA供体,并且可以通过重组DNA技术以质粒的形式组装大型供体进而介导复杂的基因编辑。在Cas9-THAP11/THAP11.BS-dsDNA系统中,将灭活的转录因子THAP11与Cas9 C-端融合[15],可以提高HDR敲入效率2倍。Cas9-N57/N57.BS-dsDNA系统[16]采用了来自睡美人转座子SB100X的N57 DNA结合域,该系统具有上述相似的供体适配特征,但使用了非同源依赖靶向整合(homology-in-dependent targeted integration,HITI)供体[51],可以在分裂和非分裂细胞中提高靶向敲入效率。
最后,值得注意的是灭活的转录因子THAP11、转座酶SB100X的N57 DNA结合域和本研究使用的酵母源Gal4BD长度分别为105 aa、57 aa和146 aa,均远小于Cas9蛋白(1368 aa)。此外,Cas9-N57系统采用了HITI供体进行非同源依赖靶向基因敲入,表明CRISPR/Cas9 DASs能够同时适用于HDR和HITI介导的基因编辑。在BS-dsDNA供体方面,Cas9- Gal4BD系统和Cas9-THAP11系统具有更短BS (17 bp和19 bp)的相对优势。
综上所述,本研究开发了一种新型提高HDR效率的CRISPR/Cas9-Gal4BD供体适配基因编辑系统,并在Cas9-Gal4BD融合表达和BS-dsDNA供体设计方面进行了优化。最终利用优化后的CRISPR/ Gal4BD-Cas9 DAS实现了对和位点的HDR增强编辑,为进一步利用该系统进行动物分子设计育种研究提供了参考和借鉴。
[1] Zhou SW, Yu HH, Zhao XE, Cai B, Ding Q, Huang Y, Li YX, Li Y, Niu YY, Lei AM, Kou QF, Huang XX, Petersen B, Ma BH, Chen YL, Wang XL. Generation of gene-edited sheep with a defined Booroola fecundity gene (FecB ) mutation in bone morphogenetic protein receptor type 1B (BMPR1B) via clustered regularly interspaced short palindromic repeat (CRISPR)/CRISPR-associated (Cas) 9., 2018, 30(12): 1616–1621.
[2] Niu YY, Zhao XE, Zhou JK, Li Y, Huang Y, Cai B, Liu YT, Ding Q, Zhou SW, Zhao J, Zhou GX, Ma BH, Huang XX, Wang XL, Chen YL. Efficient generation of goats with defined point mutation (I397V) in GDF9 through CRISPR/Cas9., 2018, 30(2): 307–312.
[3] Park KE, Kaucher AV, Powell A, Waqas MS, Sandmaier SES, Oatley MJ, Park CH, Tibary A, Donovan DM, Blomberg LA, Lillico SG, Whitelaw CBA, Mileham A, Telugu BP, Oatley JM. Generation of germline ablated male pigs by CRISPR/Cas9 editing of the NANOS2 gene., 2017, 7: 40176.
[4] Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering., 2014, 157(6): 1262–1278.
[5] Chu VT, Weber T, Wefers B, Wurst W, Sander S, Rajewsky K, Kühn R. Increasing the efficiency of homology-directed repair for CRISPR-Cas9-induced precise gene editing in mammalian cells., 2015, 33(5): 543–548.
[6] Decottignies A. Alternative end-joining mechanisms: a historical perspective., 2013, 4: 48.
[7] Vasquez KM, Marburger, K, Intody Z, Wilson JH. Manipulating the mammalian genome by homologous recombination., 2001, 98(15): 8403–10.
[8] Ruff P, Koh KD, Keskin H, Pai RB, Storici F. Aptamer-guided gene targeting in yeast and human cells., 2014, 42(7): e61.
[9] Savic N, Ringnalda FC, Lindsay H, Berk C, Bargsten K, Li YZ, Neri D, Robinson MD, Ciaudo C, Hall J, Jinek M, Schwank G. Covalent linkage of the DNA repair template to the CRISPR-Cas9 nuclease enhances homology- directed repair., 2018, 7: e33761.
[10] Gu B, Posfai E, Rossant J. Efficient generation of targeted large insertions by microinjection into two-cell-stage mouse embryos., 2018, 36(7): 632–637.
[11] Ma M, Zhuang FF, Hu XB, Wang BL, Wen XZ, Ji JF, Xi JZJ. Efficient generation of mice carrying homozygous double-floxp alleles using the Cas9-Avidin/Biotin-donor DNA system., 2017, 27(4): 578–581.
[12] Carlson-Stevermer J, Abdeen AA, Kohlenberg L, Goedland M, Molugu K, Lou M, Saha K. Assembly of CRISPR ribonucleoproteins with biotinylated oligonucleotides via an RNA aptamer for precise gene editing., 2017, 8(1): 1711.
[13] Aird EJ, Lovendahl KN, Martin AS, Harris RS, Gordon WR. Increasing Cas9-mediated homology-directed repair efficiency through covalent tethering of DNA repair template., 2018, 1: 54.
[14] Ali Z, Shami A, Sedeek K, Kamel R, Alhabsi A, Tehseen M, Hassan N, Butt H, Kababji A, Hamdan SM, Mahfouz MM. Fusion of the Cas9 endonuclease and the VirD2 relaxase facilitates homology-directed repair for precise genome engineering in rice., 2020, 3(1): 44.
[15] Li GL, Wang HQ, Zhang XW, Wu ZF, Yang HQ. A Cas9- transcription factor fusion protein enhances homology- directed repair efficiency., 2021, 296: 100525.
[16] Ma SF, Wang XL, Hu YF, Lv J, Liu CF, Liao KT, Guo XH, Wang D, Lin Y, Rong ZL. Enhancing site-specific DNA integration by a Cas9 nuclease fused with a DNA donor-binding domain., 2020, 48(18): 10590–10601.
[17] Maruyama T, Dougan SK, Truttmann MC, Bilate AM, Ingram JR, Ploegh HL. Increasing the efficiency of precise genome editing with CRISPR-Cas9 by inhibition of nonhomologous end joining., 2015, 33(5): 538–542.
[18] Hu Z, Shi Z, Guo X, Jiang B, Wang G, Luo D, Chen Y, Zhu YS. Ligase IV inhibitor SCR7 enhances gene editing directed by CRISPR-Cas9 and ssODN in human cancer cells.. 2018 19(8):12.
[19] Riesenberg S, Maricic T. Targeting repair pathways with small molecules increases precise genome editing in pluripotent stem cells., 2018, 9(1): 2164.
[20] Riesenberg S, Chintalapati M, Macak D, Kanis P, Maricic T, Pääbo S. Simultaneous precise editing of multiple genes in human cells.. 2019 47(19):e116.
[21] Robert F, Barbeau M, Éthier S, Dostie J, Pelletier J. Pharmacological inhibition of DNA-PK stimulates Cas9-mediated genome editing., 2015, 7(1): 93.
[22] Li G, Liu D, Zhang X, Quan R, Zhong C, Mo J, Huang Y, Wang H, Ruan X, Xu Z, Zheng E, Gu T, Hong L, Li Z, Wu Z, Yang H. Suppressing Ku70/Ku80 expression elevates homology-directed repair efficiency in primary fibroblasts., 2018, 99: 154–160.
[23] Nambiar TS, Billon P, Diedenhofen G, Hayward SB, Taglialatela A, Cai KH, Huang JW, Leuzzi G, Cuella-Martin R, Palacios A, Gupta A, Egli D, Ciccia A. Stimulation of CRISPR-mediated homology-directed repair by an engineered RAD18 variant., 2019, 10(1): 3395.
[24] Paulsen BS, Mandal PK, Frock RL, Boyraz B, Yadav R, Upadhyayula S, Gutierrez-Martinez P, Ebina W, Fasth A, Kirchhausen T, Talkowski ME, Agarwal S, Alt FW, Rossi DJ. Ectopic expression of RAD52 and dn53BP1 improves homology-directed repair during CRISPR-Cas9 genome editing., 2017, 1(11): 878–888.
[25] Tran NT, Bashir S, Li X, Rossius J, Chu VT, Rajewsky K, Kühn R. Enhancement of precise gene editing by the association of Cas9 with homologous recombination Factors., 2019, 10: 365.
[26] Hackley CR. A novel set of Cas9 fusion proteins to stimulate homologous recombination: Cas9-HRs., 2021, 4(2): 253–263.
[27] Shao SM, Ren CH, Liu ZT, Bai YC, Chen ZL, Wei ZH, Wang X, Zhang ZY, Xu K. Enhancing CRISPR/Cas9- mediated homology-directed repair in mammalian cells by expressing Saccharomyces cerevisiae Rad52., 2017, 92: 43–52.
[28] Richardson CD, Ray GJ, DeWitt MA, Curie GL, Corn JE. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA., 2016, 34(3): 339–344.
[29] Shy BR, Vykunta V, Ha A, Roth TL, Talbot A, Nguyen DN, Chen YY, Blaeschke F, Vedova S, Mamedov MR, Chung JY, Li H, Wolf J, Martin TG, Ye LM, Eyquem J, Esensten JH, Marson A. Hybrid ssDNA repair templates enable high yield genome engineering in primary cells for disease modeling and cell therapy manufacturing., 2021, doi:10.1101/2021.09.02.458799.
[30] Cruz-Becerra G, Kadonaga JT. Enhancement of homology-directed repair with chromatin donor templates in cells., 2020, 9: e55780.
[31] Hirotsune S, Kiyonari H, Jin MY, Kumamoto K, Yoshida K, Shinohara M, Watanabe H, Wynshaw-Boris A, Matsuzaki F. Enhanced homologous recombination by the modulation of targeting vector ends., 2020, 10(1): 2518.
[32] Liang X, Potter J, Kumar S, Ravinder N, Chesnut JD. Enhanced CRISPR/Cas9-mediated precise genome editing by improved design and delivery of gRNA, Cas9 nuclease, and donor DNA., 2017, 241: 136–146.
[33] Nguyen DN, Roth TL, Li PJ, Chen PA, Apathy R, Mamedov MR, Vo LT, Tobin VR, Goodman D, Shifrut E, Bluestone JA, Puck JM, Szoka FC, Marson A. Polymer- stabilized Cas9 nanoparticles and modified repair templates increase genome editing efficiency., 2020, 38(1): 44–49.
[34] Ling X, Xie B, Gao X, Chang L, Zheng W, Chen H, Huang Y, Tan L, Li M, Liu T. Improving the efficiency of precise genome editing with site-specific Cas9-oligonucleotide conjugates., 2020, 6(15): eaaz0051.
[35] Lomova A, Clark DN, Campo-Fernandez B, Flores- Bjurström C, Kaufman ML, Fitz-Gibbon S, Wang XY, Miyahira EY, Brown D, DeWitt MA, Corn JE, Hollis RP, Romero Z, Kohn DB. Improving gene editing outcomes in human hematopoietic stem and progenitor cells by temporal control of DNA repair., 2019, 37(2): 284–294.
[36] Wienert B, Nguyen DN, Guenther A, Feng SJ, Locke MN, Wyman SK, Shin J, Kazane KR, Gregory GL, Carter MAM, Wright F, Conklin BR, Marson A, Richardson CD, Corn JE. Timed inhibition of CDC7 increases CRISPR- Cas9 mediated templated repair., 2020, 11(1): 2109.
[37] Zhang JP, Li XL, Li GH, Chen W, Arakaki C, Botimer GD, Baylink D, Zhang L, Wen W, Fu YW, Xu J, Chun N, Yuan W, Cheng T, Zhang XB. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage., 2017, 18(1): 35.
[38] Gutschner T, Haemmerle M, Genovese G, Draetta GF, Chin L. Post-translational regulation of Cas9 during G1 enhances homology-directed repair., 2016, 14(6): 1555–1566.
[39] Matsumoto D, Tamamura H, Nomura W. A cell cycle- dependent CRISPR-Cas9 activation system based on an anti-CRISPR protein shows improved genome editing accuracy., 2020, 3(1): 601–601.
[40] Li G, Zhang X, Zhong C, Mo J, Quan R, Yang J, Liu D, Li Z, Yang H, Wu Z. Small molecules enhance CRISPR/ Cas9-mediated homology-directed genome editing in primary cells., 2017, 7(1): 8943.
[41] Takayama K, Igai K, Hagihara Y, Hashimoto R, Hanawa M, Sakuma T, Tachibana M, Sakurai F, Yamamoto T, Mizuguchi H. Highly efficient biallelic genome editing of human ES/iPS cells using a CRISPR/Cas9 or TALEN system., 2017, 45(9): 5198–5207.
[42] Li GL, Zhang XW, Wang HQ, Liu DW, Li ZC, Wu ZF, Yang HQ. Increasing CRISPR/Cas9-mediated homology- directed DNA repair by histone deacetylase inhibitors., 2020, 125: 105790.
[43] Keegan L, Gill G, Ptashne M. Separation of DNA binding from the transcription-activating function of a eukaryotic regulatory protein., 1986, 231(4739): 699–704.
[44] Marmorstein R, Carey M, Ptashne M, Harrison SC. DNA recognition by GAL4: structure of a protein-DNA complex., 1992, 356(6368): 408–414.
[45] Lohr D, Venkov P, Zlatanova J. Transcriptional regulation in the yeast GAL gene family: a complex genetic network., 1995, 9(9): 777–787.
[46] Xu K, Ren CH, Liu ZT, Zhang T, Zhang TT, Li D, Wang L, Yan Q, Guo LJ, Shen JC, Zhang ZY. Efficient genome engineering in eukaryotes using Cas9 from., 2015, 72(2): 383–399.
[47] Liang SD, Marmorstein R, Harrison SC, Ptashne M. DNA sequence preferences of GAL4 and PPR1: how a subset of Zn2 Cys6 binuclear cluster proteins recognizes DNA., 1996, 16(7): 3773–3780.
[48] Bram RJ, Lue NF, Kornberg RD. A GAL family of upstream activating sequences in yeast: roles in both induction and repression of transcription., 1986, 5(3): 603–608.
[49] Selleck SB, Majors JE. In vivo DNA-binding properties of a yeast transcription activator protein., 1987, 7(9): 3260–3267.
[50] Yan NN, Sun YS, Fang YY, Deng JR, Mu L, Xu K, Mymryk JS, Zhang ZY. A universal surrogate reporter for efficient enrichment of CRISPR/Cas9-mediated homology- directed repair in mammalian cells., 2020, 19: 775–789.
[51] Suzuki K, Tsunekawa Y, Hernandez-Benitez R, Wu J, Zhu J, Kim EJ, Hatanaka F, Yamamoto M, Araoka T, Li Z, Kurita M, Hishida T, Li M, Aizawa E, Guo SC, Chen S, Goebl A, Soligalla RD, Qu J, Jiang TS, Fu X, Jafari M, Esteban CR, Berggren WT, Lajara J, Nuñez-Delicado E, Guillen P, Campistol JM, Matsuzaki F, Liu GH, Magistretti P, Zhang K, Callaway EM, Zhang K, Belmonte JCI. In vivo genome editing via CRISPR/Cas9 mediated homology-independent targeted integration., 2016, 540(7631): 144–149.
A CRISPR/Cas9-Gal4BD donor adapting system for enhancing homology-directed repair
Xiaojun Zhang, Kun Xu, Juncen Shen, Lu Mu, Hongrun Qian, Jieyu Cui, Baoxia Ma, Zhilong Chen, Zhiying Zhang, Zehui Wei
The fast-rising CRISPR-derived gene editing technologies has been widely used in the fields of life science andbiomedicine, as well as plant and animal breeding. However, the efficiency of homology-directed repair (HDR), an important strategy for gene knock-in and base editing, remains to be improved. In this study, we came up with the term Donor Adapting System (DAS) to summarize those CRISPR/Cas9 systems modified with adaptor for driving aptamer-fused donor DNA. A set of CRISPR/Cas9-Gal4BD DAS was designed in our study.In this system, Gal4 DNA binding domain (Gal4BD) is used as adaptor to fuse with Cas9 protein, and Gal4 binding sequence (Gal4BS) is used as aptamer to bind to the double-stranded DNA (dsDNA) donor, in order to improve the HDR efficiency. Preliminary results from the HEK293T-HDR.GFP reporter cell line show that the HDR editing efficiency could be improved up to 2-4 times when donor homologous arms under certain length (100-60 bp). Further optimization results showed that the choice of fusion port and fusion linker would affect the expression and activity of Cas9, while the Cas9-Gal4BD fusion with a GGS5 linker was the prior choice.In addition, the HDR efficiency was likely dependent on the aptamer-dsDNA donor design, and single Gal4BD binding sequence (BS) addition to the 5′-end of intent dsDNA template was suggested. Finally, we achieved enhanced HDR editing on the endogenousandsites by using the CRISPR/Gal4BD-Cas9 DAS, which we believe can be applied to facilitate animal molecular design breeding in the future.
CRISPR/Cas9; gene editing; donor DNA; donor adapting; homology-directed repai
2022-04-05;
2022-05-11;
2022-05-23
国家自然科学基金项目(编号:32172736),陕西省重点研发计划项目(编号:2021NY-027)资助[Supported by the National Natural Science Foundation of China (No. 32172736), and the Shaanxi Key R&D Program (No. 2021NY-027)]
张潇筠, 在读硕士研究生,专业方向:动物生物技术。E-mail: mshn15@163.com
魏泽辉,博士,副教授,研究方向:动物分子设计育种,E-mail:weizehui7848@163.com
徐坤,博士,副教授,研究方向:动物基因编辑技术,E-mail: xukunas@nwafu.edu.cn
10.16288/j.yczz.22-118
(责任编委: 谷峰)

