加系大白猪乳头数性状的全基因组关联分析
周佳伟,张 宇,吴俊静,乔 木,徐 忠,李梓芃,杨前平,彭先文,梅书棋
(动物胚胎工程及分子育种湖北省重点实验室,湖北省农业科学院畜牧兽医研究所,湖北武汉 430064)
乳头数是评估母猪泌乳能力的重要指标,影响仔猪断奶存活率,是提高断奶仔猪头数的有效途径。在选育过程中,随着母猪产仔数提高,母猪乳头数性状的选育也引起了育种人的重视。不同品种猪的乳头数差异较大,大白猪的总乳头数14 个左右,杜洛克在10~12 个,而我国地方猪种二花脸猪的总乳头数最高达到25个。乳头数遗传力在0.1~0.4,属于中等遗传力性状。Hong 等报道大白猪总乳头数遗传力为0.22,Lee 等报道大白猪总乳头数遗传力为0.41。不同品系杜洛克猪总乳头数遗传力在0.1~0.4,我国地方猪种清平猪的总乳头数遗传力为0.36。
全基因组关联分析(GWAS)被广泛应用于重要经济性状相关SNP 的挖掘与鉴定。Moscatelli 等在大白猪SSC7 鉴定到2 个影响总乳头数的SNP,1 个影响左乳头数的SNP,2 个影响右乳头数的SNP。Uzzaman等在大白猪SSC8 和SSC13 上分别鉴定到2 个和1 个与乳头数相关的SNP。Zhuang 等在SSC18 之外的17 对染色体上均鉴定到了与杜洛克猪乳头数相关的SNP,其中SSC7 上鉴定到的位点数最多,筛选到等候选基因。本研究以944 头加系大白猪为研究材料,对左、右和总乳头数进行GWAS 分析,鉴定与加系大白母猪乳头数性状相关的SNP 及候选基因,为乳头数性状的育种提供新的分子标记,加快乳头数性状的选育。
1 材料与方法
1.1 实验材料和表型数据 本研究中的944 头加系大白猪来源于贵州某场,出生日期为2020 年1 月—2020 年6 月,来自12 个血统,每个血统的头数、左乳头数、右乳头数和总乳头数的均值见表1。母猪分娩时采集耳组织样品,记录其左乳头数、右乳头数和总乳头数。
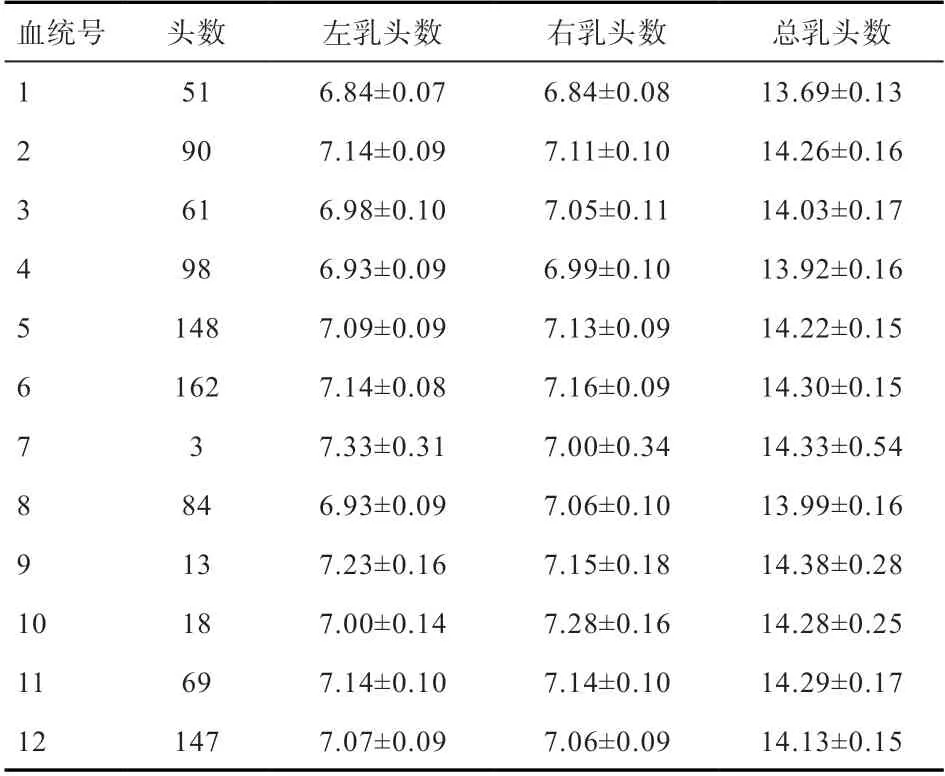
表1 944 头大白猪群体的基本信息
1.2 基因分型 采用动物组织DNA 提取试剂盒(天根生化科技有限公司,货号DP304)提取耳组织的基因组DNA。使用NanoDrop 2000 分光光度计检测DNA 浓度,利用1.5% 的琼脂糖凝胶电泳检测DNA 质量。将检测合格样品的DNA 稀释至50 ng/μL 左右,送样至北京康普森生物技术有限公司“中芯一号”芯片检测。
1.3 基因型数据质控 采用PLINK v1.9进行质量控制。筛选标准为:①剔除最小等位基因频率MAF<5%的位点;②剔除样本检出率小于95%,SNP 检出率小于90%;③剔除没有对应到猪参考基因组位置的位点和Y 染色体上的位点。共保留944 头猪和39 653 个SNP用于后续研究。
1.4 遗传参数估计模型 本研究使用以下模型进行乳头数性状的遗传力估计,使用GCTA 软件进行GREML分析:
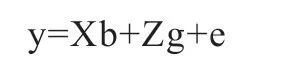
式中,y 表示乳头数性状的表型向量,b 表示固定效应向量,g 表示每个个体的随机多基因效应向量,X 和Z分别对应b 和g 的关联矩阵,e 表示乳头数性状的残差。
1.5 全基因组关联分析 本研究采用rMVP 软件进行全基因组关联分析,利用软件中的广义线性模型(GLM),将前5 个PCA、初生年、季节、血统因素分别作为固定效应加在关联分析模型中,本实验采用的关联分析模型如下:
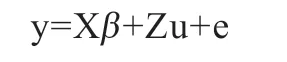
2 结果与分析
2.1 乳头数统计及遗传力分析 通过对944 头大白猪的乳头数进行统计分析,结果如表2 所示,左乳头数在6~10,右乳头数在5~9,总乳头数在11~19;左、右、总乳头数的平均值分别为(7.05±0.53)、(7.08±0.57)、(14.14±0.92)。进一步对以上3 个性状进行遗传力分析,发现遗传力均在0.1~0.2,其中左乳头数遗传力为0.1,右乳头数遗传力为0.12,总乳头数遗传力为0.16(表3)。
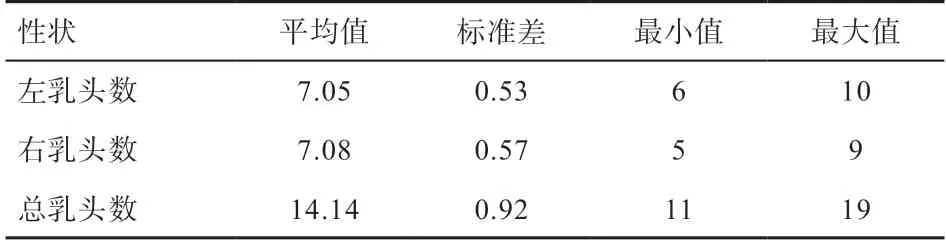
表2 左、右和总乳头数的统计结果

表3 左、右和总乳头数遗传力
2.2 主成分分析 利用“中芯一号”芯片检测数据经PLINK 质控后得到944 个有效个体及39 653 个有效SNP 位点,用于后续分析。通过主成分分析,绘制基于前两个特征向量的主成分分析图(图1),结果表明群体结构稳定,仅有少数个体表现出群体分层。因此,须在广义线性模型(GLM)中加入相应主成分进行矫正,以减少关联分析出现假阳性。
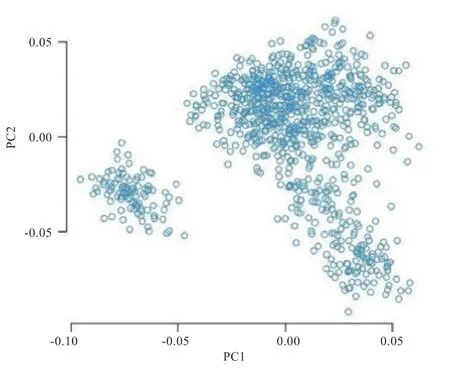
图1 群体结构PCA 图
2.3 全基因组关联分析 利用rMVP 软件包中的GLM方法对左乳头数、右乳头数和总乳头数3 个性状进行全基因组关联分析,结果如图2 所示,左乳头数、右乳头数、总乳头数分别得到4、1、4 个基因组显著的SNP,6、8、11 个潜在显著的SNP,去除重复的SNP 后,共获得4 个基因组显著的SNP,21 个潜在显著的SNP。这些SNP 分布在SSC3、SSC4、SSC6、SSC7、SSC9、SSC11、SSC13 上,4 个基因组显著的SNP 位点,均位于SSC7 上。
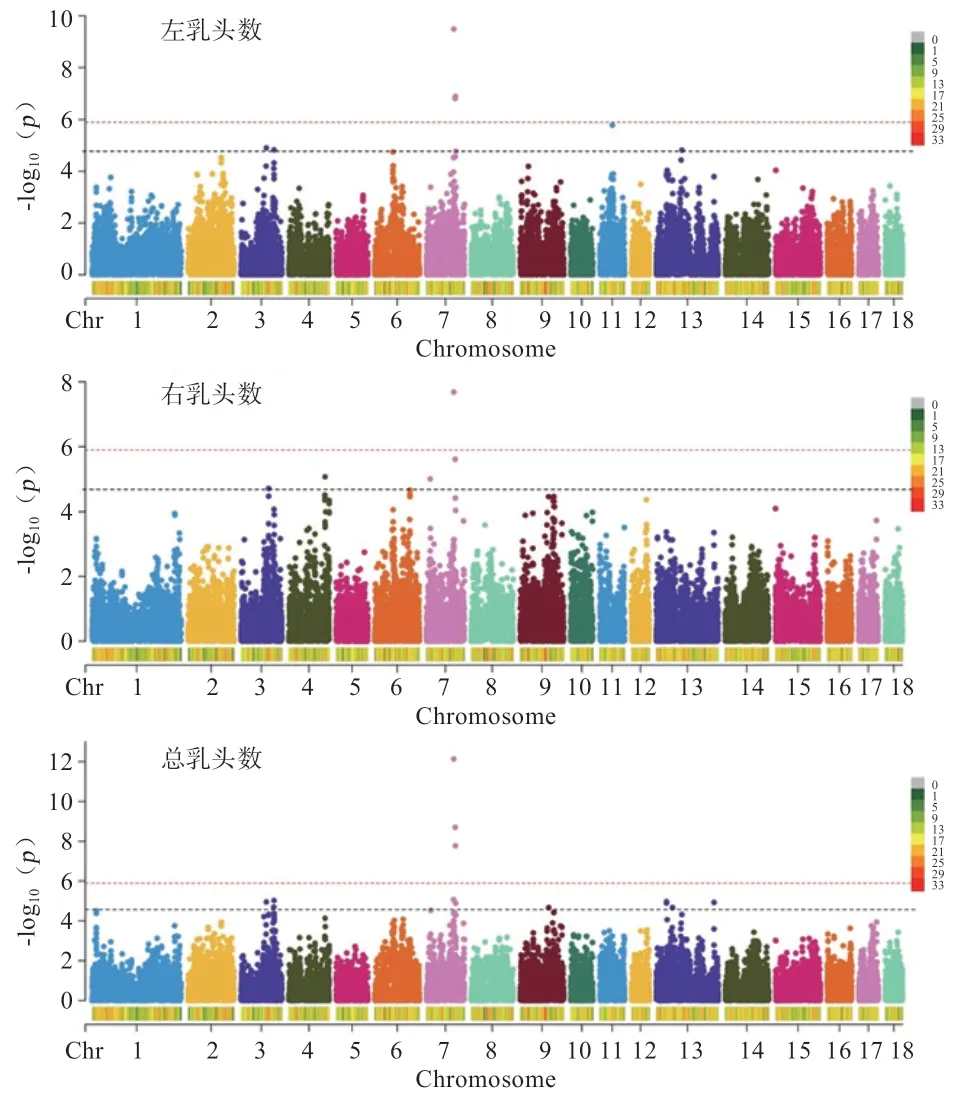
图2 乳头数性状全基因组关联分析曼哈顿图
进一步对SSC7 上4 个基因组显著和5 个潜在显著的SNP 位点上下游200 kb 进行候选基因分析。如表4~6所示,1 个SNP 位于SSC7 的14 124 612 bp 处,剩余的8个位点的距离较近,从96 646 705 bp 至104 914 125 bp。其中chr7:14124612 位点附近存在等5 个基因,chr7:96646705、chr7:97430037、chr7:97614602 位点附近存在等12 个基因,chr7:101888771、chr7:102041850、chr7:103232787、chr7:103574383、chr7:104914125 等5 个位点附近发现等22 个基因,其中被广泛报道与乳头数性状密切相关。
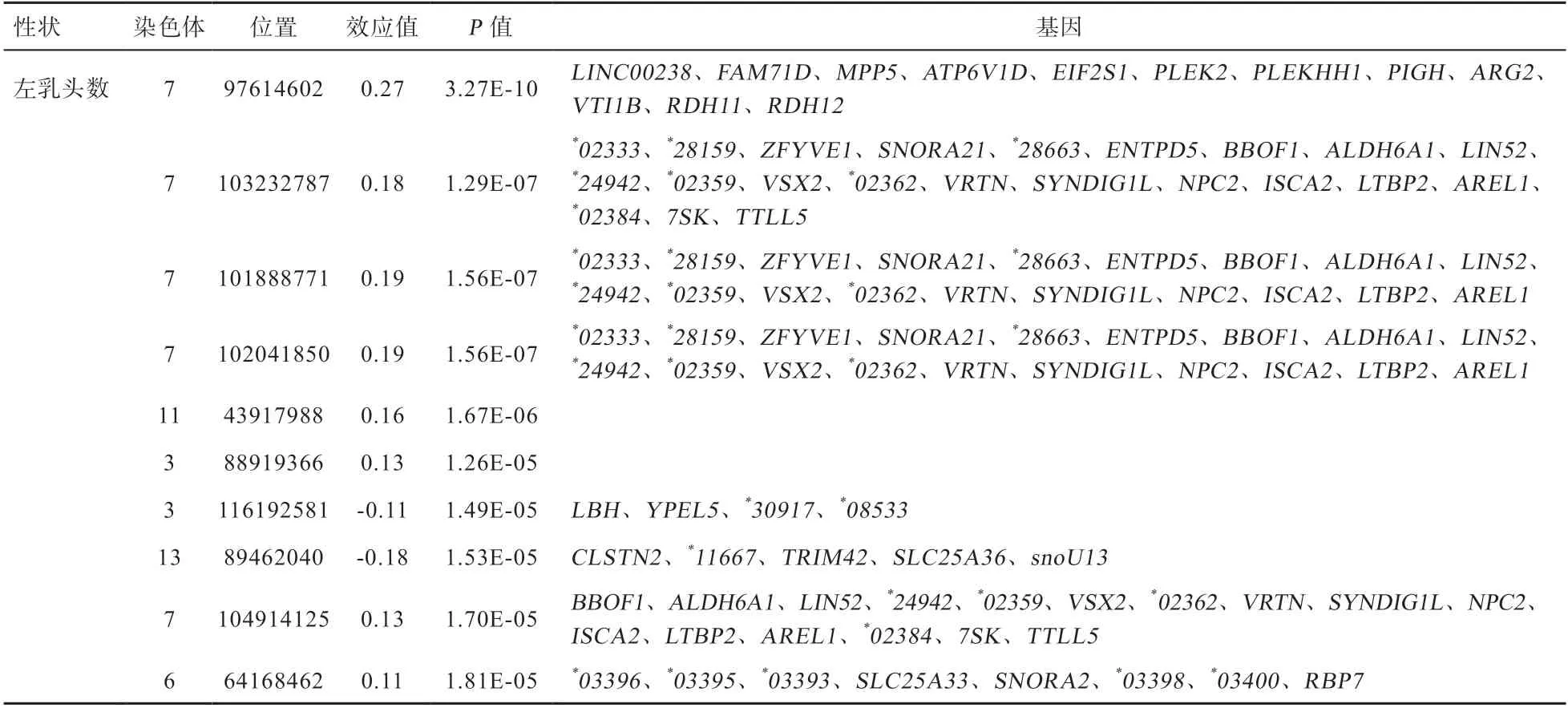
表4 左乳头数显著关联SNP 位点及候选基因信息
3 讨 论
乳头数是猪重要经济性状,与母猪繁殖性状密切相关。国内外研究者对不同品种和品系的猪相继开展了乳头数性状相关QTL 的鉴定,但关于加系大白猪乳头数相关QTL 的研究较少。本研究对944 头加系大白母猪乳头数性状开展了GWAS 研究,旨在找到影响加系大白猪乳头数的SNP,为加系大白猪乳头数选育提供新的分子标记和候选基因。
本研究使用的加系大白猪群体左、右和总乳头数的平均值分别为7.05、7.08 和14.14 个,与前人报道的大白猪乳头数相似。猪乳头数性状的遗传力为中等遗传力,遗传力为0.1~0.4。本研究加系大白猪群体左、右和总乳头数的遗传力分别为0.1、0.12 和0.16,略低于前人报道的乳头数遗传力,可能与大白猪的品系差异有关。
在主成分分析中发现左下角个体表现出群体分层,进一步查看这部分个体的系谱信息,发现这些个体同属于11 号血统,通过乳头数性状与血统因素的广义线性模型分析,发现不同血统个体的乳头数存在差异。因此,在GWAS 分析模型中,将排名前5 的PCA、初生年、季节及血统作为固定效应加在关联分析模型中。
通过全基因组关联分析,在SSC3、SSC4、SSC6、SSC7、SSC9、SSC11、SSC13 上找到了25 个与乳头数性状基因组水平显著相关或潜在显著相关的SNP。本研究发现的4 个基因组水平显著相关的SNP 均位于SSC7上,其中3 个SNP 位于SSC7 的97.6~102.0 Mb。值得注意的是,SSC7 上97614602(A
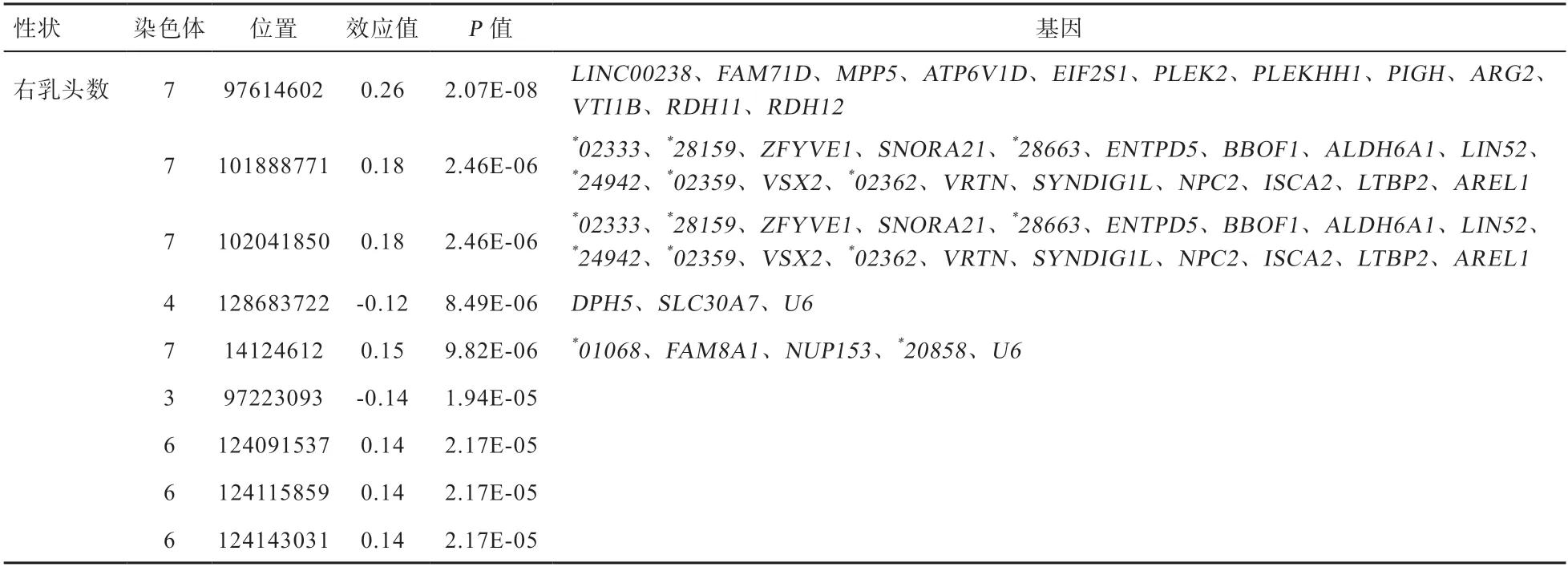
表5 右乳头数显著关联SNP 位点及候选基因信息
综上,猪乳头数性状的调控机制复杂,在多个染色体上均定位到与其相关的SNP,并获得了多个重要候选基因,但具体的分子机制仍有待后续研究,本研究将为乳头数的遗传改良提供参考。
4 结 论
本研究利用“中芯一号”芯片分析加系大白猪乳头数性状的遗传机制,发现左、右和总乳头数的遗传力分别为0.1、0.12 和0.16,属于中低遗传力。通过全基因组关联分析的方法,在SSC7 上鉴定到4 个与乳头数性状基因组水平显著相关的SNP,并在SSC3、SSC4、SSC6、SSC7、SSC9、SSC11、SSC13 上共鉴定到21个与乳头数性状潜在显著相关的SNP。进一步扫描25个显著相关的位点上下游200 kb 内的基因,获得84个候选基因,SSC7 上获得39 个候选基因,其中包括等候选基因,关于部分候选基因的功能及分子机制还有待进一步研究。
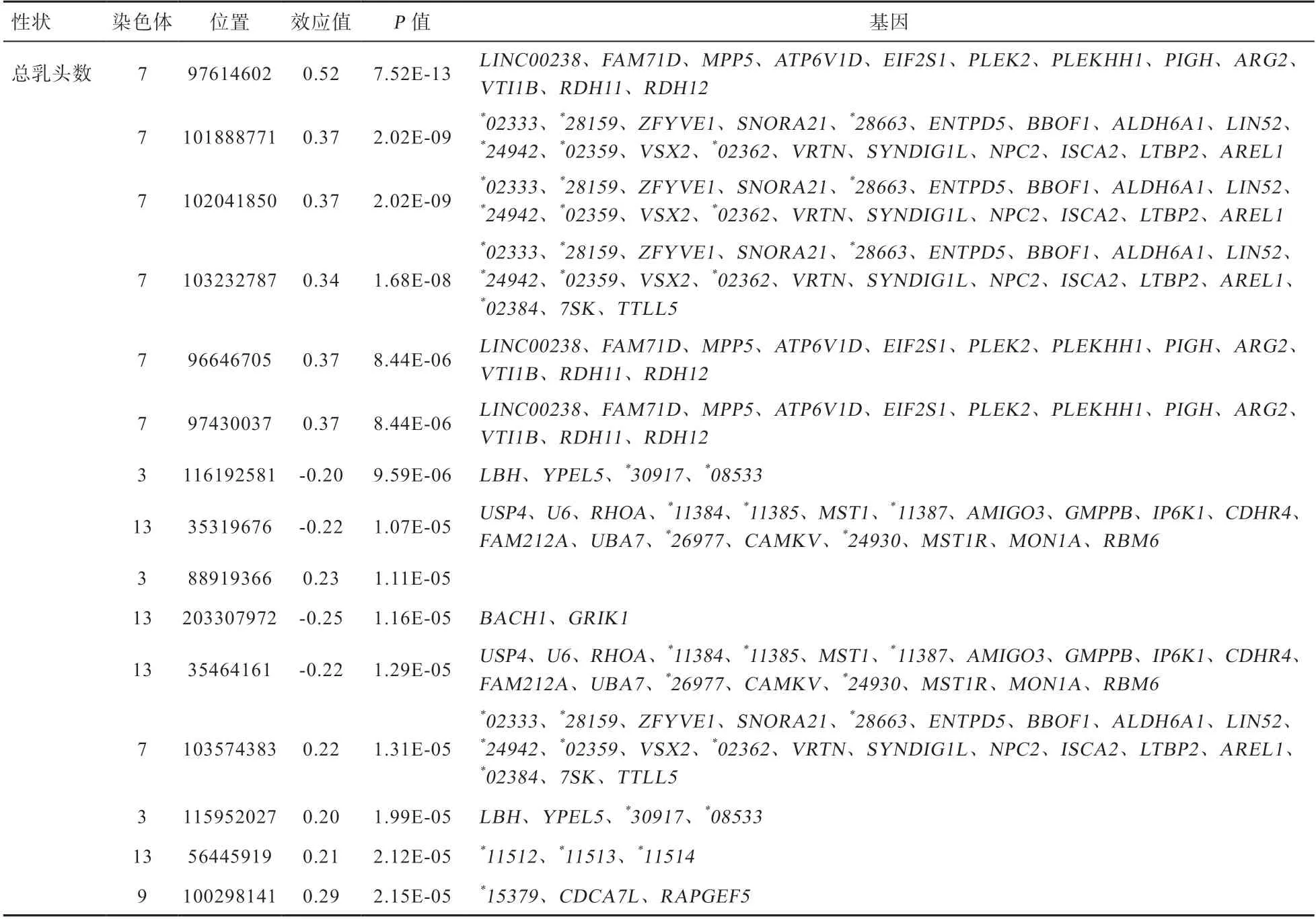
表6 总乳头数显著关联SNP 位点及候选基因信息

