平菇单核体与双核体差异的RAPD分析
张朝辉,王 勋,陈天航,梅会元,韩盼盼,
刘天翔1,王振河2,邱立友1 *
(1.河南农业大学生命科学学院/农业农村部农业微生物酶工程重点实验室,河南 郑州 450002;2.河南科技学院生命科技学院,河南 新乡 453003)
0 引言
【研究意义】平菇(Pleurotus ostreatus),又称糙皮侧耳,属于食药用真菌[1],在我国具有较长的栽培历史,种类繁多[2],具有营养价值高、适应性强、产量高的特点,目前已成为世界性的食用菌品种[3]。平菇属于典型的异宗结合担子菌,即单核菌株不能形成子实体,两个具有亲和作用的异性单核菌丝体结合后,才具备出菇能力[4],但单核体与双核体之间的差异尚不明确。【前人研究进展】随着分子生物学的发展,DNA分子标记技术被广泛应用于食用菌的遗传多样性分析中。DNA分子标记包括限制性片段长度多态性(Restrction Fragment Lengh Polyorhism,RFLP)标记[5]、相关序列扩增多态性(Sequence-related amplified polymorphism,SRAP)标 记[6]、RAPD标记[7]、简单重复序列区间(Inter-simple sequence repeats,ISSR)标记[8]等,它是直接在DNA水平上反映遗传变异的一种技术。其中RAPD标记由于其具有检测范围广[9]、检测速度快、操作简单等优点,现已被广泛应用于菌种鉴定、遗传多样性分析、遗传图谱的建立等方面[10],并取得了显著成效。詹才新等利用RAPD标记对金针菇不同亲本菌株及其杂交菌株进行分析后,得出该技术可用于鉴别真伪杂种的结论[11]。赵光辉等利用RAPD标记对20株草菇进行遗传多样性的分析,结果表明所有供试菌株间均存在遗传差异[12]。RAPD标记技术在食用菌遗传多样性分析中发挥了极大的作用[13]。【本研究切入点】双核体是一种独特的生物,它的菌丝每个隔间包含两个单倍体核,每个单倍体核来自不同的亲本[14],两个相容的单核菌丝体交配形成新的双核菌丝体,仅发生质配而没有进行核配[15],这种有性繁殖方式与二倍体明显不同。通过来自糙皮侧耳或香菇(Lentinus edodes)的同一亲本的交配亲和性担孢子自交,已选育出多个优良菌种[16−19],但其机理还不清楚。目前对于双核体及其亲本单核体之间的差异研究较少,通过比较单核体和双核体的菌丝生长状况、出菇产量和遗传多样性是研究两者之间差异的有效方法。【拟解决的关键问题】从平菇TD300的担孢子中分离得到MK13和MK3,二者交配得到DK300,通过对单双核菌株及其亲本进行菌丝生长状况和出菇产量的测定,利用RAPD法检测单双核菌株的遗传差异,以期为食用菌自交育种提供理论依据。
1 材料与方法
1.1 菌种
TD300:平菇天达300,河南农业大学农业农村部农业微生物酶工程重点实验室保藏菌株;单核菌株MK13和MK3:TD300担孢子分离获得。选择品质优良、大小合适、约八成熟的TD300子实体通过钩悬法收集孢子并制成孢子液,稀释涂布于PDA平板,25 ℃培养至出现单菌落。挑取单菌落接种至PDA平板培养基中央,继续培养。选择能够配合为优良双核菌丝的MK13和MK3作为试验的单核菌株;双核菌株DK300:MK13和MK3交配所得。
1.2 培养基的配制
PDA培养基:马铃薯200 g,葡萄糖20 g,琼脂20 g,水 1000 mL,pH 自然,分装后高压灭菌;棉籽壳栽培料培养基:棉籽壳78%,麸皮20%,石灰1%,石膏1%,含水量65%,pH自然,装袋后高压灭菌。
1.3 菌丝生长状况对比
将MK13、MK3、DK300和TD300分别接种在含有等体积PDA培养基的培养皿上进行活化,3~5 d后,打孔接种到新的平板上培养,在培养期间从菌落形态、菌丝颜色、长势、满板天数等方面观察并记录4个菌株的菌丝生长状况。
1.4 出菇情况及产量对比
常规栽培试验:使用15 cm ×32 cm聚丙烯折角袋,每袋装0.9 kg栽培料。高压灭菌2.5 h,每处理数量为20袋。常规管理,在此期间分别记录菌株性状和出菇产量。
1.5 菌株 RAPD 分析
RAPD标记技术是以基因组DNA为模板,随机选择一个长度通常为10个碱基对的寡聚核苷酸序列作为引物,进行PCR扩增反应,得到不连续的DNA产物,用于检测DNA序列的多样性[20]。本试验PCR扩增所用的引物来自上海生工的十碱基随机引物,如表1所示。

表1 供试随机引物Table 1 Random primers applied
菌丝DNA提取:称取新鲜的平板菌丝0.1~0.2 g,利用CTAB法[21]提取菌株DNA。随机引物的PCR扩增 :PCR体系:HSTMMix(购自广州东盛公司)12.5 μL,随机引物(上海生工合成)1 μL,DNA模板 0.5 μL,ddH2O 8.5 μL。PCR 扩增程序为:94 ℃,5 min;94 ℃,1 min;36 ℃,1 min;72 ℃,2 min;72 ℃,10 min,进行 30 Cycles。用 1.2% 的琼脂糖凝胶电泳检测。
2 结果与分析
2.1 菌丝生长状况分析
将直径为6 mm的TD300、DK300、MK13和MK3菌丝块接种在PDA平板中培养,观察记录4个菌株菌丝的生长情况。如表2所示,从菌落形态来看,TD300与DK300为半气生型,MK13和MK3为匍匐型。从菌丝长势来看,TD300的菌丝比其他3个菌株菌丝纤细稀疏,MK13和MK3的菌丝长势最好,均表现为粗壮浓密。而从菌丝颜色来看,DK300、MK13和MK3的菌丝均为白色,TD300的菌丝为浅白色。结果表明,4个菌株菌丝形态差异明显。
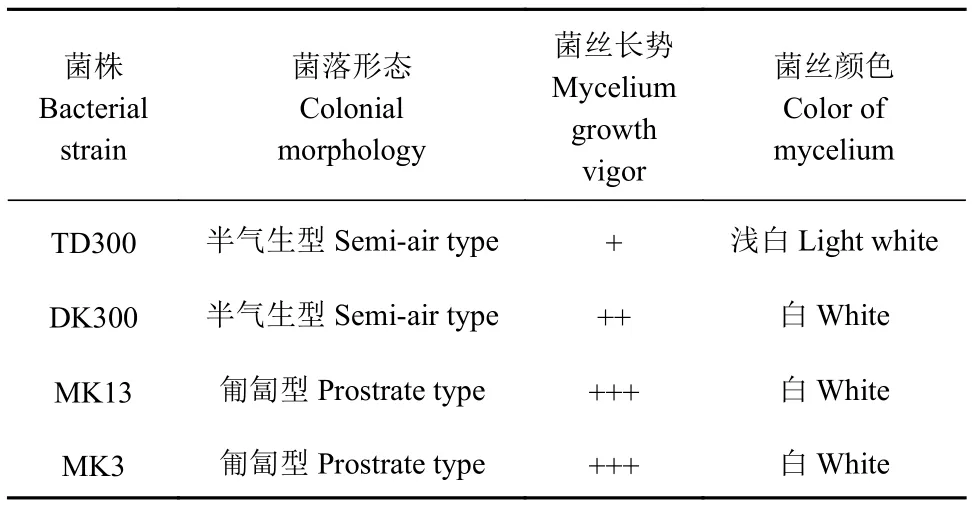
表2 菌株的菌丝形态Table 2 Mycelium morphology of tested strains
通过观察记录,对4个菌株菌丝在不同培养基中的生长状况进行统计分析。如图1所示,从菌丝满板天数来看,TD300与DK300最快满板,均为4 d,MK13和MK3长速较慢,其中MK13最慢,长满整个平板需要近20 d。在栽培试验中,TD300菌丝最先满袋,MK13最晚满袋,TD300比MK13提前20 d满袋,这与4个菌株菌丝在平板中的生长状况一致。结果表明,在不同培养料中,TD300和DK300菌丝生长状况较好,MK13和MK3菌丝生长状况较差。
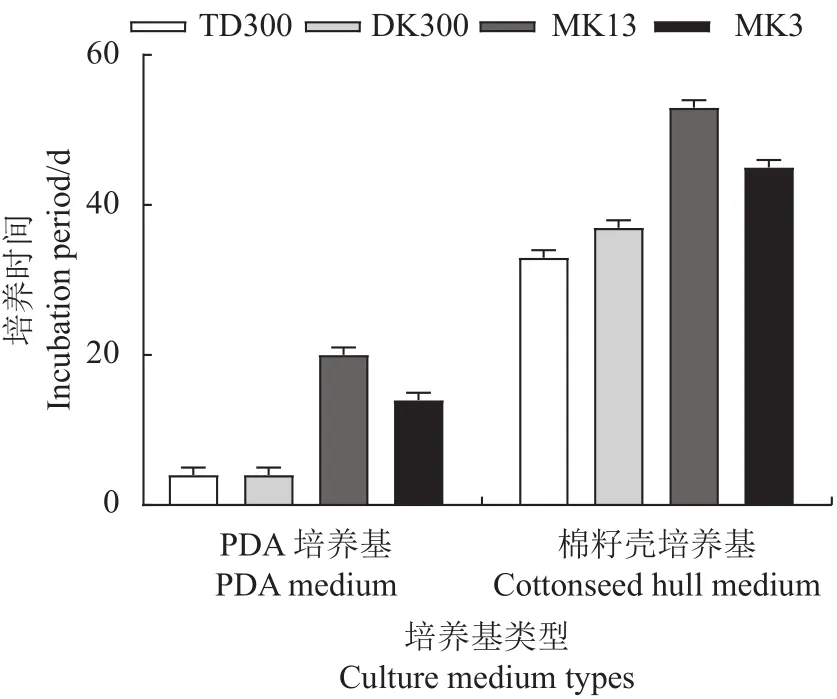
图1 菌株在不同培养基中生长状况Fig.1 Mycelial growth of strains in different culture media
2.2 出菇情况及产量分析
如表3所示,TD300和DK300均可以出菇,MK13和MK3均不出菇。从子实体形状来看,TD300为贝壳状,DK300为扇叶状。而从子实体颜色来看,TD300为深褐色,DK300为浅褐色。结果表明,TD300与DK300子实体性状差异明显。

表3 菌株出菇情况及子实体性状Table 3 Germination and fruiting body properties of mushrooms
如表4所示,从第一茬均产量来看,DK300平均每袋产量高于TD300,每袋高出4.40%,但不具有显著性差异;从第二茬均产量来看,TD300平均每袋产量比DK300高出21.93%,且具有显著性差异;而从平均每袋总产量来看,TD300的平均每袋总产量比DK300高出3.26%。
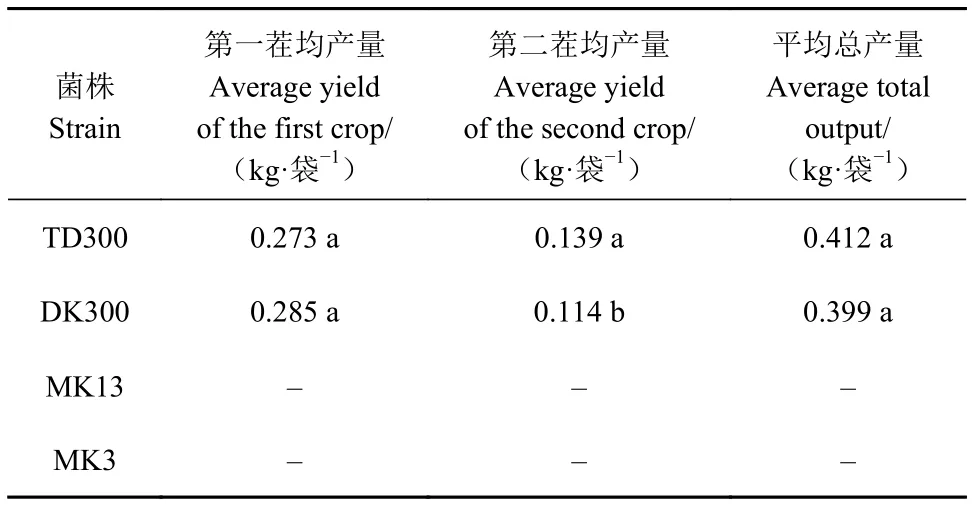
表4 菌株出菇产量Table 4 Yields of mushrooms
2.3 基于 RAPD 标记的菌株间亲缘关系分析
对提取的单、双核菌丝DNA进行电泳,结果如图2所示,DNA条带清晰且明亮,无明显弥散荧光区出现,适合用来做RAPD分析。本试验对提取的TD300、DK300、MK13和MK3的菌丝DNA进行RAPD分析。
图2 DNA电泳检测结果Fig.2 DNA electrophoresis of 4 mushroom strains
对13条随机引物进行PCR扩增,通过凝胶电泳发现,12条引物能扩增出清晰且重复性好并能用于RAPD分析的条带。如图3所示,共有86个条带被扩增,其中多态性片段69条,且特异性较高。如引物S36扩增出多态性片段8条,4个菌株共同条带2条,在DK300、MK13和MK3这3个菌株中也有两条共同条带,其中DK300拥有的条带均能够在MK13和MK3中找到,说明DK300与MK13和MK3的亲缘关系最近。引物S62共扩增出6条多态性片段,4个菌株中出现3条共同条带,在DK300、MK13和MK3中有两个共同条带出现。所有条带中,出现共同条带17条,说明这四个菌株在DNA水平上具有丰富的遗传多样性和一定的遗传均一性。
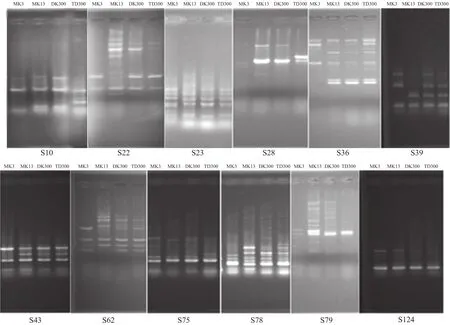
图3 随机引物扩增电泳图谱Fig.3 PCR electrophoresis using random primers
2.4 RAPD 结果聚类分析
为进一步研究菌株间的亲缘性关系,根据RAPD产生的条带作出“0-1”矩阵图并利用NTSYS软件的Jaccard聚类分析法制作聚类图谱(图4)。可以看出,在遗传相似系数为0.53时,4个菌株分为两个类群,TD300为一类,MK13、DK300、MK3为一类。当遗传相似系数为0.80时,4个菌株被分为3大类,其中MK13为第一类,DK300和MK3为第二类,TD300为第三类。这表明,DK300、MK13、MK3与TD300之间的亲缘关系较远,MK13和MK3与DK300的亲缘关系较近,其中MK3与DK300的亲缘关系最近。而从遗传差异性来看,遗传相似系数越大说明二者之间的遗传差异越小,由图4可知,MK13、MK3和交配所得的DK300与亲本TD300间具有较大的遗传差异性,而MK13、MK3和DK300间遗传差异性较小,尤其是MK3和DK300遗传差异性最小。
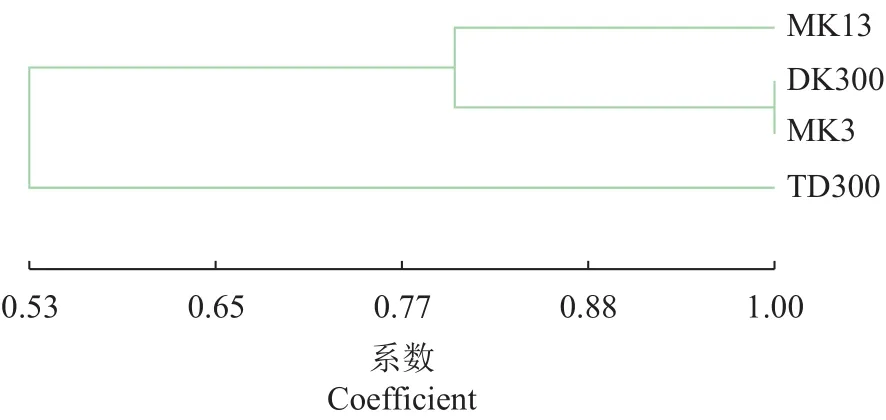
图4 RAPD聚类分析Fig.4 RAPD clustering on mushroom strains
3 讨论
平菇属于异宗结合食用菌,由担孢子直接萌发形成的菌株是不孕的,只有双核化的菌株才能形成子实体[4]。根据这一原理,本试验从TD300的担孢子中分离得到MK13和MK3,二者交配得到DK300,通过比较4个菌株的菌丝生长状况和出菇产量发现,TD300与其子代DK300的菌丝生长状况和产量没有显著性差异[15],这一结果也证实了由单核菌丝交配产生的子代双核菌株遗传了亲本的优良性状,对杂交育种提供一定的借鉴。
RAPD技术能够准确鉴定菌株的亲缘关系[22]。这是由于PAPD技术利用随机引物对不同物种或个体的DNA序列进行随机扩增[7],而引物与序列结合位点存在的差异能导致扩增的多态性DNA片段在DNA水平上反映出物种或者个体间的遗传差异[23]。本试验通过RAPD技术检测4个菌株间的遗传差异性,结果表明两个单核菌株MK13、MK3与亲本TD300和单核菌株交配所得的双核菌株DK300间具有遗传差异性,而单核菌株MK13和MK3与其交配所得的双核菌株DK300间遗传差异较小,尤其是MK3与DK300间差异更小。表明经过减数分裂形成担孢子时可能发生有基因重组,而通过两个单核菌株交配得到双核菌株过程则较少发生基因重组。前期同工酶谱结果与之类似。NADH脱氢酶同工酶谱中双核菌株DK300和单核菌株MK3有2条NADH脱氢酶同工酶谱带,而单核菌株MK13有3条NADH脱氢酶同工酶谱带;NADPH脱氢酶同工酶中双核菌株DK300、单核菌株MK3只有1条同工酶谱带,而单核菌株MK13的NADPH脱氢酶同工酶谱带为4条[24]。
端粒酶发挥作用能够维持端粒长度。由于体细胞中缺乏端粒酶活性,因此端粒长度会随DNA复制逐渐缩短。在生殖细胞中则有端粒酶,能够进行端粒修复,端粒长度因端粒酶活性的存在保持不变,保护染色体遗传物质的完整性[25]。担子菌食用菌保藏、制种和复壮时,往往通过菌丝和组织块的无性繁殖进行的,因此,染色体端粒将不断缩短,进而导致菌种活力下降,产量降低。通过有性繁殖形成担孢子时,担孢子中的染色体可能存在有端粒修复,由担孢子萌发形成的单核菌丝交配形成的双核菌丝其染色体端粒可能得以修复,菌种活力得到真正的复壮。这种机制可能是担子菌食用菌自交育种的另一个机理,但还有待进一步的实验验证。
本研究通过RAPD分析和聚类结果,并结合同工酶分析,可以快速检测出4个菌株间的遗传差异性,得出结论:MK13、MK3和交配所得的DK300与亲本TD300间具有较大的遗传差异性,而MK13、MK3和DK300间遗传差异性较小,尤其是MK3和DK300遗传差异性最小。这一结果是由于减数分裂过程中产生的遗传变异引起的,可能是担子菌食用菌自交育种的重要机制之一。

