CHIP2020评测任务1概述: 中文医学文本命名实体识别
李雯昕,张坤丽,关同峰,张 欢,朱田恬,常宝宝,陈清财,4
(1. 郑州大学 计算机与人工智能学院,河南 郑州 450001; 2. 北京大学 计算语言学教育部重点实验室,北京 100871;3. 哈尔滨工业大学(深圳) 计算机科学与技术学院,广东 深圳 518055;4. 鹏城实验室人工智能研究中心,广东 深圳 518055)
0 引言
随着医疗信息化的发展,医学领域积累了大量医学文本数据,例如,医学期刊、医学教材、医学百科、医学检验报告和用药诊断等,这些信息大多为叙述性文本摘要,是进行疾病预测、用药推荐、辅助诊疗的重要资源。
医学命名实体识别是医学领域文本挖掘的一个基础任务,该任务旨在自动识别医学文本中具有特殊意义的医学实体,并将抽取的信息以结构化的形式存储,供研究者们做进一步分析使用。医学命名实体在医学文本中普遍存在,例如,“疾病”实体被广泛用于病因探索、病情诊断、疾病预防以及后遗症的医学研究中。
医学领域文本数据具有信息密集、数据量庞大等特征,而且医学文本中往往包含大量半结构化和非结构化的数据,文本用词不受约束,使用简写/缩写、近义词、未登录词的现象普遍存在,导致医学命名实体本身具有复杂性与歧义性。现有的医学领域命名实体识别研究不免存在数据利用效率低、对现有数据挖掘和分析能力较弱等问题,与通用领域的命名实体识别相比还具有一定差距。通过自然语言处理技术与医学专业领域结合,从多元异构的医学文本中挖掘重要信息,有利于提高临床科研的效率和质量,并服务于下游子任务。
CHIP2020的主题为“数据和知识驱动的医疗AI”,会议组织了中文医疗信息处理相关的六项评测任务,其中评测任务一为中文医学文本命名实体识别任务,该评测鼓励参赛队伍使用基于自然语言处理技术和深度学习算法对中文医学文本进行命名实体识别研究。本次评测任务使用的数据集是由北京大学计算语言学教育部重点实验室、郑州大学计算机与人工智能学院、哈尔滨工业大学(深圳)计算机科学与技术学院以及鹏城实验室人工智能研究中心联合构建的中文医学实体抽取数据集CMeEE(Chinese Medical Entity Extraction)。总计开放了28 618条被标注的中文医学文本,包含定义的9类医学实体类别,如疾病(dis)、身体部位(bod),任务要求给定一条真实的医学教材文本,需要模型返回文本中可能的医学实体位置和医学实体类型。评测任务最终排名指标为微平均F1值,示例数据如表1所示。
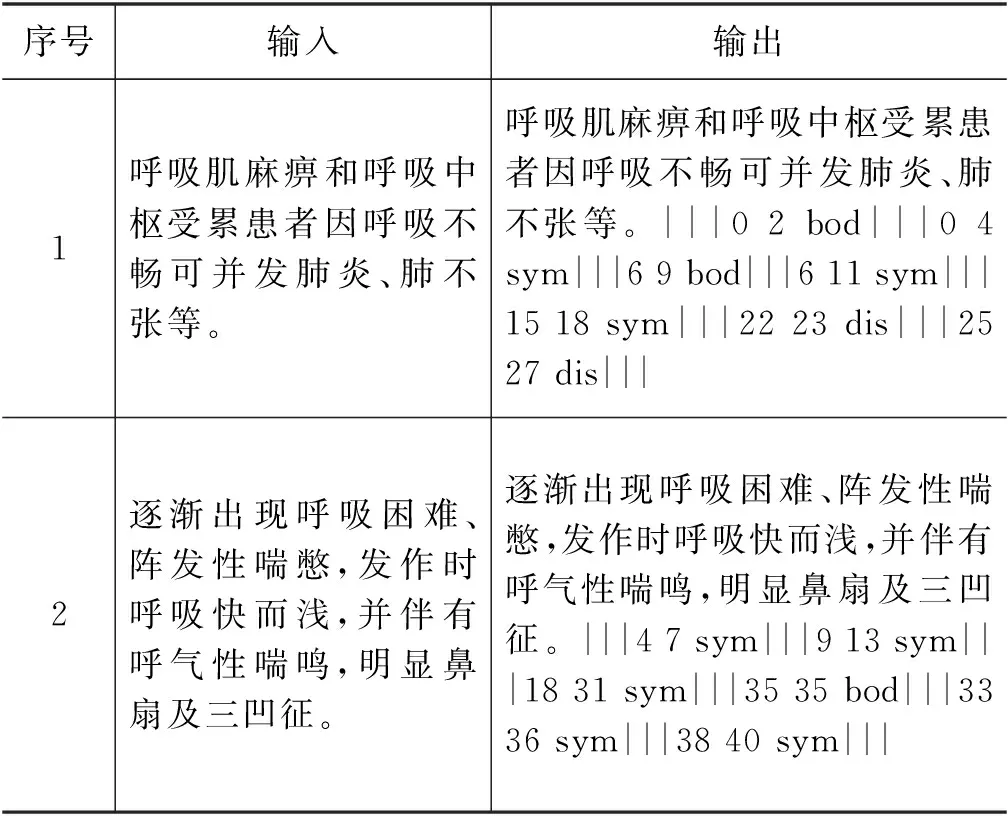
表1 评测任务示例
1 相关工作
1.1 医学命名实体识别研究
医学命名实体识别是医学领域信息抽取的关键任务,是医学关系抽取、医学文档分类和健康问答系统等技术研究中的重要步骤。医学命名实体识别方法可分为基于规则的方法、基于词典的方法、基于机器学习的方法和基于深度学习的方法。
基于词典的方法和基于规则的方法往往需要医学领域专家结合语料特点人工构建词典和规则模板[1],然后采取字符串匹配模式,从医学文本中检索相似单词进行命名实体识别,如统一语言系统(Unified Medical Language System,UMLS)[2]。Etzioni等[3]提出了一个基于规则的实体识别系统,通过规则模板能够自动化地提取网页中存在的实体。Hanisch等[4]通过处理同义词词典构建一个用于识别生物医学文本中实体名的系统。基于词典和规则的方法易于实现,但识别性能受限于构建的词典和规则的质量,所制定的规则仅对特定数据集有效,很难用单个词典或规则涵盖所有可能的表述,无法应用于其他数据集。对于新出现或不在专业医学词典中的命名实体,字符串匹配方法很难进行识别,且匹配精度不高。
基于机器学习的方法需要标准的标注数据集,利用样本数据统计特征和模型参数来构建模型。常用的机器学习模型有隐马尔可夫模型HMM(Hidden Markov Model)、支持向量机SVM(Support Vector Machine)和条件随机场CRF(Conditional Random Field)等。Wang等[5]采取模型融合策略,将SVM、CRF等模型组合,结果显示融合的模型比单一模型效果更好。
基于深度学习的方法利用词向量作为输入,通过深度学习框架自动学习语料特征,有效降低了人工的干预。Qiu等[6]提出了一种基于条件随机场的残差扩张卷积神经网络RD-CNN-CRF(Residual Dilated ConVolutional Neural Network with the Conditional Random Field),使模型在计算上具有异步性,从而加快了训练速度。Xue等[7]通过动态范围注意机制集成变换器双向编码表征模型BERT(Bidirectional Encoder Representations from Trans-formers),提高了共享参数层的特征表示能力,在曙光医院冠脉造影文本命名实体识别F1值达到96.89%。Wang等[8]针对识别嵌套实体的任务设计了次优序列学习的算法,并采用从外部到内部迭代提取实体的方式进行解码预测,取得不错的效果。
1.2 医学命名实体评测会议
国内外组织了多个与医学命名实体识别相关的评测任务,如I2B2[9]评测任务一要求从电子病历中识别并提取出医疗疾病、检查和治疗等实体,JNLPBA2004[10]要求自动识别五类生物医学命名实体,以及BioCreative IV化合物和药品实体识别任务[11]等。在国内,全国知识图谱与语义计算大会CCKS(China Conference on Knowledge Graph and Semantic Computing)于2017年发布了中文电子病历命名实体评测任务,识别并抽取电子病历中与医学临床相关的实体提及,并于2018~2020年在此基础上进行数据集更新和规范的补充。此次CHIP评测任务一基于医学教学文本聚焦于中文医学文本命名实体识别,希望验证最新的自然语言处理技术和算法,为生物医学领域命名实体识别研究提供最新实验结果。
2 评测数据
本次评测任务使用的数据集(CMeEE数据集)的标注语料来自《临床儿科学》[12],全书共分为17篇,介绍了儿科各系统疾病、传染病、肿瘤以及新生儿生长发育、营养、监护和急救等基础理论知识。该教材由儿科专家编写,文本叙述简洁,内容具有权威性,可用于专业科学研究。
CMeEE参考面向医学教材的医学实体标注规范[13],将医学实体划分为9类,并对每种类别定义了实体边界和标注规则,如表2所示。18名经过培训的标注者被分为9组,采用多轮迭代的模式进行规范的修订和标注工作[14],根据科恩卡帕评分对每个类别进行标注一致性计算,总体的一致性分数为0.869。
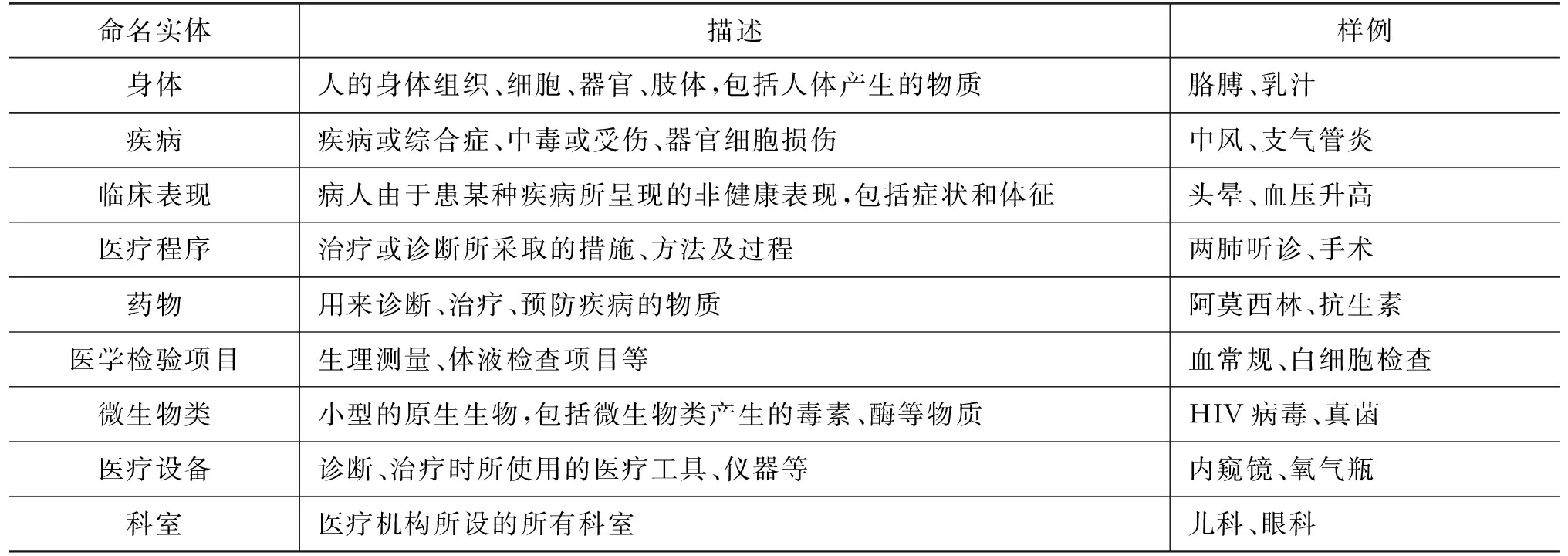
表2 9类医学实体简述及分类
CHIP2020评测任务一最终公布的数据集包括9种实体类别信息和28 618条医学文本,包括训练集15 000条,验证集5 000条,测试集8 618条,各实体类别的分布如表3所示。
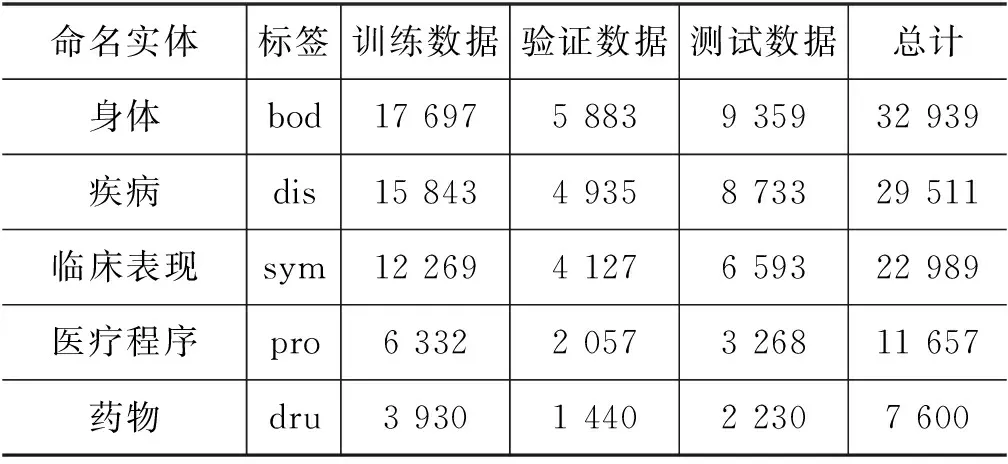
表3 训练集、验证集、测试集中各命名实体数量分布情况
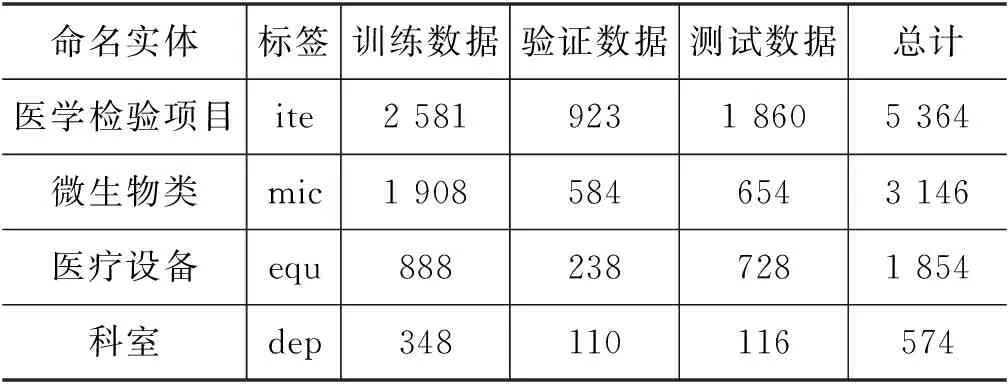
续表
3 评测结果
CHIP2020评测任务一于2020年7月12日开放注册,参赛团队通过邮箱进行报名。评测任务的训练集、验证集、测试集A以及评价脚本于2020年7月20日发布,供参赛队伍搭建并测试各自开发的模型。测试集B于2020年9月28日发布,每支队伍在测试集B公布期间最多提交两次结果文件。CHIP2020评测任务一于2020年9月29日截止,总共253支队伍报名参加评测,共计614人,其中,128支队伍来自科研院校等机构,41支队伍来自企业,84支队伍为个人报名。最终有91支队伍提交了测试集A模型评测结果,37支队伍提交了测试集B模型评测结果。参赛队伍的评测方法和结果,由评测组织者进行学术评测分析研究。
3.1 评估指标
在评估单个实体类别预测性能时采用准确率P、召回率R、F1值为评估指标,计算如式(1)~式(3)所示。本次评测使用的评价指标包括微平均准确率(Micro_P)、微平均召回率(Micro_R)和微平均F1值(Micro_F1), 最终排名以微平均F1值为基准。假设有n个类别:C1,…,Ci…,Cn,微平均F1值计算如式(4)~式(6)所示,其中,TP、FP、TN和FN分别代表真正类、假正类、真负类和假负类。
3.2 方法分析
本次评测队伍解决中文医学文本命名实体识别任务的主流思路为引入预训练语言模型和神经网络模型,进行多种模型集成,并在此基础上引入词汇增强等方案提升性能。评测任务一的参赛队伍使用了多种预训练语言模型,主要包括BERT[15]、强力优化变换器双向编码表征模型RoBERTa(Robustly Optimized BERT Pretraining Approach)[16]、面向中文理解的神经语境表征模型NEZHA(Neural Contextualized Representation for Chinese Language Understanding)[17]、高效替代令牌检测分类编码器Electra(Efficiently Learning an Encoder that Classifies Token Replacements Accurately)[18],其中,RoBERTa模型使用次数最多。用大规模领域文本语料训练预训练语言模型,可以提供丰富的语义表示知识。此外有部分队伍采用多种机器学习算法通过与预训练语言模型拼接进行分类,如门控循环单元GRU(Gate Recurrent Unit)[19]、长短时记忆网络模型LSTM(Long Short-term Memory)[20]、双向长短期记忆网络模型BiLSTM(Bi-Directional Long Short-Term Memory)[21]、CRF[22]、空洞卷积神经网络IDCNN(Iterated Dilated Convolutional Neural Networks)[23]等。提交的最终结果显示,神经网络模型在序列标注问题中具有更好的性能表现,多数队伍利用BiLSTM得到上下文特征,结合CRF对标注结果进一步优化。
由于评测任务一是嵌套类命名实体识别问题,传统的基于序列标注的命名实体模型不能很好地解决嵌套命名实体识别的任务,此次评测中前三名队伍采用的策略包括以下几点:
(1) 结合机器阅读理解任务MRC(Machine Reading Comprehension)的研究方法,对每一个实体类别构建问题,以问答的方式利用预训练模型提取实体,同时获得实体首尾可能位置的预测结果;
(2) 层叠式模型,每一层采取指针标注,通过堆叠多个识别层的方式来识别命名实体中的层次化结构;
(3) 多头选择机制,结合关系抽取任务中的关系线性分类器,将其适用于嵌套实体抽取任务;
(4) 双仿射机制,通过双仿射机制捕获边界信息,并采取传统的序列标注任务强化嵌套结构的内部信息交互;
(5) 词汇增强,在模型中结合词级别的信息,将文本的编码结果和实体的词向量作为抽取层的最终向量;
(6) 采用动态规划的思想对超长的句子进行切分。
结果显示,这些策略单独使用和组合使用时对模型最终的性能表现有不同程度的提升。此外,在特定领域对预训练模型进行进一步的训练也能提升模型效果。
3.3 结果分析
对37支队伍提交的评测结果进行分析,微平均F1值的平均数为0.603 8,最大值为0.683 5,最小值为0.001 8,中位数为0.661 8。排名前三的队伍提交的各自最优的结果信息如表4所示,包括参赛单位、方法描述和微平均F1值。
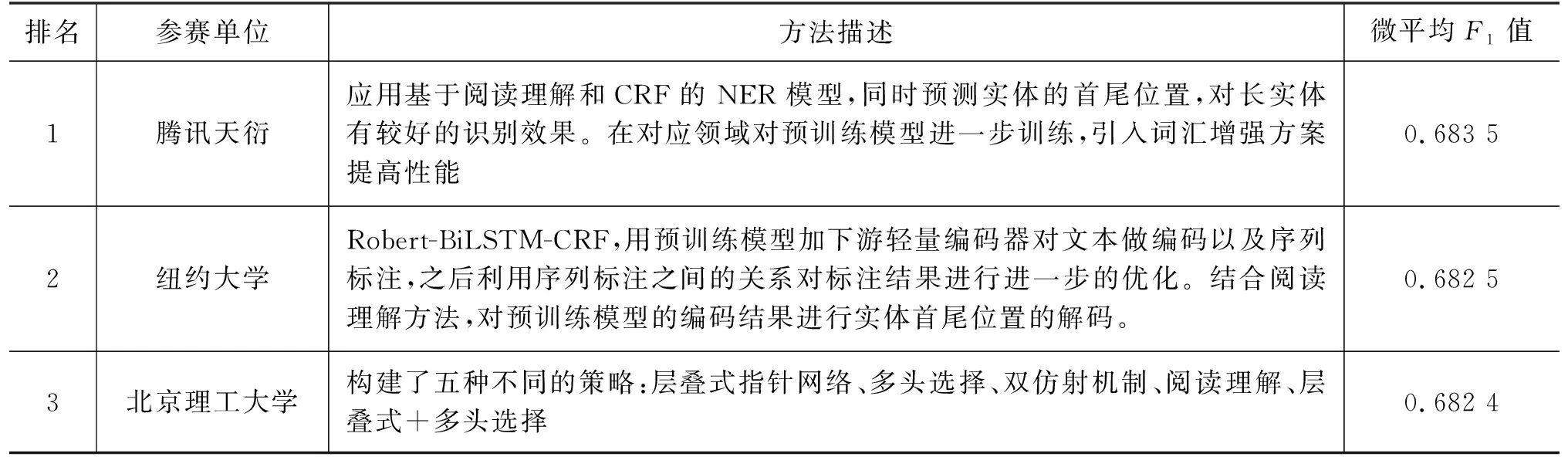
表4 排名前三参赛队伍的系统结果
排名前三的队伍提交结果在9种实体类别的性能表现如图1所示,本文使用F1值评估每一个类的性能指标,纵坐标表示为F1值,横坐标表示9类实体类别,其中6种类别的F1值在0.60以上。药物(dru)识别效果最优,所有队伍结果F1值都在0.82以上,可能原因为药物这类实体数据量较多,句子长度较短,且语义信息辨识性高。临床表现(sym)和医学检验项目(ite)识别效果最差,成绩在0.48~0.50之间。可能原因在于医学检验项目实体往往嵌套出现在临床表现实体中,这在一定程度上影响了模型的识别能力。而临床表现是指病人患病时的异常变化以及异常结果,实体普遍较长,且构成规律复杂,模型的识别能力有待提高。并且临床表现实体与疾病实体易混淆,相同的实体在不同语境下可能具有不同的实体类别。例如,“病人患高血压”以及“病人检测出高血压、高血糖”,“高血压”在前者语境中应被识别为疾病,后者被识别为临床表现。

图1 排名前三的队伍最终结果分布在9类实体类别的性能表现
将评测结果结合9类医学实体的数据量和文本描述分析,可以发现数据量丰富,具有独特描述特征的实体类别识别效果最好,数据稀疏,构成规律复杂的实体类别识别效果较差。模型识别效果较好的实体,如药物、疾病、微生物类,以上类别文本往往是医学领域专业名词,文本描述信息独特性高,这些都会帮助模型表现出更好的识别效果。识别效果较差的实体类别,如科室、临床表现、医学检验项目,由于数据量少、实体长度不确定以及存在实体嵌套等原因而使识别效果表现一般。
结合不同队伍的系统方案分析,预训练语言模型结合深度学习网络模型是大部分队伍采用的方案,排名较好的队伍将多个模型的实验结果相融合,相比于单个模型取得较好的性能提升。如排名第3的队伍与排名第7的队伍均采用RoBERTa预训练模型,但第3名在此基础上构建了5种不同策略进行模型融合识别实体,总体效果取得一定提升,同时在识别药物、微生物类、科室这3种实体中取得最好效果。在识别表现较好的实体类别中,如药物、疾病,各模型的表现差异不大。在识别效果较差的实体类别中,各模型表现差异较为明显。因此提升数据辨识度低、数据量小的实体识别效果有助于提升模型性能。
4 结语
CHIP2020中国健康信息处理会议中的评测任务一为中文医学文本命名实体识别,总共开放了28 618条中文医学文本,以及预先定义好的9类医学实体。一共37支队伍提交了最终结果,排名第1的模型在对应领域再次训练预训练语言模型BERT并结合MRC,微平均F1值达到了0.683 5。大部分参赛队伍都使用了预训练语言模型,结合深度学习模型,然后针对任务进行词汇增强,最后进行模型融合,提高模型总体性能。结果分析排名靠前的队伍模型总体表现很接近,但在不同类别的实体间识别效果差异明显,F1值最低为0.487 5,最高为0.859 0。其中,数据规模大、描述文本辨识度强、实体边界明显的类别识别效果较好。
未来的研究中,在对应领域训练预训练模型,提高小类别和嵌套实体的表现,可进一步提高中文医学文本命名实体识别模型的性能。

