基于生物信息学对食管鳞癌潜在枢纽基因的筛选
房佳慧 王金胜
食管癌(Esophageal cancer,ESCA)在全球肿瘤中的发病率及死亡率分别为第6位和第8位[1-2]。其中,近90%的食管癌病例为食管鳞状细胞癌(Esophageal squamous cell carcinoma,ESCC)。由于食管鳞癌的早期症状不明显,导致大多数病人发现时已是中晚期,预后不良。缺乏早期诊断指标仍然是食管鳞癌治疗和预后面临的巨大挑战。近年来,为了挖掘出与人类肿瘤演进有关的差异表达基因(Differentially expressed genes,DEGs),识别生物标志物和潜在的治疗靶点,越来越多的微阵列和高通量测序技术结合生物信息学分析被广泛应用[3]。但只有很少的生物标志物和药物靶点被转化为临床实践,主要原因是由于独立的微阵列分析、不同的技术平台会造成假阳性。
在本研究中,我们从基因表达汇编(Gene expression omnibus,GEO)数据库中下载3个包含ESCC与非癌组织的原始数据集进行分析,使用维恩图工具重叠3个数据集,获得223个共有的DEGs进行进一步的生物信息学分析,包括基因本体(Gene ontology,GO),京都基因和基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)分析和蛋白-蛋白相互作用(Protein-protein interaction,PPI)网络构建,并验证枢纽(hub)基因的相对表达水平及其与食管鳞癌患者存活的关系,以帮助我们了解食管鳞癌发生和转移相关枢纽基因和关键通路。
1 材料与方法
1.1 材料
从GEO(http://www.ncbi.nlm.nih.gov/geo/)数据库[4]下载GSE161533、GSE100942、GSE45670的ESCC及邻近正常组织基因表达谱,关键词“食管”和“人”用作查询。其中GSE161533包括28个ESCC组织和28个非肿瘤组织;GSE100942包括4个ESCC组织和4个非肿瘤组织;GSE45670包括28个ESCC组织和10个非肿瘤组织。
1.2 筛选差异基因
使用GEO2R(http://www.ncbi.nlm.nih.gov/geo/geo2r)对ESCC和非肿瘤组织之间的差异基因进行筛选。以logFC(fold change)>1和P<0.05为有统计学意义。对每个数据集进行统计分析,并使用维恩图web工具(https://bioinformatics.psb.ugent.be/webtools/venn/)识别相交部分。
1.3 差异基因的KEGG和GO富集分析
将从3个数据集中筛选出的共有的差异基因用DAVID数据库[5](http://david.ncifcrf.gov)先进行GO功能富集分析,包括生物过程(Biological processes,BP)、分子功能(Molecular function,MF)、细胞成分(Cell component,CC);再进行KEGG通路富集分析,寻找其关键的信号通路。以P<0.05为差异有统计学意义。
1.4 PPI 网络构建和hub基因筛选
利用相互作用基因检索工具STRING(http://string-db.org)在线数据库[6]构建DEGs的PPI网络,一个组合交互>0.4的评分被认为有统计学意义。利用Cytoscape平台[7]绘制PPI网络,并使用其cytohubba插件筛选食管鳞状细胞癌hub基因[8]。Genclip(http://ci.smu.edu.cn/genclip3/analysis.php)绘制hub基因的火山图。
1.5 hub基因的表达验证及患者生存分析
应用GEPIA(cancer-pku.cn)[9]及UCSC癌症基因组浏览器(http://genome-cancer.ucsc.edu)[10]验证hub基因在肿瘤样本与正常组织之间的表达差异。并采用Kaplan Meier-plotter(KM plotter,http://kmplot.com/analysis/)[11]在线工具进行hub基因的生存分析,以识别食管鳞癌生存相关的生物标志物。
2 结果
2.1 食管鳞癌中DEGs的鉴定
经芯片结果标准化后,GSE161533中有850个DEGs,GSE100942中有409个DEGs,GSE45670中有6 413个DEGs。3个数据集的重叠部分包含227个基因,223个基因有相同的表达趋势,其中食管鳞癌组织与非癌组织间上调基因91个,下调基因132个。见图1。

图1 GSE161533、GSE100942、GSE45670中DEGs的韦恩图Fig.1 Venn diagram of DEGs common to all 3 GEO datasets
2.2 GO和KEGG富集分析
生物过程富集分析显示,DEGs主要与RNA聚合酶II启动子转录的正调控、细胞黏附、转录的正调控、DNA 模板化、细胞增殖的正/负调控等过程有关(见图2A)。DEGs的细胞组成主要包括细胞质、胞外外泌体、胞外区、细胞外间隙、蛋白质细胞外基质等(见图2B)。DEGs分子功能(MF)的变化主要集中在蛋白质结合、锌离子结合、相同的蛋白质结合、肌动蛋白结合等(见图2C)。在KEGG功能通路中,DEGs主要参与趋化因子信号通路、甲状腺激素信号通路、心肌细胞中的肾上腺素信号等(见图2D)。

A.GO生物学过程分析;B.GO细胞学组成分析;C.GO分子功能分析;D.KEGG通路分析图2 DEGs的GO和KEGG分析Fig.2 GO and KEGG analysis of DEGs
2.3 PPI网络构建
本研究利用STRING数据库结合Cytoscape软件构建PPI网络。红色标记为上调基因,蓝色标记为下调基因,见图3。
2.4 hub基因鉴定
利用Cytoscape其中的模块cytoHubba筛选hub基因并进行可视化,按照最大团中心性(Maximal clique centrality,MCC)方法排名前10位的hub基因分别为激光激酶A(Aurora kinase A,AURKA)基因、泛素结合酶E2C(Ubiquitin-conjugating enzyme E2C,UBE2C)基因、Xklp2靶向蛋白(Targeting protein for Xklp2,TPX2)基因、甲状腺激素受体互作体13(Thyroid hormone receptor interactor 13,TRIP13)基因、拓扑异构酶 2A(Topoisomerase 2A,TOP2A)基因、异常纺锤体样小头相关蛋白(Abnormal spindle-like microcephaly-associated,ASPM)基因、中心体蛋白(Centrosomal protein 55,CEP55)基因、无齿E3泛素蛋白连接酶同源物(Denticleless E3 ubiquitin protein ligase homolog,DTL)基因、细胞分裂周期6(Cell division cycle 6,CDC6)基因、细胞周期蛋白依赖性激酶抑制剂3(Cyclin dependent kinase inhibitor 3,CDKN3)基因。随后构建了10个枢纽hub基因的PPI网络(见图4A)。genclip绘制相关hub基因的火山图(见图4B),发现10个hub基因在ESCC中均属于上调基因。

红色:上调基因; 蓝色:下调基因图3 DEGs的PPI网络构建Fig.3 PPI network construction of DEGs

A.利用 Cytoscape中cytoHubba插件筛选10个hub基因网络图;B.hub基因火山图。红点代表上调基因,绿点代表下调基因图4 鉴定hub基因Fig.4 Identification of hub gene
2.5 hub基因在食管癌中的表达情况
使用在线数据库GEPIA与UCSC数据库,对Hub基因进行验证,发现其表达水平均明显高于正常组织,差异有统计学意义(P<0.05),见图5、图6。
2.6 hub基因与肿瘤分期、患者生存时间的关系
采用Kaplan-Meier在线数据库分析ESCC中10个枢纽基因的预后情况。其中7个基因,包括ASPM、DTL、CDKN3、AURKA、TPX2、TOP2A和CDC6基因高表达组与食管鳞癌病人总体生存率相关,风险率(Hazard Rate,HR)分别为0.35,0.30,2.45,0.32,0.33,0.31和0.29,差异有统计学意义(P<0.05)。通过GEPIA分析发现筛选出的hub基因均与食管癌临床分期无显著关系。
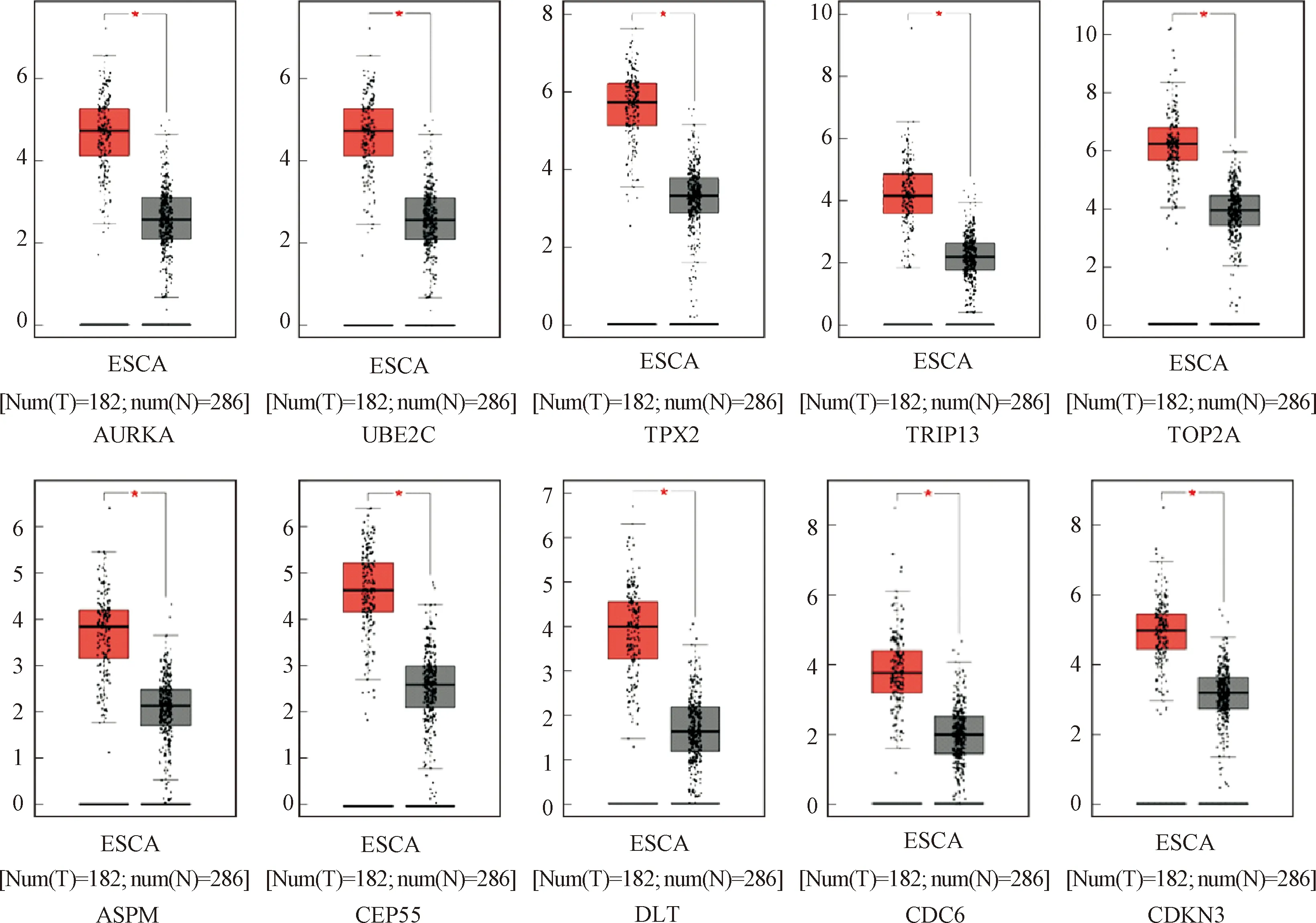
*P<0.05; ■ Tumor; ■ Normal图5 食管癌肿瘤组织与正常组织之间hub基因表达差异Fig.5 The expression difference of hub gene between esophageal cancer tumor tissue and normal tissue

蓝色:食管癌样本;红色:非癌样本;紫色:发生转移样本图6 基因表达差异情况Fig.6 Hub gene expression differences
3 讨论
虽然近年来已有大量研究探讨食管鳞癌的发病机制,但在世界范围内食管鳞癌的发病率和死亡率仍呈上升趋势。与以往仅关注几个基因或单个队列的研究相比,本研究选择了来自不同研究团队的3个高质量基因谱数据集,对ESCC中的驱动基因和生物学途径进行了综合研究。最后鉴定出223个DEGs(91个上调,132个下调);通过GO、KEGG富集分析、PPI网络构建等方法探索DEGs之间的相互作用;构建了包括165个节点和534条边的PPI网络,鉴定出10个与ESCC具有高度连通性的hub基因,包括ASPM、DTL、CDKN3、AURKA、UBE2C、TPX2、TRIP13、TOP2A、CEP55和CDC6。这些基因的生物学功能主要富集在RNA聚合酶Ⅱ启动子转录的正调控、细胞黏附、转录的正调控、DNA 模板化、细胞增殖的正/负调控等过程。富集途径主要为趋化因子信号通路、甲状腺激素信号通路、心肌细胞中的肾上腺素信号等。这些基因可能在肿瘤微环境内与各种趋化因子参与复杂的信号传导过程,促进恶性细胞通过基质迁移,促进肿瘤进展。
此外,与正常组织相比,食管癌组织中的所有hub基因表达水平均上调。通过文献查询,9/10的hub基因都有食管癌相关的实验及临床研究,证实参与了食管癌的发生发展。其中TOP2A是本研究中连接节点最高的基因,它是一种参与DNA复制的基因编码酶,与蒽环类化合物对多种癌症的反应相关[12]。它的过度表达在包括ESCA在内的多种癌症中很常见。此外,TOP2A抑制剂已被用于多种实体肿瘤,如小细胞肺癌、食管癌等[13-14]。关于ASPM在食管癌中的基础研究未见报道。ASPM是一种中心体蛋白,Wnt信号通路的正调节因子,在有丝分裂纺锤体调节、神经发生和大小脑调节中起着至关重要的作用[15]。有研究表明,ASPM在多种癌症中高表达,与临床预后差和复发密切相关。PAI等[16]报道前列腺癌组织中ASPM的表达增加,并通过增强Wnt-β-catenin信号促进癌细胞的增殖、迁移和侵袭。最近研究显示,在子宫内膜癌患者中ASPM的表达水平上调[17]。为进一步探讨研究ASPM与食管鳞癌的关系,评价其是否可作为判断食管鳞癌患者临床治疗的靶基因及预后的独立指标提供依据。
与单数据集研究相比,本研究采用综合生物信息学分析可能提供更准确的结果,为ESCC的研究提供了新的发现。本研究仍存在一定的局限性,首先,基因表达谱数量只有3个;其次,我们主要探讨了hub基因的功能和潜在作用,而没有深入分析其他基因;另外,我们仅使用Gepia和UCSC数据验证hub基因的表达水平,以上结果还需要进一步的实验研究证实。
综上所述,本研究通过数据库筛选鉴定出与食管鳞癌发展和预后相关的的10个基因,为探讨食管鳞癌的发病机制提供了线索。这些结果为全面了解食管鳞癌特征提供了重要思路,仍需进一步研究来验证并阐明这些基因在食管鳞癌中的具体分子机制。

