基于迭代卷积神经网络的肝脏MRI图像分割
陆雪松,闫书豪
(中南民族大学 生物医学工程学院,武汉 430074)
目前,肝脏疾病已经成为腹部较为常见的疾病之一.临床上,各种不同的成像手段,如CT(Computed Tomography)、MRI(Magnetic Resonance Imaging)和超声等被用来进行肝病的诊断和治疗.腹部MRI由于具备较好的软组织对比度、无辐射等优势,在临床检查中逐步占据主导地位[1].随着成像技术的发展,从医学图像中提取出肝脏组织已成为精准医疗的基本前提.但是,依靠人工的手动分割耗时耗力,效率低下,且一致性较差.因此,开发一种快速准确的自动分割技术,更好地服务于临床工程,越来越受到研究者的关注.
腹部MRI对软组织和内脏的成像能力高,且多序列图像(如T1、T2)能提供病灶解剖和功能方面的互补信息.但是,由于腹部组织结构复杂,这些脏器之间的黏连容易导致成像结果中出现浸润现象[2].而且,肝脏与周围器官如胃、心脏等有着相似的灰度值,边界较为模糊,使得提取任务极具挑战性.为了克服这些困难,研究者们提出了一些自动分割方法.吕晓琪等[3]提出一种基于水平集的三维肝脏磁共振图像混合分割方法,采取阈值分割等算法进行预处理改善分割结果精度.谢勤岚等[4]提出自适应形状约束Graph cuts算法应用于腹部CT图像分割,该方法基于多图谱配准技术,将初始分割结果作为形状先验加入到Graph cuts算法的能量函数中.同时根据配准分割得到的目标概率图自适应地选择形状约束项系数,通过最大流最小割算法执行最终分割.刘伟等[5]将图谱与待分割CT图像依次进行仿射和B样条配准,同时加入搜索算法自动搜索肝脏区域,进而计算出肝脏体积.
近年来,深度学习[6]被广泛应用于计算机视觉、图像分割等领域.郭雯等[7]阐述了使用率较高的深度学习基础网络结构和常用改进方法,分析了医学图像中脑部、肝部和前列腺等器官自动分割的研究进程,展望了一些图像分割网络的未来方向.KAREN等[8]提出VGG16深度卷积网络用于大规模图像识别,该网络非线性学习能力强[9],能够很好地实现图像块的区分.OLAF等[10]提出基于U-Net的图像分割模型并得到了广泛的应用,它采用复制连接的方式融合全局和局部特征,依此引导模型实施分割.VIJAY等[11]提出SegNet方法用于语义像素分割,它是一种基于图像分割的深度卷积编解码器结构体系,能够对输入特征映射进行上采样,但当对低分辨率的特征图进行池化时,会忽略邻近的上下文信息.WANG等[12]将用于序列图像检测的递归单元嵌入到U-Net框架中,利用前次分割结果对目标进行精确提炼.但是相较于标准的卷积神经网络单元,其计算量偏大.
针对传统U-Net中下采样时容易丢失细节信息的问题,本文引入一种基于深度卷积编解码的迭代网络结构,用于腹部MRI的肝脏分割.该网络将含有语义信息的概率特征图与浅层特征图进行多尺度融合,实现上下文信息的整体贯通而非个别单元,更深层次地为丢失信息提供补偿.通过ISBI 2019 liver-chaos挑战赛[13]上数据集的验证实验,这种方法能够自动准确地完成肝体组织的提取.
1 方法
1.1 网络结构
对于腹部MRI中的肝脏分割挑战,传统U-Net采用池化层进行下采样容易导致信息丢失,这些图像细节的损失在上采样时很难被弥补.为了提升网络的分割性能,拟将高水平的语义特征(分类概率图)与来自编码器的低水平特征进行复制连接,依此对下采样的图像微小结构进行定位,迭代地训练网络参数.具体网络结构如图1所示.

图1 迭代网络结构图Fig.1 Iterative network structure diagram
通常,U型网络编码阶段主要由卷积层与最大池化层[14]组成,其输入为二维的MRI图像数据.在我们的网络设计中,编码器部分需要增加另外一个输入,即前一次分割结果的概率图,以此定位到图像中的微小结构和细节.具体是两个输入分别经过两个卷积层后被复制连接.此外,多尺度操作是前次分割结果的概率图经过四个卷积层和一个池化层后被再次复制连接到对应层.在卷积层中,设定卷积核尺寸大小为3×3,移动步长为1,为了使卷积之后的特征图尺寸不变,每个卷积层都加入了Padding操作.即分别在输入特征图最外层的行和列填充0,同时在卷积层之后采用ReLU非线性激活函数.池化层采用了最大值池化方式,步长为2,池化核尺寸为2×2,每经过一个池化层,特征图尺寸变为之前的一半.每个层的特征通道数与U-Net类似,即第一层的卷积核数量为64,之后每经过一个池化层,特征通道数扩大一倍.
在网络解码阶段,反卷积层是重要组件.它采用了步长为2的2×2卷积核,每经过一次反卷积层,特征通道数减少一半,特征图的尺寸会扩大一倍.反卷积之后的特征图与编码器对应的特征图进行拼接操作,接着采用2个3×3卷积层和ReLU激活层.最后一层采用1×1的卷积和Sigmoid激活函数,将64维通道特征图映射成一个像素值在0~1之间的概率图,从而获取最终的分割结果.
1.2 迭代训练
为了在编码阶段更好地定位图像微小结构,我们将前次分割输出的概率图引入到网络浅层,网络参数以迭代的方式完成更新.在初始迭代中,概率图的像素值被设置为0.5.在网络开始下一次迭代时,将上一次网络生成的分类概率图替换当前所用概率特征图.与训练时相同,验证集也会在每一次的验证中进行概率图的迭代.为了判断网络训练是否收敛,我们加入了迭代终止条件,如果前一次迭代的输出和当前迭代的输出之间的差异小于预设阈值:

网络则终止迭代.其中,P为像素点的总个数,t为迭代次数,Si代表了输出概率图的像素值,Th为收敛阈值.网络训练和验证的流程如算法1所示.
1.3 损失函数
在神经网络的学习过程中,反向传播主要依靠损失函数来完成,它是用来估量模型的预测值与真实值的不一致程度.二元交叉熵损失函数是用来评价真实的概率分布和训练得到的概率分布之间的差异,该函数适用于二元分类任务,其数学定义如下:


其中,yn为样本标签,xn为模型输出的概率值,正样本标签为1,负样本标签为0.
2 实验
2.1 实验数据集
本文实验采用ISBI 2019 liver-chaos[13]分 割挑战数据集,该数据集包含20个病例的MRI多序列图像数据,肝脏区域已被专业医师人工勾画,被视为ground truth.每个病例都被两个MRI序列(T1-DUAL,T2-SPIR)扫描,其中T1-DUAL是双回波序列,能够在in和out两个相位成像.图像大小为256×256×26~320×320×50,图像空间分辨率为1.359 mm×1.359 mm×9 mm~1.895 mm×1.895 mm×5.5 mm.图2展示了一些切片.
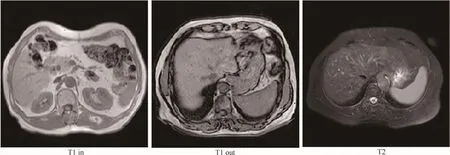
图2 肝脏MRI图像示例Fig.2 Examples of hepatic MRI images
将这三组数据分别按照8∶1∶1进行训练集、验证集和测试集划分,每组20个病例实现5-折交叉验证进行肝体组织分割.为了避免样本训练过拟合,采用平移、旋转、翻转和弹性形变等手段扩增数据.图3展示了弹性形变增广图像.最终,平均有3470幅二维切片被用于训练,在输入到网络之前,图像大小被重采样为256×256,像素值被归一化为零均值和单位方差.

图3 弹性形变增广示例Fig.3 Examples of MRI images from elastic deformation
2.2 实验坏境及参数设置
实验采用基于Pytorch 1.7.1深度学习平台,Python 3.8.3编码语言.计算机配置如下:Intel(R)Core(TM)i7-10700@2.90GHz,16GB memory,网络训练使用NVIDIA GeForce RTX 3080 Ti GPU进行加速.本文方法使用Adam优化器,设置学习率为1e-5,Batch size为1,Th为500.在传统U-Net方法中,学习率为1e-5,Batch size为4,Epoch为120.
2.3 实验评价指标
为了评估分割算法的性能,我们使用了4个度量指标[15].它们包括Dice系数、相对体积误差(Relative Volume Difference,RAVD)、平均表面距离(Average Symmetric Surface Distance,ASSD)和最大表面距离(Maximum Symmetric Surface Distance,MSSD).假设VA表示预测分割结果中的体素,VB表示ground truth中的体素,于是有:

假设SA表示预测分割结果所构成的面,SB表示ground truth所构成的面,于是有:

其中,d(SA,SB)表示SA中的某点到SB的最近欧式距离.Dice=0表示两种分割没有重叠,Dice=1表示两种分割完全一致.相反地,剩余3个度量指标越小,表明分割质量越高.当两种方法进行比较时,双边的Wilcoxon检验被执行.一个p<0.05的值表示在统计上有明显的差异.
3 结果
图4显示了采用传统U-Net和本文方法进行单次分割任务的学习曲线.在图4(a)中,两种方法在前几次epoch中训练loss下降都很快,到达60次左右模型逐渐收敛.相对应地,图4(b)给出了测试图像Dice系数随epoch进程的变化曲线,可以发现两种方法在最初上升都较快,然后逐渐收敛.但是本文方法的收敛速度更快,且最终的Dice值要高于传统U-Net.这也表明本文方法具备优良的学习和优化能力.

图4 (a)训练过程中的Loss曲线;(b)测试过程中的Dice曲线Fig.4 (a)Training loss curve over time;(b)Testing dice curve over time
图5对多序列MRI肝脏分割的Dice结果进行了比较.可以看出,在每个序列数据上本文方法比传统U-Net都有明显提升.对于总体60个待分割图像,Dice中值从0.9195增长到0.9325(p=1.355×10-7).表1列出了多序列数据肝脏分割的所有度量指标平均结果,从中可以发现本文方法在RAVD、ASSD和MSSD方面同样要好于传统U-Net.图6展示了一些分割结果图像.可以看到,由于腹部MRI中存在的肝脏分割难题造成传统U-Net出现欠分割或过分割.采用本文方法的分割结果尽管不完美,但是更接近于专业医师的人工勾画结果.

图6 分割结果图像示例Fig.6 Examples of segmentation result images

表1 多序列MRI肝脏分割的所有度量指标均值对比Tab.1 Comparison of average metrics for liver segmentation on multisequence MRI

图5 多序列MRI肝脏分割Dice对比箱线图(一个星号表示该列方法与前列方法相比在统计上有明显差异)Fig.5 The boxplot of Dice of liver segmentation on multi-sequence MRI(A star indicates a statistical significant difference between current column and previous column)
由于本文方法将高水平概率特征图以多尺度形式引入到网络编码器中,为更深层次地调查其性能,我们进行了消融实验.对照网络结构图1,我们将前次分割所获的概率图做单尺度引入,具体为仅经过两个卷积层后进行复制连接(被命名为Proposed-L1).表2利用所有分割结果的度量均值在总体上将本文方法与其他方法进行对比.可以发现,单尺度操作所获得的分割精度要低于多尺度迭代,但仍好于传统U-Net.另外,将本文方法与目前流行的自动分割方法进行对比.其中,MAS(Multi-Atlas Segmentation)是以图像配准技术为基础的多图谱分割方法.在elastix工具包上进行初始全局仿射和B样条局部形变配准[16],然后利用基于图像块的标签融合策略形成最终结果[17],所用参数均为标准配置.ISDUE、CIR-MPerkonigg和Mountain方法都来自ISBI 2019 liver-chaos挑战赛的参加者,它们均是采用卷积神经网络进行学习的分割方法,直接引用其在线测试结果[13].综合四个评价指标来看,本文方法能够达到目前领先的分割水平,在临床工程上具有潜在的价值.

表2 不同方法的总体分割结果度量指标均值对比Tab.2 Comparison of average metrics for overall segmentations using different methods
4 讨论
本文针对腹部MRI图像的肝脏分割挑战,提出了一种改良的U-Net框架进行迭代训练和学习.该网络由编码区、解码区和补偿区三个部分组成.编码区利用腹部MRI切片、中间分割结果、卷积层和最大池化层对分层特征进行编码.在解码区部分,采用反卷积层和跳跃连接来预测要分割的目标区域.为了提高网络的图像细节分割性能,在补偿区通过迭代输入概率图的方法弥补丢失的信息.
从表1中可以看到,相比于传统U-Net,本文提出的方法获得了更好的分割结果.这要归功于高水平的形状上下文特征融入到低水平的特征图中,对学习过程中细节的丢失实施了补偿.另外,表2中显示单尺度融合的分割性能比多尺度融合的差,这也证明了在多个尺度上进行特征融合是有效的.将来可以利用UNet++技术[18],将深浅层特征以多尺度的方式融合,以期望进一步提升网络性能的效果.
对于医学图像分割任务,特别是三维体数据中的多目标分割,前人的研究大都集中在MAS分割技术领域.该技术主要由图像配准和标签融合两个组件构成,其成败也依赖于它们的执行效能.从表2中可以看到,本文方法的分割结果在四个评价指标上都远超过MAS分割结果,而且不需要进行图像配准.基于深度学习的分割方法主要利用在训练过程中学习得到的局部和形状特征,在测试时对像素进行分类,其性能一定程度上依赖于训练样本[19].
本文方法所构建的神经网络采用二维卷积核,腹部磁共振数据以二维切片的形式被输送到网络中,模型参数量约为10M.如果采用三维卷积核,体数据可以直接输入到网络中,但是将会消耗更多的GPU内存.对于多目标分割任务,基于深度学习的方法主要考虑训练样本在各目标上的平衡性,再辅以合适的损失函数[20].下一步工作重点是将本文方法扩展到采用三维网络结构的多目标分割中.

