模式动物果蝇的基因调控前沿技术
韩玉婷,许博文,李羽童,卢心怡,董习之,邱雨浩,车沁耘,朱芮葆,郑丽,李孝宸,司绪,倪建泉
模式动物果蝇的基因调控前沿技术
韩玉婷,许博文,李羽童,卢心怡,董习之,邱雨浩,车沁耘,朱芮葆,郑丽,李孝宸,司绪,倪建泉
清华大学医学院,基因表达与调控实验室,北京 100084
转基因调控技术在生命医学研究中扮演了重要的角色,是探究个体发育和致病机制的必备工具。常用的转基因调控技术包括基因突变、基因干扰和基因转录激活等。果蝇由于具有基因的保守性、遗传工具的多样性以及不受伦理限制等优势,成为生命科学研究中经典模式动物之一,并由此开发出了多种时间和组织特异性的基因调控工具。本文主要介绍了目前在果蝇中常用的基因调控技术,包括CRISPR/Cas9介导的基因突变系统、基于miRNA的新一代转基因干扰系统以及基于CRISPR/dCas9的转录激活(flySAM)系统,希望通过对这几种系统设计原理、操作过程、相关的技术工具及相关资源品系的介绍,提升人们对这些前沿的果蝇遗传学基因表达调控技术及构建的相关果蝇资源品系重要性认识,进而推动生命医学研究发展。
果蝇;基因调控;CRISPR/Cas9;基因编辑;转基因RNAi;CRISPRa
转基因调控技术是基于分子生物学原理设计相关基因调控元件,并结合转基因技术实现降低、去除或增强靶基因活性或表达水平的一项前沿生物技术。转基因调控技术是现代生物技术的核心,对生命医学的发展和提升人民健康水平具有重大意义。例如,通过转基因调控技术建立的动物疾病模型有助于推动神经退行性疾病病理生理学的研究和大规模小分子化合物以及中草药物的筛选[1,2];转基因技术可修复患者原有突变基因或将正常基因引入,实现对疾病进行治疗,为包括地中海贫血症、脊索性肌萎缩在内的多种遗传疾病提供了新的治疗手段[3]。因此,掌握并研发转基因调控技术,正成为应对西方国家技术壁垒、避免“卡脖子”的战略措施。
与其他模式动物相比,果蝇有着易于饲养、繁殖力强、生长周期短、遗传背景明确等优势。正是因为这些得天独厚的优势,果蝇吸引了越来越多的研究者,许多生命现象和规律在果蝇中也被揭示,如胚胎发育、先天性免疫和昼夜节律等成果均先后获得了诺贝尔生理医学奖,有效推动了生命医学发展[4~6]。按照世界果蝇大会和美国Bloomington的注册信息,目前全世界有近3000个果蝇实验室,其中绝大多数在美国。根据复旦大学举办的2017年第四届中国果蝇大会统计,目前国内也已有200多个独立果蝇实验室。
为促进生命科学的快速发展,西方发达国家最先建立了相关的果蝇资源库:美国建有Bloomington 果蝇存储中心,主要储存果蝇转基因干扰品系(哈佛大学和清华大学构建)、果蝇突变体、定点激活品系(哈佛大学和清华大学构建)和Gal4品系;欧洲维也纳VDRC果蝇存储中心,主要构建和储存转基因干扰品系;日本NIG存储中心,构建并储存转基因干扰和突变体品系。目前,国内主要有三家单位构建和存储果蝇品系:上海果蝇平台,主要储存转基因干扰、突变体和Gal4品系;广州医科大学,主要构建和存储突变体品系;清华大学果蝇中心(Tsinghua Fly Center),主要研发转基因技术,构建及储存转基因干扰、转基因激活以及突变体品系。目前,国内外果蝇资源库的建立主要依靠3个转基因技术:(1)基于CRISPR/Cas9的基因突变系统,实现基因特异性敲除(knock-out)或敲入(knock-in);(2)基于RNA干扰(RNA interference,RNAi)原理研发的条件性转基因干扰技术,在特定组织、器官或细胞中高效敲低(knock-down)目的基因的表达;(3)基于CRISPR/ dCas9的flySAM转录激活(CRISPR transcriptional activation,CRISPRa)系统,提高目的基因的转录水平[7~13]。
本文主要介绍位于领域前沿的CRISPR/Cas9基因突变系统、转基因RNAi和转录激活系统的原理、流程以及未来发展方向,希望让更多的果蝇同行了解并掌握这些前沿的果蝇遗传学基因表达调控技术,了解构建相关果蝇资源品系的重要性,进而推动生命医学研究发展。
1 CRISPR/Cas9基因突变系统
基因是传递遗传信息的基本单位,在个体发育、疾病和衰老中起决定性作用。对基因进行突变是研究其功能的基本方法之一。基因突变分为自然突变和诱导突变。自然突变指自然条件下发生的突变,虽在生物界广泛存在,但突变的频率较低,为提高基因突变率,加快基因功能的研究,发展出化学诱变(甲基磺酸乙酯或N-乙基-N-亚硝基脲等致癌剂)、物理诱变(γ/x-射线等高能射线)或转座子转座等方式,广泛用于正向遗传筛选。然而以上这些诱变具有随机性,无法定点突变,增加了后续确定突变位点的难度[14,15]。早期锌指核酸酶(Zinc-finger nucleases, ZFNs)系统和转录激活子样效应物核酸酶(transcription activator-like effector nucleases, TALENs)系统等靶向性突变的工具可在特定位置引入双链DNA的断裂,引发细胞内部的DNA修复机制。但这两种系统的靶向性依赖于特异性结合DNA序列的蛋白模块,因此其构建成本高,操作步骤繁琐[15,16]。与上面两种系统相比,2013年出现的CRISPR/Cas9技术具有显著的优势[17]。CRISPR/Cas9
来自于细菌与古细菌的获得性免疫系统,用于抵御病毒的侵染。当噬菌体入侵时,crRNA、tracrRNA与Cas9蛋白形成复合体,识别噬菌体DNA序列的前间区序列邻近基序(protospacer adjacent motif,PAM,序列为NGG三碱基区段),其中crRNA以碱基互补的方式与PAM相邻的DNA序列结合,使双链结构打开,tracrRNA激活Cas9切割活性,在PAM位点上游第三个核苷酸附近进行切割使DNA 双链断裂,从而抵抗病毒入侵。基于此系统研发出的CRISPR/Cas9基因突变系统只包含两个重要成分,一个是具有DNA双链切割活性的Cas9蛋白,另一个是具有导向功能的sgRNA (small guide RNA)。Cas9蛋白可以与sgRNA结合,在sgRNA的引领下通过碱基互补配对靶向目的DNA,借助Cas9内切酶活性使靶位点发生DNA双链断裂,随后借助细胞的DNA修复引起基因突变(图1),如细胞利用非同源性末端链接(non-homologous end joining,NHEJ)途径可使基因发生移码突变或片段缺失和插入,而利用同源重组(homologous recombination, HR)修复途径通过提供供体DNA可实现基因的定点编辑或特定基因的插入[18,19]。
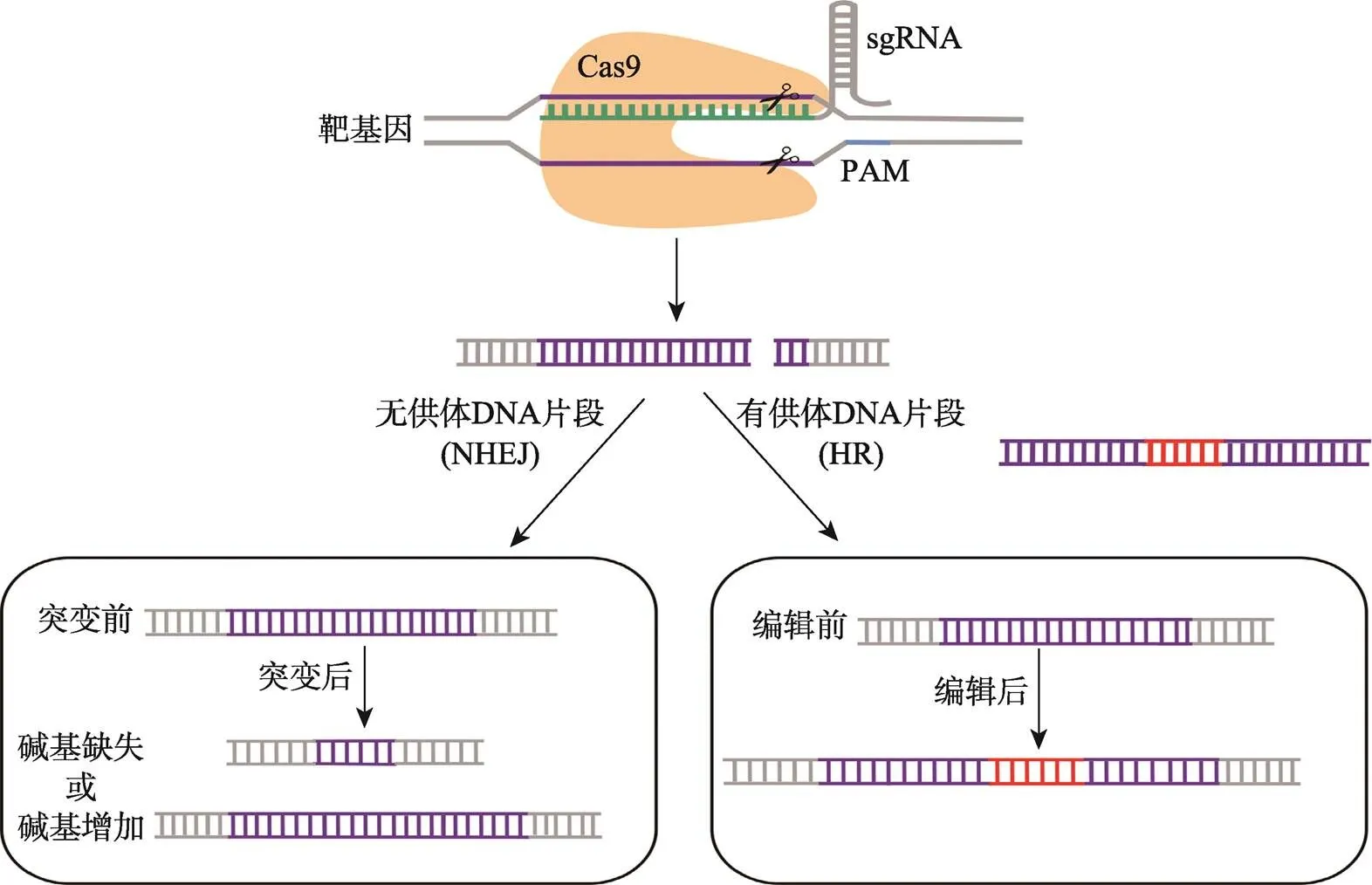
图1 CRISPR/Cas9介导的基因突变原理
目前,在模式动物果蝇中主要有4种基于CRISPR/Cas9构建突变体的途径[9]:(1)卵中共注射Cas9 mRNA和sgRNA;(2)卵中共注射两个可以分别表达Cas9和sgRNA的质粒;(3)在Cas9转基因果蝇卵中注射sgRNA质粒;(4)构建sgRNA转基因品系,然后和现存的Cas9转基因果蝇杂交。在这些方法中,方法(1)的成本高,方法(2)的突变效率低,方法(4)获得突变体的时间长。相比之下,方法(3)通过收集Cas9转基因果蝇卵,向其注射sgRNA质粒是最简单的突变体构建方式[20]。Cas9 转基因果蝇又分为两大类:一类是体细胞与生殖细胞均表达Cas9,比如-Cas9品系;另一类是仅生殖细胞特异表达Cas9,如-Cas9和-Cas9品系。其中,利用第二类转基因果蝇可以避免体细胞突变引起的致死,所以构建可遗传突变体的效率更高,同时sgRNA质粒可以高通量构建,便于突变体资源库的建立[20]。对于sgRNA元件,其转录可由不同的启动子调控,比如等,经过测试,用调控sgRNA转录、调控Cas9表达是构建果蝇突变体的最佳选择[10]。基于此,清华大学果蝇中心已建立成熟的突变体构建体系[20,21]:根据CRISPR/ Cas9系统原理,选择PAM位点上游20 nt 序列作为sgRNA序列,也可利用网站(http://www.flyrnai. org/crispr/)设计,并合成相应的寡核苷酸链,通过退火形成双链DNA片段并与酶切后的载体连接,构建-sgRNA质粒并测序确认,纯化后用注射缓冲液溶解(附表1);收集生殖细胞特异表达Cas9的-Cas9转基因果蝇(;-Cas9;或;;-Cas9)卵并注射-sgRNA质粒,注射了质粒的果蝇为G0,G0与携带平衡子的果蝇杂交,其后代G1挑选携带平衡子的个体并进行PCR检测,筛选得到突变体,之后再经过遗传杂交去除Cas9,即可得到相应的突变体品系,突变体品系经过测序确认后,编号(TH代表清华,M代表突变)并存储资源库(图2)。经过系统分析发现,sgRNA质粒注射浓度会影响突变效率:浓度低,突变效率低,但浓度高,毒副作用大,所以一般选择其注射浓度约为75 ng/μL。另外,sgRNA的GC含量也会影响突变效率,特别是临近PAM区域6个碱基的GC含量,与突变效率有极高的相关性,高突变率sgRNA,其GC含量往往大于60%。实际应用中,单个sgRNA往往效率低或突变后的基因仍有功能,因此为了获得基因功能丧失突变体(null mutant)并提高突变效率,可利用两个sgRNA,引导Cas9在同一基因的不同位置发生切割,达到去除部分基因序列的目的。上述优化条件,为进一步提高突变体构建效率提供了依据[22]。
利用CRISPR/Cas9系统,不仅可以进行基因突变,也可以借助带有左右同源臂的供体,将外源片段引入基因组或敲除特定片段,实现基因的精准编辑。但突变体的筛选除了利用敲入的标记基因外,绝大多数依靠提取基因组、PCR甚至测序,且不同基因可能由于所处染色质位置不同,或者对发育的作用不同,突变效率差异很大,对于这些基因,筛选其突变体成本高,且耗时费力。因此如何便捷快速筛选所需突变体仍然是该系统亟需解决的问题,这也是这套系统应用以来还没有建立其大规模突变资源库的原因。在研究基因在组织器官的功能时,也有一些实验室利用Gal4/UAS系统,条件性控制Cas9的表达,在sgRNA的作用下产生突变,但是这套系统仍有较大的局限性,比如在组织器官中产生突变的细胞数量少,检测确认困难,且由于突变细胞的随机性产生了表型的多样性,分析困难,所以条件性突变系统应用范围较小[23,24]。
2 条件性转基因RNA干扰系统
RNA干扰是指细胞中的单链或双链RNA,将与其互补的内源性mRNA降解,从而导致基因表达沉默的现象。模式动物果蝇中主要有两个RNAi通路,小干扰RNA(small interference RNA,siRNA)通路和微小RNA(microRNA,miRNA)通路[25,26]。具体来说:(1) siRNA通路,某些转录产物、病毒入侵产生的RNA以及人工DNA载体转录的RNA,可以形成长双链RNA(long dsRNA),经过Dicer2切割,产生21~23 bp的siRNA,其中一条RNA链整合到含有Ago2的RNA沉默复合体(RNA-induced silencing complex,RISC)中,通过碱基配对找寻目的mRNA,结合并将其降解;(2) miRNA通路,内源或人工DNA载体转录出带茎环结构的pri-miRNA,经过核酶Drosha切割去除首末端,产生的pre-miRNA随后出核被Dicer1加工,形成22~24 nt的miRNA,其中一条RNA链可以整合到含有Ago1的沉默复合体里,如果其序列与mRNA 3′端UTR结合可以抑制该基因的翻译;如果其序列与mRNA的可编码序列高度匹配,则同siRNA通路类似,可以将mRNA降解。
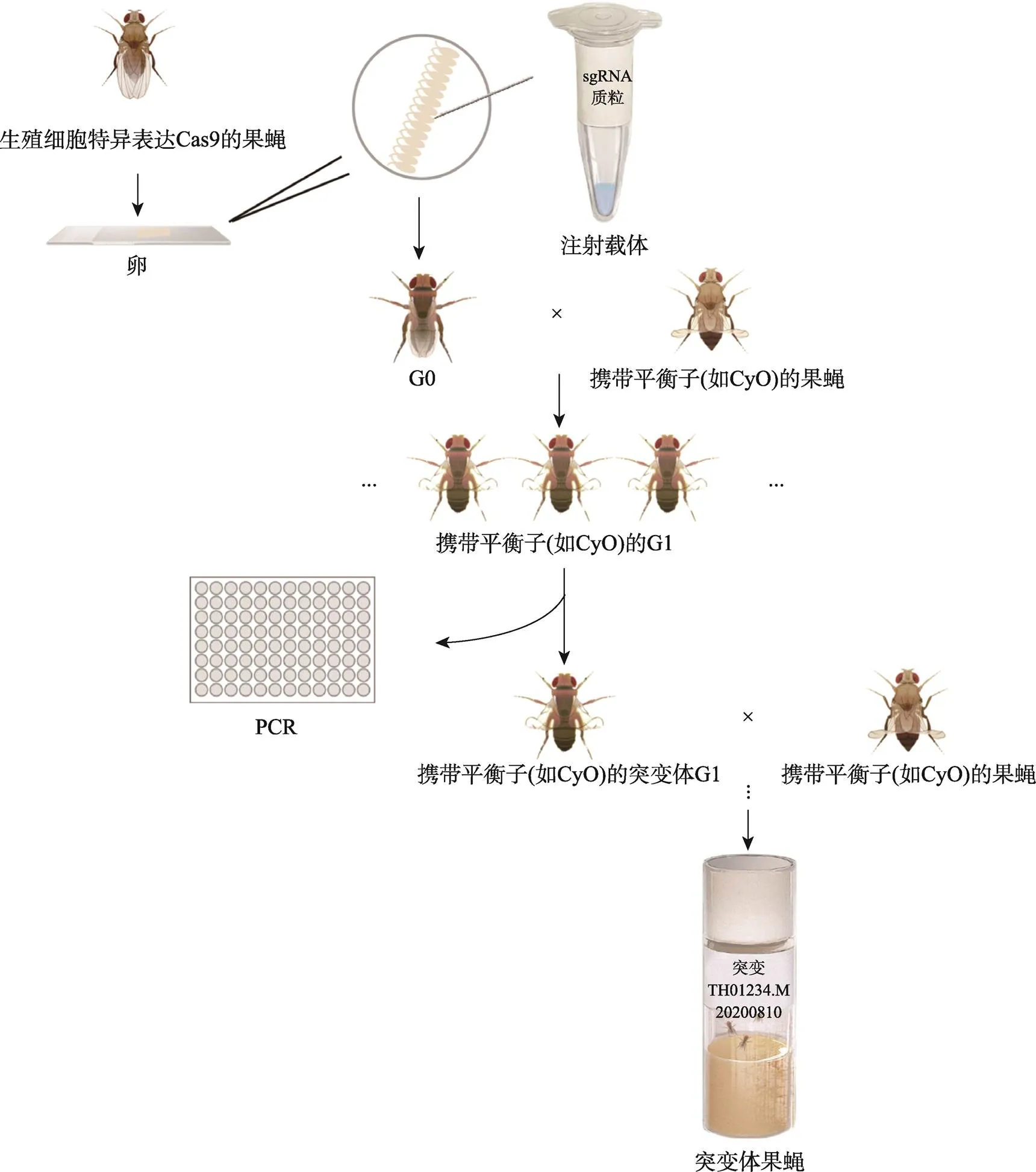
图2 突变体果蝇构建体系
果蝇中转基因RNAi技术的利用主要依赖于Gal4/UAS二元系统,Gal4作为转录激活因子能够激活UAS下游DNA序列的转录。因此构建转基因干扰品系后,与在细胞、组织或器官中特异表达Gal4果蝇品系杂交,即可实现条件性敲低目的基因[27]。此二元系统,只有当Gal4和UAS同时存在于同一果蝇中时,干扰才会发生,所以单独构建转基因干扰品系便于品系的保存和相应资源库的建立。借助其他调控因子,可以实现对目的基因的时间特异性调控。例如,Gal80是Gal4的负调控因子,能够抑制Gal4的转录激活作用[27]。温度敏感型Gal80ts,是将intein序列整合进Gal80,形成的融合蛋白在18℃时会释放出intein,从而保持Gal80活性,抑制Gal4;而在29℃时,intein稳定存在于Gal80中,导致Gal80失活,从而解除Gal80对Gal4的抑制,激活下游DNA的转录。因此借助于温度的改变,Gal80ts的应用可以控制RNA干扰发生的时间[28]。
转基因干扰技术是近些年研究基因功能重要的工具,由第一代逐步发展出二代、三代、新一代转基因干扰技术。第一代转基因干扰技术最初由VDRC和NIG研发,依据siRNA途径原理构建能够转录长双链RNA (300~600 bp)的载体,通过转座酶介导载体随机整合到果蝇基因组中[29,30]。日本、奥地利分别利用此技术构建了大量相关品系,建立了各自资源库。由于长双链RNA能够在Dicer2的切割下产生数百个siRNA,该技术容易有较高的假阴性和假阳性。假阴性的产生是由于:(1)长双链RNA干扰效率低;(2)整合位点转录水平低;(3)载体设计缺陷。假阳性的产生是由于:(1)长双链RNA脱靶效应;(2)整合时破坏了发育相关重要基因;(3) 载体本底转录水平高。二代技术由霍华德休斯医学研究所和哈佛大学合作研发,采用phiC31整合酶介导的attP/attB定点整合系统。phiC31整合酶是序列特异性重组酶,能够识别attB和attP位点特定序列并切割,经过连接后载体整合到基因组上,排除了转座酶介导的随机整合引起的位置效应[31]。三代技术主要由哈佛大学主导研发,采用miRNA途径,即以miR1的构型设计shRNA,调控目的基因。依赖miRNA通路,借助高效核酸内切酶,可以提高成熟miRNA的数量,从而显著提升干扰效率。另外,shRNA设计简单,转录产物均一,便于与基因组比对,大大降低脱靶率。尤其重要的是,该技术首次实现对果蝇雌性生殖细胞基因的干扰调控[12,31]。哈佛大学、清华大学和日本NIG利用此三代技术,构建了上万株品系,并在其国家的资源库保存。近些年,此技术和相关品系的应用大大促进了发育、神经、干细胞等相关学科发展。
然而,在使用三代转基因干扰技术调控目的基因时,本研究团队发现该技术存在以下缺点:(1)不能调控高表达基因;(2)不能同时调控多个基因;(3)该系统存在本底转录水平高的现象,在没有Gal4驱动时,某些组织仍有相当高的转录水平,从而影响果蝇发育,造成某些重要基因的转基因干扰果蝇无法纯合。为了解决这些问题,清华大学果蝇实验室研发了新一代转基因干扰技术[11,32]。新一代干扰技术拥有以下特点:(1)使用代替基因作为筛选标记,避免对果蝇神经系统及行为造成影响;(2)利用attP/attB系统,将转基因干扰载体定点整合到染色体上,避免了载体随机插入造成的位置效应和假阳性结果;(3)使用两个gypsy序列,可以加倍增强shRNA转录水平;(4)该系统使用人工设计合成的启动子DSCP(synthesized core promoter)替代三代技术的启动子,降低了系统的本底转录水平,显著降低了该系统的假阳性[33],提升了纯合体的比例,将纯合子比例从85%提升到99%,便于保存和早期发育遗传学分析;(5)采用miR1构型调控目的基因,干扰效率高,脱靶率低,降低了假阳性和假阴性;(6)该系统可应用于雌性生殖细胞基因的调控;(7)突破性地实现了在同一个载体上克隆多个shRNA,可同时有效调控多个不同基因,如多个乙酰化基因或PRC1复合体等,也可针对同一基因,设计多个shRNA,提高干扰效率,实现对高转录基因的干扰,如组蛋白或微管蛋白等。
利用此系统构建转基因干扰果蝇品系,首先需要在网站(biodev.cea.fr/DSOR/DSIR.html)预测shRNA序列,并构建shRNA质粒,收集表达整合酶(phiC31)和携带attP位点果蝇(; attP40;或;;attP2)的卵并注射shRNA质粒,卵孵化后的果蝇即G0为浅红眼,G0与浅红眼果蝇()杂交,去除整合酶并根据眼睛颜色深浅确定转基因果蝇,深红眼果蝇是携带转基因的果蝇,但由于深红眼与浅红眼色差不大,初学者挑选深红眼需要在孵化成虫后6个小时内进行。深红眼转基因果蝇(G1)进一步与携带平衡子的果蝇杂交,得到转基因干扰果蝇品系,经提取DNA测序确认后,编号(TH代表清华,R代表RNAi)并存储资源库(图3)。利用此技术,清华大学果蝇中心针对果蝇中与人类基因高度同源的致病基因构建了数千株品系。
与基因突变相比,构建转基因干扰果蝇周期短、成本低、便于保存,特别是在研究因基因缺失而产生组织器官的表型时,转基因干扰表型均一,可重复性高,所以转基因干扰是研究基因功能的重要工具。虽然新一代转基因干扰技术相比于第三代技术有了多维度的改进,但其单个shRNA调控基因表达的效率较之第三代并无显著提升,这是由新一代系统使用的DSCP启动子本身转录水平决定的。因此,继续研发下一代转基因干扰技术,提升干扰效率,对发育生物学研究具有重要意义。
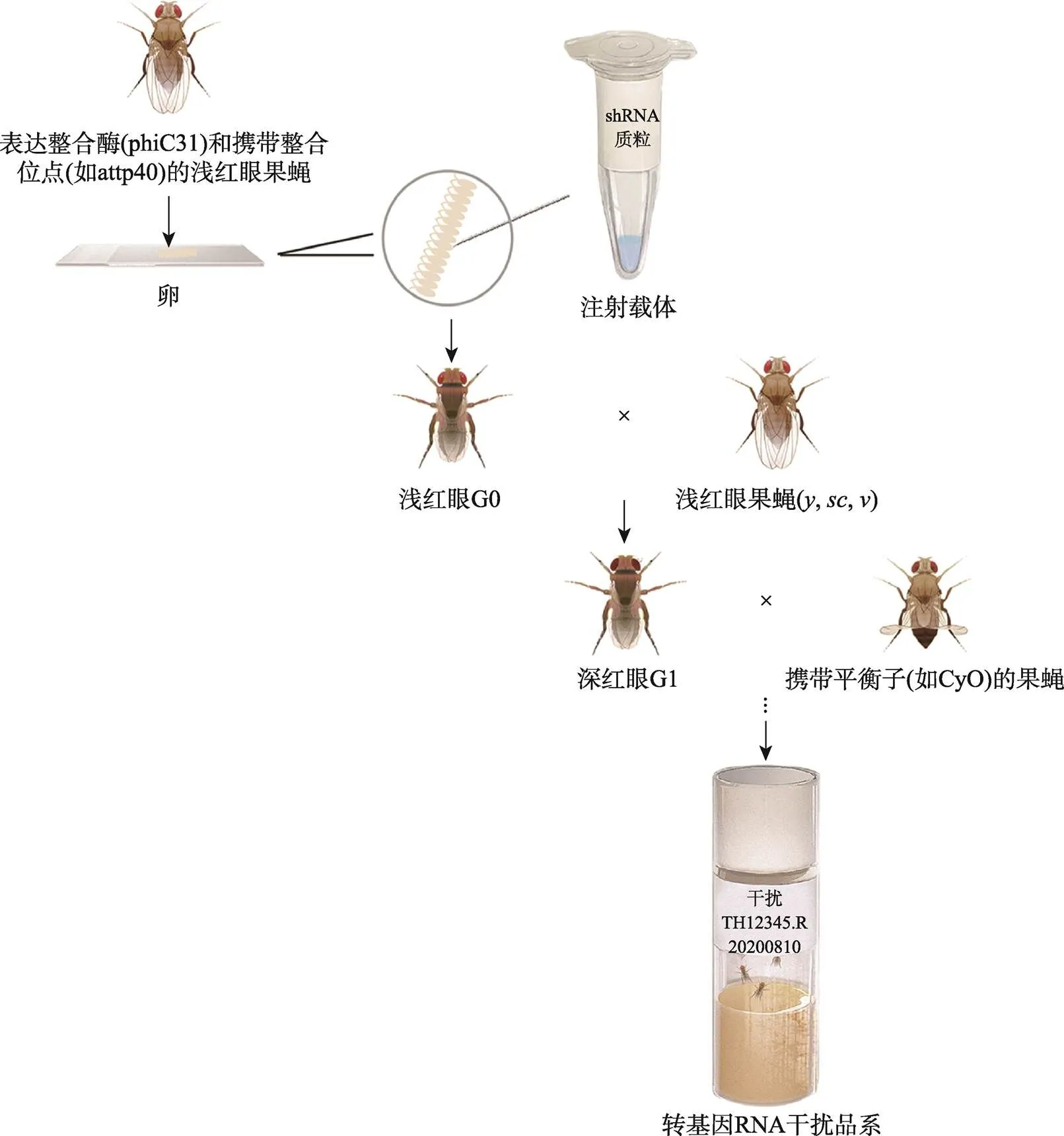
图3 RNA干扰果蝇构建体系
3 flySAM转录激活系统
研究基因的功能、挽救敲低或敲除基因产生的表型,都离不开目的基因的过表达。传统的过表达方式主要利用Gal4/UAS系统,组织特异性驱动目的基因cDNA的转录,实现过表达的目的[34],但是,此方式存在相关载体构建复杂,时间成本高,难以同时实现多个基因的过表达等缺点,尽管利用现有cDNA质粒资源库可省略克隆步骤,但过表达所用序列只能依靠现有质粒资源库,且仍未解决难以同时实现多个基因过表达的问题[35],因此亟需一种简单的基因过表达或基因激活系统。
近年来,对CRISPR/dCas9系统的深入研究为转录激活系统(CRISPR/Cas9-based transcriptional activation,CRISPRa)的开发奠定了基础[36]。通过对Cas9蛋白的内切酶活性位点进行点突形成dCas9,使其失去DNA双链切割能力,但仍可以与sgRNA结合,引起其构象的改变,如果dCas9羧基端融合转录激活因子,则能够在sgRNA引导下靶向特定位置并激活相应基因的转录。与构建CRISPR/Cas9突变系统类似,该激活系统也仅需20 bp sgRNA就可实现基因的转录激活。
为了在果蝇中建立CRISPRa系统,许多实验室进行了不同的尝试。VPR(VP64-p65-Rta)系统可应用于果蝇体外细胞系或个体体内,能够激活靶基因的转录并产生相应的发育表型,但其效率较低,且需要两个sgRNA才能完成对基因的激活,增加了载体构建的成本及时间,此外,应用时需要提前将两个元件(如Gal4和UAS-dCas9-VPR、Gal4和sgRNA、UAS-dCas9-VPR和sgRNA)整合在果蝇上,整合周期长[37,38]。为了进一步提高激活效率,降低毒副作用,简化遗传操作步骤,通过对比各种转录激活因子及其组合分析,清华大学果蝇中心主导研发了flySAM(synergistic activation mediator)系统[13,39]。此系统包括UAS-dCas9激活元件和sgRNA元件(图4)。对于UAS-dCas9激活元件,dCas9转录激活融合蛋白受Gal4/UAS二元系统调控,可以表达dCas9-VP64和T2A介导的MCP-p65-HSF1。sgRNA元件除了有靶向目的基因的引导序列外,还有能被MCP识别的MS2序列。MCP序列能够结合MS2,因此在sgRNA的带领下,dCas9-VP64结合到靶位点处,同时,MCP-p65-HSF1与MS2结合,从而增加了转录激活元件,提高效率。为了简化遗传操作的步骤,将以上两个元件整合到同一载体上,其注射产生的转基因品系直接与现存的Gal4品系杂交,一步实现基因表达的激活。
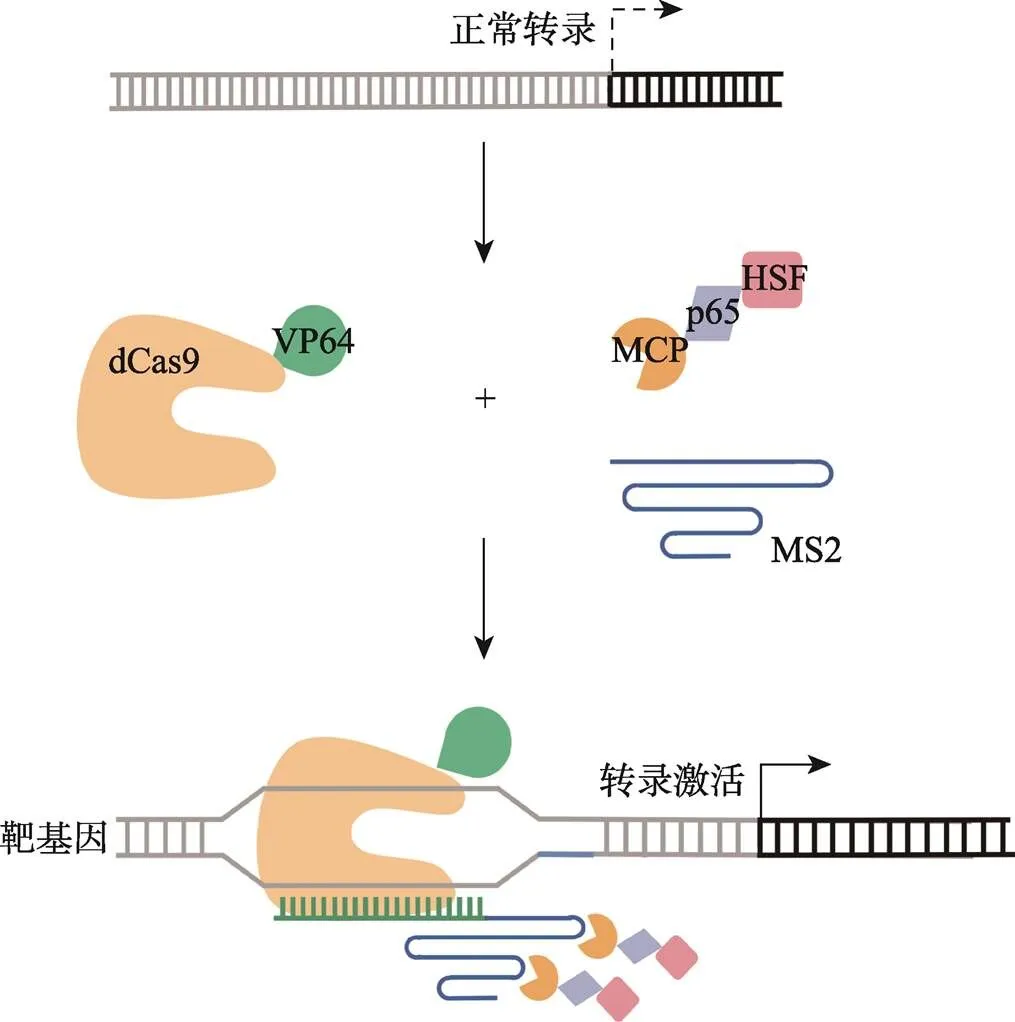
图4 flySAM基因转录激活模式图
与传统方法比,flySAM有显著的优势:(1)载体仅需克隆20 bp DNA,成本低,操作简单;(2)实现原位基因的转录激活,与传统Gal4/UAS强制过表达相比,能够减少副作用;(3)可以同时克隆多个sgRNA,便于激活多个基因;(4)许多基因如泛素化连接酶经过转录后剪切修饰,形成多个不同的成熟转录本,进而翻译出多个蛋白异构体,不同异构体可能发挥不同功能。使用传统过表达方式只能过表达其中一种异构体,为研究基因功能造成了一定障碍。使用flySAM系统在基因组水平上激活基因表达,可同时提高目的基因所有转录本的转录水平,在研究基因功能时有得天独厚的优势。
此系统的效率高度依赖于sgRNA中20 bp靶向序列的选择,为了提高flySAM的效率,对sgRNA参数进行探究后发现:(1)通常sgRNA GC碱基含量愈高,激活愈显著;(2)存在位置效应,激活有效区主要集中在转录起始位点上游150 bp左右,当sgRNA远离起始位点上游600 bp时,尽管GC含量高,也失去激活能力;(3) sgRNA靶向DNA非模板链时,与靶向模板链相比激活效果更加显著[40]。完备的工作效率测试和工作条件探索极大地提升了flySAM系统应用的高效性和便利性,完整的sgRNA设计标准对所有希望使用该系统的科研工作者开放,帮助他们实现高效的基因转录激活。构建转录激活果蝇时首先根据已知优化条件设计高效sgRNA,并构建质粒,质粒的注射及转基因激活果蝇的筛选与转基因干扰果蝇构建流程相似,得到转录激活品系后,测序确认,编号(TH代表清华,A代表激活)并存储资源库(图5)。基于该系统清华大学构建了丰富的转基因品系资源库,与基因突变和基因干扰技术互相补充,为疾病模型构建、药物功能筛选等科研工作提供了极大助力。
目前flySAM系统仍存在一定的缺陷,比如利用flySAM系统激活某些基因时,与转基因干扰或传统过表达相比,出现表型的概率较低。另外,虽然该系统可以在体细胞中实现高效、特异的基因转录激活,但在生殖系统中效果较差并有降低胚胎孵化率的副作用。因此为了进一步提高该系统的激活效率,还可以对载体进行改造,如测试其他的转录激活因子等。

图5 flySAM转基因果蝇构建体系
4 结语与展望
果蝇是生物医学领域经典的遗传、发育模式动物,各项转基因调控技术可与其他模式动物相互借鉴。得益于果蝇得天独厚的优势,以及遗传学工具的多样性,特别是基因编辑、转基因干扰和基因过表达的应用,在发育、神经、代谢以及免疫等领域相关的科研成果不断涌现。近10年来,国内果蝇实验室迅猛发展,科研成果大幅度增加,对转基因品系需求日益增长。尽管果蝇饲养简单,但品系维持成本非常高,限制了果蝇转基因资源库的规模。清华大学果蝇中心(https://thfc.zzbd.org) (图6)及实验室研发的包括基因突变、转基因干扰和转基因激活系统在内的世界前沿技术,被国内外科研工作者广泛应用,但各项技术仍然有改进和提升空间。在生命科学领域高速发展、高通量技术逐渐泛用化的时代背景下,各类模式生物的品系资源库的构建愈发重要。目前已有的品系构建技术基本可以覆盖科研需求,但仍存在调控部分基因效率较低,构建品系周期较长,人力成本较高等问题。在提升系统工作效率的同时,优化品系构建的方法、缩短品系构建周期是未来研发工作的重点所在。清华果蝇资源库与国内其他品系资源库的建立大大改善了之前国内果蝇品系构建周期长、运输成本高、运输途中果蝇死亡率高等众多不利情况,更重要的是,利用自身研发的前沿转基因技术,可免受国外技术的知识产权限制。

图6 清华果蝇中心平台
附加材料详见文章电子版www.chinagene.cn。
附表1 果蝇培养所需试剂耗材及注射缓冲液配方
Supplementary Table 1 Materials forculture and formulations of injection buffer

项目备注 果蝇食物混合玉米面(济南佳硕生物技术有限公司)、红糖(网山)、白砂糖(龙二)、酵母(安琪)、琼脂(科百奥)、乙醇(沪试)、丙酸(沪试)、抗生素(Amresco) 果蝇瓶塞济南佳硕生物技术有限公司 果蝇管绍兴上虞八达通信电器有限公司 注射缓冲液0.1 mM磷酸钠缓冲液(pH 6.8)、5 mM KCl
[1] Esquerda-Canals G, Montoliu-Gaya L, Güell-Bosch J, Villegas S. Mouse models of Alzheimer's disease., 2017, 57(4): 1171–1183.
[2] Jin HL, He R, Oyoshi M, Geha RS. Animal models of atopic dermatitis., 2009, 129(1): 31–40.
[3] High KA, Roncarolo MG. Gene therapy., 2019, 381(5): 455–464.
[4] Nüsslein-Volhard C, Wieschaus E. Mutations affecting segment number and polarity in., 1980, 287(5785): 795–801.
[5] Hoffmann JA. The immune response of., 2003, 426(6962): 33–38.
[6] Konopka RJ, Benzer S. Clock mutants of., 1971, 68(9): 2112–2116.
[7] Housden BE, Muhar M, Gemberling M, Gersbach CA, Stainier DYR, Seydoux G, Mohr SE, Zuber J, Perrimon N. Loss-of-function genetic tools for animal models: cross-species and cross-platform differences., 2017, 18(1): 24–40.
[8] Xu RG, Wang X, Shen D, Sun J, Qiao HH, Wang F, Liu LP, Ni JQ. Perspectives on gene expression regulation techniques in., 2019, 46(4): 213–220.
[9] Xu J, Ren XJ, Sun J, Wang X, Qiao HH, Xu B-W, Liu LP, Ni JQ. A toolkit of CRISPR-based genome editing systems in., 2015, 42(4): 141–149.
[10] Mohr SE, Smith JA, Shamu CE, Neumüller RA, Perrimon N. RNAi screening comes of age: improved techniques and complementary approaches., 2014, 15(9): 591–600.
[11] Qiao HH, Wang F, Xu RG, Sun J, Zhu R, Mao D, Ren XJ, Wang X, Jia Y, Peng P, Shen D, Liu LP, Chang ZJ, Wang GR, Li S, Ji JY, Liu QF, Ni JQ. An efficient and multiple target transgenic RNAi technique with low toxicity in., 2018, 9(1): 4160.
[12] Ni JQ, Zhou R, Czech B, Liu LP, Holderbaum L, Yang-Zhou D, Shim HS, Tao R, Handler D, Karpowicz P, Binari R, Booker M, Brennecke J, Perkins LA, Hannon GJ, Perrimon N. A genome-scale shRNA resource for transgenic RNAi in., 2011, 8(5): 405–407.
[13] Jia Y, Xu RG, Ren XJ, Ewen-Campen B, Rajakumar R, Zirin J, Yang-Zhou D, Zhu RB, Wang F, Mao D, Peng P, Qiao HH, Wang X, Liu LP, Xu BW, Ji JY, Liu QF, Sun J, Perrimon N, Ni JQ. Next-generation CRISPR/Cas9 transcriptional activation inusing flySAM., 2018, 115(18): 4719–4724.
[14] Anderson P. Mutagenesis., 1995, 48: 31–58.
[15] Ren XJ, Holsteens K, Li HY, Sun J, Zhang YF, Liu LP, Liu QF, Ni JQ. Genome editing in: from basic genome engineering to the multipurpose CRISPR-Cas9 system., 2017, 60(5): 476–489.
[16] Bak RO, Gomez-Ospina N, Porteus MH. Gene editing on center stage., 2018, 34(8): 600–611.
[17] Gaj T, Gersbach CA, Barbas III CF. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering., 2013, 31(7): 397–405.
[18] Musunuru K. The hope and hype of CRISPR-Cas9 genome editing: a review., 2017, 2(8): 914–919.
[19] Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering., 2014, 157(6): 1262–1278.
[20] Ren XJ, Sun J, Housden BE, Hu YH, Roesel C, Lin SL, Liu LP, Yang ZH, Mao D, Sun LZ, Wu QJ, Ji JY, Xi JZ, Mohr SE, Xu J, Perrimon N, Ni JQ. Optimized gene editing technology forusing germ line-specific Cas9., 2013, 110(47): 19012–19017.
[21] Peng P, Wang X, Shen D, Sun J, Jia Y, Xu RG, Zhu LF, Ni JQ. CRISPR-Cas9 mediated genome editing in., 2019, 9(2): e3141.
[22] Ren XJ, Yang ZH, Xu J, Sun J, Mao DC, Hu YH, Yang SJ, Qiao HH, Wang X, Hu Q, Deng P, Liu LP, Ji JY, Li JB, Ni JQ. Enhanced specificity and efficiency of the CRISPR/Cas9 system with optimized sgRNA parameters in., 2014, 9(3): 1151–1162.
[23] Port F, Chen H, Lee T, Bullock S. Optimized CRISPR/Cas tools for efficient germline and somatic genome engineering in., 2014, 111(29): E2967–E2976.
[24] Xue ZY, Wu MH, Wen KJ, Ren MD, Long L, Zhang XD, Gao GJ. CRISPR/Cas9 mediates efficient conditional mutagenesis in., 2014, 4(11): 2167–73.
[25] Liu QH, Paroo Z. Biochemical principles of small RNA pathways., 2010, 79: 295–319.
[26] Williams L, Carles CC, Osmont KS, Fletcher JC. A database analysis method identifies an endogenous trans-acting short-interfering RNA that targets theARF2, ARF3, and ARF4 genes., 2005, 102(27): 9703–9708.
[27] Brand AH, Perrimon N. Targeted gene expression as a means of altering cell fates and generating dominant phenotypes., 1993, 118(2): 401–415.
[28] McGuire SE, Le PT, Osborn AJ, Matsumoto K, Davis RL. Spatiotemporal rescue of memory dysfunction in., 2003, 302(5651): 1765–1768.
[29] Lam G, Thummel CS. Inducible expression of double- stranded RNA directs specific genetic interference in., 2000, 10(16): 957–963.
[30] Kennerdell JR, Carthew RW. Heritable gene silencing inusing double-stranded RNA., 2000, 18(8): 896–898.
[31] Ni JQ, Markstein M, Binari R, Pfeiffer B, Liu LP, Villalta C, Booker M, Perkins L, Perrimon N. Vector and parameters for targeted transgenic RNA interference in., 2008, 5(1): 49–51.
[32] Wang F, Qiao HH, Xu RG, Sun J, Zhu RB, Mao DC, Ni JQ. pNP transgenic RNAi system manual in., 2019, 9(3): e3158.
[33] Pfeiffer BD, Jenett A, Hammonds AS, Ngo T-TB, Misra S, Murphy C, Scully A, Carlson JW, Wan KH, Laverty TR, Mungall C, Svirskas R, Kadonaga JT, Doe CQ, Eisen MB, Celniker SE, Rubin GM. Tools for neuroanatomy and neurogenetics in., 2008, 105(28): 9715–9720.
[34] Rørth P. A modular misexpression screen indetecting tissue-specific phenotypes., 1996, 93(22): 12418–12422.
[35] Wei P, Xue W, Zhao Y, Ning G, Wang JW. CRISPR-based modular assembly of a UAS-cDNA/ORF plasmid library for more than 5500genes conserved in humans., 2020, 30(1): 95–106.
[36] Sapranauskas R, Gasiunas G, Fremaux C, Barrangou R, Horvath P, Siksnys V. TheCRISPR/Cas system provides immunity in., 2011, 39(21): 9275–9282.
[37] Lin SL, Ewen-Campen B, Ni XC, Housden BE, Perrimon N.transcriptional activation using CRISPR/Cas9 in., 2015, 201(2): 433–442.
[38] Ewen-Campen B, Yang-Zhou D, Fernandes VR, González DP, Liu LP, Tao R, Ren XJ, Sun J, Hu YH, Zirin J, Mohr SE, Ni JQ, Perrimon N. Optimized strategy forCas9-activation in., 2017, 114(35): 9409–9414.
[39] Jia Y, Shen D, Wang X, Sun J, Peng P, Xu RG, Xu BW, Ni JQ. FlySAM transgenic CRISPRa system manual., 2019, 9(2): e3147.
[40] Mao DC, Jia Y, Peng P, Shen D, Ren XJ, Zhu RB, Qiu YH, Han YT, Yu JC, Che QY, Li YT, Lu XY, Liu LP, Wang Z, Liu QF, Sun J, Ni JQ. Enhanced efficiency of flySAM by optimization of sgRNA parameters in., 2020, 10(12): 4483–4488.
The cutting edge of gene regulation approaches in model organism
Yuting Han, Bowen Xu, Yutong Li, Xinyi Lu, Xizhi Dong, Yuhao Qiu, Qinyun Che, Ruibao Zhu, Li Zheng, Xiaochen Li, Xu Si, Jianquan Ni
Transgenic gene regulatory techniques play a critical role in biomedical fields since they are essential for researchers to study the molecular mechanisms of development and diseases. The currently prevalent transgenic techniques include specific gene mutation, transgenic RNAi and targeting activation, etc. Specifically, various gene regulatory tools have been developed to temporarily and spatially modulate genes in, which is a typical model organism with evolutionally conserved genome and without ethical issues in scientific research. Here, we introduce several gene regulatory techniques commonly used in, includingthe principle, procedure, toolbox and related transgenic resource of CRISPR/Cas9-triggered heritable gene mutation system, next-generation transgenic RNAi and CRISPR/dCas9-based transcriptional activation (flySAM) approach. We wish to take this opportunity to raise the awareness of the importance of transgenic gene regulatory techniques development and related resource construction and thus to promote biomedical research progression.
; gene regulation; CRISPR/Cas9; gene editing; transgenic RNAi; CRISPRa
2021-10-07;
2021-12-21;
2022-01-04
国家自然科学基金项目(编号:20181300988,20201300797)和科技部国家重点研发计划(编号:2016YFE0113700)资助[Supported by the National Natural Science Foundation of China (Nos. 20181300988, 20201300797) and the National Key Technology Research and Development Program of the Ministry of Science and Technology of the People’s Republic of China (No. 2016YFE0113700)]
韩玉婷,在读博士研究生,研究方向:基因调控技术。E-mail: hanyt19@mails.tsinghua.edu.cn
倪建泉,博士,研究员,研究方向:发育遗传学。E-mail: nijq@mail.tsinghua.edu.cn.
10.16288/j.yczz.21-347

倪建泉课题组以果蝇为模式动物,开发遗传工具,研究表观遗传蛋白与信号通路在干细胞稳态中发挥的作用。课题组开发了包括基于CRISPR的基因编辑技术、基因定点激活技术和新一代转基因干扰技术在内的一系列果蝇遗传学新技术,实现在个体水平上特异调控细胞组织器官基因的表达。利用这些技术,建立转基因果蝇资源库,并进行了发育与干细胞相关的研究,主要成果包括确立组蛋白H1对干细胞的直接和间接调控作用;发现HP1c直接参与Notch信号的抑制,调控消化道干细胞稳态;确定了3个组蛋白乙酰化酶在果蝇眼睛发育中的协同作用及机制。相关研究成果发表在、、等期刊上。现作为课题组长,参与科技部支撑计划和发育代谢重大研发计划等。
(责任编委: 韩俊海)

