桑细菌性枯萎病病原菌的分离鉴定与全基因组序列分析
罗龙辉 王继承 刘吉平


摘要
为明确华南蚕区桑树细菌性枯萎病主要病原菌的种类,本研究从广西、广东等地桑园采集了多份桑树细菌性枯萎病病样,采用分离培养、形态学鉴定和分子生物学鉴定的方法,对病原菌进行了分离鉴定,并通过药敏试验分析病原菌的耐药性。结果表明,结合柯赫氏法则,将分离到的桑枯萎病病原菌鉴定为产酸克雷伯氏菌Klebsiella oxytoca,编号AKKL001(菌种保藏号GDMCC No.1.1602)。该病原菌属革兰氏阴性菌,基因组全长6 149 586 bp,含有5 792个基因,GC含量55.80%;药敏试验结果表明,病原菌对氨苄西林、头孢唑啉、头孢呋辛、头孢噻吩、链霉素、麦迪霉素等抗生素具有耐受性。本研究报道了桑细菌性枯萎病病样上分离的产酸克雷伯氏菌,为桑细菌性枯萎病病原菌的研究提供理论依据,全基因组测序的結果为桑细菌性枯萎病的相关致病机制研究奠定了基础。
关键词
桑树;细菌性枯萎病;产酸克雷伯氏菌;全基因组
中图分类号:
S888.7, S436.5
文献标识码:A
DOI:10.16688/j.zwbh.2020522
Isolation, identification and wholegenome sequence analysis of the pathogen causing mulberry bacterial wilt disease
LUO Longhui,WANG Jicheng,LIU Jiping*
(College of Animal Science, Regional Sericulture Training Center For AsiaPacific,
South China Agricultural University, Guangzhou510642, China)
Abstract
In order to identify the main pathogens of mulberry bacterial wilt in the silkwormrearing area of South China, multiple mulberry samples with mulberry bacterial wilt were collected from mulberry fields in gardens in Guangxi and Guangdong. The pathogen was isolated and identified by culture separation, morphological and molecular identification. Meanwhile, drug sensitivity test was conducted to analyze the drug resistance of pathogenic bacteria. The results showed that, combined with Koch’s postulates, a strain of Klebsiella oxytoca, the pathogen of mulberry wilt disease, was isolated, designated as AKKL001 (GDMCC strain deposit no. 1.1602). The pathogen was a gramnegative bacterium with a fulllength genome sequence of 6 149 586 bp, including 5 792 genes, with a GC content of 55.80%. The results of drug sensitivity test showed that the pathogen was resistant to antibiotics such as ampicillin, cefazolin, cefuroxime, cephalothin, streptomycin and midecamycin. This study reported Klebsiella oxytoca isolated from samples of mulberry bacterial wilt disease, which provided a theoretical basis for the research on the pathogen of mulberry bacterial wilt, and the results of wholegenome sequencing provided the basis for the identification of the related pathogenic mechanism of mulberry bacterial wilt.
Key words
mulberry;bacterial wilt;Klebsiella oxytoca;genome
桑叶是寡食性昆虫家蚕的主要食物来源,作为药食两用的桑树,在我国蚕桑产业多元化中有着举足轻重的地位[1]。桑树病害一直影响着桑树产业的健康稳定发展,其中桑细菌病害,特别是桑细菌性枯萎病、桑青枯病、桑疫病等作为危害我国桑树产业的三类主要桑细菌性病害[2],近些年在各大蚕区的发生日趋严重。 桑细菌性枯萎病自2006年在浙江杭州发现以来,近几年迅速蔓延到整个华南蚕区,已严重影响蚕桑产业健康稳定的发展。桑细菌性枯萎病田间症状与茄科雷尔式菌Ralstonia solanacearum引起的桑青枯病极为相似,生产上多将二者混淆。Zhu等[3]2010年发现一种引起桑枯萎病的病原,但并非是茄科雷尔式菌,后经鉴定确定为肠杆菌属Enterobacter 细菌,从此开启了桑细菌性枯萎病病原的新篇章。之后Wang等[4]从浙江杭州桑枯萎病病样中分离到肠杆菌属的阿氏肠杆菌E.asburiae 和 Enterobacter sp.,再次明确了肠杆菌属是桑细菌性枯萎病病原的主要类群。2012年,戴凡炜等[5]从广东、广西收集的47份样本中分离到10株桑枯萎病病原菌,经鉴定分别为肠杆菌属 Enterobacter的阴沟肠杆菌E.cloacae和阿氏肠杆菌、克雷伯氏菌属 Klebsiella 的肺炎克雷伯氏菌K.pneumoniae、K.variicola和产酸克雷伯氏菌 K.oxytoca以及泛菌属Pantoea的菠萝泛菌P.ananatis等病原菌,首次提出了其他种属的细菌也可引起桑细菌性枯萎病。蒙娇荣等[6]从桑枯萎病病样中除分离到以上几种病原菌外,还分离到未确定种的3株肠杆菌属细菌和6株克雷伯氏菌属细菌,极大地丰富了桑细菌性枯萎病病原种类。由此可见,桑细菌性枯萎病病原菌种类复杂多样,探讨其病原种类对防治该细菌性病害具有重要的意义。
目前,研究者对桑细菌性枯萎病病原的研究多集中于肠杆菌属,已初步明确肠杆菌属病原菌的系统分类与致病机理,但对于其他属的病原菌研究较少。为了准确诊断桑细菌性枯萎病病原以控制其蔓延传播,本研究对采集于广西、广东等地多份桑枯萎病样本的病原菌进行分离,获得1株强致病力菌株,编号AKKL001。并对病原菌AKKL001进行形态特征观察、生理生化测定、致病性测定和全基因组分析,明确其系统发育特征,从而为桑细菌性枯萎病的防控提供科学依据。
1材料与方法
1.1材料
1.1.1桑枯萎病病株及其根际土壤样本
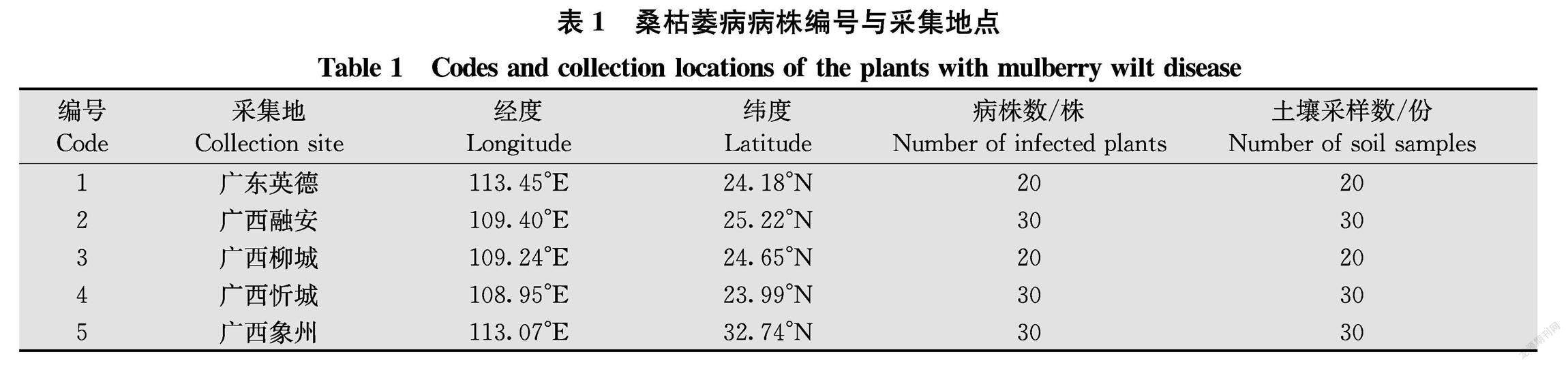
桑枯萎病病株及其根际土壤样本采集情况见表1。
1.1.2主要试剂
NA培养基、LB肉汤培养基、肠杆菌科细菌生化常规鉴定试剂盒购于广东环凯微生物科技有限公司。植物、土壤、细菌基因组DNA提取试剂盒购于生工生物工程(上海)股份有限公司; PCR反应的相关试剂购于北京擎科生物工程科技有限公司,引物由生工生物工程(上海)股份有限公司合成。
1.2方法
1.2.1桑树根部病原菌的分离纯化
取新采集的桑树根部组织用自来水冲洗干净后剪成约3~4 cm的根段,用75%乙醇漂洗15 s,无菌水冲洗3次;0.1%的升汞处理 5 min,再用无菌水冲洗3次。风干后,将根部样本置于无菌的研钵内,加入约5 mL无菌水充分研磨;取1 mL研磨液加入9 mL无菌水中,10倍梯度稀释5 次;分别取100 μL 10 4和10 5的研磨稀释液均匀涂布于NA培养基上,每个稀释度重复涂板3次。24 h后挑取单菌落于NA培养基上划线扩大培养,隔日挑取单菌落接种于LB斜面和平板,并在甘油中保存置于 80℃冰箱。
1.2.2AKKL001致病性测定
用接种环挑取一环斜面保存的单菌落于盛有100 mL LB培养液的250 mL三角瓶中,置于恒温振荡器,28℃、150 r/min培养24 h,3 000 r/min离心5 min,去上清,将菌体稀释为108cfu/mL的菌悬液备用。从桑园采集桑嫩枝(品种:‘伦40’)扦插到50 mL锥形瓶中,每瓶扦插2株,加入30 mL无菌水,用海绵封住瓶口。设试验组与空白对照两组,每组15个重复。24 h后,以第1瓶为空白对照往瓶中注入3 mL无菌水, 其余株注入3 mL上述菌液,置于恒温27℃的人工气候箱,设置光周期20 h(L∥D= 12 h∥8 h),湿度为80%RH,每天观察并记录发病情况,并对枯萎植株进行病原菌的再分离与PCR检测。选用生长40 d的桑树小苗,洗净,消毒并剪去1/3须根,移植到无菌土壤中,取5 mL 108 cfu/mL菌悬液灌根,置于27℃的人工气候箱(条件与上述条件相同),每天观察并记录发病情况。对回接后枯萎病株进行病原菌的分离与PCR验证(PCR引物 KLF1/KLR1:5′ATTGCGTAGAAGAG CCTGAA3′/ 5′CAGGACACGCAGTTAAGACC3′;拟扩增片段长度:926 bp)[7]。
1.2.3 AKKL001的形态学观察及生理生化性质的测定
从斜面挑取病原菌菌落接种于LB培养液,28℃培养24 h进行电镜制样观察,另取10 μL菌液进行革兰氏染色;参照《伯杰细菌鉴定手册》[8],选择12项肠杆菌科生理生化鉴定指标对病原菌进行测定。
1.2.4AKKL001全基因组测序
将分离的病原菌扩大培养后提取总DNA送至广州瑞科基因科技有限公司进行质量检验,质量检测合格的样品根据Illumina DNA文库构建标准流程,构建插入片段大小为500 bp的双末端测序文库。对质检合格的高通量测序文库,采用Illumina Hiseq高通量测序平台进行测序,测序模式:PE150(Pairend150)。由广州瑞科基因科技有限公司完成对基因组的测序、组装与基因的注释。
1.2.5AKKL001系统进化分析
细菌rRNA全长包括16S rRNA、23S rRNA、5S rRNA以及他们之间的间隔区序列。根据1.2.4的全基因组注释结果,将AKKL001核糖体RNA全长序列上传至NCBI(https:∥blast.ncbi.nlm.nih.gov/Blast)进行 BLAST比对,从GenBank中下载克雷伯氏菌属的rRNA基因序列用MUSCLE 3.8.31软件进行多序列比對,并利用MEGA X[9]软件,采用Maximum Composite Likelihood 模型通过邻接法(neighborjoining)构建系统发育树,bootstrap值设置为1 000。
平均核苷酸一致性(ANI)是反应基因组之间遗传距离的最有效指标之一,利用在线软件PATRIC(3.6.5)[10]的Similar Genome Finder查找并下载相近的细菌基因组,并利用OAT软件[11]基于ANI分析基因组之间的遗传距离。
1.2.6毒力因子与耐药基因分析
利用IslandViewer 4[12]对AKKL001的基因组岛(genomic island,GI)进行预测分析;通过对比毒力因子数据库VFDB[13]对AKKL001的毒力基因进行注释;利用CARD数据库[14]对耐药基因进行预测。
1.2.7药敏试验
选取氨苄西林、头孢唑啉、头孢呋辛、头孢噻吩、链霉素、麦迪霉素、头孢噻肟、氨曲南、头孢哌酮、头孢吡肟、头孢曲松、头孢他啶、氧氟沙星、哌拉西啉、头孢西丁、妥布霉素、卡那霉素等17种抗生素的药敏纸片(按YY/T11912011抗菌剂药敏纸片标准制备),参照革兰氏阴性杆菌常用药敏试剂盒(杭州滨和微生物试剂有限公司)说明书,采用药敏纸片法[15]测定病原菌株的耐药性,试验重复3次。
2结果与分析
2.1病原菌的分离培养及形态学观察
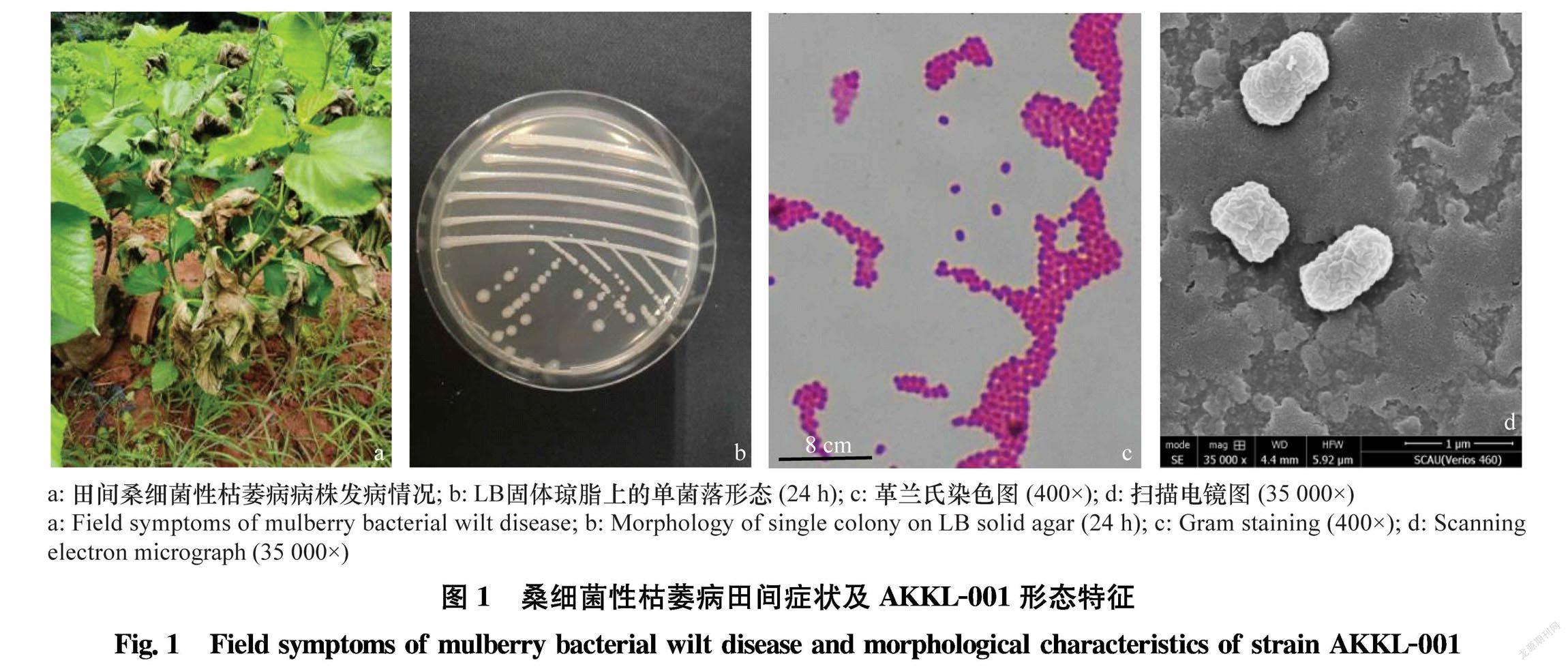
从广西融安隘口村的桑(‘桂桑12’)枯萎病病株(图1a)根部分离到1株桑枯萎病菌,编号AKKL001,病原菌在LB培养基上呈乳白色,表面光滑,流动性强,有刺激性气味(图1b)。吸取10 μL菌液进行革兰氏染色,在光学显微镜下观察,为革兰氏阴性菌,且都为椭圆至短杆状(图1c)。电镜观察病原菌呈短杆状(图1d),无鞭毛,表面不光滑。菌种送广东省微生物菌种保藏中心保藏,菌种保藏号为(GDMCC No.1.1602)。
2.2致病性鉴定与检测
分别对水培扦插桑嫩枝和土壤栽培的桑苗接种AKKL001,测定其致病性。结果表明,水培桑枝接种AKKL001菌悬液后72 h桑枝叶片出现黄化和轻微的枯萎,失水等症状(图2b),接种后96 h扦插枝条已完全枯萎,空白对照组未出现枯萎症状。桑苗经伤根灌根接种AKKL001菌液后3 d出现轻微的枯萎现象,6 d后部分桑苗已完全枯萎,表现为桑苗由下至上逐渐黄化,萎蔫,易脱落(图2d),而接种清水对照组表现正常(图2c)。
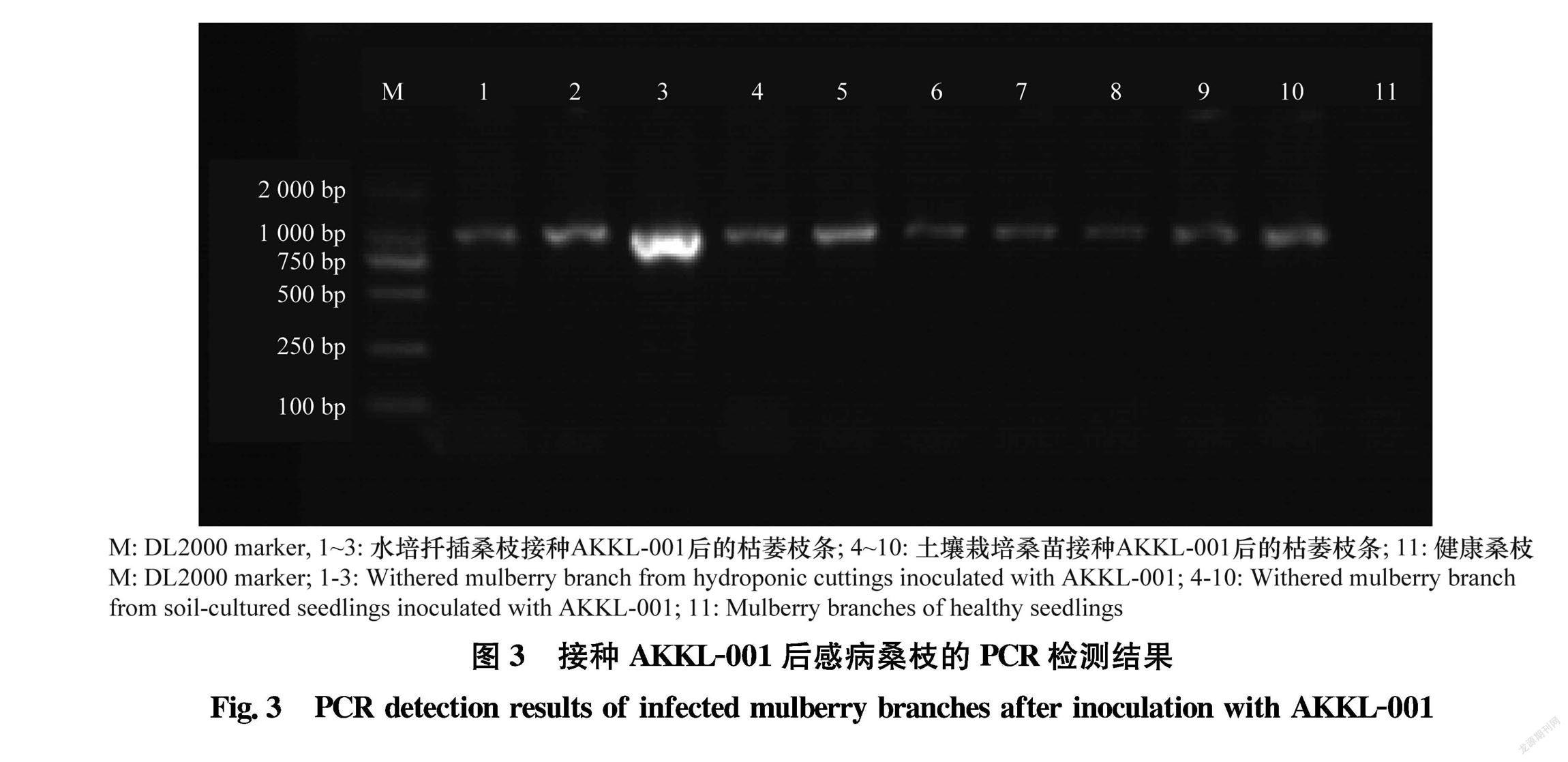
对水培扦插嫩枝组和土壤栽培苗木组的枯萎枝条的总DNA进行特异性PCR检测(图3), 均检测到AKKL001的特异条带,大小约为926 bp,并从苗木组的枯萎病株根部成功分离到产酸克雷伯氏菌。
2.3生理生化性质的鉴定
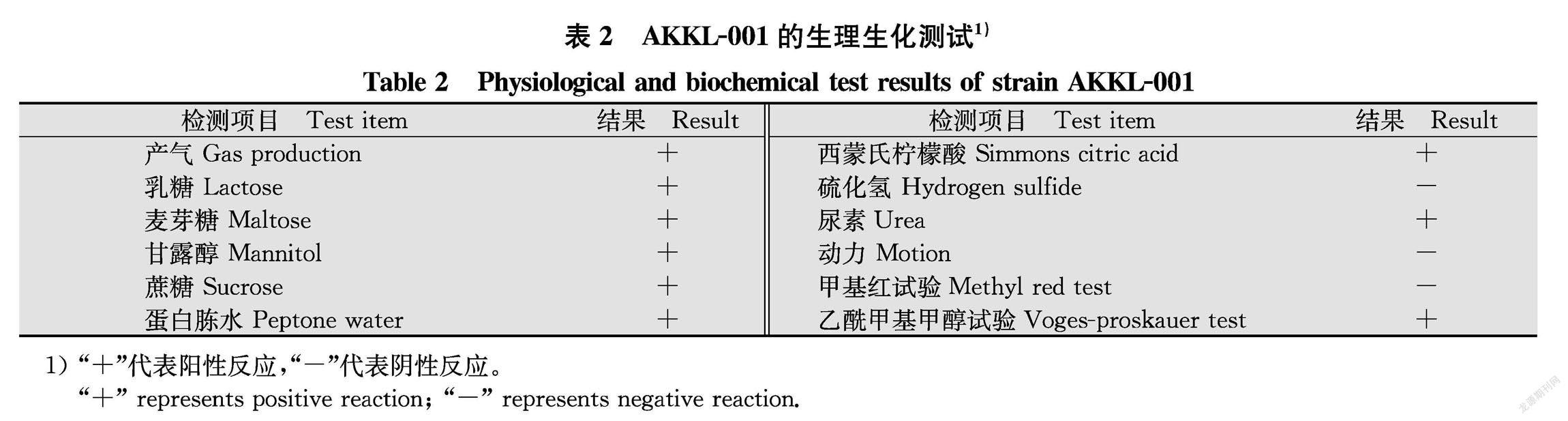
生理生化测试是细菌的常见鉴别方法,利用肠杆菌科的12种生理生化指标对分离纯化的培养物进行验证,将AKKL001鉴定为产酸克雷伯氏菌Klebsiella oxytoca。具体测试结果见表2。
2.4全基因组测序结果
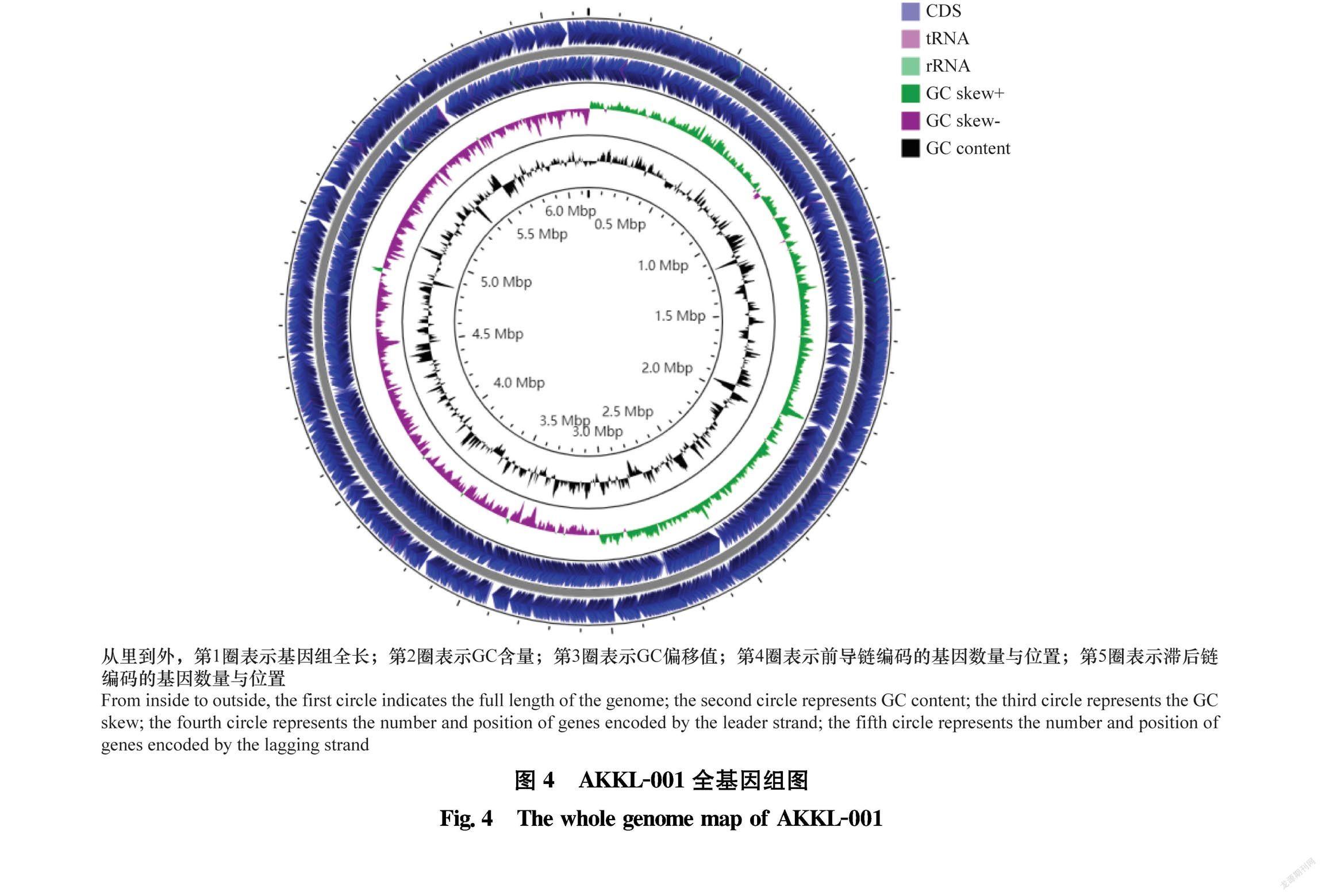
经基因组测序组装后,得到AKKL001基因组草图(图4),基因组全长6 149 586 bp,包含5 792个基因,66个重复区段,rRNA数量为25个,tRNA数量为87个,GC含量55.80%。
2.5系统进化分析
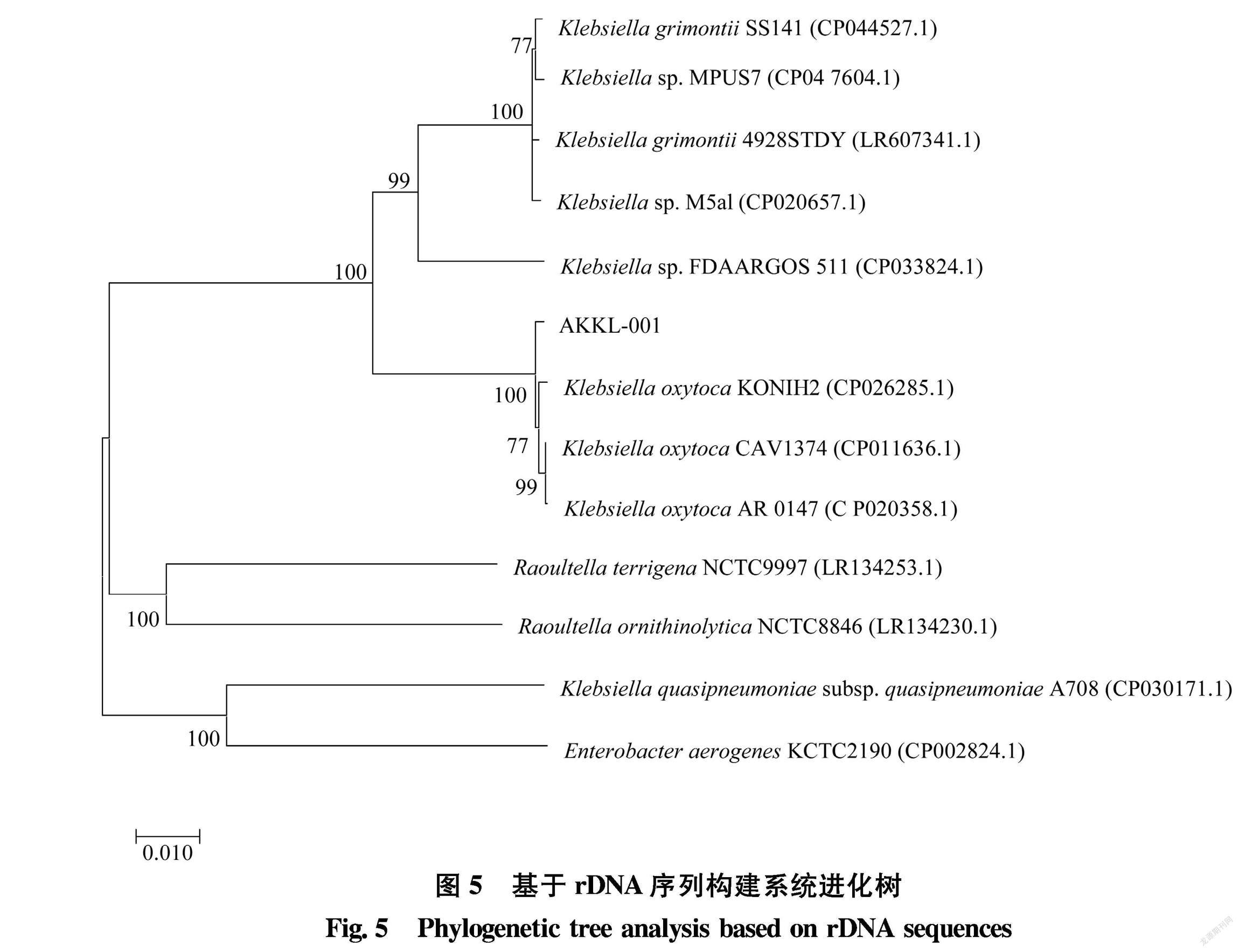
本研究获得的全基因组测序结果提交到GenBank数据库后获得登录号CP060111。基于rRNA基因全长序列的系统进化树结果(图5)显示,AKKL001与产酸克雷伯氏菌Klebsiella oxytoca聚为一支,与人源产酸克雷伯氏菌K.oxytoca CAV1374(CP011636.1) 遗传距离最为接近,二者rDNA序列相似性达99.74%。
为了更好地确定AKKL001的系统发育地位,利用在线软件OAT基于ANI分析基因组之间的遗传距离。结果(图6)显示,AKKL001与产酸克雷伯氏菌CAV1374(CP011636.1)最为接近(ANI值达99.09%),结合形态观察与生化鉴定的结果,最终将AKKL001鉴定为产酸克雷伯氏菌。
2.6基因组组分分析
经基因组预测分析,AKKL001染色体共有58个 基因岛,其中主要有糖基转移酶家族,纤维素酶家族等与病原菌侵染与定殖相关的基因簇。基因组中包含42个与植物细胞壁降解酶(plant cell wall degrading enzymes,PCWDEs)相关的基因,包括编码果胶裂解酶(pectate lyase)的pelX、pelY基因;果胶酯酶(pectinesterase)的pemL、pemK、pemB基因,编码多聚半乳糖醛酸酶(polygalacturonase)的pehX基因,以及与纤维素合成酶(cellulose synthase)相关的celY、bcsZ、bcsE、bcsF基因;编码β葡萄糖苷酶(βDglucosidase)的bglH、bglA、bglK、bglG、bglX、bglB、bglY、bglJ与nagZ基因。另外,从AKKL001基因组中注释到多种与分泌系统相关的基因,Ⅰ型分泌系统(type Ⅰ secretion system,T1SS)作为非Sec(secretoryprotein translocation)依赖途径,可将胞内蛋白直接运送到胞外。AKKL001中存在HlyD家族,ATPase家族,Tolc家族等多个Ⅰ型分泌系统相关蛋白。除此之外,AKKL001基因组中注释到高度保守的Ⅱ型分泌系统(type Ⅱ secretion system,T2SS)基因簇(gspCDEFGHIJKLMN),覆盖10 818个碱基,GC含量约为63%,T2SS主要负责将胞外蛋白从细胞周质转运到细胞外膜[16]。
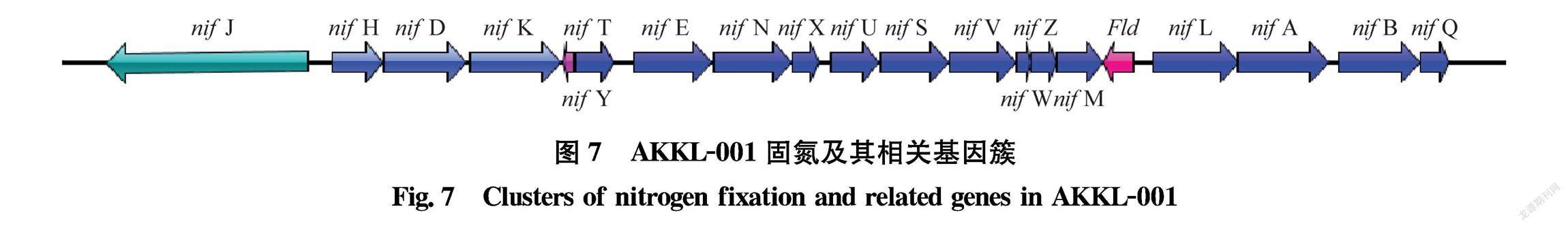
克雷伯氏菌属的固氮基因是研究的热点之一。对AKKL001基因组分析发现其存在一个完整的固氮基因簇(nitrogenfixation gene cluster),共由20个基因組成,长23.279 kb,GC含量62%。相应的基因排列如下(图7),包括nifH、nifD、nifK固氮酶结构基因与编码固氮相关的调节因子nifJ、nifT、nifY、nifE、nifN、nifX、nifU、nifS、nifV、nifW、nifZ、nifM、nifL、nifA、nifB、nifQ及Fld基因等,与肺炎克雷伯氏菌Klebsiella pneumoniae中的nif基因簇模式系统相一致。
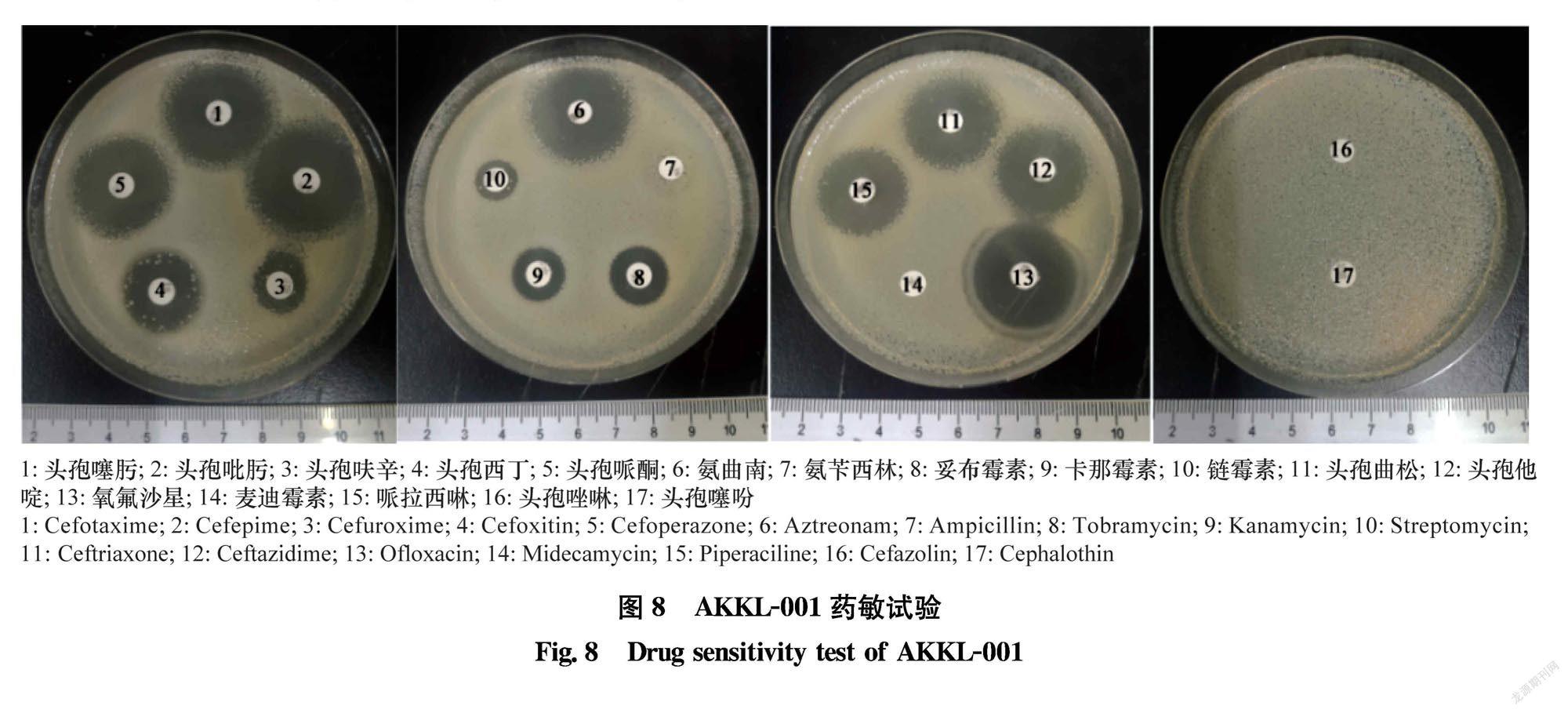
对比CARD抗性基因数据库预测到59个抗生素抗性基因,其中编码抗生素外排泵基因16个,包括patA、oqxB、oqxA、mdtM、mdtK、mdtH、mdtG、mdtD、mdtC、mdtA、mdfA、emrD、emrB、emrA、 acrD、acrB等;外排泵调节基因7个,包括emrR、baeR、baeS、robA、HNS、cpxR、cpxA等;多种抗生素抗性基因19个,如gyrA、parC、parE、soxS、nfsA、folP、mfd、PmrF、bacA、LeuO、FosA5、OXY11、gyb、cysB、alaS、UhpT、murA、GlpT、fyuA等;其他抗性基因如marR、soxR、acrR等共14个。 药敏试验(图8)表明AKKL001对头孢噻肟,氨曲南,头孢哌酮,头孢吡肟,头孢曲松,头孢他啶,氧氟沙星,哌拉西啉等8种抗生素敏感,而对氨苄西林,头孢唑啉,头孢呋辛,头孢噻吩,链霉素、麦迪霉素等抗生素均耐药。
3讨论
桑细菌性枯萎病为桑树土传病害,近几年在华南蚕区常有报道,严重影响桑叶产量。部分蚕区通过使用抗生素或种植抗病品种来控制桑细菌性枯萎病的大面积发生,虽取得了一定的成效[17],但桑枯萎病的发生仍然普遍存在。其中,病原复杂多样是桑细菌性枯萎病防控的主要困难之一。
目前,桑细菌性枯萎病的病原研究多集中于肠杆菌属细菌,包括阴沟肠杆菌、阿氏肠杆菌和桑肠杆菌3种病原[3 -4,18- 19],戴凡炜等[5]通过对广东、广西桑细菌性枯萎病病样进行分离鉴定, 增加了2种新的属,克雷伯氏菌属Klebsiella与泛菌属Pantoea。本课题组前期在对广东、广西等地的桑细菌性枯萎病病样的研究中也发现阴沟肠杆菌与菠萝泛菌,甚至在部分样本中还检测到茄科雷尔氏菌R.solanacearum[20],因此推测桑细菌性枯萎病是多种病原菌之间通过复杂的群体效应相互调控,复合侵染。桑细菌性枯萎病病原多样,目前尚未明确该病的主要病原菌,因此对桑细菌性枯萎病病原的多样性、主要致病菌、病原菌的传播途径及协同作用等都需要进一步研究。
克雷伯氏菌属病原菌多为动物源致病菌,部分研究表明其也可以导致植物病害[21-22]。2019年Huang[23]等确定了肺炎克雷伯氏菌 KpC4引起玉米细菌性顶腐病,揭示了由KpC4引起的玉米细菌性顶腐病的病程以及KpC4的生态适应性和寄主范围。桑源克雷伯氏菌主要报道有:肺炎克雷伯氏菌、K.variicola、产酸克雷伯氏菌及未命名的Klebsiella sp., 主要导致桑细菌性枯萎病。这类克雷伯氏菌通常多见于肠道微生物种群。本研究前期对样本收集地区的桑园调查发现,部分桑园存在使用动物排泄物作为有机肥的情况,因此下一步工作需要考虑不同来源的克雷伯氏菌是否对桑树具有致病性。
随着新一代测序技术的发展,测序成本逐渐降低,克雷伯氏菌的全基因组序列也陆续被报道[24 -25]。通过病原菌的全基因组测序,可以揭示病原菌的遗传规律,并在此基础上挖掘新的功能基因。ReynaFlores等[26]从甘蔗茎中获得了内生菌Klebsiella variicola KvMx2和肺炎克雷伯氏菌KpMx1,全基因组测序结果揭示了这2株内生克雷伯氏菌中与促进植物生长、发育及毒力因子等相关的基因差异。Rosenblueth等[27]在总DNADNA双杂交的基础上,基于rpoB,gyrA,mdh,infB,phoE和nifH基因的系統发育分析及不同的表型特征,发现克雷伯氏菌属的Klebsiella variicola,广泛存在于香蕉、水稻、甘蔗和玉米等农作物中。本研究测定了AKKL001全基因组进行了注释分析,结果表明基因组中存在编码荚膜、脂多糖、外膜蛋白等与植物病原菌侵染过程相关的基因,同时也存在固氮基因簇与部分抗生素的耐受性基因,但具体基因功能与致病机理也还需进一步研究。通过药敏试验测定了AKKL001对部分抗生素的耐药性,为桑枯萎病抗生素的使用提供参考,后期可考虑将部分蚕用抗生素运用到桑枯萎病的防控研究中。
桑细菌性枯萎病危害严重、病原多样,本研究从桑细菌性枯萎病病样中分离到1株克雷伯氏菌(AKKL001),结合形态学观察、生化鉴定及全基因组测序结果将其鉴定为产酸克雷伯氏菌Klebsiella oxytoca。华南蚕区是桑细菌性枯萎病的多发区,后期对桑细菌性枯萎病的防控可以考虑加强对克雷伯氏菌的检测与防治。
参考文献
[1]廖森泰, 向仲怀. 论蚕桑产业多元化[J]. 蚕业科学, 2014, 40(1): 137 -141.
[2]李磊, 唐翠明, 王振江, 等. 桑树细菌性病害研究概况[J]. 广东蚕业, 2012, 46(4): 36 -40.
[3]ZHU Bo, WANG Guofen, XIE Guanlin, et al. Enterobacter spp.: A new evidence causing bacterial wilt on mulberry [J]. Science China Life Sciences, 2010, 53(2): 292 -300.
[4]WANG Guofen, XIE Guanlin, ZHU Bo, et al. Identification and characterization of the Enterobacter complex causing mulberry (Morus alba) wilt disease in China [J]. European Journal of Plant Pathology, 2010, 126 (4): 465- 478.
[5]戴凡炜, 罗国庆, 王振江, 等. 华南蚕区桑枯萎病病原菌的分离与分子鉴定[J]. 蚕业科学, 2012, 38(6): 981- 987.
[6]蒙姣荣, 欧阳秋飞, 岑贞陆, 等. 广西桑树细菌性枯萎病菌致病力和遗传多样性分析[J]. 基因组学与应用生物学, 2014, 33(5): 998 -1006.
[7]刘吉平, 罗龙辉. 一组桑树细菌性枯萎病病原克雷伯氏菌的PCR检测引物及应用: CN111073988A [P]. 2020 -04- 28.
[8]BUCHANAN R E, GIBBONS N E. 伯杰细菌鉴定手册[M]. 北京: 中国科学出版社, 1984.
[9]KUMAR S, STECHER G, LI M, et al. MEGA X: Molecular evolutionary genetics analysis across computing platforms [J]. Molecular Biology and Evolution, 2018, 35(6): 1547 -1549.
[10] WATTAM A R, DAVIS J J, RIDA A, et al. Improvements to PATRIC, the allbacterial bioinformatics database and analysis resource center [J]. Nucleic Acids Research, 2017, 45(D1): D535 -D542.
[11] LEE I, KIM Y O, PARK S C, et al. OrthoANI: An improved algorithm and software for calculating average nucleotide identity [J]. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(2): 1100 -1103.
[12] CLAIRE B, LAIRD M R, WILLIAMS K P, et al. Islandviewer 4: expanded prediction of genomic islands for largerscale datasets [J]. Nucleic Acids Research, 2017(W1): W30- W35.
[13] LIU Bo, ZHENG Dandan, JIN Qi, et al. VFDB 2019: A comparative pathogenomic platform with an interactive web interface [J]. Nucleic Acids Research, 2018, 47(D1): D687 D692.
[14] ALCOCK B P, RAPHENYA A R, LAU T T Y, et al. CARD 2020: antibiotic resistome surveillance with the comprehensive antibiotic resistance database [J]. Nucleic Acids Research, 2020, 48(D1): D517- D525.
[15] ANDREWS J M. BSAC Working Party on susceptibility testing ft. BSAC standardized disc susceptibility testing method [J]. Journal of Antimicrobial Chemotherapy, 2001, 48(S1):43- 57.
[16] KOROTKOV K V, SANDKVIST M, HOL W G J. The type Ⅱ secretion system: biogenesis, molecular architecture and mechanism [J]. Nature Reviews Microbiology, 2012, 10(5): 336 -351.
[17] 羅国庆, 唐翠明, 王振江, 等. 桑树杂交组合抗青枯病能力鉴定及与抗病相关酶活性的研究[J]. 蚕业科学, 2010, 36(2): 300-303.
[18] 朱勃. 桑细菌性枯萎病病原的定名、分子检测及肠杆菌基因水平转移的研究[D]. 杭州: 浙江大学, 2010.
[19] 姜星. 桑枯萎病病原细菌的分离、鉴定及分子检测方法的建立[D]. 镇江: 江苏科技大学, 2012.
[20] 杨宏宇. 桑枯萎病病原鉴定及分子生物学研究[D].广州:华南农业大学,2018.
[21] LOGANATHAN M, THANGAVELU R, PUSHPAKANTH P, et al. First report of rhizome rot of banana caused by Klebsiella variicola in India [J]. Plant Disease, 2021, 105(7): 2011.
[22] FAN H C, ZENG L,YANG P, et al. First report of banana soft rot caused by Klebsiella variicola in China [J]. Plant Disease, 2016, 100(2): 517.
[23] HUANG Min, HE Pengfei, MUNIR S, et al. Ecology and etiology of bacterial top rot in maize caused by Klebsiella pneumoniae KpC4 [J/OL]. Microbial Pathogenesis, 2019, 139: 103906. DOI: 10.1016/j.micpath.2019.103906.
[24] LIN Li,WEI Chunyan,CHEN Mingyue,et al.Complete genome sequence of endophytic nitrogenfixing Klebsiella variicola strain DX120E [J/OL]. Standards in Genomic Sciences,2015,10(1): 22.DOI:10.1186/s40793-015-0004-2.
[25] FULTON J C,KLEIN J M,BEC S, et al.Draft genome sequences of plantpathogenic Klebsiella variicola strains isolated from plantain in Haiti [J/OL]. Microbiology resource announcements,2020,9(29):e00336-20. DOI:10.1128/MRA.00336-20.
[26] REYNAFLORES F, BARRIOSCAMACHO H, DANTNGONZLEZ E, et al. Draft genome sequences of endophytic isolates of Klebsiella variicola and Klebsiella pneumoniae obtained from the same sugarcane plant [J/OL]. Genome Announcements, 2018, 6(12): e0014718. DOI:10.1128/genomeA.00147-18.
[27] ROSENBLUETH M, MARTNEZ L, SILVA J, et al. Klebsiella variicola, A novel species with clinical and plantassociated isolates [J]. Systematic and Applied Microbiology, 2004, 27(1): 27-35.
收稿日期:2020-09-30修订日期:2020 -11- 22
基金项目:
财政部和农业农村部:国家现代农业产业技术体系(CARS-18-ZJ0304);2017年省级农业发展和农村工作专项(2017LM4168);2018年度广东省农业标准化计划项目
* 通信作者
E-mail:liujiping@scau.edu.cn

