大豆百粒重相关基因的全基因组发掘分析
宿洋杨静郭勇杜维俊邱丽娟
(1山西农业大学农学院,030801,山西晋中;2中国农业科学院作物科学研究所/农作物基因资源与遗传改良国家重大科学工程/农业农村部北京大豆生物学重点实验室,100081,北京)
大豆是富含蛋白质和油脂的重要粮食和经济作物[1],提高大豆产量一直是我国大豆育种最重要的目标之一。百粒重是大豆重要的产量性状,发掘调控籽粒大小/粒重相关的基因,对大豆籽粒大小/粒重的分子调控机制开展研究,可为提高大豆产量提供理论基础。同时,大豆百粒重还是重要的驯化性状,野生大豆和栽培大豆的百粒重差异很大,野生大豆百粒重在0~3g之间,而栽培大豆在5~55g之间,因此大豆种质资源中基因的变异为百粒重基因的发掘提供了重要线索。
植物的种子是由受精卵发育成熟而形成的,主要由胚、胚乳和种皮3部分组成。种子发育成熟后最终的大小也主要取决于胚、胚乳和种皮的发育程度。目前在模式植物拟南芥中已明确了多个调控种子大小的基因,其中与胚发育相关的基因有ANGUSTIFOLIA3(AN3)、MINISEED3(MINI3)、YODA(YDA)和 ETHYLENE-INSENSITIVE3(EIN3)等。突变体分析表明,an3突变体种子中胚细胞的体积增大,而mini3突变体种子中胚细胞的数量减少。mini3和an3双突材料的种子大小与mini3单突材料种子大小一致。因此,MINI3与AN3相比具有上位性,在种子大小调控路径中AN3位于MINI3的上游[2];YODA(YDA)编码MAPKK激酶,研究发现功能缺失突变体yda的种子变小,而ein3突变体的种子变大。遗传分析表明,YDA和EIN3是糖介导的代谢级联系统的组成部分,通过母系控制胚的大小来调节种子大小[3]。与胚乳发育有关的基因有ENDOSPERM DEFECTIVE1(EDE1)和DET2等。对突变体的研究发现,ede突变体胚乳存在核分裂缺陷,与野生型胚乳相比缺少有序的微管阵列,最终导致种子败育[4];det2突变体中,种子胚乳的细胞化过程与野生型相比延迟1~2d,造成种子腔和胚乳体积减小,因此导致种子变小[5]。与种皮发育有关的基因有EOD3/CYP78A6和APETALA2(AP2),对突变体的研究发现,eod3影响珠被细胞体积的扩大,导致eod3外珠被细胞显著小于野生型外珠被细胞,因此eod3的种子变小[6];ap2突变体与野生型相比具有更长的珠被细胞,产生了比野生型更大的种子,因此AP2负调控种子大小[7]。种子的粒形性状包括粒长、粒宽和粒厚,这些性状与种子的质量密切相关。在水稻中已对GW2、GW5和GL4等多个与粒形和粒重相关的基因开展深入研究。GW2编码具有E3泛素连接酶活性的环形蛋白,它的功能缺失突变体与野生型相比增加了颖壳中的细胞数量,导致水稻籽粒变宽,粒重增加[8]。GW5编码钙调结合蛋白,是BR信号通路中的正向调控因子,它的表达水平的提高能够增加谷粒宽度,从而使粒重增加[9]。GL4调控内外颖壳中纵向细胞的伸长,在发生移码突变后,谷粒的长度变短,粒重变小[10]。
在大豆中,与粒重相关的研究大多仍处在QTL定位阶段,已报道的关于大豆百粒重的QTL有304个(http://www.soybase.org,2021年3月数据),分布于20条染色体上。陈强等[11]利用冀豆12×黑豆(ZDD03651)构建的重组自交系(RIL)群体,共检测到5个百粒重QTL,遗传贡献率(R2)为7.68%~12.83%。郭洁等[12]利用东农46和L-100衍生的RIL群体,检测到5个与百粒重相关的QTL,分别位于9号、12号、14号和18号染色体上,可解释的表型贡献率为2.30%~7.59%。Dhungana等[13]利用Williams 82×PI 366121构建的RIL群体,定位到9个百粒重QTL。Liu等[14]利用Jackson×JWS156-1构建的染色体片段代换系群体定位到9个百粒重QTL,其中12号染色体上的qSW12.1位点连续3年被检测到,是一个稳定的主效QTL。Li等[15]对来自3个生态区的146个大豆品种进行全基因组关联分析,定位到21个与粒重相关联的位点,其中9号染色体上的SW9-1位点是一个新发现的与籽粒大小显著相关的QTL。从这些研究中可以看出,大豆百粒重是受多个位点调控的复杂数量性状,在不同群体中或不同环境下所检测到的QTL都有所差异,且定位到的目标区间较大。在候选基因的研究方面,Wang等[16]通过关联分析发现了1个与大豆籽粒大小/粒重相关的基因GmCYP78A10,该基因在大豆驯化和育种过程中经历了人工选择,等位基因GmCYP78A10a主要分布在野生大豆中,而GmCYP78A10b主要分布于栽培大豆中。该研究发现携带GmCYP78A10b等位基因的大豆品种在粒重、粒宽和粒厚方面均明显大于携带GmCYP78A10a等位基因的品种,但该基因的作用机理尚不清楚。Lu等[17]利用ZYD7×HN44构建的RIL群体定位到野生大豆ZYD7中的phosphatase 2C(PP2C)基因,该基因参与调控种子重量/大小,通过与转录因子GmBZR1结合,促进去磷酸化的GmBZR1蛋白积累,导致籽粒大小/粒重的增加。
1 材料与方法
1.1 植物粒重相关基因的信息收集
从相关文献中收集在拟南芥和水稻中已明确生物学功能的和调控籽粒大小/粒重的基因,基因信息均来自已发表文章。
1.2 大豆基因组中粒重相关直系同源基因的检索
在Ensembl数据库(http://plants.ensembl.org/index.html)中,根据拟南芥和水稻中基因的ID编号查找对应大豆基因组中的直系同源基因。
1.3 大豆直系同源基因SNP位点查找和筛选
在包括栽培大豆和野生大豆的56份大豆种质资源的重测序数据中[18]查找并筛选SNP位点,计算每个SNP在野生大豆和栽培大豆两大类群之间的Fst值,依据Fst>0.45筛选在两大类群中差异显著的SNP,根据基因注释信息筛选导致非同义变异的SNP。
1.4 大豆粒重相关基因的表达谱分析
从Phytozome数据库(https://phytozome.jgi.doe.gov/pz/portal.html)中获得基因在不同组织中的表达量数据,包括的组织有大豆的根、根毛、根瘤、茎、叶、分生组织、花、荚和种子等。利用软件TBtools对基因在不同组织的表达数据绘制表达谱并进行聚类分析,将表达量超过所有组织平均值2倍的组织认为是特异性表达的组织。
1.5 候选基因分析
利用上述56份大豆资源中的26份验证位于编码区的非同义突变SNP,包括8份野生大豆和18份栽培大豆。利用CTAB法提取基因组DNA,根据SNP位点两侧的基因组序列设计特异性引物,扩增SNP位点所在片段,用琼脂糖凝胶电泳检测目标条带后进行测序。根据测序结果中SNP变异类型在野生大豆和栽培大豆中的分布情况,分析得到与大豆百粒重相关的SNP位点。
利用2368份资源的重测序数据[19]以及其中1695份资源的百粒重表型数据,分析验证得到的SNP位点在野生型和突变型材料中的百粒重表型分布情况。按照 0.1~6.0g、6.1~12.0g、12.1~18.0g和>18.0g的分布划分出百粒重表型范围,统计每个范围内的材料数量,比较不同SNP变异类型在野生大豆和栽培大豆及不同百粒重材料中的分布。
2 结果与分析
2.1 大豆中粒重相关同源基因的检索
2.1.1 拟南芥和水稻中粒重相关基因的收集 通过文献检索共得到59个调控籽粒大小/粒重的基因(表1),其中拟南芥基因37个,水稻基因22个,这些基因都有明确的生物学功能,根据其功能进行分类,其中参与激素信号转导的基因有24个,参与泛素代谢途径的基因有6个,参与其他代谢途径的基因有29个。
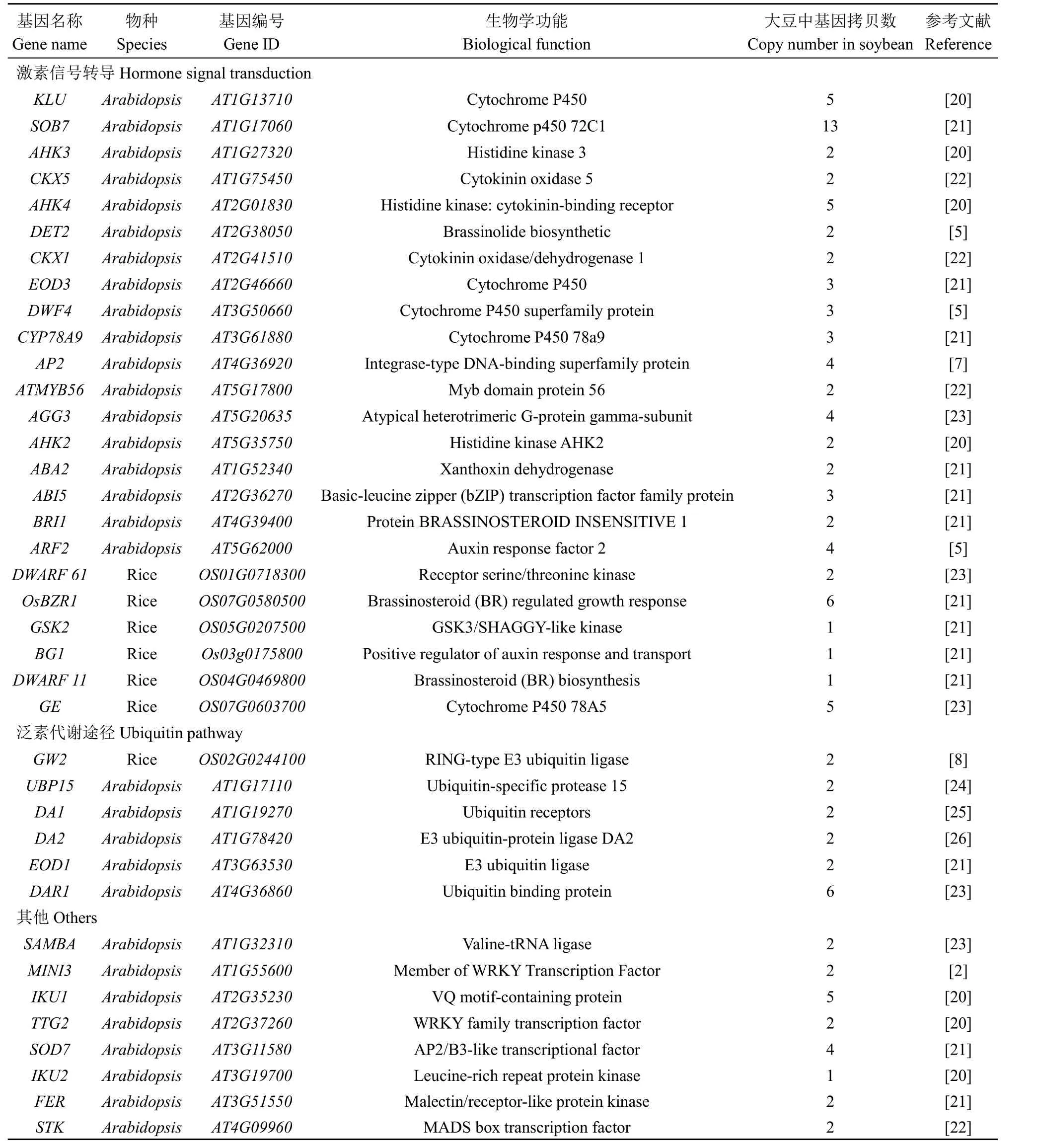
表1 调控植物籽粒大小/粒重的功能基因Table 1 Functional genes related to seed size/weight in plants

续表1 Table 1(continued)
2.1.2 大豆百粒重相关基因的全基因组检索 基于序列相似和结构域相同的原则,查找水稻和拟南芥中与粒重相关的59个基因在大豆中的同源基因,共获得了175个基因(表2),由表2可知,其中174个基因分布于大豆20条染色体上,1个基因位于基因组的Scaffold上,这些基因在10号染色体上分布最多(22个),12号染色体上最少(2个),其他染色体上基因数目在3~13之间。从代谢途径来看,泛素代谢途径相关基因主要分布在1号、2号和10~17号染色体上;激素信号转导途径相关基因在除10号、12号、18号和20号染色体的其他染色体上均有分布。由表1可知,在59个调控植物籽粒大小/粒重的基因中,SHB1、GS2、BG1、GL3和GLW7基因在大豆中没有找到同源基因;IKU2、D11和GSK2基因在大豆中为单拷贝,其他基因均为多拷贝。
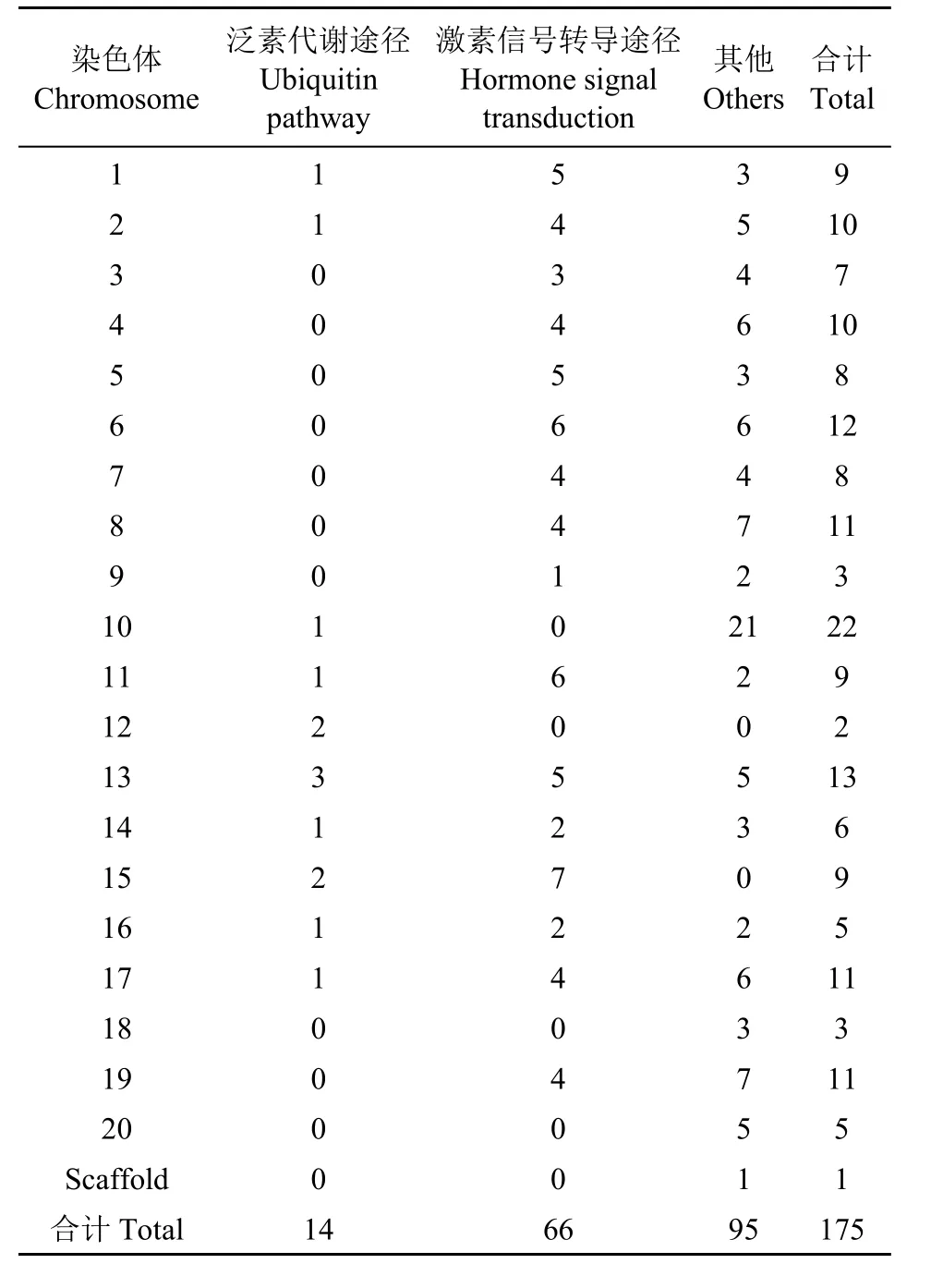
表2 大豆百粒重相关基因在染色体上的定位Table 2 Chromosome location of genes related to 100-seed weight in soybean
2.1.3 大豆百粒重相关基因表达谱分析 175个大豆同源基因中,有171个基因在Phytozome网站上有表达量数据,对这171个基因的表达丰度进行聚类分析,可以将这些基因分成6个亚群(图1),A亚群基因主要在花中表达,B亚群基因主要在根中表达,C亚群基因主要在根毛、根、根瘤和顶端分生组织中表达,D亚群基因主要在荚中表达,E亚群基因主要在种子和顶端分生组织中表达,F亚群基因主要在叶片、顶端分生组织和茎中表达。
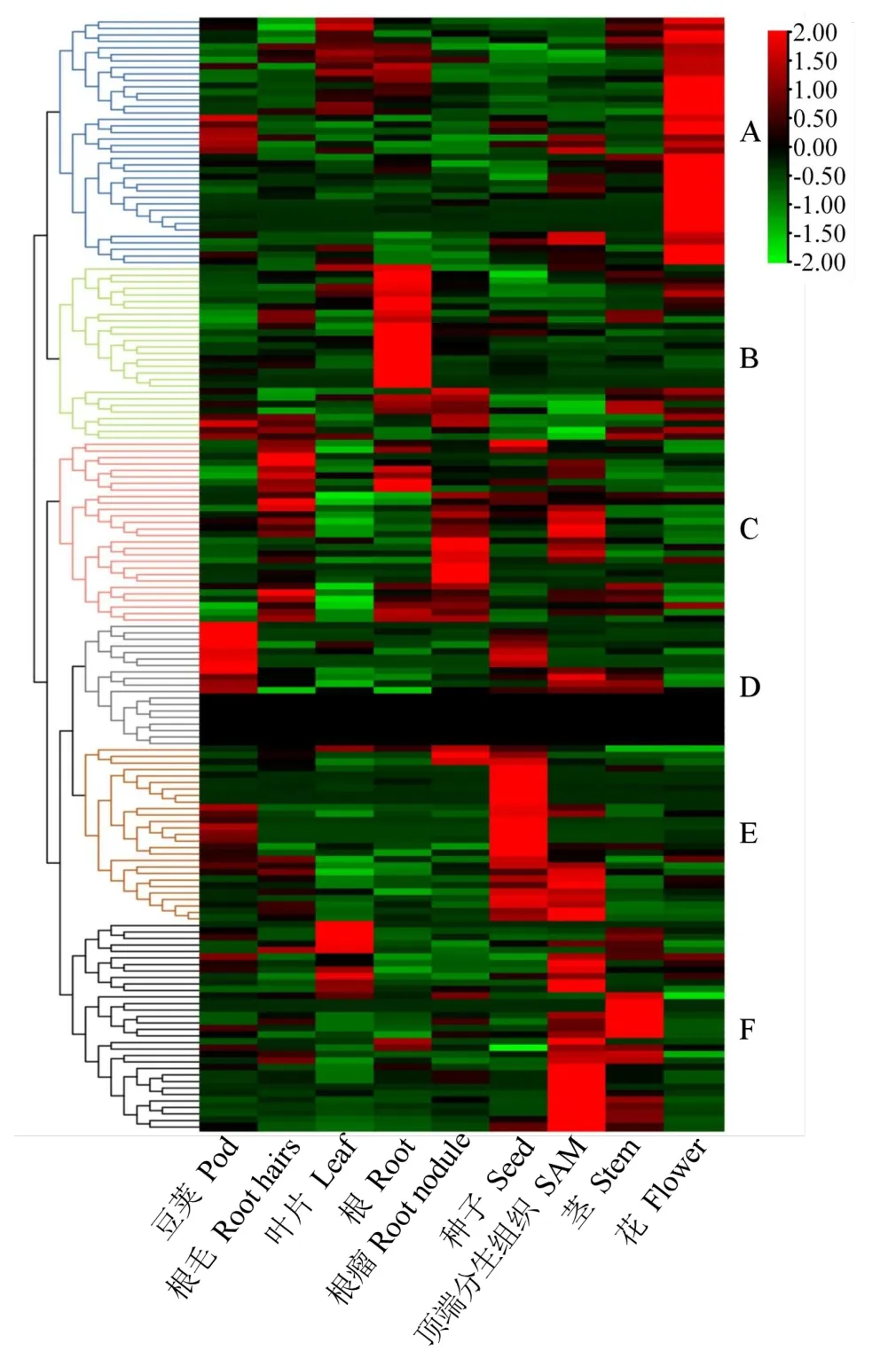
图1 大豆百粒重相关基因的电子表达谱Fig.1 Expression profiling of genes related to 100-seed weight in soybean
组织特异表达分析结果(表3)表明,不同的基因在不同组织中存在特异性表达,其中在花中特异表达的基因最多(35个),在叶片中最少(7个)。在种子中特异性表达的基因有21个,在荚中特异性表达的基因有11个。在花中,参与激素信号转导的特异性表达的基因有14个,参与泛素代谢途径特异性表达的基因有5个;在种子中,参与激素信号转导特异性表达的基因有6个,参与泛素代谢途径特异性表达的基因有3个;在荚中,参与激素信号转导特异性表达的基因有4个。从代谢途径来看,参与激素信号转导途径的62个基因在各个组织中均有表达,其中有14个基因在花中特异性表达,有6个基因在种子中特异性表达;参与泛素代谢途径的14个基因,在除了荚、根毛和顶端分生组织以外的各组织中均有表达,其中5个基因在花中特异性表达,有3个基因在种子中特异性表达,且都属于DA家族基因。

表3 大豆不同组织中特异性表达的基因Table 3 Specifically expressed genes in different tissues of soybean
2.2 基于大豆资源中的SNP位点鉴定百粒重相关基因
2.2.1 SNP位点的查找 利用56份大豆资源(31份栽培大豆和25份野生大豆)的重测序数据,以Williams 82为参考基因组,在175个大豆同源基因中共获得2769个SNP位点,其中121个SNP位点在野生和栽培大豆2个类群间的Fst值大于0.45。在这121个SNP位点中,由表4可知,有16个SNP为非同义变异,分布于2号、5号、7号、13号和17号染色体上,位于11个基因中。
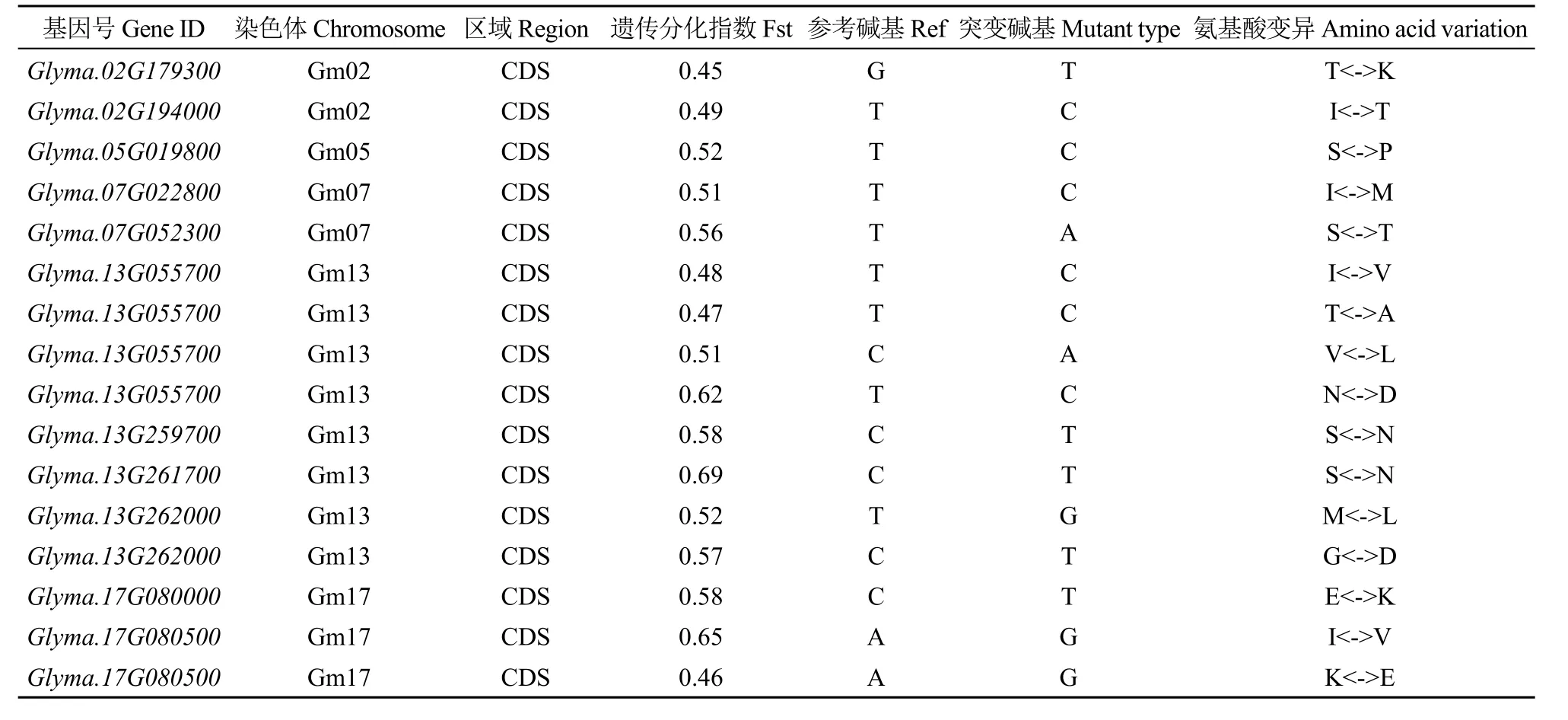
表4 非同义变异SNP位点分布情况Table 4 Distribution of non-synonymous variation SNP loci
2.2.2 SNP位点的验证 利用Williams 82参考基因组的序列,对这16个位于编码区的SNP设计特异性扩增引物。从56份大豆资源中选择26份资源(18份栽培大豆和8份野生大豆)进行PCR扩增测序,鉴定每份材料中16个SNP的碱基类型。通过分析16个SNP位点在26份资源中的碱基类型分布(图2),发现有5个SNP在26份资源中的不同变异类型在野生大豆和栽培大豆间存在显著性差异,在野生大豆中主要为一种变异,而在栽培大豆中为另一种变异。这5个SNP位点分别位于Glyma.05G019800、Glyma.07G022800、Glyma.07 G052300、Glyma.13G259700和Glyma.13G261700这5个基因中。
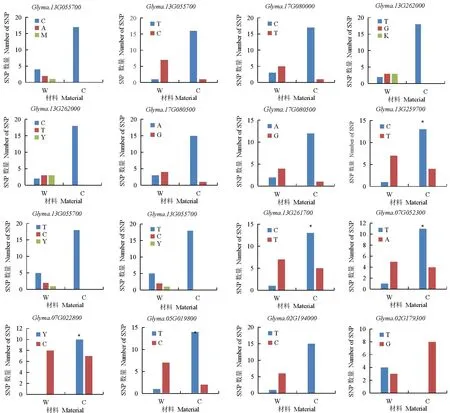
图2 16个SNP在26份大豆资源中的分布Fig.2 Distribution of 16 SNPs in 26 soybean accessions
2.3 候选基因分析
2.3.1 SNP位点在重测序数据中的表型分布 在2368份大豆资源的重测序数据中查找上述5个SNP,获得了其中的4个SNP数据,分别位于Glyma.05G019800、Glyma.07G022800、Glyma.13 G259700和Glyma.13G261700这4个基因中。对这4个SNP位点的不同变异类型在野生和栽培大豆中的分布情况进行分析发现,基因Glyma.13G259700上SNP野生型变异在野生大豆中分布频率为1%,
在栽培大豆中频率为99%;突变型变异在野生大豆中分布频率为28%,在栽培大豆中频率为72%。基因Glyma.07G022800上SNP野生型变异在野生大豆中分布频率为1%,在栽培大豆中频率为99%;突变型变异在野生大豆中分布频率为27%,在栽培大豆中频率为73%。基因Glyma.05G019800上SNP野生型变异在野生大豆中分布频率为1%,在栽培大豆中频率为99%;突变型变异在野生大豆中分布频率为32%,在栽培大豆中频率为68%。基因Glyma.13G261700上SNP野生型变异在野生大豆中分布频率为1%,在栽培大豆中频率为99%;突变型变异在野生大豆中分布频率为30%,在栽培大豆中频率为70%。更进一步说明这4个SNP在野生和栽培大豆中存在明显分化。
此外,在2368份重测序材料中,有1695份材料有百粒重表型数据。对上述获得数据的4个SNP在野生型和突变型材料中的百粒重表型分别进行显著性分析(图3),发现每个SNP在野生型和突变型变异材料对应的百粒重表型间存在极显著差异。
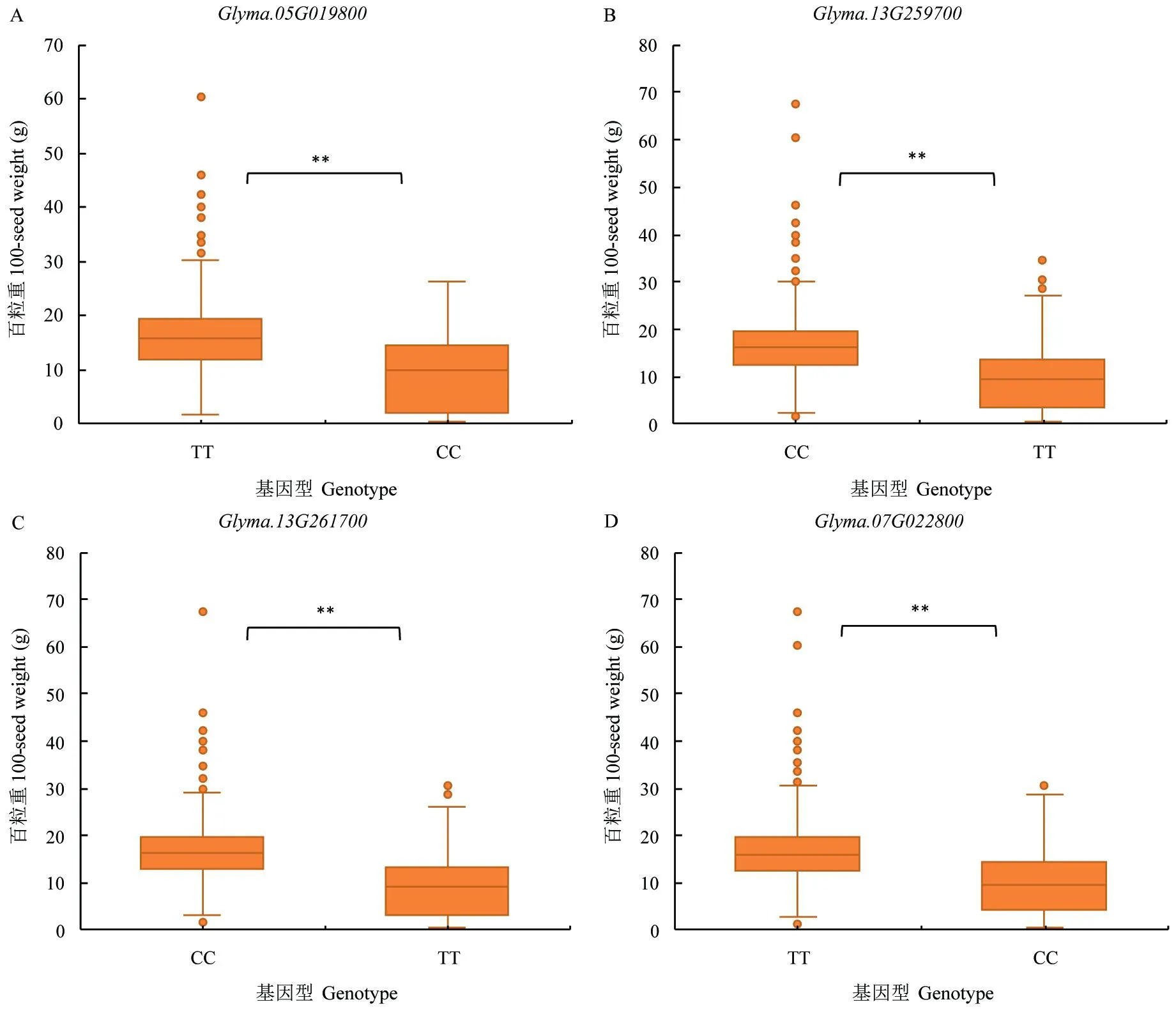
图3 不同SNP类型材料对应的百粒重表型分析Fig.3 Phenotypic analysis of 100-seed weight corresponding to different types of SNP
进一步按照 0.1~6.0g、6.1~12.0g、12.1~18.0g及>18.0g的分布范围来划分百粒重数据,统计4个SNP不同变异类型的材料中,不同百粒重范围的资源数量占总资源数量的比例。结果(图4)表明,在每个SNP中携带野生型变异的大豆资源百粒重≥12.0g的材料占总材料比例超过60%,而在每个SNP中携带突变型变异的大豆资源百粒重<12.0g的材料占总材料的比例也超过了60%。因此,这4个SNP所在的候选基因Glyma.05G019800、Glyma.07G022800、Glyma.13G259700和Glyma.13 G261700可能都与籽粒大小/粒重的调控有关。

图4 不同百粒重范围内大豆资源数量分布Fig.4 Distribution of soybean accessions in different 100-seed weight phenotypic ranges
2.3.2 候选基因功能注释 为进一步了解这4个候选基因的功能,在NCBI数据库(https://www.ncbi.nlm.nih.gov)中查找候选基因的功能注释,由表5可知,基因Glyma.05G019800为水稻GW7基因的同源基因,编码未知功能蛋白,参与单细胞的生长调节过程。基因Glyma.07G022800为水稻GW6a基因的同源基因,编码乙酰基转移酶,参与氨基酸的运输和代谢等生理活动。基因Glyma.13G259700为拟南芥UBP15/SOD2基因的同源基因,编码泛素水解酶,参与无机离子的转运与代谢等生理活动。基因Glyma.13G261700为拟南芥SOB7基因的同源基因,编码氧化还原酶,包含细胞色素P450蛋白结构域,参与次生代谢物的生物合成、运输和分解代谢等生理活动。

表5 候选基因功能注释Table 5 Functional annotation of candidate genes
3 讨论
3.1 反向遗传学加速大豆基因功能研究
大豆在进化过程中,经过了染色体的多倍体化和基因组复制,且基因组较大,利用正向遗传学方法定位控制大豆农艺性状基因耗时较长。随着大豆全基因组测序的完成,利用反向遗传学方法进行大豆基因定位和功能研究已取得了诸多进展。Zhong等[32]利用拟南芥开花途径中在结构和功能上都高度保守的1个MADS-box基因AGL20/SOC1,同源克隆到大豆中与之序列高度同源的基因GmGAL1,将该基因转入拟南芥中发现,转基因植株与野生型相比较早进入了开花期,并提出该基因在大豆的发育过程中可能是1个多功能基因。Deshmukh等[33]利用水稻和拟南芥等植物中的主嵌入蛋白(MIP)家族基因,通过序列比对同源克隆到大豆中的结瘤嵌入蛋白基因GmNIP2,该基因表达的蛋白能够转运硅元素,是豆科植物中首次报道的硅转运蛋白的基因。薛晨晨等[34]利用拟南芥中已公布的表达L-半乳糖酸-1,4-内酯脱氢酶(GLDH)的基因AtGLDH序列,在大豆中同源克隆到1个与之功能一致的基因GmGLDH(Glyma.02G166300),并对其进行生物信息学分析及非生物胁迫下的表达分析,为进一步研究该基因的功能提供帮助。大豆百粒重基因受到多个位点和环境条件的共同作用[35],利用图位克隆进行相关基因定位工作量大且耗时较长,本研究利用水稻和拟南芥中的已知功能基因鉴定出大豆中调控籽粒大小/粒重相关基因,通过生物信息学分析来挖掘相关基因,可为候选基因的筛选提供参考。
通过筛选共鉴定出至少有4个基因可能与大豆的百粒重相关,这4个基因中Glyma.13G259700在种子中的表达量最高,Glyma.13G261700和Glyma.07G022800在花中的表达量最高,Glyma.05G019800在顶端分生组织中表达量最高。查找基因功能注释发现,Glyma.07G022800编码乙酰基转移酶,在水稻中已报道了基因GW6a编码组蛋白H4乙酰基转移酶,表达该基因能增加粒重[23];Glyma.13G259700编码的蛋白质中包含泛素特异性蛋白酶结构域,在拟南芥中已报道了泛素特异性蛋白酶UBP15/SOD2参与了种子大小的调控[24];Glyma.13G261700编码细胞色素P450蛋白,在拟南芥中已报道了SHK1和KLU等基因编码细胞色素P450蛋白,它们都与种子大小的调控有关[20]。水稻和拟南芥的相关研究报道,加速了大豆百粒重候选基因的筛选鉴定。
3.2 重测序数据在大豆基因组研究中的应用
大豆是古四倍体,基因组曾发生过多次复制,导致约75%的基因以多拷贝形式存在,因此对大豆功能基因的研究较为困难。随着2010年大豆全基因组测序工作的完成,以及高通量测序技术带来的高效便捷,通过对来自不同地区和不同类型的大豆品种进行重测序,利用检测到的变异位点进行基因分型,并结合材料表型可进行基因定位等大豆功能基因组研究。Zhou等[36]通过对62个野生大豆、130个地方大豆和110个改良大豆品种进行重测序,结合全基因组关联分析(GWAS)揭示了10个被选择区域和9个驯化或改良性状相关联,并鉴定出包括含油量、株高和茸毛形态等13个农艺性状相关位点。该研究为提高对大豆驯化遗传学认知提供了资源,并为今后遗传资源库中相关性状的等位变异研究提供了参考,从而为大豆作物改良提供了依据。Kulkarni等[37]利用全基因组重测序数据进行比较基因组分析,鉴定出6个与蛋白含量和脂肪酸组分相关的SNP的基因,其中Glyma.10G275800中的单个SNP与蛋白质含量升高以及棕榈酸、油酸和亚油酸含量有关,其他5个基因为包含与该研究分析的各个性状相关的SNP的新基因。本研究利用56份大豆资源(包括野生大豆和栽培大豆)的重测序数据对大豆中与籽粒大小/粒重相关基因进行基因分型,2种类型材料的百粒重表型差异较大,结合基因型分析获得与性状相关的SNP位点;在对筛选到的SNP验证分析中用到了2368份资源重测序数据及部分材料已有的表型数据,进一步验证相关的SNP位点与大豆百粒重的相关性。通过对大量重测序数据及百粒重表型数据的分析,能够较为精准高效地获得与百粒重相关的候选基因,为相关基因的功能研究奠定基础。
4 结论
利用文献检索得到的59个在拟南芥和水稻中已明确生物学功能的与籽粒大小/粒重相关的基因,经过同源分析,在大豆基因组中鉴定到175个同源基因。对基因的表达谱进行分析,发现有22个基因在大豆的种子中特异性表达。利用56份大豆种质资源的重测序数据鉴定出121个SNP位点在野生大豆和栽培大豆间存在分化,其中16个SNP为非同义变异位点,分布于11个基因中。通过扩增测序验证这些非同义变异位点,结合1695份有百粒重表型的资源的重测序数据分析,发现有4个SNP在野生型和突变型所对应的百粒重表型间存在极显著差异,并且野生型变异材料的百粒重≥12g,而突变型变异材料的百粒重<12g。这4个SNP分别位于Glyma.05G019800、Glyma.07G022800、Glyma.13G259700和Glyma.13G261700这4个大豆基因中,可作为大豆百粒重候选基因,为后续相关的基因功能研究奠定基础。

