基于SLAF-seq技术的苗药八爪金龙遗传分析
刘畅 潘婕 刘雄伟 丁晶鑫 周英


摘 要: 为探究苗药八爪金龙群体遗传进化和亲缘关系的远近,该研究利用简化基因组测序技术(SLAF-seq)对42份八爪金龙样品进行测序,获得多态性SLAF 标签。同时采用GATK 和SAMtools软件在多态性SLAF中检测单核苷酸多态性(SNP)分子标记,并利用SNP分子标记分析八爪金龙样品间的遗传分化关系。结果表明:(1)42份八爪金龙共获得246.35 Mb reads,测序质量值Q30 的平均值为95.66%,GC 含量的平均值为41.14%。(2)通过生物信息学的分析,获得1 769 265个SLAF 标签,其中379 829个多态性SLAF 标签,共开发2 299 640个SNPs分子标记。(3)利用开发的SNPs数据构建八爪金龙的系统发育树, 42份八爪金龙分成两个大的类群。第一类群为细柄百两和原变种百两金;第二类群由贵州朱砂根、红凉伞、湖北朱砂根和江西朱砂根组成。江西朱砂根与其余群体关系较远,有明显的分群现象。该研究从基因组水平揭示不同地区八爪金龙资源之间的遗传关系,为八爪金龙种质资源的鉴定和遗传多样性分析提供了理论基础,所开发的SNP 位点可进一步用于挖掘与品质、抗逆性等相关的基因。
关键词: 八爪金龙, SLAF-seq, SNP, 遗传进化, 亲缘关系
中图分类号: Q943 文献标识码: A 文章编号: 1000-3142(2021)07-1145-10
Abstract: In order to explore the genetic evolution and relationship in Radix Ardisia, the 42 materials were used to sequencing base on specific loci amplified fragment sequencing (SLAF-seq). Based on polymorphic SLAF tags, single nucleotide polymorphisms (SNPs) were identified by the GATK and SAMtools softwares, and to analysis the genetic differentiation. The results were as follows: (1) A total of 246.35 Mb reads data were obtained by SLAF-seq, the average of Q30 and GC content was 95.66% and 41.14%, respectively. (2) In total, 1 769 265 high quality SLAF tags were obtained, including 379 829 polymorphic SLAF tags, and a total of 2 299 640 SNPs were obtained. (3)Through the SNPs data, Radix Ardisia were divided into two groups. The first group contained BLX1-8 and BLY1-8, the second group contained ZSG1-8, HL, ZSH1-6 and ZSJ1-6. The results revealed the genetic relationships in the genomic level and provided theoretical basis for germplasm identification and genetic diversity analysis of Radix Ardisia and developed SNPs could be further used for excavating the gene related resistant, quality and so on.
Key words: Radix Ardisia, SLAF-seq, SNP, genetic evolution, genetic relationship
八爪金龙为贵州省苗族常用药物,其药用部位为根及根茎,具有清热解毒、祛风除湿、散瘀止痛等功能,被苗族奉为喉科良药,是苗药验方开喉剑喷雾剂(赵欧等,2016)和养阴口香合剂(吴筑华,2012)的主要成分。八爪金龙药材来源多样,其基原植物为紫金牛科(Myrsinaceae)紫金牛属(Ardisia)植物朱砂根(A. crenata)、红凉伞(A. crenata var. bicolor)、百两金(A. crispa)、大叶百两金(A. crispa var. amplifolia)和细柄百两金(A. crispa var. dielsii) (中国科学院中国植物志编辑委员会, 2002)。研究表明,不同来源的八爪金龙药材化学成分存在差异,其主要成分岩白菜素的含量在不同来源的八爪金龙中存在差异,朱砂根中的皂苷数量远远丰富于百两金(Liu et al., 2016);而百两金中发现的4种黄酮类在朱砂根未被鉴定到(黄伟等,2010)。化学成分的形成与品质、产地、生态等密切相关,而化学成分决定其质量的好坏及其药理作用。
八爪金龙资源主要以野生为主,其地理分布、生态环境、生长周期、采收与产地初加工存在的差异,为八爪金龙用药准确及质量均一带来了难题;其药材使用比较混乱,也使得质量难以控制,严重影响其质量控制以及临床使用的“疗效同等性”。因此,建立有效的鉴定方法,對其遗传结构进行深入分析,明确八爪金龙的起源和亲缘关系,优选出质量较好且稳定的居群,保证其正确使用,对于规范八爪金龙流通和临床有效利用具有重要的指导意义。目前主要的鉴别方法有性状鉴别、显微鉴别、DNA条形码等(陈新莲等,2017;陈文婷等,2020;潘婕等,2020),但这些方法具有片面性强或实验过程复杂等缺点。高通量测序的技术,如简化基因组测序(SLAF-seq)已经被运用于药用植物的种质资源鉴定、遗传定位分析、多态性分析、系统进化分析等(Liu et al., 2016;Du et al., 2019)。
本研究利用SLAF-seq技术对贵州、湖北、江西3个地点的42份八爪金龙种质资源进行测序,获得SLAF多态性标签,应用GATK 和SAMtools软件对SNP 进行检测。基于SNP分子标记,利用生物信息学进行系统进化树、群体结构和PCA分析,从基因组水平揭示不同个体之间的遗传分化关系,评估八爪金龙遗传资源的亲缘关系,为八爪金龙种质资源的鉴定和育种提供理论基础。
1 材料与方法
1.1 试验材料
42份八爪金龙种质资源来源于贵州、湖北和江西,其中贵州30份(8份原变种百两金BLY、8份细柄百两金BLX、8份朱砂根ZSG、6份红凉伞HL),湖北恩施朱砂根ZSH 6份,江西武功山朱砂根ZSJ 6份,对叶片形态进行拍照观察。具体分布地点如表1所示。
1.2 基因组DNA 的制备
取约40 mg八爪金龙叶片,用75%医用酒精棉球擦拭叶片,将其剪碎后,加入液氮研磨,利用试剂盒提取八爪金龙基因组总DNA,用1%琼脂糖凝胶电泳和Nanodrop 2000检测八爪金龙基因组DNA的质量和浓度。
1.3 Illumina HiSeq 测序及产出数据的质量分析
由于八爪金龙目前尚无基因组序列信息发布,同时也无其近缘物种的基因组作参考,根据八爪金龙基因组的大小以及GC 含量等信息。选取水稻日本晴(Oryza sativa ssp. japonica)参考基因组进行酶切预测,选择最适酶切方案,以期获得足够标签数的酶切片段(SLAF标签)(Davey et al., 2013)。选取HaeIII+ScaI-HF酶进行酶切,对质量合格的八爪金龙个体基因DNA 分别进行酶切。将酶切片段的3′端加A,加 Dual-index接头(Kozich et al., 2013),然后进行PCR扩增、纯化、建库,质检合格的文库,利用Illumina HiSeq 进行测序。
1.4 SLAF 标签和SNP 标记的开发
利用Dual-index识别测序得到的原始数据,获得样品的reads,对低质量的reads及read接头进行过滤,获得高质量的reads。对高质量的reads进行聚类分析,聚在一起的为同一个SLAF 标签,其不同个体间序列的差异为多态性SLAF 标签。以测序深度最高的SLAF标签序列作为参考序列,利用BWA软件(Li et al., 2009)将高质量的reads比对到参考基因组上,结合GATK(McKenna et al., 2010)和SAMtools(Li et al., 2009)软件计算SNP分子标记,取两个软件的交集得到最终的SNP分子标记,MAF>0.05的SNP被认为是代表性的SNP。
1.5 遗传结构分析
将具有代表性的高质量SNP使用MEGA X(Kumar et al., 2018)软件,基于邻接法(neighbor-joining),采用Kimura 2-parameter模型,bootstrap重复1 000次,构建各样品的系统发育树。基于SNP数据,利用admixture软件(Alexander et al., 2009),分析样品的群体结构,假設样品的分群数(K值)为1~10,进行聚类分析,利用交叉验证法验证聚类结果,交叉验证错误率最小时为最佳分群数。同时利用EIGENSOFT软件(Price et al., 2006),进行主成分分析(principal components analysis)(PCA),得到样品的主成分聚类情况。
2 结果与分析
2.1 八爪金龙的形态特征
苗药八爪金龙由朱砂根、红凉伞、百两金和细柄百两金等的根茎加工而成。朱砂根(ZS)、红凉伞(HL)、原变种百两金(BLY)及细柄百两金(BLX)在叶片形态上存在差异,细柄百两金叶片较狭窄,朱砂根的叶片边缘有齿,红凉伞叶片背面是红色的(图1)。百两金及其变种主要区别在于叶片长度及宽度。朱砂根变种红凉伞的叶背、花梗、花萼及花瓣均带紫红色(图1)。
2.2 建库评估
利用SLAF-predict 软件对八爪金龙基因组进行酶切方案预测,选择HaeⅢ+ScaI-HF酶对八爪金龙基因组进行酶切,SLAF 标签长度为414~464 bp,预测得到222 509个SLAF 标签(表2)。将水稻日本晴的测序reads与其参考基因组进行比对,水稻日本晴双端比对率为96.96%,酶切比例为95.79%(表3)。
2.3 测序数据统计与评估
经Illumina HiSeq 测序平台进行测序,共获得246.35 Mb reads,测序后各样品的reads数目在1 491 644~11 923 328 bp 范围内(图2)。测序质量值Q30平均值为95.66%,测序碱基错误率低,所获数据合格;测序获得GC 含量平均值为41.14%,达到测序要求。
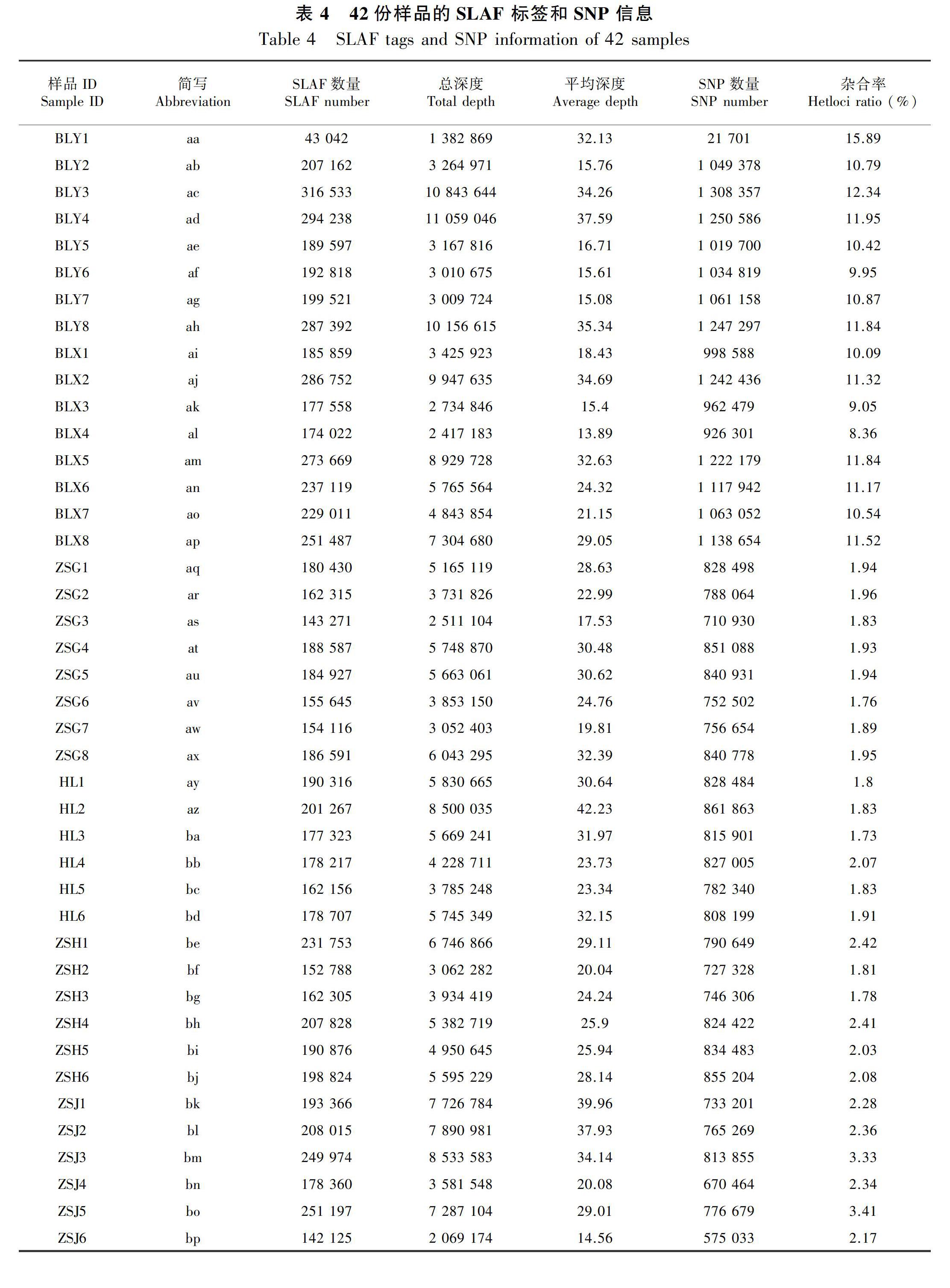
2.4 SLAF 标签与SNP 标记的鉴定
通过序列分析,从42份八爪金龙中共获得了1 769 265 个SLAF 标签(表4)。标签的平均测序深度为26.48,鉴定到379 829个多态性SLAF标签,对多态性SLAF标签进行分析,共开发得到2 299 640个SNP 位点(表4)。
2.5 系统发育分析
基于开发的2 299 640个SNP,采用 Kimura 2-parameter 模型,构建所有个体的 NJ 系统发育树(图3)。42份样品可分成2 个大的类群,第一类群由细柄百两金(BLX1-8)和原变种百两金(BLY1-8),显示它们之间有更近的亲缘关系,细柄百两金可能由原变种百两金上溯而来。第二类群由贵州朱砂根(ZSG1-8)、红凉伞(HL)、湖北朱砂根(ZSH1-6)和江西朱砂根(ZSJ1-6)组成,表明它们之间有较近的亲缘,其分化有着明显的地域特征。
2.6 遗传结构分析
基于开发出的2 299 640个SNP 位点,通过admixture软件分析群体结构。K 值为 1~10 的聚类情况(图4)及各个 K 值对应的交叉验证错误率(图 5),交叉验证错误率最小时为最优分群组。当 K=6时,交叉验证错误率最小,因此最佳分群数为6,将42 份八爪金龙种质资源分为6个亚组,表明八爪金龙种间遗传差异较大,形成明显的种群遗传分化。当K=2时,百两金和朱砂根明显分开;当K=3时,朱砂根、百两金和江西武功山朱砂根区分开;当K=4时,将细柄百两金、原变种百两金、江西武功山朱砂根和贵州朱砂根区分开;当K=5时,将细柄百两金、原变种百两金、江西武功山朱砂根、贵州朱砂根和湖北朱砂根区分开;当K=6时,将细柄百两金、原变种百两金、江西武功山朱砂根、贵州朱砂根、湖北朱砂根区分开和红凉伞区分开,但存在基因渗入的情况。
2.7 PCA分析
用EIGENSOFT软件对2 299 640个SNP位点进行PCA分析,得到42份八爪金龙主成分聚类分析结果(图6)。通过PCA 分析将个体聚为三维,PC1 代表第一主成分,PC2 代表第二主成分,PC3 代表第三主成分,且 PC1、PC2、PC3 的方差贡献率分别为39.21%、18.58%、7.71%。从图6可以看出,样品间存在差异,来自同一地方的朱砂根/百两金基本上聚在一起,说明它们之间的亲缘关系比较近。图中不同颜色表示所属的群体,能够反映个体的聚类情况,群体之间的距离能够反映亲缘关系的远近。群体之间的距离越远说明遗传背景越远,将 42 份材料大致分为 A、B、C、D 四组。A 组为原变种百两金(除BLY1外);B 组为细柄百两金(除BLX5、BLX6、BLX8外);C组为贵州朱砂根(ZSG1-8)、湖北朱砂根(ZSH)和红凉伞(HL),说明这三个种或变种的遗传背景差异较小;D 组为江西朱砂根(ZSJ1-6),与其余群体关系较远,有明显的分群现象。
3 讨论与结论
近几年,RAPD、ISSR 等分子标记已应用到八爪金龙种质资源的评价和遗传分析,采用RAPD分子标记技术对朱砂根遗传多样性进行分析,朱砂根群体在DNA 水平上存在着丰富的遗传多态性(江香梅等,2006)。骆亮等(2020)分析了20个朱砂根野生居群的255个样品的遗传多样性,发现5条ISSR 引物共扩增出96条多态性条带。但由于八爪金龙无参考基因组,常规方法开发的特异分子标记数量不多。单核苷酸多态性(single nucleotide polymorphism,SNP)是一种较为有效的分子标记(Liu et al., 2012),可用于动植物的群体遗传和进化关系研究。赵俊生等(2016)利用SNP分子标记分析化橘红的种质资源,从中筛选出21个多态性SNP位点,为化橘红种质资源鉴定提供了有效的分子标记。孙清明等(2013)利用SNP分子标记对363份荔枝种质资源进行亲缘关系鉴定,为荔枝新种质的亲本来源鉴定提供分子标记。
简化基因组测序(SLAF-seq )技术能够在无参基因组物种中实现SNP特异性分子标记开发,可用于植物遗传多样性的研究。有研究利用SLAF-seq 技术已经构建了枸杞果实和叶片性状的SNP高密度遗传图谱(Zhao et al., 2019)和丹參高密度遗传连锁图(Liu et al., 2016)。Du et al.(2019)采用SLAF-seq方法,对7个中国本地山楂属植物和2个欧洲、1个北美外来山楂属植物的53个家系进行了分析,共鉴定出933 450个SNP,用于物种基因组进化研究。姜涛等(2020)利用SLAF-seq测序技术对39份连翘种质资源遗传多样性和SNP分子标记进行研究,共获得180 974 1个SNP分子标记,将连翘资源分为4组,为其遗传结构分类提供新的信息。唐晓敏等(2020)利用SLAF-seq 技术对广金钱草样品进行测序,开发得到SNP多态标记220 704个,为种质保护和遗传改良提供参考。本研究利用SLAF-seq 技术对42份八爪金龙种质资源进行测序,共开发得到2 299 640个SNP位点,为后期八爪金龙特异性SNP开发提供基础,同时为八爪金龙遗传育种改良,提高育种效率提供数据参考。
植物的生长环境、地理位置、海拔高度对物种的遗传分化和种群结构能够产生影响。地理分布和海拔高度的差异影响浙江产车前草种群的遗传分化(郭水良等,2002)。不同地理位置的广金钱草种源间存在一定的遗传分化(唐晓敏等,2020)。系统发育树主要是描述物种之间的分类学和进化关系以及具有共同祖先的物种之间的进化关系的树形图(韩凤侠等,2008)。本研究构建了42份八爪金龙样品的系统发育树,系统发育树可以将百两金和朱砂根区分开。课题组前期对八爪金龙基原植物DNA 条型码进行筛选,ITS 序列能区别百两金和朱砂根,但无法区分朱砂根和红凉伞(潘婕等,2020)。因此,百两金和朱砂根亲缘关系较远,化学成分存在差异,需进一步明确其药理作用的差异,保证其正确使用。
叶片形态表明,贵州朱砂根和湖北恩施州朱砂根叶片形态相近,而与江西武功山朱砂根叶片形态差距较大。系统发育树结果表明,贵州朱砂根和湖北恩施州朱砂根亲缘关系较近,与江西武功山朱砂根关系较远;红凉伞与贵州的朱砂根和湖北恩施州的朱砂根亲缘关系较近。贵州和湖北恩施州同属苗族聚集区,江西武功山与贵州和湖北恩施州在人为环境和地理环境方面存在差异。因此,江西武功山朱砂根种源在进化过程中受到了人文环境、地理环境隔离的影响,与贵州、湖北恩施州的朱砂根种源基因交流较少,逐渐出现分化。
朱砂根和红凉伞叶片形态相近,但红凉伞的叶背、花梗、花萼及花瓣均带紫红色,DNA条形码无法区分朱砂根和红凉伞(陈新莲等,2017;潘婕等,2020)。当K=6 时的群体遗传结构分析表明,将细柄百两金、原变种百两金、江西武功山朱砂根、贵州朱砂根、湖北朱砂根和红凉伞区分开,说明八爪金龙种源间存在一定的遗传分化。基于SNP的分子标记,能够区分红凉伞和朱砂根,因此,我们将进一步开发红凉伞和朱砂根特异的SNP分子标记。
基于SNP 标记构建的系统发生树为研究八爪金龙品种之间亲缘关系提供了证据,虽然八爪金龙野生种可能具有相似的起源种,但在演化过程中逐渐出现分化;同时本研究开发的SNP 位点为后续八爪金龙品种间共有或特异性标记的开发提供基础,理清归纳八爪金龙品种间生物学特征。
参考文献:
ALEXANDER DH, NOVEMBRE J, LANGE K, et al., 2009. Fast model-based estimation of ancestry in unrelated individuals [J]. Genome Ees, 19(9): 1655-1664.
CHEN WT, TONG JY, ZHONG HY, et al., 2020. Character and microscopic identification of ardisiae crenatae radix and its adulterants [J]. J Chin Med Mat, 43(1): 52-55. [陈文婷, 童家赟, 钟慧怡, 等, 2020. 朱砂根等四种易混淆中药的性状和显微比较鉴别[J]. 中药材, 43(1): 52-55.]
CHEN XL, ZHOU JG, MA SJ, et al., 2017. Molecular identification of ardisiae crenatae radix and its adulterants based on ITS2 sequence [J]. Mod Chin Med, 19(7): 939-943. [陈新连, 周建国, 马双姣, 等, 2017. 基于ITS2序列的朱砂根及其混伪品分子鉴定[J]. 中国现代中药, 19(7): 939-943.]
Chinese Flora Editorial Board, Chinese Academy of Sciences, 2002. Flora Reipublicae Popularis Sinicae [D]. Beijing: Science Press. [中国科学院中国植物志编辑委员会, 2002. 中国植物志 [D]. 北京: 科学出版社.]
DAVEY JW, CEZARD T, FUENTES-UTRILLA P, et al., 2013. Special features of RAD Sequencing data: Implications for genotyping [J]. Mol Ecol, 22(11): 3151-3164.
DU X, ZHANG X, BU HD, et al., 2019. Molecular analysis of evolution and origins of cultivated hawthorn (Crataegus spp.) and related species in China [J]. Front Plant Sci, 10: 443.
GUO SL, ZHANG DX, CAO T, 2002. Principal axes analyses on population genetic differentiation of Plantago asiatica in Zhejiang, Yingyong Shengtai Xuebao [J]. Chin J Appl Ecol, 13(10): 1283-1286 [郭水良, 张东旭, 曹同, 2002. 浙江产车前(Plantago asiatica)种群遗传分化的主坐标分析 [J]. 应用生态学报, 13(10): 1283-1286.]
HAN FX, 2008. On the principle of common ancestry to understand the phylogenetic tree [J]. Bull Biol, 43(9): 14-15. [韩凤侠, 2008. 共同祖先原则和系统发育树的解读[J]. 生物学通报, 43(9): 14-15.]
HUANG W, TAN GS, XU KP, et al., 2010. Cytotoxic constituents from the root of Ardisia crispa [J]. Nat Prod Res Dev, 22(6): 949-951. [黃伟, 谭桂山, 徐康平, 等, 2010. 百两金细胞毒活性成分研究[J]. 天然产物研究与开发, 22(6): 949-951.]
JIANG T, WEN CX, TIAN W, et al., 2020. SNP molecular markers development and genetic diversity analysis of forsythia suspensa based on SLAF-seq technology [J/OL]. Mol Plant Breed, (2020-07-20)https://kns.cnki.net/kcms/detail/46.1068.S.20200720.1130.010.html.] [姜涛, 温春秀, 田伟, 等, 2020. 基于SLAF-seq 技术连翘SNP 分子标记开发及遗传多样性分析[J/OL]. 分子植物育种, (2020-07-20) https://kns.cnki.net/kcms/detail/46.1068.S.20200720.1130.010.html.]
JIANG XM, WEN Q, YE JS, et al., 2006. RAPD analysis of genetic diversity in Ardisia crenata Smis [J]. Acta Agric Univ Jiangxi, 5(28): 762-765. [江香梅, 温强, 叶金山, 等, 2006. 朱砂根群体遗传多样性RAPD分析 [J]. 江西农业大学学报, 5(28): 762-765.]
KOZICH JJ, WESTCOTT SL, BAXTER NT, et al., 2013. Development of a dual-index sequencing strategy and curation pipeline for analyzing amplicon sequence data on the MiSeq Illumina sequencing platform [J]. Appl Environ Microbiol, 79(17): 5112-5120.
KUMAR S, STECHER G, LI M, et al., 2018. MEGA X: Molecular evolutionary genetics analysis across computing platforms [J]. Mol Biol Evol, 35(6): 1547-1549.
LI H, DURBIN R, 2009. Fast and accurate short read alignment with Burrows-Wheeler transform [J]. Bioinformatics, 25(14): 1754-1760.
LI H, HANDSAKER B, WYSOKER A, et al., 2009. The sequence alignment/map format and SAMtools [J]. Bioinformatics, 25(16): 2078-2079.
LIU DL, ZHANG X, ZHAO Y, et al., 2016. Three new triterpenoid saponins from the roots of Ardisia crenata and their cytotoxic activities [J]. Nat Prod Res, 30(23): 2694-2703.
LIU J, HUANG SM, SUN MY, et al., 2012. An improved allele-specific PCR primer design method for SNP marker analysis and its application [J]. Plant Methods, 8(1): 34.
LIU T, GUO LL, PAN YL, et al., 2016. Construction of the first high-density genetic linkage map of Salvia miltiorrhiza using specific length amplified fragment (SLAF) sequencing [J]. Sci Rep, 6: 24070.
LU H, REDUS MA, COBURN JR, et al., 2005. Population structure and breeding patterns of 145 U.S. Rice cultivars based on SSR marker analysis [J]. Crop Sci, 85: 484-492.
LUO L, ZHANG WC, LI L, et al., 2020. Genetic diversity analysis of Ardisia crenata in different populations by fluorescence ISSR [J/OL]. Mol Plant Breed, (2020-08-07) https://kns.cnki.net/kcms/detail/46.1068.S.20200807.1415.006.html. [駱亮, 张文春, 李龙, 等, 2020. 不同居群朱砂根Ardisia crenata的荧光ISSR 遗传多样性分析[J/OL]. 分子植物育种, (2020-08-07) https://kns.cnki.net/kcms/detail/46.1068.S.20200807.1415.006.html.]
MCKENNA A, HANNA M, BANKS E, et al., 2010. The genome analysis toolkit: A mapreduce framework for analyzing next-generation DNA sequencing data [J]. Genome Res, 20(9): 1297-1303.
PAN J, LIU C, FENG TT, et al., 2020. Screening and identification on ITS sequences of original plants from Radix Ardisia[J]. Mol Plant Breed, 18(24): 8187-8195. [潘婕, 刘畅, 俸婷婷, 等, 2020. 苗药八爪金龙基原植物DNA 条型码的筛选与鉴定[J]. 分子植物育种, 18(24): 8187-8195.]
PRICE AL, PATTERSON NJ, PLENGE RM, et al., 2006. Principal components analysis corrects for stratification in genome-wide association studies [J]. Nat Genet, 38(8): 904-909.
SUN QM, LI YZ, XIANG X, et al., 2013. A novel litchi germplasm (Litchi chinensis Sonn.), Yujinqiu, identified by EST-SSR and SNP analysis[J]. Mol Plant Breed, 11(3): 403-414. [孙清明, 李永忠, 向旭, 等, 2013. 利用SNP 和EST-SSR 分子标记鉴定荔枝新种质御金球[J]. 分子植物育种, 11(3): 403-414.]
TANG XM, ZHANG CR, ZHOU LY, et al., 2020. SNP loci development and genetic analysis of Desmodium styracifolium(Osbeck) Merr based on SLAF-Seq technology [J]. Mol Plant Breed, 18(18): 6101-6107. [唐晓敏, 张春荣, 周良云, 等, 2020. 基于SLAF-Seq技术的广金钱草SNP位点开发及遗传分析[J]. 分子植物育种, 18(18): 6101-6107.]
WU ZH, 2012. Prevention and treatment of metabolic syndrome with miao medicine Yangyin Kouxiang Mixture [J]. J Med Pharm Chin Minor, (8): 41-43. [吴筑华, 2012. 苗药养阴口香合剂对代谢综合征的防治[J]. 中国民族医药杂志, (8): 41-43.]
XU LL, LI TJ, ZHANG MY, et al., 2009. Interspecific relationships and variation of 12 species in Ardisia Sw. (Myrsinaceae) based on ITS and trnL-F data sets [J]. Acta Hortic Sin, 36 (10): 1531-1537. [徐玲玲, 李同建, 張美云, 等, 2009. 基于核ITS与叶绿体trnL-F序列分析12种紫金牛属植物的种间关系与变异[J]. 园艺学报, 36(10): 1531-1537.]
ZHAO JH, XU YH, LI HX, et al., 2019. A SNP-based high-density genetic map of leaf and fruit related quantitative trait loci in wolfberry (Lycium Linn.) [J]. Front Plant Sci, 10: 977.
ZHAO JS, YANG XY, ZENG XY, et al., 2016. Analysis on germplasm resources of exocarpium citri grandis using SNP molecular markers [J]. Mol Plant Breed, 14(5):1203-1211. [赵俊生, 杨晓燕, 曾祥有, 等, 2016. 利用SNP 分子标记分析化橘红种质资源[J]. 分子植物育种, 14(5): 1203-1211.]
ZHAO O, DU Y, WEI WL, 2016. Analysis of volatile oils of kai hou sword and its major constituted radix sophorae tonkinensis and Ardisia crispa (Thunb.) A. DC by GC-MS [J]. Hubei Agric Sci, 55(6): 1568-1571. [赵欧, 杜莹, 韦万丽, 2016. 开喉剑及组方药材山豆根、八爪金龙挥发油的GC-MS 分析[J]. 湖北农业科学, 55(6): 1568-1571.]
(责任编辑 周翠鸣)

