耐力运动对人外周血微RNA表达谱的影响
孙庭汉,刘美,孙晓
(中国医科大学 1.体育部;2.药学院离子通道药理研究室,沈阳 110122)
科学适宜的耐力运动对慢阻肺、糖尿病等具有较好的保健和治疗效果[1-2]。对骨骼肌全基因组表达分析后发现,运动能够显著上调三羧酸循环相关的基因表达,说明运动能够调控能量代谢[3]。运动对身心健康的积极影响已被广泛研究。目前关于遗传信息与体育活动相关性以及运动影响体内分子过程的研究仍然有限。
微RNA(microRNA,miRNA)是一类普遍存在于动植物体内的非编码小分子RNA,miRNA在基因转录后调控中发挥核心作用,可通过调节靶基因的表达发挥多种生物学功能[4]。机体内的miRNA在运动诱导的肌肉结构重塑、能量代谢和免疫调节等作用中起关键作用[5-6]。不同训练模式可能导致体内循环miRNA发生改变,由于miRNA在血浆或血清中的稳定性,循环miRNA可以作为一种潜在的生物标志物。
本研究通过生物信息学方法分析经8周耐力运动前后健康受试者外周血中miRNA的表达谱变化,预测差异表达miRNA作用的靶基因及可能的靶向信号通路,为后续的机制研究提供依据。
1 材料与方法
1.1 芯片数据来源
本研究从美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)公 共基因芯片数据平台(gene expression omnibus,GEO)数据库(https://www.ncbi.nlm.nih.gov/geo/)下载基因芯片数据集GSE133910,该研究共纳入23例健康且未经锻炼的受试者,其中男13例,女10例,平均年龄(47.9±8.3)岁(范围为30~63岁),体质量为(82.0±13.1)kg,身高为(173.9±7.0)cm。未经锻炼的纳入标准为:既往无竞技耐力训练,入组前1年每周运动时间<1 h,男性最大摄氧量(maximal oxygen consumption,VO2max)< 45 mL/(min·kg),女 性VO2max <40 mL/(min·kg)。受试者经8周的耐力运动,每周进行4次45 min运动训练,可以选择持续步行或慢跑,通过心率监测方式保证整个运动过程中,心率储备(heart rate reserve,HRR)强度保持在70%以上。然后对受试者外周血中的miRNA表达谱进行分析,对照耐力运动前后外周血中miRNA水平的变化。PAXGene Blood miRNA试剂盒(德国Qiagen公司)分离出包括小RNA在内的总RNA。通过采集全血,应用PAXGene全血miRNA分离试剂盒(德国凯杰公司)分离出包括小RNA在内的总RNA。检测的基因芯片平台为GPL25134 Agilent-070156 Human_miRNA_V21.0_Microarray 046064(miRNA ID version),芯片共检测了2 549个miRNA。
1.2 差异表达miRNA筛选
用R语言软件读取下载矩阵文件,使用limma包对耐力运动后和运动前受试者的外周血miRNA表达谱进行分析,得到差异表达miRNA。筛选标准为log2基因表达差异倍数(fold change,FC)绝对值≥1,由于miRNA和靶基因的关系为一对多,为了降低出现假阳性结果的概率,在miRNA靶基因筛选时,以P< 0.01作为统计学差异表达的判定标准。利用R语言ggplot2包绘制耐力运动前后外周血差异表达miRNA的火山图。
1.3 差异miRNA靶基因筛选
miRNA在体内主要结合在靶基因的3’UTR,下调靶基因的表达。miRWalk(http://mirwalk.umm.uni-heidelberg.de/)是用于预测miRNA靶基因的开源在线分析工具[10]。首先应用miRWalk中的外联数据库miRDB对耐力运动前后差异表达的miRNA进行靶基因预测,设定阈值为0.9,获得靶基因数据集A;再利用DIANA tools中的MicroT-CDS数据库(http://diana.imis.athena-innovation.gr/DianaTools/)对差异表达的miRNA进行靶基因预测[11],获得靶基因数据集B。然后,不同数据获得的差异表达miRNA靶基因取交集作为差异miRNA的靶基因。利用Cytoscape软件对筛选的差异表达miRNA和靶基因进行网络分析。
1.4 差异miRNA靶基因的GO和KEGG分析
本研究通过DAVID(https://david.ncifcrf.gov/)数据库[12]在线分析提供所需的基因本体论(gene ontology,GO)生物功能富集数据,GO按照生物途径(biological process,BP)、细胞定位(cellular component,CC)和分子功能(molecular function,MF)对基因进行注释和分类。除此之外,进行京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)信号通路分析[13],从而发现差异miRNA可能参与的生物学通路。采用EASE Score或Fisher Exact统计方法,GO和KEGG各项筛选条件为P< 0.05。应用R语言ggplot2包根据分析结果绘制气泡图。
1.5 蛋白间相互作用(protein-protein interaction,PPI)网络构建和关键基因筛选
STRING(https://string-db.org/)网站是用于评估PPI的在线分析工具[14]。将本研究筛选出的所有差异表达miRNA靶基因导入STRING(版本11.0)分析工具,探讨它们之间可能的相互作用关系。设置筛选条件为互作最大值=0,最小互作得分≥ 0.40。通过STRING得到PPI网络后,把STRING的计算结果导入Cytoscape(版本3.7.2)[15],应用Cytoscape插件cyto-Hubba依据Closeness值大小,筛选出PPI网络中处于关键位置的前10个基因进行分析。
2 结果
2.1 耐力运动前后外周血中差异miRNA的筛选
本研究选用基因表达芯片GSE133910,经统计分析,健康受试者经8周耐力运动后,外周血中共筛选出8个差异表达的miRNA,其中2个miRNA表达上调,6个miRNA表达下调(FC≥ 2,P< 0.01),其中hsamiR-8052、hsa-miR-6889-5p和hsa-miR-4457变化最为明显,见表1。利用火山图分析和观察FC值2倍以上变化的miRNA分布及统计学差异,其中标注为灰色的是FC值2倍以下或者P> 0.05的基因,表达下调的基因标注为蓝色,表达上调的基因标注为红色,见图1。
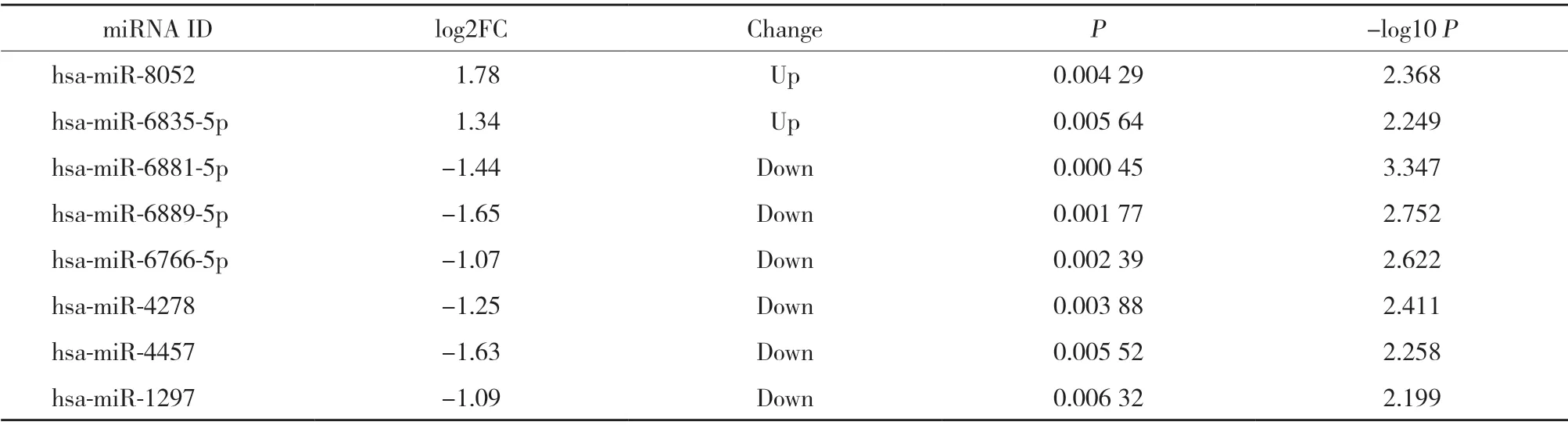
表1 耐力运动后外周血差异表达的miRNA分析Tab.1 Analysis of miRNA differentially expressed in peripheral blood after endurance exercise
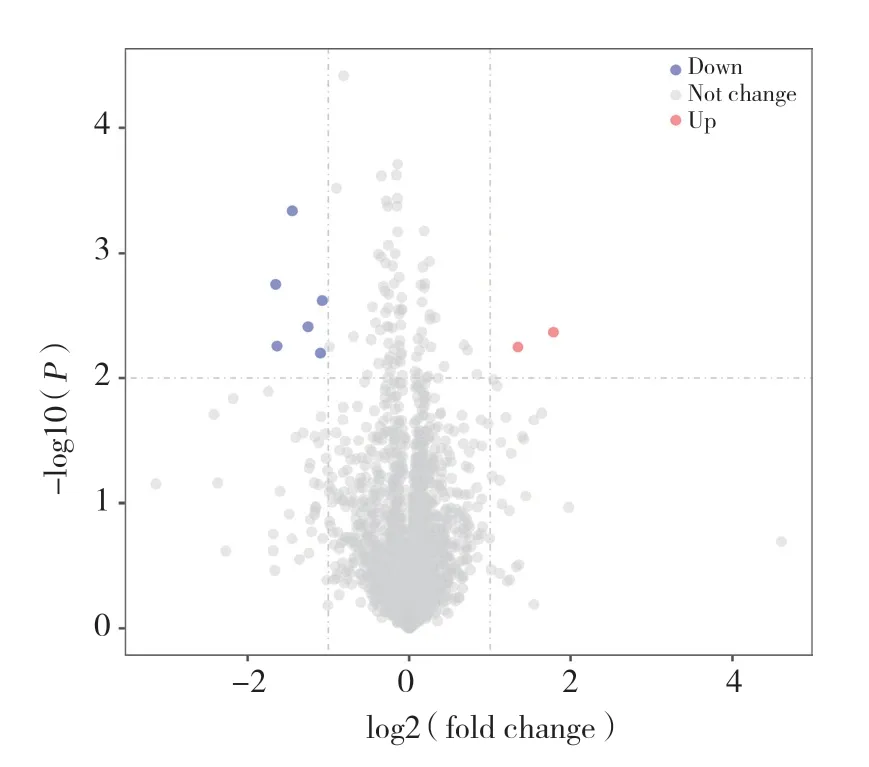
图1 8周耐力运动后血中miRNA表达变化火山图Fig.1 Volcano plot profiling miRNA differential expression in human peripheral blood between trained and untrained groups
2.2 差异miRNA靶基因的筛选
分别通过miRWalk及microT-CDS数据库筛选出差异表达miRNA靶基因进行分析(图2),由miRWalk数据库筛选出靶基因1 424个,由microT-CDS数据库筛选出靶基因1 030个。利用R语言的VennDiagram包对2组数据进行韦恩图分析,可见2个数据库共同预测的靶基因个数为251个。韦恩图结果显示(图2),可差异miRNA有较多共同的靶基因。将miRNA及其调控的靶基因,通过Cytoscape软件进行可视化,见图3。

图2 miRWalk和microT-CDS数据库预测miRNA靶基因的韦恩图Fig.2 Venn diagram demonstrating the overlap between target genes of differentially expressed miRNA predicted with the miRWalk and microT-CDS databases
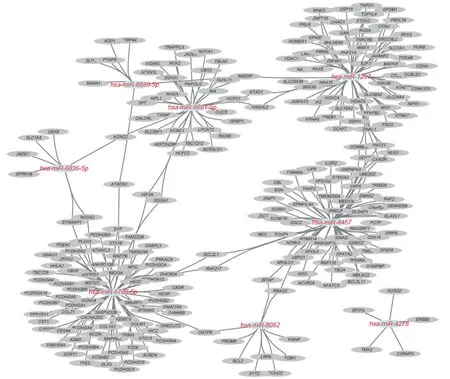
图3 差异miRNA靶基因及通路互作图Fig.3 Visualization of interactions between differentially expressed miRNAs and their corresponding target genes
2.3 差异miRNA靶基因GO和KEGG富集分析
采用DAVID数据库对预测的差异表达miRNA靶基因进行GO和KEGG信号通路富集分析,见图4。GO富集分析发现,差异表达miRNA靶基因主要参与离子通道结合、突触后致密蛋白结构域结合、mRNA结合等分子功能;BP主要涉及外在凋亡通路、GTP酶激活、同种细胞黏附、抑制RNA聚合酶Ⅱ启动子等;CC在质膜、核膜等部位。KEGG通路分析结果显示,miRNA靶基因主要与多个肿瘤信号通路、谷氨酸能突触、Wnt信号通路、甘油磷脂代谢通路等有关。

图4 差异表达miRNA靶基因GO和KEGG通路富集分析Fig.4 GO and KEGG pathway enrichment analysis of differential miRNA target genes
2.4 差异表达miRNA靶基因PPI网络分析
将251个差异表达miRNA靶基因导入STRING进行分析后得到PPI网络,该网络由251个节点蛋白和285条相互作用关系构成(PPI enrichmentP=1.29×10-4)。将STRING分析结果导入Cytoscape,利用插件cytoHubba对候选miRNA靶基因的closeness筛选出的前十个关键基因分别为PRKACA、SMURF1、RHOA、GNAO1、CBL、UBE2D2、GSK3B、GNAQ、BCL2L1和RRAS2(图5),关键基因用黄色、橙色、红色标注,颜色越深表明该基因在网络中的作用越为重要。筛选出的关键基因主要分布在能量代谢平衡、细胞增殖等通路。

图5 CytoHubba插件筛选出PPI网络中处于关键位置的基因Fig.5 The top 10 hub genes in the PPI network selected by cytoHubba
3 讨论
miRNA是机体内广泛分布的一类小RNA,主要通过诱导mRNA的降解或干扰蛋白质的翻译过程,发挥转录后调控作用。耐力运动能够通过调控相关miRNA的表达,有效提高机体肌肉的强度,增强心血管功能,同时对改善呼吸、血液、能量代谢等功能具有积极影响[7]。
在本研究中,通过对经8周耐力运动后的健康受试者外周血miRNA水平进行检测,共发现8个miRNA(FC≥2,P< 0.01),分别是miR-8052、miR-6835-5p、miR-6881-5p、miR-6889-5p、miR-6766-5p、miR-4278、miR-4457和miR-1297。其中,研究[8]发现miR-6835的表达与血管内皮的炎症和脂滴形成有关,脂联素受体1(adiponectin receptor 1,AdipoR1)被证明是miR-6835-5p的直接靶基因,而miR-6835-5p通过抑制AdipoR1的表达可刺激胰岛素的分泌,对于2型糖尿病可能是一种有效的治疗方法[9]。在耐力运动后,健康受试者外周血的miR-6835-5p的表达显著升高,从而抑制AdipoR1的表达,从而影响糖类和脂肪的代谢过程,进而影响机体的能量代谢[10]。研究[11-12]发现miR-6881的表达与人脂肪干细胞的成骨分化有关,血清miR-1297可作为蛛网膜下腔出血及多种肿瘤的生物标志物,并可能影响肿瘤的发生发展。
通过数据库对差异表达的miRNA靶基因进行预测,结果显示共有251个靶基因可能受到差异miRNA的靶向作用。本研究富集的KEGG信号通路及通路中富集的基因为后续运动对肿瘤作用机制的研究提供一定的参考。KEGG通路还涉及细胞增殖、能量代谢(GTP酶激活通路)和脂质代谢通路等。1项对健康老年人肌肉的全基因组分析研究[13]发现,经耐力运动后与能量代谢、物质代谢、肌肉质量、肌肉收缩、肌肉成分信息转导和亚铁血红素的生物合成等相关基因表达改变。
此外,本研究经cytoHubba筛选出的关键基因中PRKACA(cAMP依赖性蛋白激酶A催化亚基α)能够调节多种蛋白的翻译表达,包括离子通道蛋白、转录激活蛋白和糖原代谢调节酶的表达[14]。SMURF1与多种重要的生物学途径相关,包括骨生成途径、非经典的Wnt途径和促分裂原活化的蛋白激酶途径等,并且研究[15]发现SMURF1在骨骼形成和肿瘤细胞侵袭方面与年龄和生理状态有关。GSK3B除能够调控糖原合成酶的活性外,还能作用于众多信号蛋白、结构蛋白和转录因子[16]。通过本研究结果及查阅相关文献结合既往研究,猜测耐力运动可能通过miR-6835-5p/PRKACA通路调控机体的能量代谢平衡,通过miR-6766-5p/SMURF1通路促进骨骼生长,或通过miR-6881-5p/GSK3B通路调控糖代谢等。本课题由于未进行相应的验证,具有一定的局限性,后续将开展进一步的机制研究。
综上,本研究发现耐力运动显著影响外周血miRNA的表达水平,差异表达的miRNA可能通过肿瘤、能量代谢、糖代谢、骨生成等通路介导耐力运动对机体的生物学作用,通过综合多平台数据库分析能够有效预测相关靶蛋白富集的生物功能和通路,为进一步研究耐力运动对体内分子过程影响提供参考。

