柑橘黄化脉明病毒RT-RPA检测方法的建立
马志敏,许建建,段玉,王春庆,苏越,张琦,宾羽,周常勇,宋震
柑橘黄化脉明病毒RT-RPA检测方法的建立
马志敏,许建建,段玉,王春庆,苏越,张琦,宾羽,周常勇,宋震
西南大学柑桔研究所/国家柑桔工程技术研究中心,重庆 400712
【】利用逆转录重组酶聚合酶扩增技术(reverse transcription-recombinase polymerase amplification,RT-RPA),结合侧向流层析(lateral flow dipstick,LFD)试纸条,建立一种快速简便、特异、灵敏、裸眼可视的柑橘黄化脉明病毒(citrus yellow vein clearing virus,CYVCV)检测新方法。根据CYVCV外壳蛋白基因的保守序列设计5对引物,通过样品检测筛选出扩增效果好、特异性强的引物对。将选出的引物进行标记物修饰,并设计对应的特异性探针,分别设置6个反应时间梯度(5、10、20、30、40、50 min)和8个反应温度梯度(37、38、39、40、41、42、43、44℃),对RT-RPA反应条件进行优化,建立CYVCV的RT-RPA检测体系。分别以仅感染了CYVCV、柑橘叶斑驳病毒(citrus leaf blotch virus,CLBV)、柑橘衰退病毒(citrus tristeza virus,CTV)、柑橘碎叶病毒(citrus tatter leaf virus,CTLV)、柑橘裂皮病类病毒(citrus exocortis viroid,CEVd)、柑橘鳞皮病毒(citrus psorosis virus,CPV)、温州蜜柑萎缩病毒(satsuma dwarf virus,SDV)、柑橘黄龙病菌(Liberibacter asiaticus,Las)和柑橘溃疡病菌(subsp.,)柑橘材料的总核酸为模板,采用所筛选的最佳引物、探针和反应条件进行检测,评价所建立RT-RPA检测体系的特异性。将感染了CYVCV的柑橘总RNA样品进行10倍梯度稀释,以原液和10-1、10-2、10-3、10-4、10-5、10-6、10-7稀释液作为模板,平行进行RT-PCR及RT-RPA的检测,评价所建立检测方法的灵敏度。随机采集来自田间9个不同品种柑橘的叶片,同时进行RT-RPA和RT-PCR检测,检验所建立RT-RPA检测方法的适用性。建立了CYVCV的RT-RPA检测体系:最佳引物为CY1-F/R,对应探针为CY1-Probe(47 bp),最佳反应条件为39℃,30 min,特异扩增目的片段为177 bp,检测结果可通过侧向流层析试纸条直接判读。特异性检测结果显示,利用该检测体系仅对感染了CYVCV的样品检测结果为阳性,其余均为阴性;灵敏度检测结果显示,RT-RPA与RT-PCR方法均最低可检测到10-4稀释液,两种方法检测灵敏度相当。随机采集的45株田间柑橘样品中,RT-PCR与RT-RPA均检测出37个阳性样品,检出率均为82.2%,表明该方法检测效果稳定可靠。建立了CYVCV的RT-RPA检测方法,该检测方法操作简单、反应快速,结果裸眼可视,适用于基层条件不足的实验室或者植检站现场快速检测。
柑橘;柑橘黄化脉明病毒;RT-RPA;侧向流层析试纸条;快速检测
0 引言
【研究意义】柑橘黄脉病是由柑橘黄化脉明病毒(citrus yellow vein clearing virus,CYVCV)引起的一种新发虫传病害[1]。CYVCV能够侵染绝大多数柑橘种类,柠檬和酸橙受感染后,叶片黄化、扭曲、明脉,叶脉背面呈水渍状,造成大量减产[2-3],部分杂柑和温州蜜柑受侵染后表现为叶片卷曲等症状,甜橙在实验条件下也可表现明脉等症状。柑橘黄脉病于1988年首次在巴基斯坦发现[4-5],2003年Alshami等[6]通过电子显微镜观察到弯曲的丝状病毒颗粒,并将其病原命名为柑橘黄化脉明病毒。2009年,首次于我国云南省发现该病[7],随后Zhou等[8]对中国11个主要柑橘种植省份进行检测调查,发现该病害在其中9个省份已广泛分布。由于CYVCV可通过带毒接穗远距离传播,并可通过虫传和污染农具等高效传播,近几年来该病害流行呈加速态势,严重威胁柑橘产业,特别是柠檬产业发展。早期检测和应用脱毒苗木是有效防止柑橘黄脉病危害的重要方法,因此建立简便快速、有效的CYVCV检测方法,可为柑橘黄脉病的防控提供重要的技术支持。【前人研究进展】CYVCV属于线性病毒科()印度柑橘病毒属()的正义单链RNA病毒。CYVCV基因组由7 529个核苷酸组成,包含6个开放阅读框(open reading frame,ORF)[9-10]。CYVCV可通过嫁接、绣线菊蚜()和柑橘粉虱()等在柑橘之间传播,可通过机械摩擦接种到苋色藜()、昆诺藜()、菜豆()和豇豆()等草本植株[1,5,11-13]。以往CYVCV的检测技术如指示植物检测法[5]、电子显微镜检测法[6]、血清学检测法[6, 9,14-15]和核酸分子检测技术[9,16-19]等大多具有操作复杂、检测时间长、需要复杂的实验仪器、对实验操作者要求较高等问题。逆转录重组酶聚合酶扩增(reverse transcription-recombinase polymerase amplification,RT-RPA)是一种新型等温核酸扩增技术,该技术的原理主要模仿T4噬菌体的核酸复制机制,依赖重组酶、单链结合蛋白和链置换聚合酶,在恒温条件下实现与模板的特异性识别和扩增,其目标产物可通过凝胶电泳或添加特异性探针通过试纸条呈现[20-22]。由于具有快速、灵敏、操作简便等特点,该技术现已广泛应用于多种动植物病害的检测[23-28]。在柑橘上,该技术已应用于柑橘黄龙病菌(Liberibacter asiaticus,Las)的快速检测[29]。【本研究切入点】目前,国内外尚未有利用RT-RPA对CYVCV进行检测的报道。【拟解决的关键问题】利用RT-RPA技术结合侧向流层析试纸条,通过引物筛选和反应条件优化,建立一种简易、快速、裸眼可视的CYVCV检测新方法,为基层条件不足的实验室或植检站现场快速检测CYVCV提供新的选择。
1 材料与方法
试验于2020年在西南大学柑桔研究所脱毒中心完成。
1.1 材料
1.1.1 供试材料 本试验所使用的感染了柑橘黄化脉明病毒、柑橘叶斑驳病毒(citrus leaf blotch virus,CLBV)、柑橘衰退病毒(citrus tristeza virus,CTV)、柑橘碎叶病毒(citrus tatter leaf virus,CTLV)、柑橘裂皮病类病毒(citrus exocortis viroid,CEVd)、柑橘鳞皮病毒(citrus psorosis virus,CPV)、温州蜜柑萎缩病毒(satsuma dwarf virus,SDV)、柑橘黄龙病菌和柑橘溃疡病菌(subsp.,)的柑橘材料及健康柑橘材料,均由柑桔研究所脱毒中心提供。田间检测样品采于柑桔研究所展示园和母本园。
1.1.2 主要试剂与仪器 Trizol reagent(Ambion)、RT-RPA核酸扩增试剂(基础型)(众测)、RT-RPA-nfo核酸扩增试剂(试纸条型)(众测)、HybriDetect试纸条(安普未来)、Prime ScriptTMOne Step RT-PCR Kit Ver.2(TaKaRa)、Nanodrop2000分光光度计(Thermo)、PCR仪(Biometra Tgradient)、水平电泳槽(Sub-cell GT wide Mini,BioRad)、凝胶成像仪(立德赛,成都)。
1.2 方法
1.2.1 样品总RNA制备 取50—100 mg待测样品的叶片,使用Trizol法提取植柑橘样品的总RNA,使用琼脂凝胶电泳检测RNA完整性,用分光光度计测定其浓度和纯度,将纯度良好的样品核酸保存于-80℃冰箱备用。
1.2.2 引物筛选以及探针设计 利用CLC软件对已报道的CYVCV基因组序列(GenBank登录号:MG878869、KX156740、KX156738、KP313240、KP313242)进行比对,寻找保守区段,按照RT-RPA引物设计原则:引物长度30—35 bp,无二级结构和单碱基重复序列,扩增片段大小100—200 bp[20],通过Primer Premier 5软件设计5对引物CY1-F/R、CY2-F/R、CY3-F/R、CY4-F/R、CY5-F/R(表1)。
用RT-RPA(基础型)试剂盒进行引物筛选试验。以感染CYVCV阳性样本的核酸为模板,清水为对照,进行RT-RPA反应,根据结果进行筛选。RT-RPA反应体系(总体积50 µL):在反应管中依次加入A buffer 41.5 µL,上下游引物(10 μmol·L-1)各2 µL,核酸2 µL,充分混匀,再加入B buffer 2.5 µL,充分振荡混匀,瞬时离心10 s,置于42℃金属浴上反应30 min。RPA 反应结束后,向反应物中加入等体积25﹕24﹕1溶液50 μL,轻轻涡旋,充分混匀,10 000 r/min离心2 min,吸取5 μL上清液进行琼脂糖凝胶电泳,观察电泳结果,选出最佳引物对。

表1 本试验所用引物
针对筛选出的最佳引物进行修饰以及设计对应探针。下游引物的5′端添加抗原标记Biotin进行修饰。探针设计原则:探针序列不与引物识别序列重叠,长度在46—52 nt,内部无二级结构和连续的重复碱基,探针的5′端用抗原标记物FAM基团修饰,将距5′端30 bp处的碱基替换为碱基类似物四氢呋喃(tetrahydrofuran,THF),探针3′端用聚合酶延伸阻断基团C3-spacer进行修饰。
1.2.3 反应产物测序鉴定 将选出引物的RT-RPA扩增产物经琼脂糖电泳分离后,切取目的条带,使用胶回收试剂盒纯化切胶产物,送往擎科公司测序,将测序结果与参考基因序列进行比对,确定反应扩增产物是否为目的片段。
1.2.4 RT-RPA体系建立及优化 利用RT-RPA-nfo(试纸条型)试剂盒,修饰后的优选引物以及探针优化反应体系。设置6个反应时间梯度(5、10、20、30、40、50 min)和8个反应梯度温度(37、38、39、40、41、42、43、44℃),根据电泳和试纸条检测结果,筛选出最佳反应时间和温度,建立RT-RPA检测体系。
RT-RPA-nfo(试纸条型)扩增反应体系(总体积50 µL):在反应管中依次加入A buffer 41.5 µL,修饰后的上下游引物(2 μmol·L-1)各2 µL,探针(2 μmol·L-1)0.2 µL,核酸2 µL,B buffer 2.5 µL。反应结束后,向反应物中加入等体积25﹕24﹕1溶液50 μL,混匀离心后,吸取5 μL上清液进行琼脂糖凝胶电泳,再吸取5 μL上清液于干净的离心管中,加入95 μL去离子水稀释20倍,将HybriDetect试纸条插入离心管中,约1 min后判读结果,根据电泳结果和试纸条检测结果,选出最佳反应时间和反应温度,最终确定最佳反应体系。
HybriDetect试纸条结果判读方法:当质控线(control line)呈蓝色或红色,且测试线(test line)呈红色,结果判定为阳性;试纸条质控线呈蓝色,检测区没有条带,结果判定为阴性;质控线和检测区均未出现条带,表示所使用试纸条失效、损坏或操作有误,检测结果无效。
1.2.5 特异性检测 提取仅携带CYVCV、CTV、CLBV、CTLV、CEVd、CPV、SDV、Las、的柑橘样品总RNA,采用优化后的方法分别对其进行检测,以携带CYVCV的阳性柑橘样品为正对照,以健康柑橘样品为负对照,根据电泳结果和试纸条检测结果,确定该检测方法的特异性。
1.2.6 灵敏度检测 将提取的阳性样品总RNA按10倍梯度稀释,以原液和10-1、10-2、10-3、10-4、10-5、10-6、10-7稀释液作为模板,平行使用RT-RPA和RT-PCR进行检测,比较两种方法的检测灵敏度。RT-RPA反应体系同1.2.4,RT-PCR检测使用一步法RT-PCR试剂盒参照陈洪明等[16]的方法及引物,检测反应体系(总体积10 μL)以及程序:Prime script 1 step enzyme 0.4 μL,2×1 step buffer 5 μL,上下游引物(10 μmol·L-1)各0.2 μL,D&R-nase free water 3.2 μL,模板1 μL;50℃反转录30 min,94℃预变性3 min,94℃变性30 s,58℃复性30 s,72℃延伸30 s,循环35次,72℃后延伸5 min,4℃保存。
1.2.7 适用性检测 以西南大学柑桔研究所内随机采摘的9个柑橘品种共45株样品为检测对象,应用所建立的RT-RPA检测方法进行CYVCV的检测,平行进行CYVCV的RT-PCR检测,携带CYVCV的阳性柑橘样品为正对照,健康柑橘样品为负对照。比较两种方法的检测结果,评价RT-RPA检测方法的适用性。
2 结果
2.1 RT-RPA检测体系建立及优化
2.1.1 引物筛选 以感染CYVCV阳性样本的核酸为模板,分别使用设计的5对RT-RPA引物进行扩增。结果显示,引物CY1-F/R、CY3-F/R、CY5-F/R均能扩增出单一明亮的条带(图1-A)。为进一步筛选最佳检测引物,取感染了CYVCV阳性植株不同部位(嫩叶、嫩皮、老叶、老皮)的样品抽提总核酸,分别用3对引物进行检测,结果也均扩增出单一的目的条带。其中CY1-F/R条带更明亮更清晰(图1-B),说明CY1-F/R扩增效果更好。对CY1-R的5′端添加抗原标记Biotin进行修饰,设计对应探针CY1-Probe(47 bp)进行后续试验(表1)。
2.1.2 扩增产物鉴定 将利用引物CY1-F/R扩增出的产物测序,测序结果与参考基因序列的比对结果显示,该产物长度为177 bp,与目标CYVCV基因片段长度一致,与参考序列的相似率为100.00%(图2),说明RT-RPA扩增产物为目标CYVCV片段。
2.1.3 反应体系优化 检测结果表明,在所设置反应时间梯度(5、10、20、30、40、50 min)下,随反应时间增加,电泳条带亮度逐渐增加,在20、30、40和50 min时,条带亮度无明显差异(图3-A),但试纸条检测时反应时间为20 min时,测试区条带不明显,当反应时间达到30 min后,试纸条测试区条带变化无明显差异(图3-B),故选择反应时间为30 min。在所设置反应梯度温度(37、38、39、40、41、42、43和44℃)下,均能扩增出单一条带,39℃时电泳条带最明亮(图4-A、4-B),试纸条测试区均出现红色条带,但是纸条无明显变化,故选择39℃为最适反应温度。

M:标准分子量,下同standard molecular weight. The same as below。A:1:阳性样品positive sample;2:ddH2O。B:1:嫩叶young leaf;2:嫩皮young bark;3:老叶old leaf;4:老皮old bark

图2 RT-RPA扩增产物的序列比对
2.2 RT-RPA检测体系评价
2.2.1 特异性检测 利用所建立的柑橘黄化脉明病毒RT-RPA检测体系分别对仅感染了CYVCV、CTV、CEVd、CTLV、CLBV、CPV、SDV、和las的样品进行检测。电泳和试纸条结果均显示,只有感染了CYVCV的样品检测呈阳性,而感染其他8种柑橘病害的样品均检测呈阴性(图5-A、5-B)。表明所建立的CYVCV RT-RPA检测体系与其他柑橘病原无交叉反应,特异性强。
2.2.2 灵敏度检测 将CYVCV检测为阳性的样品总核酸进行10倍梯度浓度稀释,原液及其梯度稀释液分别进行RT-RPA和RT-PCR反应。电泳检测结果显示,当总核酸稀释至10-4时,两种方法能检测出CYVCV(图6-A、6-B),当总核酸稀释至10-5时RT-RPA和RT-PCR电泳检测仍有微弱条带,但是RT-RPA试纸条检测结果显示测试区无条带(图6-C),表明两种检测方法的检测灵敏度基本相当。

图3 RT-RPA检测柑橘黄化脉明病毒反应时间筛选

图4 RT-RPA检测柑橘黄化脉明病毒反应温度筛选
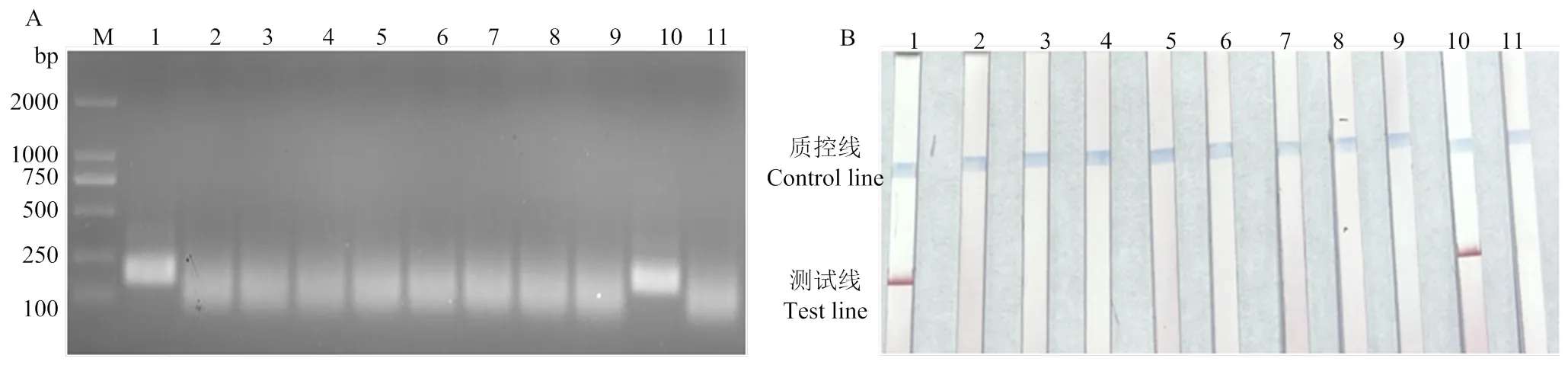
1:柑橘黄化脉明病毒CYVCV;2:柑橘衰退病毒CTV;3:柑橘叶斑驳病毒CLBV;4:柑橘碎叶病毒CTLV;5:柑橘裂皮病类病毒CEVd;6:柑橘鳞皮病毒CPV;7:温州蜜柑萎缩病毒SDV;8:柑橘溃疡病菌Xcc;9:柑橘黄龙病菌Clas;10:正对照positive control;11:负对照negative control

A:RT-PCR;B、C:RT-RPA;100—10-7:稀释倍数Degree of dilution
2.2.3 适用性检测 田间随机采集9个不同品种共45个柑橘样品,分别进行RT-PCR和RT-RPA检测。RT-PCR和所建立的RT-RPA法检均测出37个阳性样品(图7),检出率为82.2%(表2),两种检测方法检测结果相同,表明该检测方法效果良好,检测效果稳定可靠。
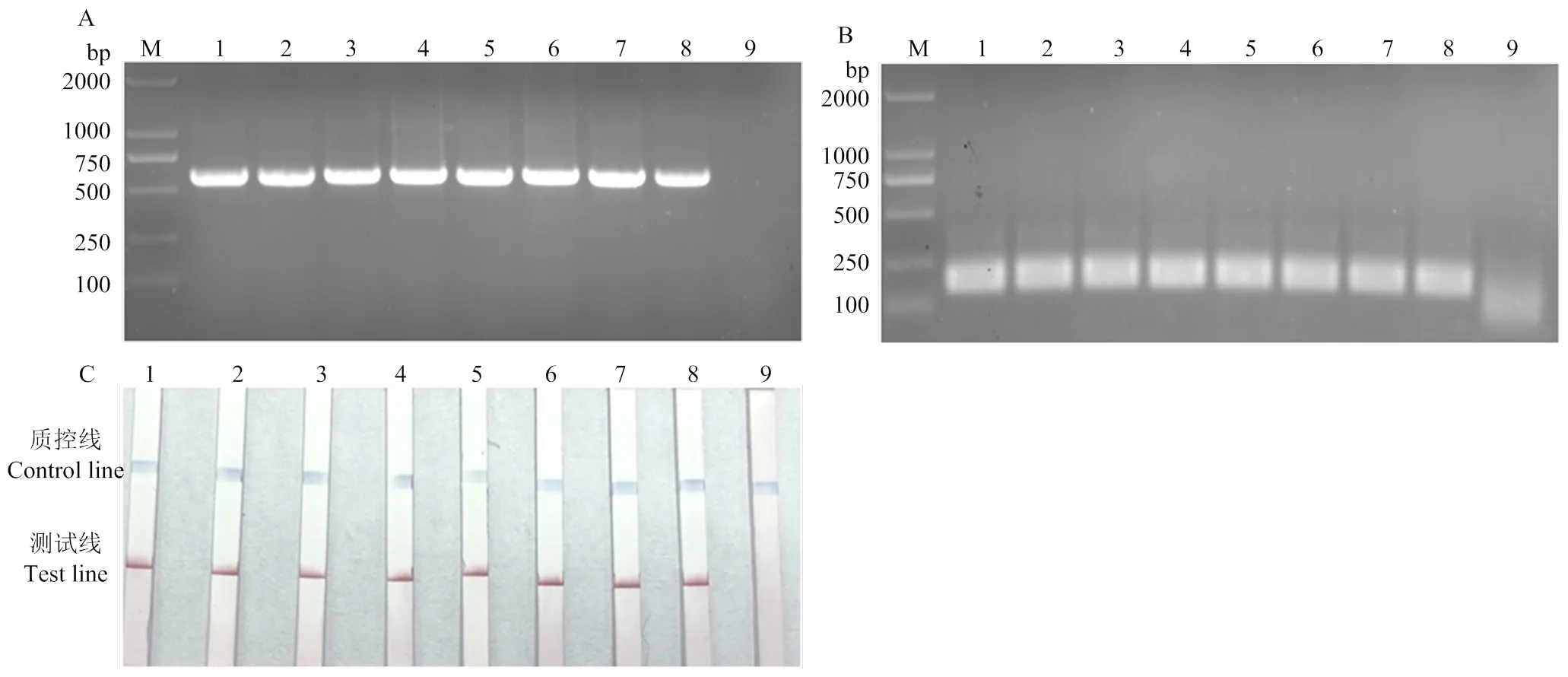
A:RT-PCR;B、C:RT-RPA;1、2:沃柑orah;3—7:爱媛38 ehime 38 hybrid citrus;8:正对照positive control;9:负对照negative control
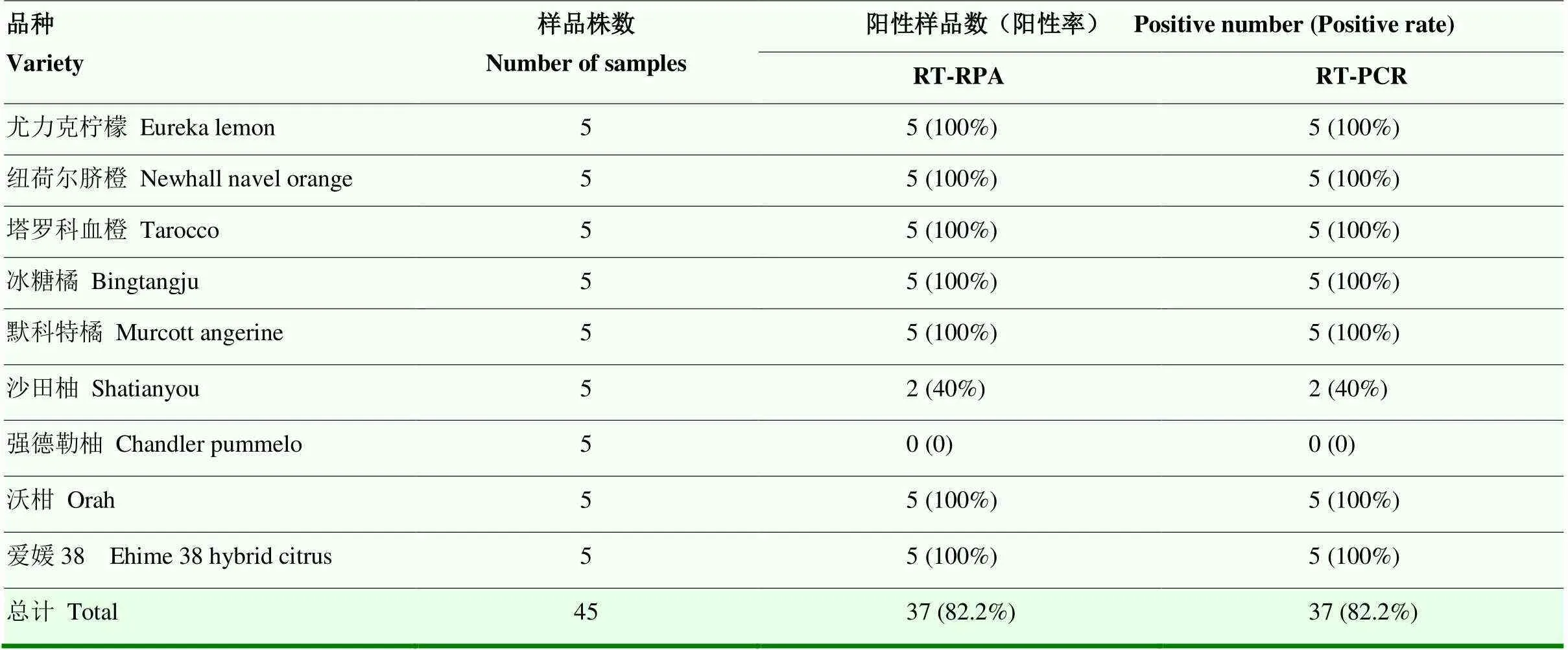
表2 不同柑橘品种的RT-RPA 及RT-PCR 检测
3 讨论
CYVCV是近年来危害柑橘尤其是柠檬的重要病毒之一。随着柑橘产业的大发展,地域间种质资源交流、苗木调运频繁,我国普遍存在其高效传播虫媒,使得该病毒的流行呈加速扩散态势[1,8]。因此,建立一种快速、准确的CYVCV检测方法对柑橘黄脉病的防控具有重要意义。
本研究将RT-RPA技术应用于柑橘黄化脉明病毒的检测,结合侧向流层析试纸条,建立了CYVCV的RT-RPA检测体系。该体系与柑橘衰退病毒、柑橘裂皮类病毒、柑橘碎叶病毒、柑橘黄龙病菌、温州蜜柑萎缩病毒等常见柑橘病原均无交叉反应,特异性强。检测灵敏度与RT-PCR灵敏度一致,并且对田间样品检测的结果稳定可靠,适用性良好。刘科宏等[19]建立的实时环介导等温核酸扩增(RT-LAMP)法和陈洪明等[17]建立的实时荧光RT-PCR法检测CYVCV的灵敏度分别是RT-PCR方法[16]的10倍和100倍,本试验所建立的RT-RPA检测方法灵敏度与RT-PCR相当,虽然灵敏度低于RT-LAMP和实时荧光RT-PCR,但不需要实时荧光PCR仪,也不需要设计RT-LAMP复杂的引物,反应温度更低,操作也更为简洁,反应时长缩短了约一半。同时通过在反应体系中加入特异性探针,增强了RT-RPA检测的特异性。本试验将RT- RPA技术与侧向流层析技术相结合,检测结果可使用试纸条呈现,更加直观、简明。同时,RT-RPA所需反应温度更低,甚至仅靠人体体温即能完成反应[30],这也促进了该技术在基层的推广使用。因而,本试验建立的RT-RPA检测方法与上述两种检测方法相比具有一定优势。
RT-RPA技术作为一种新型核酸扩增技术,具有特异性强、灵敏度高、操作快速便捷等优点,已逐渐应用于众多动植物病原的检测。不过,目前该技术方法所用试剂的成本较高,相信随着技术的不断成熟,应用范围的不断扩大,该技术的检测成本将不断降低,应用前景将更为广阔。柑橘病毒病害种类繁多,将RT-RPA技术应用于更多柑橘病害的检测,从而为基层人员提供新的快速简便又可视化的检测手段,对于相应病害的防控具有积极意义。
4 结论
建立了柑橘黄化脉明病毒的RT-RPA快速检测方法。该方法灵敏度高、特异性强、反应快速、操作简单、适用性好,可肉眼直接观察结果,无需大型精密仪器,可满足基层检测需求,具有较大的应用潜力。
[1] ZHANG Y H, LIU C H, WANG Q, WANG Y L, ZHOU C Y, ZHOU Y. Identification ofas a vector of citrus yellow vein clearing virusin ChinaPlant disease, 2019, 103(1): 65-68.
[2] GRIMALDI V, CATARA A. Association of a filamentous virus with yellow vein clearing of lemon//13th International Organization of Citrus Virologists Conference, 1996: 343-345.
[3] AHLAWAT Y S. Viruses, greening bacterium and viroids associated with citrus (Citrus species) decline in IndiaIndian Journal of Agricultural Science, 1997, 67: 51-57.
[4] CATARA A, AZZARO A, MUGHAL S M, KHAN D A. Virus, viroid and prokaryotic diseases of citrus in Pakistan//Proceedings of Sixth Conference. Tel Aviv, Israel, 1988: 957-962.
[5] CATARA A, AZZARO A, DAVINO M, POLIZZI G. Yellow vein clearing of lemon in Pakistan//12th International Organization of Citrus Virologists Conference, 1992: 364-367.
[6] ALSHAMI A A A, AHLAWAT Y S, PANT R P. A hitherto unreported yellow vein clearing disease of citrus in India and its viral etiology. Indian Phytopathology, 2003, 56(4): 422-427.
[7] CHEN H M, LI Z A, WANG X F, ZHOU Y, TANG K Z, ZHOU C Y, ZHAO X Y, YUE J Q. First report of citrus yellow vein clearing virus on lemon in Yunnan, ChinaPlant Disease, 2014, 98(12): 1747.
[8] ZHOU Y, CHEN H M, CAO M J, WANG X F, JIN X, LIU K H, ZHOU C Y. Occurrence, distribution, and molecular characterization of citrus yellow vein clearing virus in ChinaPlant disease, 2017, 101(1): 137-143.
[9] LOCONSOLE G, ÖNELGE N, POTERE O, GIAMPETRUZZI A, BOZAN O, SATAR S, DE STRADIS A, SAVINO V, YOKOMI R K, SAPONARI M. Identification and characterization of citrus yellow vein clearing virus, a putative new member of thegenusPhytopathology, 2012, 102(12): 1168-1175.
[10] SONG Z, Kurth E G, Peremyslov V V, ZHOU C Y, Dolja V V. Molecular characterization of a citrus yellow vein clearing virusstrain from ChinaArchives of virology, 2015, 160(7): 1811-1813.
[11] ÖNELGE N, SATAR S, ELIBUYUK Ö, BOZAN O, KAMBEROOLU M. Transmission studies oncitrus yellow vein clearing virus//18th International Organization of Citrus Virologists Conference, 2011.
[12] ZHOU Y, MA D D, CHEN H M, WANG X F, HE S G, ZHOU C Y. A rapid and efficient purification of citrus yellow vein clearing virus by sucrose cushion ultracentrifugationJournal of Plant Pathology, 2016, 98: 159-161.
[13] ZHOU Y, CHEN H M, WANG X F, LI Z A, TANG M, ZHOU C Y. Lack of evidence for seed transmission of citrus yellow vein clearing virus despite its frequent detection in seed tissuesJournal of Plant Pathology, 2015, 97: DOI:10.4454/JPP.V97I3.023.
[14] 宾羽, 宋震, 李中安, 周常勇. 柑橘黄化脉明病毒DTBIA检测方法的建立园艺学报, 2015, 42(9): 1843-1850.
BIN Y, SONG Z, LI Z A, ZHOU C Y. Direct tissue blot immunoassay for detection of citrus yellow vein clearing virusActa Horticulturae Sinica, 2015, 42(9): 1843-1850. (in Chinese)
[15] LIU Z, SUNZHU Y J, ZHOU X P, HONG J, WU J X. Monoclonal antibody-based serological detection of citrus yellow vein clearing virus in citrus grovesJournal of integrative agriculture, 2017, 16(4): 884-891.
[16] 陈洪明, 王雪峰, 周彦, 周常勇, 郭俊, 李中安. 尤力克柠檬上一种新病害的生物学特性及 RT-PCR 检测植物保护学报, 2015, 42(4): 557-563.
CHEN H M, WANG X F, ZHOU Y, ZHOU C Y, GUO J, LI Z A. Biological characterization and RT-PCR detection of a new disease of Eureka lemon. Journal of Plant Protection, 2015, 42(4): 557-563. (in Chinese)
[17] 陈洪明, 周彦, 王雪峰, 周常勇, 杨秀燕, 李中安. 应用实时荧光RT-PCR检测柑橘黄化脉明病毒园艺学报, 2016, 43(1): 168-174.
CHEN H M, ZHOU Y, WANG X F, ZHOU C Y, YANG X Y, LI Z A. Detection of citrus yellow vein clearing virus based on a real-time RT-PCR approach. Acta Horticulturae Sinica, 2016, 43(1): 168-174. (in Chinese)
[18] 周彦, 陈洪明, 王雪峰, 李中安, 王亮, 周常勇. 柑橘黄化脉明病毒巢式RT-PCR检测方法的建立及应用植物保护学报, 2016, 43(2): 255-259.
ZHOU Y, CHEN H M, WANG X F, LI Z A, WANG L, ZHOU C Y. development and application of nested RT-PCR assay for detection of citrus yellow vein clearing virus. Journal of Plant Protection, 2016, 43(2): 255-259. (in Chinese)
[19] 刘科宏, 陈洪明, 周彦, 李中安. 柑橘黄化脉明病毒RT-LAMP检测方法的建立园艺学报, 2015, 42(5): 997-1002.
LIU K H, CHEN H M, ZHOU Y, LI Z A. Establishment of RT-LAMP assay for detection of citrus yellow vein clearing virus. Acta Horticulturae Sinica, 2015, 42(5): 997-1002. (in Chinese)
[20] PIEPENBURG O, WILLIAMS C H, STEMPLE D L, ARMES N A. DNA detection using recombination proteinsPLoS Biology, 2006, 4(7): e204.
[21] KARAMI A, GILL P, MOTAMEDI M H K, SAGHAFINIA M. A review of the current isothermal amplification techniques: applications, advantages and disadvantagesJournal of Global Infectious Diseases, 2011, 3(3): 293.
[22] WANG R, ZHANG F, WANG L, QIAN W J, QIAN C, WU J, YING Y B. Instant, visual, and instrument-free method for on-site screening of GTS 40-3-2 soybean based on body-heat triggered recombinase polymerase amplificationAnalytical chemistry, 2017, 89(8): 4413-4418.
[23] BABU B, WASHBURN B K, MILLER S H, PODUCH K, SARIGUL T, KNOX G W, OCHOA-CORONA F M, PARET M L. A rapid assay for detection of rose rosette virus using reverse transcription- recombinase polymerase amplification using multiple gene targetsJournal of virological methods, 2017, 240: 78-84.
[24] JIAO Y B, XU C T, LI J L, GU Y, XIA C, XIE Q, XIE Y B, AN M N, XIA Z H, WU Y H. Characterization and a RT-RPA assay for rapid detection of chilli veinal mottle virus (ChiVMV) in tobaccoVirology journal, 2020, 17(1): 33.
[25] KAPOOR R, SRIVASTAVA N, KUMAR S, SARITHA R K, SHARMA S K, JAIN R K, BARANWAL V K. Development of a recombinase polymerase amplification assay for the diagnosis ofbanana bunchy top virus in different banana cultivarsArchives of virology, 2017, 162(9): 2791-2796.
[26] MIAO F M, ZHANG J Y, LI N, CHEN T, WANG L D, ZHANG F, MI L J, ZHANG J X, WANG S C, WANG Y, zhou x t, zhang y y, li m, zhang s f, hu r l. Rapid and sensitive recombinase polymerase amplification combined with lateral flow strip for detecting African swine fever virusFrontiers in microbiology, 2019, 10: 1004.
[27] SHALABY M A, EL-DEEB A N, EL-THOLOTH M, HOFFMANN D, CZERNY C P, HUFERT F T, WEIDMANN M, ABD EL WAHED A. Recombinase polymerase amplification assay for rapid detection of lumpy skin disease virusBMC veterinary research, 2016, 12(1): 244.
[28] ZENG R, LUO J Y, GAO S G, XU L H, SONG Z W, DAI F M. Rapid detection of cucumber green mottle mosaic virus by reverse transcription recombinase polymerase amplificationMolecular and cellular probes, 2019, 43: 84-85.
[29] QIAN W J, LU Y, MENG Y Q, YE Z Z, WANG L, WANG R, ZHENG Q Q, WU H, WU J. Field detection of citrus Huanglongbing associated with ‘Liberibacter asiaticus’ by recombinese polymerase amplification within 15 minJournal of agricultural and food chemistry, 2018, 66(22): 5473-5480.
[30] CRANNELL Z A, ROHRMAN B, RICHARDS-KORTUM R. Equipment-free incubation of recombinase polymerase amplification reactions using body heatPloS one, 2014, 9(11): e112146.
Establishment of RT-RPA for citrus yellow vein clearing virus (CYVCV) detection
MA ZhiMin, XU JianJian, DUAN Yu, WANG ChunQing, SU Yue, ZHANG Qi, BIN Yu, ZHOU ChangYong, SONG Zhen
Citrus Research Institute, Southwest University/National Citrus Engineering Research Center, Chongqing 400712
【】The objective of this study is to establish a fast, simple, accurate and visualized with naked eyes new detection method for citrus yellow vein clearing virus(CYVCV) using reverse transcription-recombinase polymerase amplification (RT-RPA) combined with lateral flow dipstick (LFD).【】Five pairs of primers were designed according to the conservative sequence of the coat protein gene of CYVCV. By detecting different samples, the pair of primers with the best amplification efficiency and specificity was selected. The selected primers were modified and its corresponding specific probe was designed. According setting 6 reaction gradient times (5, 10, 20, 30, 40 and 50 min) and 8 reaction gradient temperatures (37, 38, 39, 40, 41, 42, 43 and 44℃), the RT-RPA system for CYVCV detection was optimized. The specificity of the established RT-RPA was evaluated by detecting the samples infected with CYVCV, citrus leaf blotch virus (CLBV), citrus tristeza virus (CTV), citrus tatter leaf virus (CTLV), citrus exocortis viroid (CEVd), citrus psorosis virus (CPV), satsuma dwarf virus (SDV),Liberibacter asiaticus (Las) andsubsp.(), respectively. The citrus total RNA samples infected with CYVCV was diluted by 10 times. The original RNA solution and 10-1, 10-2, 10-3, 10-4, 10-5, 10-6, 10-7dilutions were used as templates for testing the sensitivity of RT-RPA, and the sensitivity was compared with RT-PCR. Leaves of different citrus varieties were randomly collected from the field. RT-RPA and RT-PCR were used at the same time to test the applicability of the established RT-RPA detection method.【】A RT-RPA detection system for CYVCV was established, with primer pairs CY1-F/R and corresponding probe CY1 (47 bp). It could specifically amplify the target fragment of CYVCV with a size of 177 bp. The best reaction conditions were 39℃, 30 min. The result could be judged by the LFD test strip directly. In the specific test, only samples infected with CYVCV were positive, and the rest were negative. In the sensitivity detection, 10-4dilution was the lowest detection sensitivity of RT-RPA and RT-PCR. The sensitivity of the two methods was equivalent. Among the 45 field citrus samples taken randomly, 37 samples were positive by RT-PCR and RT-RPA, and the positive rate was both 82.2%, indicating that the RT-RPA method established in this study was stable and reliable.【】A RT-RPA detection method for CYVCV is established. The method is convenient, rapid, and visualized. It can be applied to on-site rapid detection for the labs with insufficient basic conditions or plant protection and quarantine station.
citrus; citrus yellow vein clearing virus (CYVCV); RT-RPA; lateral flow dipstick test strip; rapid detection

10.3864/j.issn.0578-1752.2021.15.009
2020-11-12;
2020-12-28
国家自然科学基金(31972237)、国家重点研发计划(2018YFD02021500)、国家现代农业柑橘产业技术体系(CARS-26-05B)
马志敏,E-mail:mazhimin02@163.com。通信作者周常勇,E-mail:zhoucy@cric.cn。通信作者宋震,E-mail:songzhen@cric.cn
(责任编辑 岳梅)

