基于生物信息学筛选子宫内膜癌预后相关的lncRNAs分子标签
胡云双,张颖,曾海平
温州市中西医结合医院 检验科,浙江 温州 325000
子宫内膜癌是女性生殖系统中最为常见的癌症之一,目前位列发达国家女性恶性肿瘤中的第四位,我国每年也约有2万人死于该病[1-2]。虽然子宫内膜癌的手术、化疗及免疫治疗都取得了一定的进展[3],但患者的病死率仍居高不下[4]。因此,从分 子层面更深入地探索其发病机制[5],研究影响子宫内膜癌预后的分子标签来评估与预测患者生存率,具有临床实际意义[6]。
长链非编码RNA(long non-coding RNA,lncRNA) 是一类长度在200~100 000 nt之间非蛋白编码RNA分子,常被用于肿瘤诊断或预后评估,是一类极具潜力的新一代标志物[7]。研究表明,lnc RNA作为重要的调控因子,参与肿瘤细胞的增殖、分化、转 移[8],但目前关于lncRNAs在子宫内膜癌中的生物学功能和分子机制仍鲜见报道[9]。本研究提取TCGA数据库中子宫内膜癌的lnc RNA表达数据及临床相关数据,通过差异表达分析和单因素LASSO Cox回归,寻找与子宫内膜癌预后相关的IncRNA,采用多因素Cox回归模型构建预测子宫内膜癌预后相关的lncRNA分子标签,为子宫内膜癌的预后评估提供进一步支持。
1 资料和方法
1.1 子宫内膜癌lncRNA表达数据及临床资料的收集与处理 从TCGA数据库(https://p ortal.gdc.cancer.gov/)中下载子宫内膜癌和癌旁组织的RNAseq level 3转录组数据及临床相关资料,数据集截止日期为2020年2月。样本筛选纳入标准包括:①经过病理证实为原发性子宫内膜癌,并且术前未经任何放化疗;②预后信息完整无缺失,预后随访时间大于30 d。除去临床信息缺失的个体,共计523个子宫内膜癌和23个癌旁组织纳入分析。
1.2 lncRNA差异表达分析 采用R语言的“edgeR”包[10]对子宫内膜癌和癌旁组织的lncRNA进行表达差异筛选分析,筛选标准如下:log2|差异倍数(FC)| ≥1且假阳性发现率(false discovery rate,FDR)<0.05。用R语言“ggplot2”包绘制火山图,采用R语言“ComplexHeatmap”包绘制热图,显示其在癌组织和癌旁组织的差异表达情况。
1.3 LASSO Cox预后模型构建与验证 本研究采用随机数生成法将523例子宫内膜癌患者分为训练集(training set,n=262)和测试集(testing set,n=261)两个队列。样本基本特征:训练集中位年龄为63(31~90)岁,中位身高为161(142~183)cm,肿瘤分期I期160例,II期26例,III期62例,IV期14例;测试集中位年龄为64(34~90)岁,中位身高为161.5(130~178)cm,肿瘤分期I期167例,II期27例,III期54例,IV期13例。训练集用于学习特征的选择和模型的估计,测试集作为内部验证队列来评估模型的预测性能。
使用R语言的“Survival”包进行单因素Cox回归分析,获得与预后相关的lnc RNA分子。对与预后显著相关的lncRNA分子(P<0.001)进行LASSO Cox回归分析筛选变量,构建lncRNAs分子标签[11]。lncRNAs分子标签=Σ(回归系数×基因表达量),以风险值的中位数为阈值,把子宫内膜癌患者分成高、低两个风险组别。绘制Kaplan-Meier生存曲线,采用Log-rank法检验比较两组患者的总生存率差异,P<0.05为差异有统计学意义。此外,为评估lncRNAs分子标签模型的预测性能,我们在测试集中进行验证,考察相关的lnc RNAs用于预测子宫内膜癌预后的可能性。
1.4 预后模型ROC曲线评价 采用R包“Survival”和“timeROC”绘制1、3、5年总生存率的ROC曲线,计算曲线下的面积(area under the curve,AUC),评估预后模型在不同时间终点的预测性能。
1.5 基因集富集分析(gene set enrichment analysis,GSEA) 从MsigDBv6.2数据库(https://www.gsea-msigdb.org/gsea/msigdb/index.jsp)中下载“C2:经典通路”基因集,采用默认基因富集统计分析方法[12],进行GSEA,执行1 000次置换检验,P<0.05认为是显著富集。
2 结果
2.1 与预后相关的子宫内膜癌差异lncRNA的筛选 基于FDR<0.05且|log2FC|>2的筛选标准共得到2 277个差异表达的lncRNAs(见图1A),包括1 303个表达上调的lnc RNAs和974个表达下调的 lncRNAs(见图1B)。
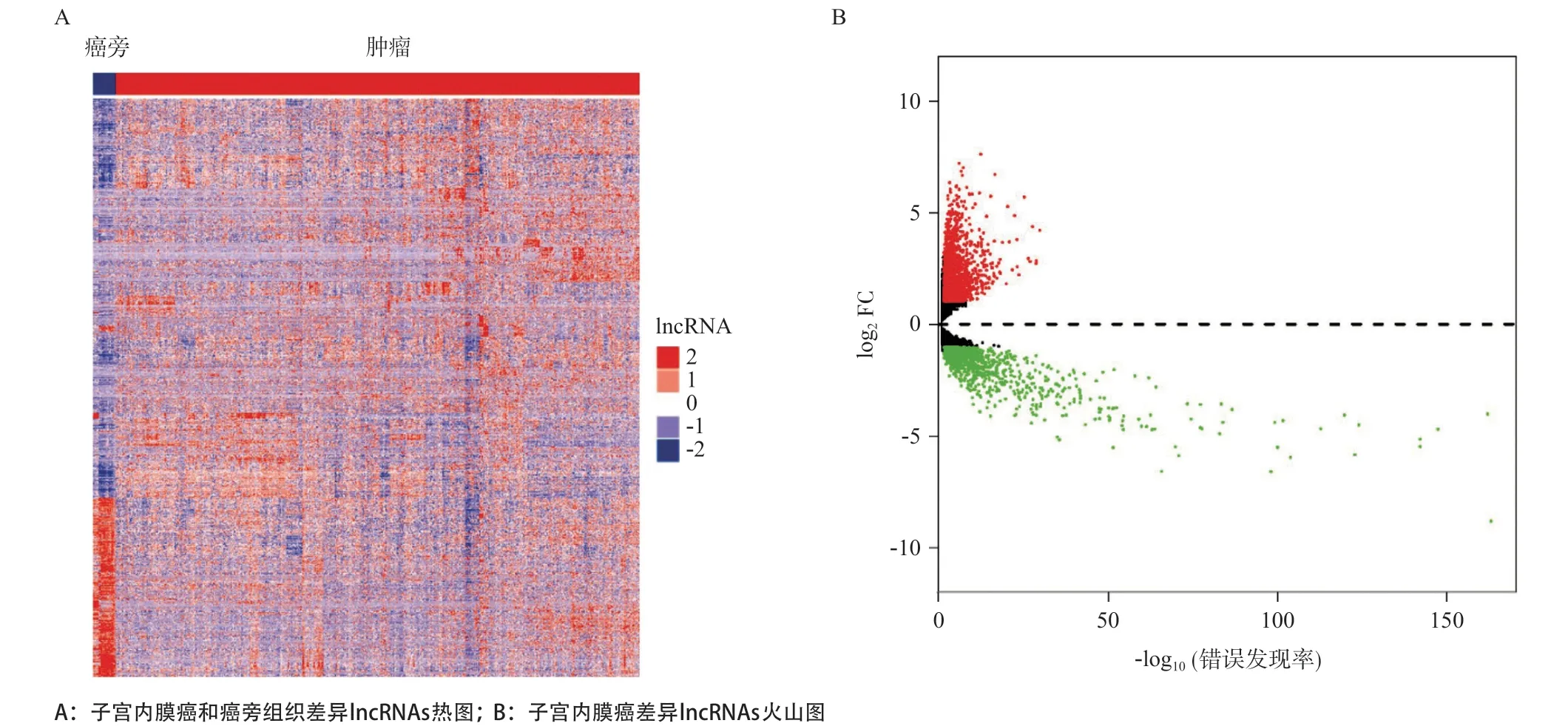
图1 子宫内膜癌差异lncRNAs的筛选
2.2 子宫内膜癌预后模型的构建与评价 单因素Cox回归初步筛选出18个和子宫内膜癌预后相关的差异lncRNAs(P<0.001)(见表1),通过LASSO回归分析(见图2A、2B)降维确定了13个关键的lncRNAs分子。采用多因素Cox回归模型进行lncRNAs的预后风险评分,Risk Score=(0.13118×AC005381.1)+(0.09103×AC009237.15)+(0.25714×AC011294.1)+ (0.21220×AC017074.1)+(0.11446×AC073842.2)+ (0.04660×AC108025.2)+(0.28335×AL121906.2)(-0.12554×AP000808.1)+(-0.19050×LINC00475)+ (0.14988×LINC01116)+(0.17987×LINC01250)+(0.21181×NCAM1-AS1)+(-0.30462×UNQ6494),以风险得分的中位值(0.92)为阈值,将训练集中的262例患者分为高风险(>0.92,n=131)组和低风险(≤0.92,n=131)组。结果发现高风险组患者平均生存时间较短,具有较多的死亡人数,其对应热图也显示出这13个lncRNAs分子的表达量在2个风险组别中差异有统计学意义(见图3A)。Kaplan-Meier生存曲线(见图3B)显示低风险组患者中位生存时间(3.03年)长于高风险组(2.59年),总生存率也高于高风险组(P<0.001)。ROC进行预测性能评估,发现子宫内膜癌患者1、3、5年总生存率AUC分别为0.771、0.868、0.864(见图3C)。
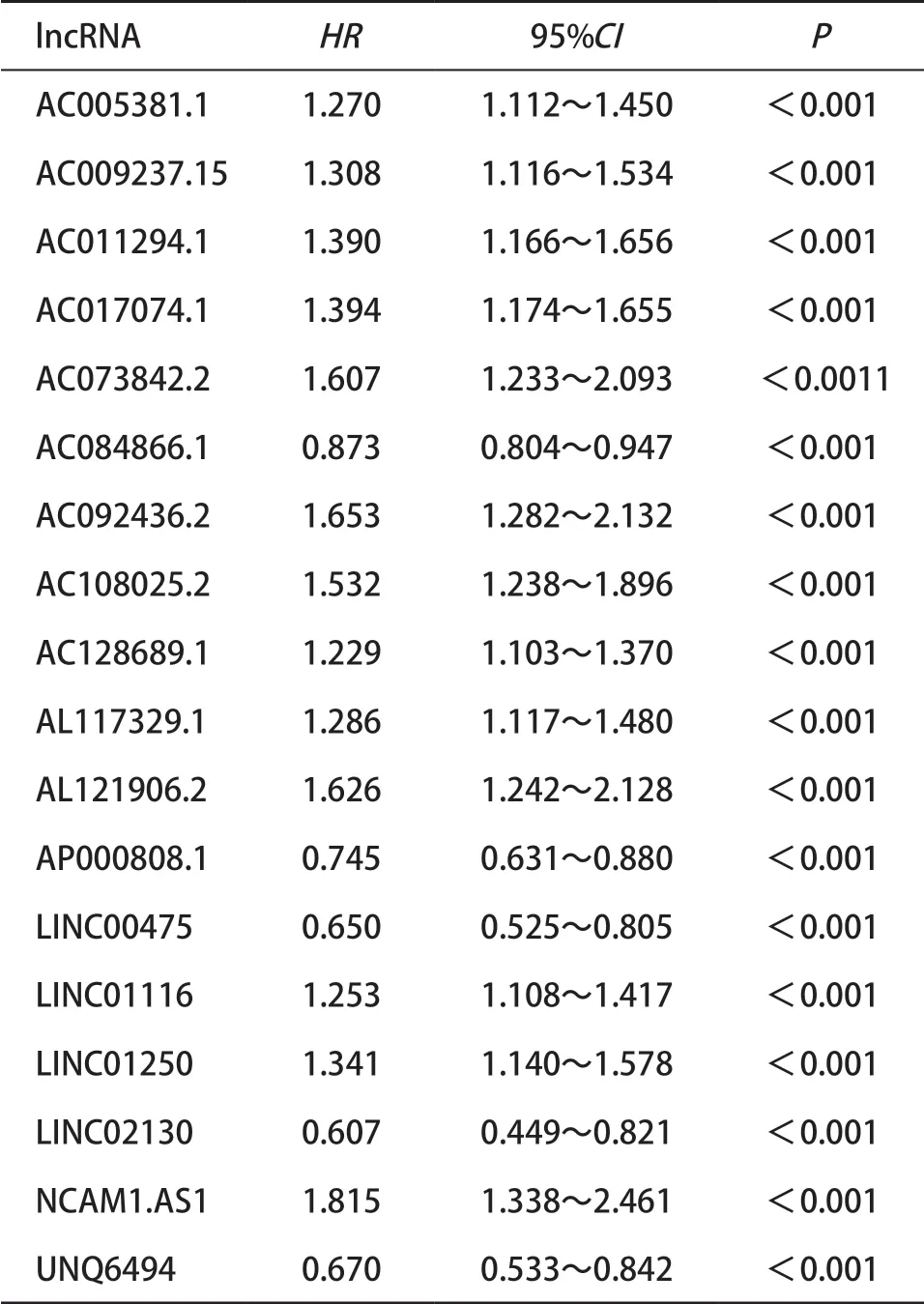
表1 单因素Cox回归分析初步筛选和子宫内膜癌预后相关的lncRNA
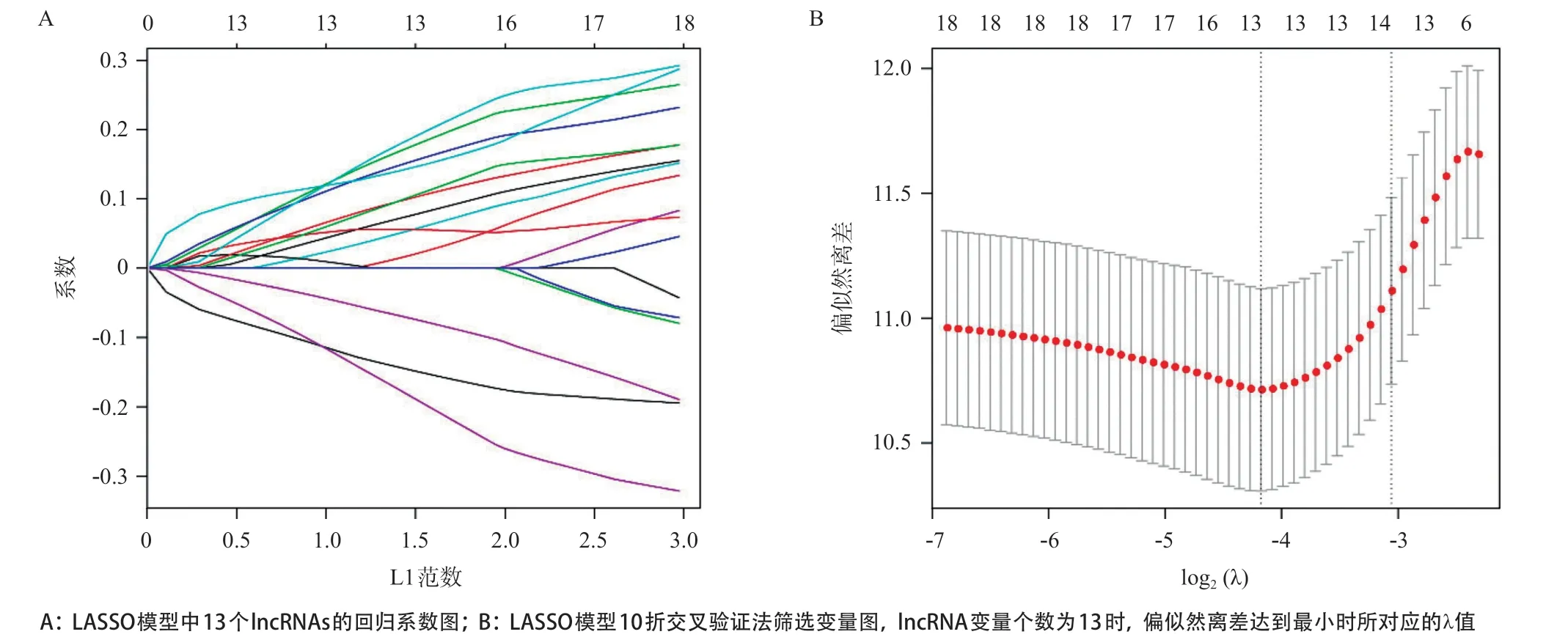
图2 LASSO回归分析
为评估预后模型的预测能力,以测试集中的风险评分中位值(0.98)为阈值,将测试集的患者分为高、低风险组。如图4A所示,与训练集一致,测试集中高风险组患者(130例)与低风险组患者(131例)相比生存时间更短,死亡人数更多,热图也显示这13个lncRNAs的表达量在高、低风险组中差异有统计学意义。Kaplan-Meier生存曲线(见图4B)表明,高风险组患者的总生存率较低(P<0.001)。ROC分析显示,测试集中子宫内膜癌患者1、3、5年总生存率AUC分别为0.751、0.757、0.730(见图4C)。
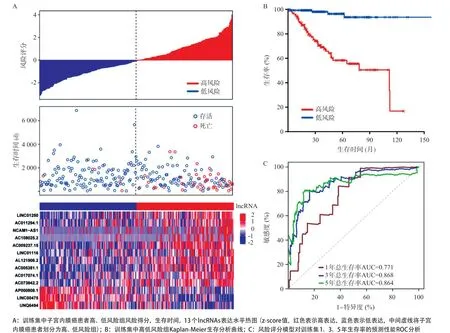
图3 训练集lncRNAs热图、Kaplan-Meier图和ROC曲线图
2.3 筛选的lncRNAs分子标签模型与临特征指标的联合分析 本研究收集了子宫内膜癌患者的临床信息,在训练集中进行单因素Cox回归分析,发现肿瘤临床分期(HR=1.98,P<0.001)、肿瘤组织学分级(HR=2.89,P<0.001)与预后相关。构建临床指标多因素Cox回归预测模型Risk Score=(0.5231×肿瘤临床分期)+(0.7608×肿瘤组织学分级),采用ROC预测模型效能,如图5A所示,在训练集中,患者1、3、5年总生存率AUC分别为0.678、0.767、0.801。进一步构建lnc RNAs分子标签联合临床指标Cox回归模型,ROC分析显示,患者1、3、5年总生存率AUC分别为0.755、0.874、0.877(见图5B)。在测试集中,ROC分析临床指标和lncRNAs分子标签联合临床指标Cox回归模型的预测效能,患者1、3、5年总生存 率AUC分别为0.828、0.720、0.719和0.802、0.767、0.772(见图5C-D)。综合来看,无论在训练集还是测试集中,lnc RNAs分子标签联合临床指标模型预测效能最好。
2.4 子宫内膜癌的GSEA GSEA结果表明(见图6),多条致癌通路在高风险组中富集,包括小分子泛素样调节蛋白化通路(SUMO pathway)、PTK6调控的细胞周期(PTK6 regulates cell cycle)、微小染色体维持蛋白通路(MCM pathway),表明它们参与了子宫内膜癌的发生发展过程。相反,免疫相关和代谢通路在低风险组富集,如苯丙氨酸和酪氨酸代谢(p henylalanine and tyrosine metabolism)、TCRA通路(TCRA p athway)、阿尔法亚麻酸代谢 (Alpha linolenic acid metabolism)等。
3 讨论
子宫内膜癌是女性生殖系统肿瘤中一种最常见的类型[13],随着对它的认识不断深入,子宫内膜癌的诊疗和预后评估也取得了一定成效,但目前尚缺乏突破性进展,子宫内膜癌的个体化治疗仍然面临巨大的挑战。
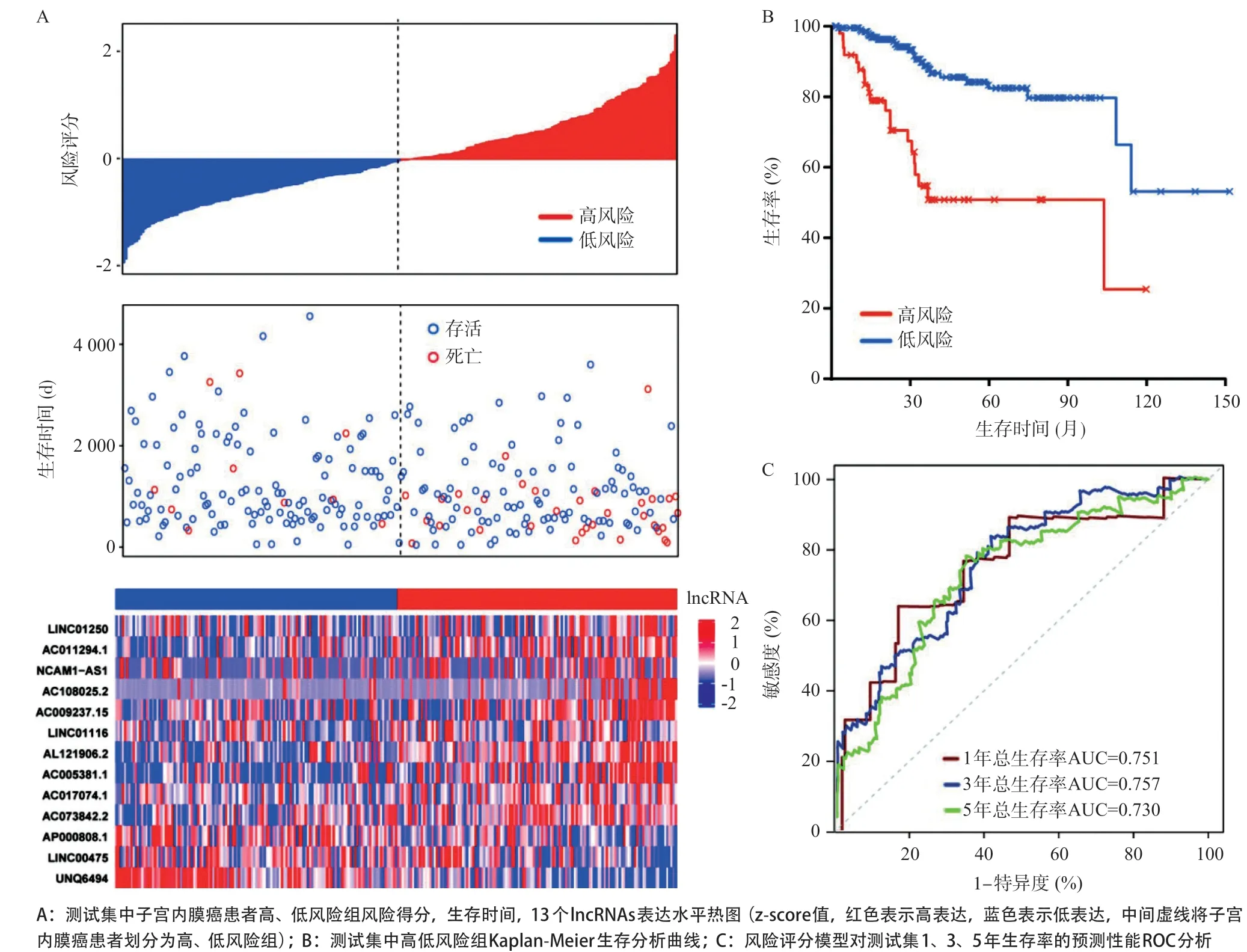
图4 测试集lncRNAs热图、Kaplan-Meier图和ROC曲线图
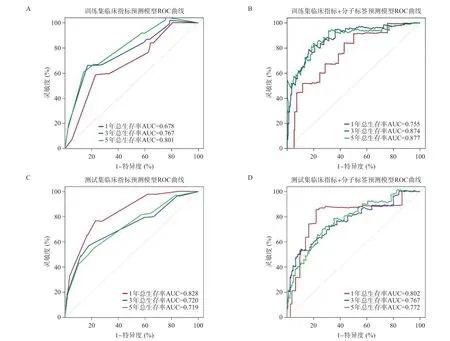
图5 ROC曲线对临床指标预测模型及lncRNAs分子标签联合临床指标模型的预测性能评价
本研究采用的LASSO Cox回归算法与传统的Cox回归相比较,可以很好地解决高维特征数据的预后分析,有效避免多个变量间的共线性问题。此外,本研究构建的lncRNA分子标签的风险评分模型综合了13个lncRNA的表达信息,显著提高子宫内膜癌预后评估能力。相关研究也表明,lncRNA分子标签模型与单个lncRNA相比可以更好地评估肿瘤的预后,更具有临床价值[14]。时间依赖的ROC曲线分析对1、3、5年的生存时间的预测也体现了该模型较强的鲁棒性。而且预测的13个lncRNA中有部分已被报道与肿瘤发生密切相关,比如LINC01116分子与肿瘤的增殖以及凋亡相关,LINC01116可以靶向miR-520a-3p,影响IL6R通过JAK-STAT信号通路促进骨肉瘤细胞的增殖和迁移[15]。LINC01116还通过调节IFI44促进非小细胞肺癌吉非替尼耐药[16]。在缺氧条件下,LINC00475沉默可以下调AGAP2,对神经胶质瘤产生抑制作用[17]。另外,本研究进一步构建了lncRNAs分子标签联合临床指标模型,ROC曲线发现lncRNAs分子标签联合临床指标模型可进一步提高预测效能。GSEA通路富集分析进一步揭示了细胞周期调控相关基因集在高风险组中有显著富集,而免疫和代谢相关通路更多地在低风险组中富集,暗示不同通路的激活可以影响子宫内膜癌患者的预后。
但本研究也存在着一些局限:①未对本研究中13个lncRNA进行相关的肿瘤机制实验研究,探索lncRNA是如何影响子宫内膜癌的增殖和凋亡,以及如何影响预后;②该数据集源于TCGA数据库,没有汉族人特有的子宫内膜癌数据,因此,未能验证构建的Inc RNA标签能否预测汉族人子宫内膜癌的预后。这些问题需要在以后的研究中进一步探讨。
综上所述,本研究利用公共数据库挖掘,筛选并构建了子宫内膜癌预后13个差异lncRNA的分子标签模型,可以把子宫内膜癌患者分为高风险组和低风险组,并为该疾病的预后预测以及患者的个性化治疗方案提供指导依据,同时本研究策略也可以对其他肿瘤预后相关标志物的筛选提供有效参考。
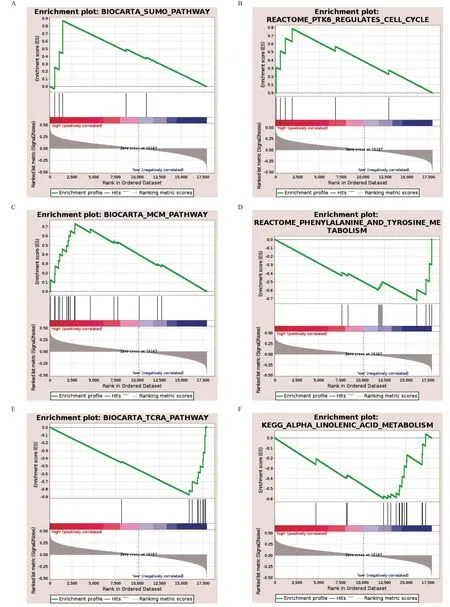
图6 子宫内膜癌的GSEA

