鳅科鱼类DNA条形码鉴定及系统进化研究
刘红艳 ,蔡 金,谢仲桂,熊 飞*,王 莹,王 沁,喻记新,翟东东,夏 明,陈元元
(1.江汉大学生命科学学院,湖北武汉 430056;2.湖北省汉江流域特色生物资源保护开发与利用工程技术研究中心,湖北武汉 430056;3.持久性有毒污染物环境与健康危害湖北省重点实验室,湖北武汉 430056;4.湖南生物机电职业技术学院,湖南长沙 410127)
【研究意义】鳅科(Cobitidae)鱼类为鲤形目中一个较大的类群,种类多、形态差异大,分布广泛,分类关系较为复杂[1-4]。一直以来,鳅科鱼类主要以可数性状和可量性状等形态特征以及一些内部特征为依据进行分类鉴定。【前人研究进展】陈景星[5]通过颊部鳞片有无和眼下刺是否分叉等特征对沙鳅亚科鱼类进行分类。赵铁桥[6]通过鳔、肠、脊椎骨等特征对条鳅属鱼类的分类关系进行了探讨。Kottelat[7]基于骨骼学特征对鳅科进行了系统性的整理,将鳅科分为3个主要的类群。然而传统的鱼类分类需要对大量标本的可量和可数性状进行统计和测量,但是由于个体差异、性别差异和地理环境差异等因素的影响,在形态分类鉴定时容易造成相似种的混淆[8-9]。在实际操作中,也往往会遇到标本量较少以及样本保存不完整等问题,给传统的物种分类和鉴定研究带来较大的困难。因而,单一的形态学鉴定已经无法满足鱼类物种鉴定的需求,选择一种更为有效、便捷的鱼类鉴定方法,并将其用于物种鉴定、分类和进化研究是非常必要的。DNA 条形码是指生物体内能够代表该物种的、有足够变异的、标准的、易扩增并且相对较短的DNA片段,可以快速、准确的进行物种鉴定[10]。DNA条形码自2003年由Hebert等[11]提出后就得到了飞速发展。线粒体DNA 的COI基因具有长度适中、进化速率适中,引物通用性好和易于扩增的特点,已经成为了对物种进行快速而准确鉴定的一个标准分子标签[12]。目前,基于COI基因的DNA 条形码技术在鱼类的分类鉴定及系统进化中得到了广泛的应用[13],如对银鲳进行物种识别[14]和对石首鱼科鱼类系统分类地位的探讨[15]。
【本研究切入点】鳅科是鱼类系统中的一个重要分类单元,近年来,一些研究者以线粒体Cytb基因片段和控制区以及ND4 和ND5 基因分析了部分鳅科鱼类的系统分类关系[16-17],但一些鳅科鱼类的系统发育关系仍未得到一致结论。Li等[18]用线粒体的COI基因条形码对高原鳅属鱼类进行鉴定,但涉及鳅科的种类较少,仅包含高原鳅属鱼类。在鳅科鱼类中,利用线粒体COI基因作为DNA 条形码对鳅科鱼类进行分类鉴定的研究还比较匮乏。【拟解决的关键问题】本研究通过对鳅科鱼类3亚科18属61种鱼类线粒体COI基因的序列进行分析,分析基于COI基因的DNA 条形码在鳅科鱼类中作为物种鉴定的有效性,探讨其系统进化关系,以期为鳅科鱼类的物种鉴定和系统分类提供科学参考。
1 材料和方法
1.1 研究材料
2016—2018年长江上游江段采集了红尾副鳅、横纹南鳅、长薄鳅、小眼薄鳅、红唇薄鳅、中华沙鳅6种鳅科鱼类样本126尾。每个样品取其肌肉组织保存于体积分数为95%乙醇中用于DNA条形码分析。物种形态学鉴定主要依据《中国淡水鱼类检索》[19]、《四川鱼类志》[20]和《云南鱼类志》[21]。另外,从GenBank(https://www.ncbi.nlm.nih.gov)下载了55种鳅科鱼类的COI基因序列232条。总共包含了鳅科鱼类的3亚科18属61种,共358尾鱼类标本(表1)。

表1 研究所用样本名录及相关信息Tab.1 The samples used in the study and related information

续表1
1.2 DNA提取、PCR扩增和序列测定
基因组DNA 采用GENEray 细胞/组织基因组试剂盒法提取,获得的DNA 用TE 缓冲液溶解保存于-20 ℃备用。
PCR扩增引物采用鱼类COI序列通用引物FishF1/FishR1。
FishF1:5'-TCAACCAACCACAAAGACATTGGCAC-3'
FishR1:5'-TAGACTTCTGGGTGGCCAAAGAATCA-3'
PCR反应体系为50µL:上下游引物各为1.0µL(浓度为10µmol/L),基因组DNA模板总量约为20 ng,2×PCRmixture溶液25.0µL,其余补足灭菌蒸馏水。PCR扩增程序为94 ℃预变性5 min,接着进行35个循环:94 ℃变性30 s,56 ℃退火45 s,72 ℃延伸45 s,最后再72 ℃延伸10 min,反应在PTC100型PCR 仪上进行。PCR 产物经10 g/L 琼脂糖凝胶电泳检测,确定PCR 产物为目的条带后,送至武汉擎科生物技术有限公司进行双向测序。
1.3 数据分析
采用DNAstar8.0 软件(DNASTAR Inc.Madison,WI,USA)中的SeqMan 程序查看测序波峰图,手工去除序列两端峰型不规则的部分,保留峰型规则的序列,然后将两条单向序列拼接成一条双向序列。使用MEGA7.0 软件[22]中的Muscle 程序对所有的序列进行同源比对,进行序列的编辑剪切,得到COI基因538 bp 的同源序列矩阵用于随后的遗传分析。利用MEGA7.0 软件统计变异位点、碱基组成情况,基于Kimura2-parameter 双参数模型对种内、种间,属内、属间和亚科内、亚科间的遗传距离进行计算。利用网络版ABGD 软件[23]确定物种的可操作单元,参数采取默认设置,距离矩阵模型选取K2P 模型,计算物种的OTUs 数。利用RAxMLv8.1.17 软件[24]依据GTR+I+G 模型构建贝叶斯(Bayesian)系统进化树,运行2 000 000 代重复。
2 结果
2.1 COI序列信息
本研究获取鳅科鱼类358 条序列,同源比对后538 bp 的线粒体COI基因序列用于条形码分析,包含鳅科鱼类3 亚科,18 属,61 个物种,所有序列都没有发现缺失、插入。A、T、C、G 平均碱基组成分别为A23.5%、T30.9%、C28.2%、G17.4%,平均碱基含量A+T(54.4%)高于C+G(45.6%),显示明显的碱基组成偏倚性,与其他硬骨鱼类线粒体COI基因碱基组成基本一致[25]。在538 pb 序列位点中,有457 个不变位点和81个变异位点,变异位点中有52个转换位点,29个颠换位点,转换与颠换比(R)为1.79。
2.2 遗传距离分析
基于K2P 计算鳅科物种之间的遗传距离关系,种内、属内和亚科内的平均遗传距离分别为0.010、0.054 和0.163,随着分类阶元的升高,遗传距离增大。亚科内的遗传距离为0.107~0.158,条鳅亚科内的遗传距离最小,花鳅亚科内的遗传距离最大。亚科间的遗传距离相差不大,为0.175~0.182。条鳅亚科与沙鳅亚科遗传距离相对较小,花鳅亚科与沙鳅亚科遗传距离相对较大(表2)。

表2 鳅科鱼类的亚科内遗传距离和亚科间遗传距离Tab.2 Genetic distance within subfamily and between subfamily of Cobitidae fishes
鳅科属内和属间遗传距离如表3,其中7个属只有1个物种,不计算属内遗传距离,其余11个属的属内遗传距离为0.013~0.112,平均值为0.054。其中中华沙鳅属属内遗传距离最小(0.013),似鳞头鳅属属内遗传距离最大(0.112)。属间遗传距离为0.092~0.210,其中副沙鳅属与薄鳅属遗传距离最小(0.092),须鳅属与马头鳅属(0.210)、瘦身鳅属与沙鳅属(0.210)遗传距离最大。

表3 鳅科鱼类属内遗传距离和属间遗传距离Tab.3 Genetic distance within genera and between genera of Cobitidae fishes
7 个物种只有1 个个体,不计算种内遗传距离,其余54 种鳅科鱼类种内遗传距离为0~0.130,平均值为0.010。除北方须鳅(0.022)、马头小刺眼鳅(0.026)、温泉似鳞头鳅(0.030)、泥鳅(0.031)、赫氏似鳞头鳅(0.032)、中华花鳅(0.090)、冈特似鳞头鳅(0.095)和瘤鳅(0.130)8 个物种的种内遗传距离大于0.02 外,其余46 个物种的种内遗传距离明显小于Hebert 等[11]提出的种内遗传距离小于0.02 的标准,其中有18 个物种的种内遗传距离为0。种间遗传距离为0.001~0.219,平均值为0.162,种间平均遗传距离是种内平均遗传距离的16.2 倍,种间平均遗传距离大于种内平均遗传距离的10 倍以上。软口高原鳅与黑背高原鳅的种间遗传距离最小,为0.001,北方须鳅与冈特似鳞头鳅种间遗传距离最大,为0.219。有7 组鱼类种间遗传距离小于0.02,它们是:软口高原鳅与黑背高原鳅(0.001)、小眼薄鳅与红唇薄鳅(0.002)、麻尔柯河高原鳅与硬刺高原鳅(0.002)、天台薄鳅与小眼薄鳅(0.002)、天台薄鳅与红唇薄鳅(0.002)、宽体沙鳅与中华沙鳅(0.004)、麻尔柯河高原鳅与贝氏高原鳅(0.009)。除软口高原鳅与黑背高原鳅的种间遗传距离(0.001)小于软口高原鳅种内遗传距离(0.003)外,其余53 种鳅科鱼类的种内遗传距离均小于其种间遗传距离。鳅科鱼类遗传距离在种内、种间重叠较少,能形成一定的DNA 条形码间隙(图1)。
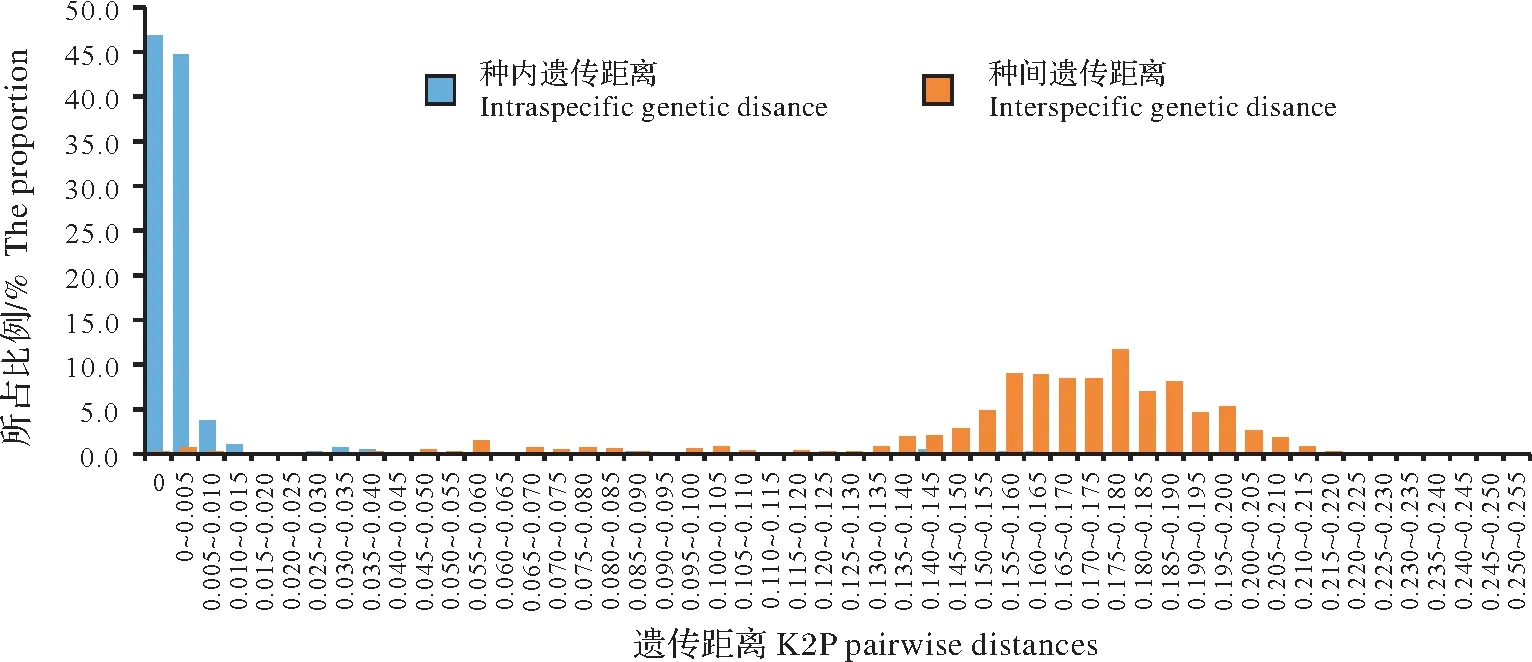
图1 种内和种间遗传距离直方图Fig.1 Histogram of genetic distance within species and between species
2.3 分子系统树及OTUs分析
基于GTR+I+G 模型构建的Bayesian 系统进化树如图2,以鲤科的草鱼(Ctenopharyngodon idellus)和青鱼(Mylopharyngodon piceus)作为外群时,整个鳅科鱼类形成一个单系。在物种水平上,有6 组鱼类彼此的个体相互混杂,不能按各自的物种聚类,它们分别是:中华沙鳅与宽体沙鳅、小眼薄鳅与红唇薄鳅和天台薄鳅、硬刺高原鳅与麻尔柯高原鳅、软口高原鳅与黑背高原鳅、花鳅与长鳅、冈特似鳞头鳅与温泉鳞头鳅。有4种鱼类分成明显的两支,它们是花鳅、冈特似鳞头鳅、泥鳅、瘤鳅。其余46种鳅科鱼类均能按照各自的形态学分类分别聚成单支。在属的水平上,泥鳅属的泥鳅与花鳅属鱼类聚在一起,瘦身鳅属的瘦身鳅和花鳅属的瘤鳅与似鳞头鳅属鱼类聚在一起,南鳅属的2个物种分别与副鳅属和须鳅属鱼类聚在一起,其它属的鳅科鱼类均能按各自的属进行聚类。在亚科的水平上,沙鳅亚科的鱼类聚成一大支,条鳅亚科的全部种类与花鳅亚科的白吻鳅、泥鳅及花鳅属(除瘤鳅外)鱼类聚成第二大支,花鳅亚科的小刺眼鳅属、马头鳅属、瘦身鳅属、柯氏鳅属、似鳞头鳅属与花鳅属的瘤鳅聚成第三大支。值得注意的是,有2个瘤鳅个体处于整个鳅科鱼类聚类的根部位置。
ABGD 分析结果显示(图3),当先验种内遗传距离在0.001 0~0.035 9 时,划分了66 个操作分类单元(OTUs)),多于形态学分类的物种。北方须鳅、花鳅、中华花鳅、泥鳅、冈特似鳞头鳅、瘤鳅分为2 个OTUs,马头小刺眼鳅、赫氏似鳞头鳅分为了3 个OTUs。OTUs 的划分大体与距离法一致,被分为多个OTUs的物种,其种内遗传距离大多大于0.02的分类标准。
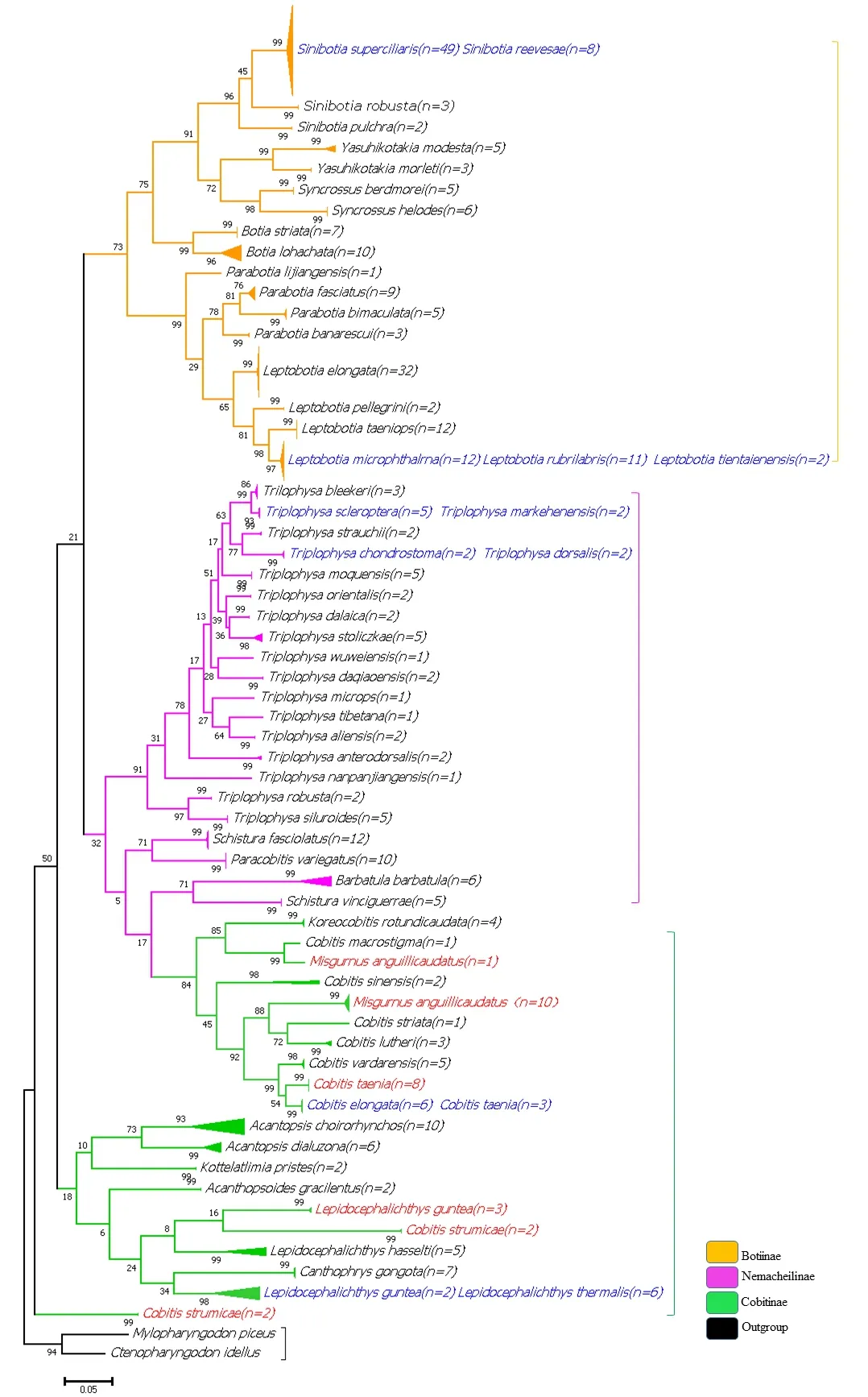
图2 COI基因基于GTR+I+G模型构建的鳅科鱼类Bayesian系统树Fig.2 Bayesian phylogenetic tree of Cobitidae based on COI gene using GTR+I+G model
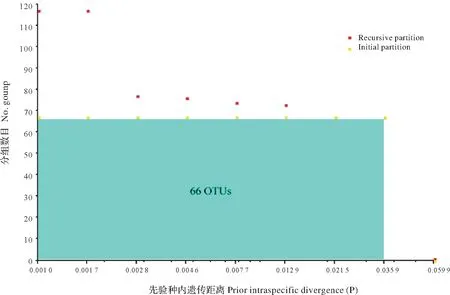
图3 ABGD划分的鳅科鱼类操作分类单元Fig.3 Operational taxonomic units of Cobitidae defined by ABGD analysis
3 讨论
在DNA 序列分析中,不同物种的种内和种间遗传距离大小是物种鉴定的一个重要判别标准。Hellberg 等[26]利用线粒体COI基因作为条形码对动物物种鉴别时,认为种内的遗传距离不大于0.02,且种间的遗传距离大于种内距离10 倍以上可有效判别物种。本研究中,种内平均遗传距离为0.010,种间平均遗传距离为0.162,种间平均遗传距离是种内平均遗传距离的16.2 倍,说明COI基因序列的遗传差异为鉴定鳅科鱼类提供了良好的基础。本研究中,除软口高原鳅与黑背高原鳅的种间遗传距离小于软口高原鳅种内遗传距离外,其余鳅科鱼类的种内遗传距离均小于其种间遗传距离,形成了一定的条形码间隙。
本研究中,鳅科鱼类的种内平均遗传距离(0.010)稍高于已报道的几种鱼类的条形码遗传距离,如大西洋邻加拿大水域的海水鱼类[27]、长江中游的鲤科鱼类[28]。较高的平均种内遗传距离主要是由于北方须鳅、马头小刺眼鳅、温泉似鳞头鳅、泥鳅、赫氏似鳞头鳅、中华花鳅、冈特似鳞头鳅、瘤鳅等鱼类中较高的种内遗传距离导致的。同一物种出现较高水平的种内遗传距离可能是由于原始物种错误鉴定,也有可能是由于不同隐存的或是没有被发现的成种事件造成的[29]。目前,已有不少分子数据证明鱼类存在数量巨大的隐存种[30-31]。如唐琼英[32]认为形态特征上比较相似的一些花鳅物种均被划分到中华花鳅这一物种中,中华花鳅可能是包含很多种类的一个复合类群。由于隐存种形态十分相似,根据外形特征不能把它们区分开,但在分子水平上表现出差异性,因此条形码相对于传统生物鉴定的优势在于可以揭示隐存种的存在。本研究中,北方须鳅、花鳅、中华花鳅、泥鳅、冈特似鳞头鳅、瘤鳅分为2 个OTUs,马头小刺眼鳅、赫氏似鳞头鳅分为了3个OTUs。另外,聚类分析中,花鳅、冈特似鳞头鳅、泥鳅、瘤鳅在聚类上已经形成了两个明显的分支。这些结果显示,可能有隐存种的存在。另外,值得注意的是,有2个瘤鳅个体处于整个鳅科鱼类的根部位置,与另外2个正常聚类到花鳅亚科的瘤鳅个体的遗传距离差异相当大,但是这4 个个体均来源于同一地理位置,因此,笔者推测处于鳅科根部位置的2 个瘤鳅个体可能是原始种的错误鉴定造成的。
一般认为,鳅科中的沙鳅亚科是分类关系相对明确的一个类群,而条鳅亚科和花鳅亚科种类繁多,分类关系复杂。先前的学者支持沙鳅亚科为一个单系类群,但关于条鳅亚科和花鳅亚科单系性的问题争议则较大[17,32-34]。本研究中,沙鳅亚科的所有个体分别按各自的属聚类,然后各属聚为一大支,研究结果也支持沙鳅亚科为一单系类群,并且各属之间关系明确。唐琼英[32]认为条鳅亚科包含大量形态上极为相似的物种,支持该亚科为一单系类群,但Nalbant[33]认为条鳅亚科为多系起源。本研究结果中,花鳅亚科的一些物种与条鳅亚科的物种聚在一起,因此,条鳅亚科并不能形成单系。Liu 等[35]认为花鳅亚科物种的多样性最为丰富也最为复杂,认为花鳅亚科分为两支,一支由花鳅属、后鳍花鳅属、副泥鳅属和泥鳅属构成,另一支由小刺眼鳅属、瘦身鳅属和潘鳅属构成,不能形成单系。本研究中花鳅亚科的一些种类虽然与Liu 等[35]研究的种类有些差异,但本研究结果与Liu 等的观点大体相同,花鳅亚科分为两支,支持花鳅为多系起源。
在鳅科中,Hora[36]将沙鳅属从薄鳅属和副沙鳅属中分离出来成为一个独立的属,Fang[37]又将沙鳅属分为沙鳅亚属(botia)、中华沙鳅亚属(Sinibotia)和鳔膜沙鳅亚属(Hymenophysa)。后来的一些学者在分类的时候并没有把这3个属进行细分,而是统一把它们归为沙鳅属[20,32,35]。本研究的材料中,只包含有沙鳅亚属和中华沙鳅亚属的种类,但在聚类关系中,这2个亚属的物种分别聚类,各自形成单系,与沙鳅亚科其它属的物种形成平行分支,并且这2 个属的遗传距离较远,本研究结果支持把这2 个亚属上升到属的地位,分别为沙鳅属和中华沙鳅属。Bananescu等[38]认为南鳅属包含了形态上差异极大的几个类群,是较为复杂的一个多系起源类群。本研究中包括了南鳅属的2 个物种,2 个南鳅属鱼类并没有聚在一起,横纹南鳅与副鳅属鱼类聚成在一起,密纹南鳅与须鳅属鱼类聚在一起,因此南鳅属物种分类关系还需要明确。陈景星等[1]指出花鳅属是分化最为明显的一个属。本研究中,花鳅属物种的聚类比较分散,花鳅属鱼类并没有聚在一起,而是分别与花鳅亚科的其它属物种聚在一起,这表明了花鳅属的分类还存在较大的问题,对其物种的归属问题还需进一步的整理和细化。
DNA条形码最重要的作用就是对物种进行准确鉴别,弥补传统形态学鉴定方法的不足,明确鱼类的种类和系统进化过程[39]。在本研究中,综合遗传距离、聚类分析和OTUs的结果,61种鳅科鱼类中有46个物种能利用COI条形码进行有效的鉴别,占总物种数的75.41%,另外,COI条形码在鳅科的属及亚科分类上也有一定的参考价值,能够明确大多数属和亚科物种的分类归属问题。

