浓香型白酒酒醅中总RNA 提取方法评价
胡晓龙,王康丽,宋丽丽,侯建光,曹振华,吴丽丽,牛广杰,马歌丽,赵书民,赵 东
(1.郑州轻工业大学食品与生物工程学院,河南 郑州 450000;2.河南仰韶酒业有限公司博士后研发基地,河南 渑池 472000;3.河南宋河酒业股份有限公司,河南 鹿邑 477200;4.河南省寿酒有限公司,河南 新乡 453000;5.河南省酒业协会,河南 郑州 450000;6.五粮液集团有限公司,四川 宜宾 644000)
浓香型白酒作为我国优势传统发酵食品的典型代表,风格独特,深受消费者青睐,其产销量均占中国白酒行业的主导地位[1]。在酿造过程中,酒醅是微生物发酵的主要载体和白酒呈香物质的直接来源,因此,研究酒醅活性微生物菌群的多样性、代谢特征及相互作用等是阐明浓香型白酒固态发酵机制及风味物质溯源的关键环节。
近年来,在RNA水平上进行的宏转录组学技术被不断运用于微生物群落研究,宏转录组学是后基因组时代最具代表性的新技术[2],通过对特定时期、特定环境样品中所有微生物群落的RNA转录本进行大规模测序,可以直接获得环境中可培养和不可培养的微生物转录组信息,能真实地反映出活性微生物状态及其动态变化[3]。例如,Urich等[4]通过对沙质土壤生态系统中微生物RNA转录本的分析得到了许多未被人们发现的基因组序列,且发现有5 种不同的优势细菌种类发生了变化,并得到了这些细菌的代谢机制变化以作为预测生物体活动的指标。Franzosa等[5]在关联人体肠道的宏基因组和宏转录组分析中,发现有部分微生物可在DNA水平上检测到但很少具有转录活性,且RNA水平功能组成的组间差异明显强于DNA水平,因此仅鉴定DNA水平上微生物的单独贡献(高或低)并不能完全反映它们在群落中的作用。以RNA为基础的宏转录组等分子生物学研究则能帮助更好地了解浓香型白酒发酵过程中活性微生物群落的特征及变化,而提取的总RNA质量好坏对后续的研究结果影响较大。
能否有效去除各种杂质和RNA酶是成功提取高质量RNA的关键,目前常见的环境样品RNA提取方法有Trizol法、十二烷基硫酸钠(sodium dodecyl sulfate,SDS)法、异硫氰酸胍法、十六烷基三甲基溴化铵(hexadecyl trimethyl ammonium bromide,CTAB)法和已商品化的试剂盒法。异硫氰酸胍是强烈的蛋白变性剂,可有效抑制RNA酶的活性[6],此方法可得到较高产量的总RNA,但耗时长,不适用于大量样品的提取[7]。Trizol是一种总RNA抽提试剂,可以直接从细胞或组织中提取出高总产量的总RNA[8-10],但需液氮研磨,实验条件苛刻、成本较高,提取富含多糖和多酚的样品总RNA时会存在严重的污染。CTAB法操作需要预热,温度过高会使得RNA酶变得活跃导致RNA降解,同时耗时较长、操作繁琐[11]。试剂盒法提取RNA虽然方便快捷,可得到高纯度的RNA,但产量较低、费用高,且对不同的样品具有选择性[12]。SDS法虽然试剂简单易配制,但其无法抑制内源性RNA酶的活性,在提取过程中RNA降解严重,将其与苯酚结合可在抑制RNA酶的同时使蛋白质等变性,再结合玻璃珠物理破碎法,可有效地裂解土壤等环境样品,用以大量提取总RNA[13-15]。
酒醅中多糖、酚类、盐类、腐殖质、腐殖酸和代谢色素等杂质大量存在,形成了酒醅复杂的环境结构[16],所以其总RNA的提取存在一定困难。另外,不同提取方法获得的微生物群落多样性及其种类、丰度均不同[17]。目前酒醅总RNA提取可采用Trizol法、月桂酸钠法[11,18],然而,有关酒醅总RNA提取方法的比较研究尚鲜见报道。
本研究建立了一种改良的SDS-苯酚法,并从提取成本及耗时、RNA质量浓度和纯度、电泳结果、反转录效果及高通量测序分析5 个方面比较其与目前常规的4 种RNA提取方法提取浓香型白酒酒醅总RNA的效果,包括试剂盒法、Trizol法、CTAB法和月桂酸钠法。
1 材料与方法
1.1 材料与试剂
酒醅样品取自河南省某知名浓香型白酒企业,发酵时间为7 d和15 d的酒醅样品各200 g,放入无菌厌氧袋中并作好标记,迅速放于冰盒中运回实验室,于4 ℃保藏。窖泥护理液(主要为Clostridia纲细菌)来自本实验室。
SDS 上海百赛生物技术股份有限公司;月桂酸钠、二硫苏糖醇、β-巯基乙醇、异戊醇、聚乙二醇(polyethylene glycol,PEG)、CTAB、焦碳酸二乙酯(diethyl pyrocarbonate,DEPC) 上海麦克林生化科技有限公司;Trizol、水饱和酚 上海源叶生物科技有限公司;异丙醇、无水乙醇 天津市富宇精细化工有限公司;Tris、聚乙烯吡咯烷酮(polyvinyl pyrrolidone,PVP)、RNase-Free Water、2×Taq Master Mix 北京索莱宝科技有限公司;氯化钠、磷酸二氢钠、磷酸氢二钠 北京北化精细化学品有限责任公司;E.Z.N.A.TMSoil RNA Kit 美国Omega公司;Recombinant DNase I、PrimeScriptTMII 1st Strand cDNA Synthesis Kit 大连TaKaRa公司;所有分离用有机溶剂均为国产分析纯。
1.2 仪器与设备
MP200A精密电子天平 上海良丰仪器仪表有限公司;C1000温度梯度聚合酶链式反应(polymerase chain reaction,PCR)仪 伯乐生命医学产品(上海)有限公司;TGL-20M高速离心机 上海卢湘仪离心机仪器有限公司;DYY-8C电泳仪 北京市六一仪器厂;Mixer4K微型涡旋混合仪 生工生物工程(上海)股份有限公司;KW-1000DC恒温水浴锅、85-2恒温磁力搅拌器江苏金坛市中大仪器厂;IMS-40全自动雪花制冰机常熟市雪科电器有限公司;LDZX-50KBS立式压力蒸汽灭菌器 上海中安医疗器械厂;SW-CJ-1F超净工作台 苏州净化设备有限公司;WD-9403C紫外仪 北京六一生物科技有限公司;NanoDrop2000微量分光光度计赛默飞世尔科技公司;Illumina MiSeq测序仪 美国Illumina公司。
提取RNA所用的枪头、离心管、烧杯、玻璃棒等均需用0.1% DEPC溶液浸泡过夜,之后1×105Pa灭菌30 min,烘干备用;电泳槽和制胶用具用3%的H2O2溶液浸泡30 min,再用DEPC溶液冲洗干净。
1.3 方法
1.3.1 酒醅总RNA的提取方法
1.3.1.1 酒醅预培养
将低温保存发酵7 d和15 d的酒醅样品各取出100 g,分别置于无菌厌氧培养袋中,每个样品均按5%接种量分别添加窖泥护理液,将接种后的培养袋置于恒温培养箱中,37 ℃培养3 d,以期获取更多有活性的微生物。
1.3.1.2 SDS-苯酚法
参照赵俊等[19]总RNA提取方法进行改良。1)取7 g酒醅于50 mL离心管中,加入5 mL 1 mol/L的CaCl2溶液和5 mL 5% PVP溶液,涡旋混匀,8 000 r/min离心5 min,弃上清液;加入10 mL 0.05 mol/L的草酸钠溶液,用相同的方法离心、弃上清液。2)加入3 颗玻璃珠(d=0.5 cm)和12 mL 0.1 mol/L磷酸盐缓冲液悬浮,涡旋振荡5 min后500 r/min离心5 min,取上清液至新的离心管中,沉淀重复洗涤一次,2 次上清液混合后离心3 min,弃去上清液收集细胞沉淀。3)在得到的细胞沉淀中加入玻璃珠(0.5 mm:0.33 g,0.1 mm:0.17 g)和300 µL SDS提取液(50 mmol/L Tris-HCl,10 mmol/L EDTANa2,10 mmol/L MgCl2,1% SDS,6%水饱和酚,0.1% β-巯基乙醇),最大转速涡旋振荡5 min,然后15 000 r/min、4 ℃离心2 min,取上清液至新的离心管中,再重复一次;4)向合并的上清液中加入500 µL水饱和酚(pH 4.5),涡旋混匀后离心2 min,取上清液;再加入500 µL的氯仿-异戊醇(24∶1),用相同的方法混匀、离心、回收上清液。5)上清液加入2 倍体积的PEG-NaCl(30% PEG,1.6 mol/L NaCl)溶液,低温静置2 h后15 000 r/min、4 ℃离心10 min,弃上清液。6)用200 µL 70%乙醇溶液清洗沉淀,最后加入50 µL RNase-free water溶解RNA。
1.3.1.3 试剂盒法
称取7 g酒醅样品于50 mL离心管中,用15 mL灭菌后的0.1 mol/L磷酸盐缓冲液悬浮,加入3 颗玻璃珠(d=0.5 cm),涡旋振荡5 min,然后500 r/min离心5 min,取上清液。沉淀用磷酸盐缓冲液重复洗涤3 次,离心后收集上清液。全部上清液于9 000 r/min离心3 min,弃去上清液,收集细胞沉淀。对预处理后的酒醅样品按照E.Z.N.A.TMSoil RNA Kit(Omega)的试剂盒说明书方法进行总RNA提取。
1.3.1.4 Trizol法
本方法与SDS-苯酚法基本相同,不同的是采用Trizol试剂抽提,将步骤3)中的SDS提取液换成Trizol试剂。
1.3.1.5 CTAB法
参照Griffiths等[20]的方法并进行适当调整。首先进行预处理(方法同试剂盒法),然后在预处理后的酒醅样品中加入0.5 mL CTAB提取液(等体积的磷酸钾缓冲液与10% CTAB溶液配制)和玻璃珠(0.5 mm:0.33 g,0.1 mm:0.17 g),在FastPrep匀浆仪上以6 m/s的速率破碎45 s;16 000×g、4 ℃离心5 min,取上清液;加入0.5 mL苯酚-氯仿-异戊醇(25∶24∶1),涡旋混匀,16 000×g、4 ℃离心5 min,取上清液;上清液加入等体积的氯仿-异戊醇(24∶1),涡旋混匀,16 000×g、4 ℃离心5 min,取上清液;上清液加入2 倍体积PEG-NaCl(30% PEG,1.6 mol/L NaCl),上下颠倒混匀,低温静置2 h,18 000×g、4 ℃离心10 min,弃上清液;加入200 µL 70%乙醇溶液清洗沉淀,16 000×g离心5 min,弃掉液体,并吹干沉淀,加入50 µL RNase-free water溶解RNA,然后保存于-80 ℃超低温冰箱备用。
1.3.1.6 月桂酸钠法
本方法与SDS-苯酚法基本相同,不同的是采用月桂酸钠抽提液提取,将步骤3)中的SDS提取液换成月桂酸钠抽提液(25 份月桂酸钠缓冲液(0.1 mol/L pH 8.0 Tris-HCl,0.1 mol/L NaCl,0.02 mol/L pH 8.0 EDTANa2,月桂酸钠10 g/L),15 份Trizol,1.5 份β-巯基乙醇,0.5 份二硫苏糖醇)[11]。
1.3.2 酒醅总RNA的质量检测
1.3.2.1 浓度及纯度测定
分别吸取利用上述5 种方法所获得的总RNA样品各2 μL,采用NanoDrop 2000超微量分光光度计测定每个RNA样品用于表征浓度的260 nm波长处OD值,以及表征样品纯度的OD260nm/OD280nm和OD260nm/OD230nm比值。
1.3.2.2 琼脂糖凝胶电泳
取20 mL 1×TAE溶液,加入0.2 g的琼脂糖粉,加热至沸腾,室温冷却至 50~60 ℃后,加入2 µL Goldview I型核酸染料,倒入制胶槽中冷却至凝固。每个RNA样品取5 µL分别与1 µL的6×DNA loading buffer均匀混合,然后将其与Marker分别点样于琼脂糖凝胶,于100 V电泳30 min。
1.3.2.3 反转录PCR(reverse transcription-PCR,RT-PCR)验证
利用Recombinant DNase I除去上述RNA样品中的DNA,以防止DNA污染导致扩增结果出现偏差。利用TaKaRa公司的PrimeScriptTMII 1st Strand cDNA Synthesis Kit反转录合成cDNA,反应操作按照试剂盒说明书进行。
参照Zhang Yuguang等[21]描述的方法,使用细菌通用引物515F和806R、相应的扩增反应体系及条件对上述cDNA进行扩增。扩增完成后对扩增产物进行琼脂糖凝胶电泳检测。
1.3.2.4 高通量测序及数据分析
针对酒醅反转录cDNA模板,采用微生物16S rRNA基因的通用引物扩增V3和V4两个高度可变区。PCR扩增参数:94 ℃预变性3 min;94 ℃变性5 s,57 ℃退火90 s,72 ℃延伸10 s;72 ℃终延伸5 min;共进行24 个循环。扩增完毕后将PCR产物进行1.5%琼脂糖凝胶电泳,在紫外灯下切割回收目标产物片段进行纯化,再进一步通过电泳检测PCR产物纯化效果。然后使用酶标仪检测文库浓度,将文库定量到10 nmol/L,按Illumina MiSeq仪器使用说明书进行PE250/PE300双端测序,由MiSeq自带的MiSeq Control Software读取序列信息。
对双端测序得到的正反向reads首先进行两两拼接,过滤拼接结果中含有N的序列,保留序列长度大于200 bp的序列。经过质量过滤,去除嵌合体序列,最终得到的序列使用VSEARCH(1.9.6)进行操作分类单元(operational taxonomic units,OTU)聚类(序列相似性设为97%),比对的16S rRNA参考数据库为Silva 132。然后利用RDP classifier(Ribosomal Database Program)贝叶斯算法对OTU的代表性序列进行物种分类学分析,并在不同物种分类水平下统计每个样本的群落组成。基于OTU得到分析结果,采用对样本序列进行随机抽平的方法,分别计算Shannon指数、Chao1指数等α多样性指数反映群落的物种丰度和多样性,再通过层次聚类中的非加权组平均法构建UPGMA(unweighted pair group method with arithmetic mean)聚类树,并基于样本OTU丰度进行主成分分析(principal component analysis,PCA)。
2 结果与分析
2.1 总RNA质量浓度及纯度、成本及耗时分析
如表1所示,5 种方法均能提取到酒醅样品中总RNA,其中试剂盒法提取的总RNA平均质量浓度最高(1 190.8 ng/µL),分别为SDS-苯酚法(647.6 ng/µL)、Trizol法(512 ng/µL)、CTAB法(385.65 ng/µL)和月桂酸钠法(62.2 ng/µL)的1.84、2.33、19.14 倍和3.09 倍。5 种方法所得总RNA样品的OD260nm/OD230nm存在较大差异,其中SDS-苯酚法和试剂盒法提取的酒醅总RNA的OD260nm/OD230nm均大于2.0,表明这两种方法所提取出的酒醅总RNA中多糖、酚类、腐殖酸和有机溶剂等物质的污染较小,纯度高[22]。而Trizol法、CTAB法和月桂酸钠法提取的酒醅总RNA样品的OD260nm/OD230nm远小于2.0,其平均值分别为0.29、1.10和0.58,表明这3 种方法所提取的RNA样品中存在多糖、酚类、腐殖酸或有机溶剂等物质的污染现象,纯度较差,尤其是CTAB法所提RNA样品纯度最差,进而会对后续分子实验(如real-time PCR、RT-PCR及电泳分析等)产生不同程度的干扰。SDS-苯酚法和Trizol法所提取酒醅总RNA的OD260nm/OD280nm均在1.8~2.0之间,表明这两种方法所提取的RNA样品中蛋白质污染较小,纯度较好。试剂盒法所提取的酒醅总RNA的OD260nm/OD280nm大于2.0,表明该方法所提取的总RNA有降解发生[23]。CTAB法和月桂酸钠法提取的酒醅总RNA样品的OD260nm/OD280nm均较低,表明RNA样品中存在蛋白质等物质的污染,纯度较差。
从提取每个酒醅样品总RNA所需要试剂的费用进行比较,SDS-苯酚法和CTAB法成本最低,试剂盒法成本最高,提取一次RNA的费用约49 元。5 种方法提取RNA耗时在3.5~5 h之间,试剂盒法耗时最短,其余方法在5 h左右。
2.2 总RNA完整性分析
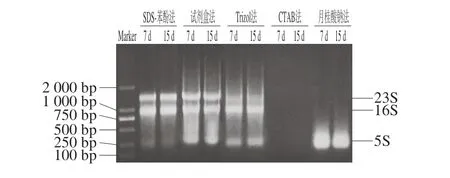
图1 5 种方法提取酒醅总RNA的琼脂糖凝胶电泳图Fig. 1 Agarose gel electrophoresis of total RNA from fermented grains extracted by five extraction methods
如图1所示,采用同种方法提取两种酒醅所得RNA的电泳条带无明显差异,但不同方法所提RNA亮度差异较大,试剂盒法和SDS-苯酚法所提取的RNA条带亮度明显亮于Trizol法,CTAB法未检测到条带,这也与RNA质量浓度检测结果一致。SDS-苯酚法提取酒醅总RNA中23S和16S条带清晰整齐,5S条带不明显,其中23S条带亮度与16S基本一致,说明RNA完整性较好。试剂盒法提取酒醅总RNA 23S和16S条带模糊不整齐,电泳条带弥散,整体提取效果一般。Trizol法提取酒醅总RNA 23S和16S条带不够清晰完整,5S条带明显,略有拖带,表明RNA部分降解、有杂质污染,提取效果一般。月桂酸钠法提取酒醅总RNA仅有5S条带清晰可见且不规整,表明RNA降解严重,完整性差。
2.3 RT-PCR检测分析
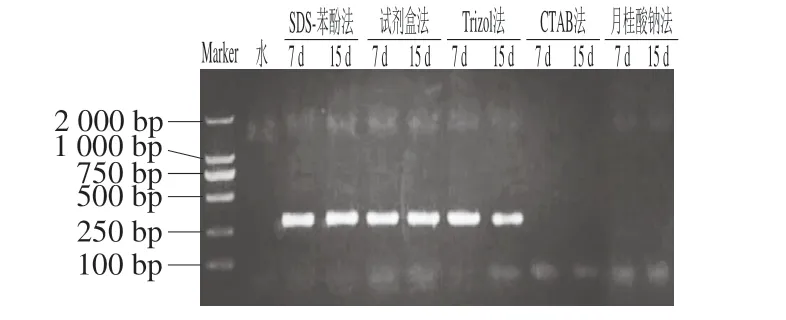
图2 5 种方法提取酒醅总RNA进行RT-PCR后的琼脂糖凝胶电泳图Fig. 2 Agarose gel electrophoresis of RT-PCR amplified total RNA extracted from fermented grains with five extraction methods
采用RT-PCR实验进一步分析上述方法所得总RNA的完整性及用于后续分子生物学实验的可行性,结果如图2所示。除CTAB法、月桂酸钠法外,SDS-苯酚法、试剂盒法和Trizol法代表的6 个泳道均出现了目的条带,表明这3 种方法所提RNA均能有效地进行反转录实验,条带大小在290 bp左右,与预期结果一致;结合RNA纯度的分析结果(表1),3 种方法所提取的总RNA基本无蛋白质污染,同时总RNA质量浓度高,对后续RT-PCR影响不大。但Trizol法提取总RNA的OD260nm/OD230nm偏低,考虑会对后续分析结果有影响,将进一步结合高通量测序结果进行分析。
2.4 高通量数据分析
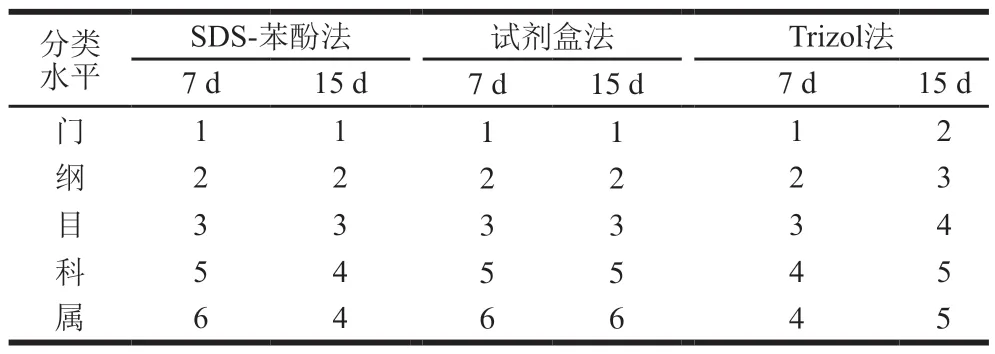
表2 SDS-苯酚法、试剂盒法和Trizol法所提RNA对酒醅活性群落种属鉴定结果的影响Table 2 Effect of RNA extraction methods on the identification of microbial communities in fermented grains
根据RT-PCR结果,本研究选取了SDS-苯酚法、试剂盒法和Trizol法所提取的RNA反转录后的cDNA样本用于高通量测序16S rRNA,结果表明,3 种方法提取的RNA对结果的影响存在差异。如图3A所示,SDS-苯酚和试剂盒法结果相似,即发酵7 d酒醅OTU数量均高于15 d的酒醅,与Trizol法所得结果相反,说明高通量测序结果与样品和RNA的提取方法均有关系[19]。采用上述3 种方法从酒醅中共检测到2 个门、3 个纲、4 个目、6 个科和7 个属,每种方法所对应每个分类水平的数量见表2。不同方法对酒醅样品中细菌不同分类水平种属鉴定数量(图3B)及细菌群落Chao1指数(图3C)没有显著差异(P>0.05),说明上述指标主要是由样品决定的。由图3D可知,微生物群落多样性的趋势为SDS-苯酚法>Trizol法>试剂盒法,其中SDS-苯酚法所得Shannon指数显著高于试剂盒法(P<0.05),说明结合高通量测序分析,SDS-苯酚法所提取的RNA能更好地反映酒醅群落的群落的多样性。
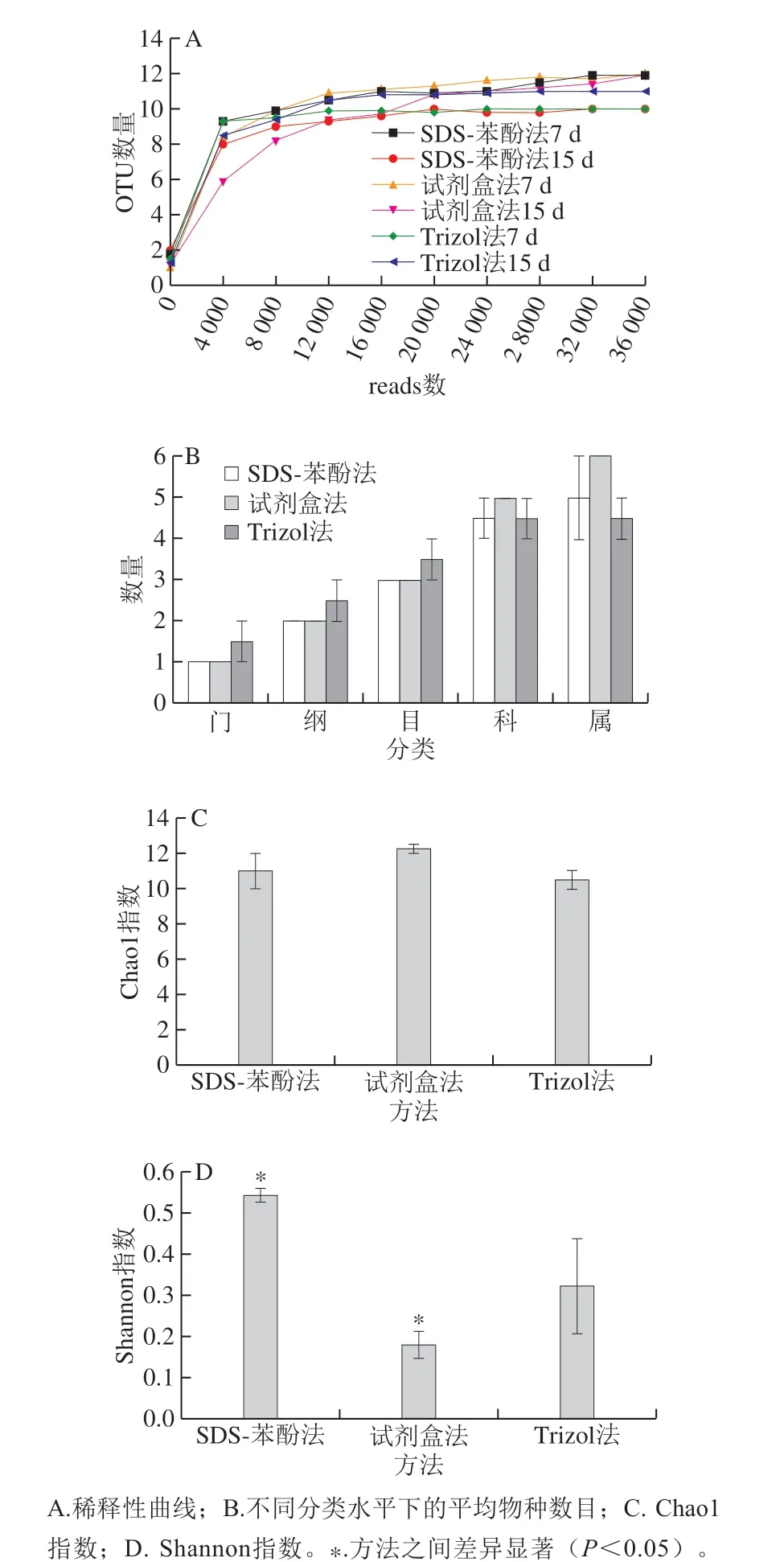
图3 SDS-苯酚法、试剂盒法和Trizol法所提RNA对酒醅活性群落α多样性分析影响Fig. 3 Effect of RNA extraction methods on α-diversity analysis of microbial communities in fermented grains
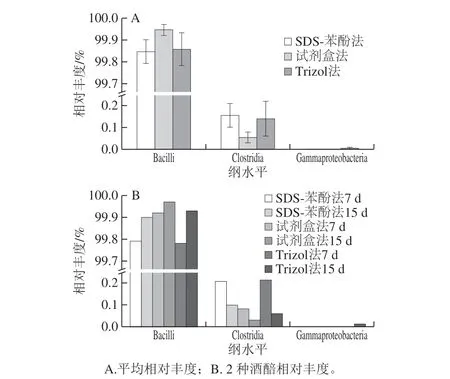
图4 SDS-苯酚法、试剂盒法和Trizol法所提RNA对酒醅活性群落纲水平组成影响Fig. 4 Effects of RNA extraction methods on microbial community composition at class level in fermented grains
在原核微生物纲水平(图4A),SDS-苯酚法和试剂盒法在2 种酒醅中提取的纲数量均一致,分别是芽孢杆菌纲(Bacilli)和梭菌纲(Clostridia),而Trizol法除了提取到芽孢杆菌纲和梭菌纲外,在15 d酒醅中还提取到γ-变形菌纲(Gammaproteobacteria),但其相对丰度仅为0.01%。随着发酵时间的延长(图4B),SDS-苯酚法、试剂盒法和Trizol法均表现出芽孢杆菌纲的相对丰度升高,涨幅分别为0.11%、0.05%、0.15%,相反,梭菌纲的相对丰度降低,降幅分别为0.2%、0.05%、0.16%,这可能与酒醅的特性有关,随着发酵过程的进行,芽孢杆菌纲的乳杆菌属作为酒醅微生物群中的优势菌不断增加。结果表明,不同方法提取的纲数量和相对丰度存在一定差异,但基本变化趋势一致。
在原核微生物属水平(图5A),两种酒醅共计检测到7 个属,分别为乳杆菌属(Lactobacillus)、梭菌属(Clostridium_sensu_stricto_10)、芽孢杆菌属(Bacillus)、赖氨酸芽孢杆菌属(Lysinibacillus)、短小芽孢杆菌(Brevibacillus)、水栖菌属(Enhydrobacter)和类芽孢杆菌属(Paenibacillus),3 种方法提取微生物的数量和相对丰度均有一定差异。在7 d酒醅中,SDS-苯酚法和试剂盒法均提取到了5 个属(图5B、C),Trizol法提取到了4 个属(图5D);在15 d酒醅中,SDS-苯酚法、试剂盒法和Trizol法分别提取到了4、6、5 个属,其中试剂盒法偏好性地提取到了类芽孢杆菌属,Trizol法偏好性地提取到了水栖菌属,但两者相对丰度均仅为0.01%。随着发酵时间的延长(图5B~D),3 种方法所得原核微生物中相对丰度排名前3的乳杆菌属、梭菌属和芽孢杆菌属的变化趋势均一致,乳杆菌属不断增加,梭菌属和芽孢杆菌属则不断下降,这些结果与纲水平上的结果一致。尽管3 种方法所得微生物丰度并不完全相同,但3 种方法均能反映出两种酒醅原核微生物群之间的差异,并且规律基本一致。
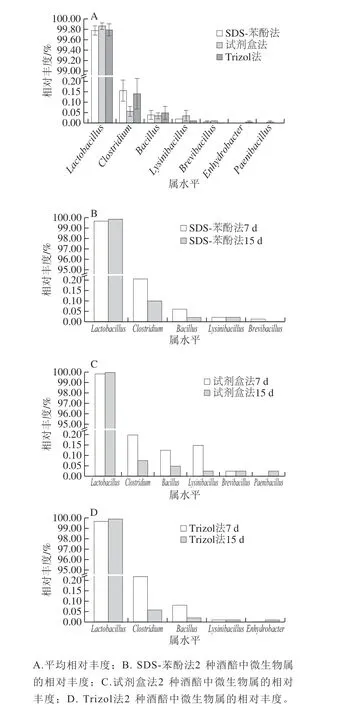
图5 SDS-苯酚法、试剂盒法和Trizol法所提RNA对酒醅活性群落属水平组成影响Fig. 5 Effects of RNA extraction methods on microbial community composition at genus level in fermented grains
根据UPGMA聚类树分析和PCA结果(图6),Trizol法在反映样本间差异方面的效果最好,SDS-苯酚法次之,试剂盒法则较差。虽然试剂盒法和Trizol法偏好性地提取到了1 个属,但其相对丰度过低,不能完全反映其在发酵过程中的变化情况。所以,决定原核微生物群的因素不仅有酒醅类型,提取方法在反映其丰度变化及样本差异方面的影响可能更大。
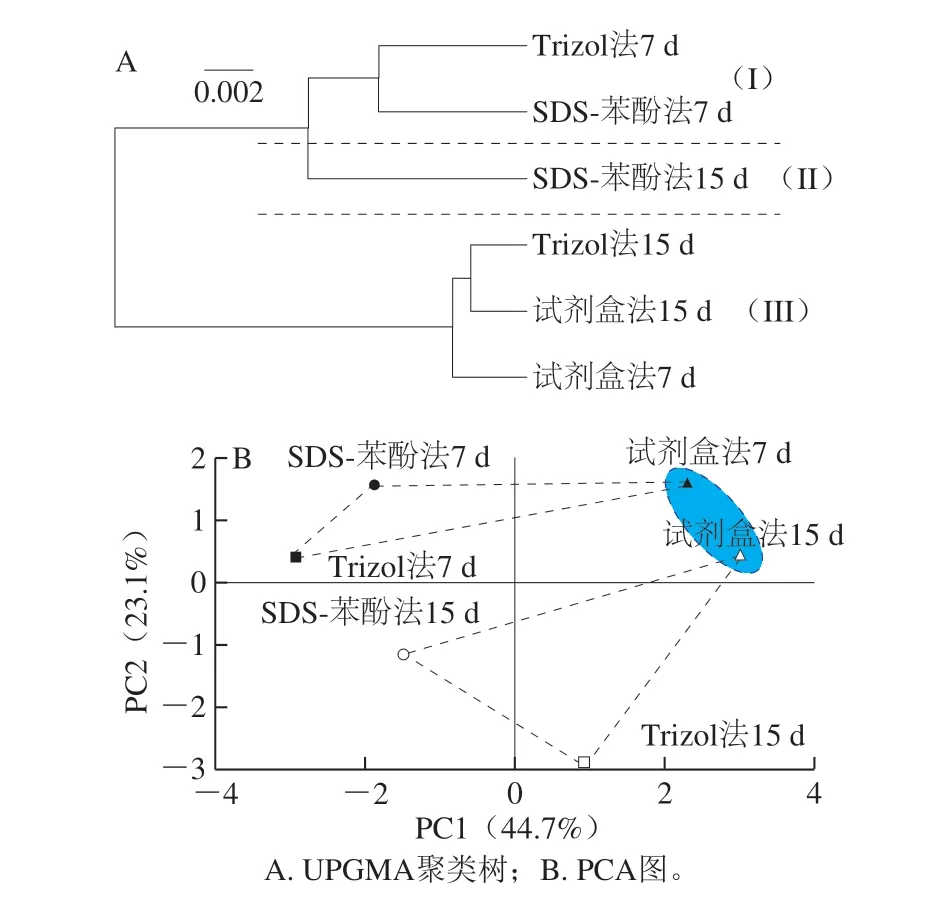
图6 SDS-苯酚法、试剂盒法和Trizol法所提RNA对酒醅活性群落β多样性影响Fig. 6 Effect of RNA extraction methods on β-diversity analysis of active microbial populations in fermented grains
3 讨 论
酒醅作为白酒酿造过程中微生物发酵的主要载体和白酒呈香物质的直接来源,影响其RNA提取质量的因素有多种,一般分为外因和内因:内因主要指酒醅环境中多糖、蛋白质、酚类及腐殖质等杂质含量较高,多糖类的溶解性质与RNA相似不容易被去除[24],而蛋白质则影响RT-PCR,酚与RNA结合会影响RNA活性[25];外因通常也影响RNA提取的质量,如实验人员的不规范操作、实验仪器以及试剂的污染等。因此,克服内因和外因对RNA的不良影响,是得到高质量RNA成败的关键。对酒醅样品进行预处理,则是一种简单有效的方法,能有效地去除大量杂质,保证后续分子生物学实验的顺利进行[16]。
CTAB法在应用于酒醅提取时,浓度及纯度都很低,可能是由于裂解液不能完全裂解细胞、操作步骤过于简单、沉淀时间较短等引起的RNA质量较差。月桂酸钠是一种生物降解性良好的阴离子表面活性剂,将其作为提取缓冲液能将植物组织中存在的多糖、色素和多酚等次生物质进行洗脱[26],但应用于酒醅中时却无法有效提取到RNA以及去除杂质,这可能需要结合液氮研磨提取效果才会更佳。Trizol试剂可直接从组织或细胞中提取总RNA,能迅速裂解细胞并抑制细胞释放出来的核糖核酸酶,从而保护RNA的完整性[27],是一种操作简单便捷的方法,但在本研究中,Trizol法对酒醅进行总RNA提取时,所得的杂质污染较严重。而本研究中选用的试剂盒为目前市场上流行的E.Z.N.A.TMSoil RNA Kit(Omega)试剂盒[12,28-31],其通过涡旋珠磨技术与苯酚-氯仿萃取相结合从样品中分离RNA[32],再通过离心柱和洗脱液去除DNA、蛋白质和其他杂质,既能有效去除杂质又同时具有浓缩功能,因而能获得浓度高、纯度好的RNA。本研究建立的SDS-苯酚法通过结合酒醅独特的预处理方法,包括CaCl2去除腐殖质、PVP沉淀多糖多酚等在抽提前去除大量杂质,再通过磷酸盐缓冲液对酒醅进行除杂和收集细胞沉淀,排除大部分杂质的干扰,有利于提升总RNA提取纯度[33];同时完善了SDS提取液配方,即在提取液中添加β-巯基乙醇,可以防止酚氧化,有助于去除蛋白质[18];并用常见的涡旋仪代替原方法[19]中昂贵的核酸裂解仪,可供更多的实验室选用。
已有的R N A 提取方法评价主要集中于R N A 提取效率,如浓度及纯度、RT-P C R 检测等方面的比较[6,20-24,27,32,34],对微生物群落结构的比较研究较少。针对两种不同时间酒醅的微生物结构和组成进行了分析,结果发现,方法本身对特定微生物专一偏好性很小。两种酒醅中共计检测到3 个纲、7 个属,Trizol法偏好性地提取到1 个纲、1 个属,占所有原核微生物类群仅为0.01%,试剂盒法偏好性地提取到1 个属,占所有原核微生物类群也仅为0.01%。其中原因可能是3 种方法均采用了剧烈振荡破碎细胞的方法提取RNA,这一方法能将所有微生物细胞裂解并提取RNA[19],因此3 种方法本身的专一偏好性并不明显。同时发现3 种RNA提取方法所得微生物组成和相对丰度并不完全相同,但3 种方法均能检测到两种酒醅中的优势微生物类群,并且能反映这些优势微生物类群在两种酒醅中的变化规律。
4 结 论
本研究建立了一种改良的SDS-苯酚法,并比较了其与试剂盒法、Trizol法、CTAB法和月桂酸钠法提取浓香型白酒酒醅总RNA的效果,其中:1)SDS-苯酚法和CTAB法成本较低,试剂盒法耗时最短但成本最高;2)5 种总提取方法所得RNA质量浓度为:试剂盒法>SDS-苯酚法>Trizol法>月桂酸钠法>CTAB法;3)SDS-苯酚法和试剂盒法提取样品总RNA的纯度较高,且SDS-苯酚法电泳条带完整性优于其他方法;4)SDS-苯酚法、试剂盒法和Trizol法所提取的RNA能有效地用于反转录实验及高通量测序分析;5)RNA提取方法及样品特性均会对酒醅微生物群落结构及多样性解析的结果产生不同程度的影响,如SDS-苯酚法获得的微生物多样性明显高于其他两种方法,以及7 d和15 d酒醅间微生物类群存在差异,且SDS-苯酚法和Trizol法能较好地反映出这种差异。
综上所述,本研究系统地比较了5 种常见的总RNA提取方法在浓香型酒醅RNA提取的应用效果,其中改良的SDS-苯酚法具有一定的综合优势,为今后以RNA为基础的酒醅分子生物学研究提供了理论依据及方法借鉴。

