利用HRM功能标记检测黑龙江省水稻抗稻瘟病基因Pita和pik的基因型分布
郭震华 黄翠红 罗文龙 黄晓群 王瑞英 陈志强 关世武 张淑华 蔡丽君
摘要:为明确Pita及pik基因在黑龙江省水稻种质资源中的分布情况,本研究以67份黑龙江省主栽品种及优异种质资源作为试验材料,通过HRM技术,利用Pita及pik的HRM功能型分子标记Pita-G/T、Pik2-C/T,对67份材料进行基因分型。结果表明,67份种质中,含纯合Pita有27份,占参试种质的40.3%;纯合pik有22份,占参试种质的32.9%,同时含有两基因的为12份,比例为17.9%。综上,Pita-G/ T及Pik2-C/T标记可以高效准确地对黑龙江水稻资源进行基因分型,同时,通过基因分型,明确了Pita及pik在黑龙江省的分布情况,为抗稻瘟病育种提供了理论基础。
关键词:水稻;高分辨率熔解曲线;Pita基因;pik基因;基因分型
中图分类号:S326文献标志码:A论文编号:cjas20190900197
基金项目:国家重点研发计划“寒地早粳稻分子设计育种”(2016YFD0101801);黑龙江省农科院重大科研成果激励项目“Pita等抗稻瘟病基因在寒地稻区的分布研究”(2018KYJL001)。
Detection of the Rice Blast Resistance Genes Pita and pik in Heilongjiang: HRM Functional Markers
Guo Zhenhua1,2, Huang Cuihong2, Luo Wenlong2, Huang Xiaoqun1, Wang Ruiying1, Chen Zhiqiang2, Guan Shiwu1, Zhang Shuhua1, Cai Lijun3(1Rice Research Institute of Heilongjiang Academy of Agricultural Sciences, Jiamusi 154026, Heilongjiang, China;
2National Engineering Research Centre of Plant Space Breeding, Guangzhou 510642, Guangdong, China; 3Jiamusi Branches of Heilongjiang Academy of Agricultural Sciences, Jiamusi 154026, Heilongjiang, China)
Abstract: To clarify the distribution of Pita gene and pik gene in rice germplasm resources of Heilongjiang, 67 major varieties and excellent germplasm resources were used as materials with HRM (high resolution melting) technology. 67 materials were used for genotyping analysis by Pita-G/T and Pik2-C/T of Pita and pik HRMbased functional markers. The results showed that, among the 67 germplasm resources, 27 germplasm resources contained the homozygous gene Pita (40.3% of the total number), 22 germplasm resources contained the homozygous gene pik (32.9% of the total number) and 12 germplasm resources contained both of the two homozygous genes (17.9% of the total number). Based on the results, the functional markers Pita-G/T and Pik2-C/T could be efficiently and accurately used in rice genotyping in Heilongjiang, meanwhile, through genotyping the materials in this research, we clarified the distribution of those two genes in Heilongjiang, which provided a theoretical basis for rice blast resistance breeding.
Keywords: Rice; High Resolution Melting (HRM); Pita Gene; pik Gene; Genotyping
0引言
稻瘟病是目前影響水稻最为严重的一种病害,每年可造成水稻减产11%~30%[1],严重制约着水稻的高产、稳产。黑龙江省作为北方稻区的第一水稻大省,稻瘟病时有发生,危害严重,严重时可造成减产15%~ 20%。稻瘟病是由子囊菌Magnaporthe grisea(hebert) barr(无性世代:Pyricularia grisea (cooke) Sacc)引起的[2],在水稻整个生育期内及各个部位均可发生,其中尤以叶瘟影响最为严重。
水稻抗稻瘟病基因的分析最早在日本于20世纪60年代展开,共鉴定了最初的8个抗性位点上的14个基因(JDCs,Japanese differential cultivars)。截至2011年2月,多于75个抗稻瘟病的主效基因已被克隆。其中,包含Pia、Pib、Pita、Pi2、Piz-t、Pi9、Pid2等共18个基因已被成功克隆(China Rice Data Center , http:// www. ricedata.cn/gene/gene_pi.htm)。
Pita作为水稻中第2个被克隆的抗稻瘟病主效基因,位于水稻第12染色体的近着丝粒区域,是一个抗谱比较广的单拷贝显性基因,在抗病品种和感病品种中都有较低的组成型表达[3]。Jia等[4]利用Pita的DNA序列上的多态性,开发的DNA显性分子标记YL155/ YL87和YL183/YL87,成功用于Pita基因的鉴定。Pik基因位于水稻第11染色体长臂近末端区域,目前已发现含有5个等位基因,分别是Pik、Pikh、Pikm、Pikp和Piks[5],其中Pikm、Pikp和Pik己被克隆。此3个基因都为NBS-LRR类抗病基因,且各自均由2个相邻基因Pikm-1和Pikm-2;Pikp-1和Pikp-2;Pik-1和Pik-2共同作用表现出稻瘟病抗性[6-8]。
高分辨熔解曲线(high–resolution melting curve, HRM)技术作为近年来新兴的一种基因分型技术,通过配套的软硬件系统,可以快速自动监测DNA熔解曲线的变化,实现基因分型,分辨率可达单碱基差异,对SSR、SNP及Indel等不同类型变异有很好的区分效果[9]。由于不需要凝胶电泳等步骤,从而真正实现了闭管操作[10],避免污染的可能。该方法目前已在玉米[11]、大麦[12]及水稻[13]等农作物中得到很好的应用。罗文龙等[14]针对Pita基因中的G/T SNP位点及Pik、Pikm、Pikp基因中共同存在的C/T SNP,分别开发设计得到Pita及Pik的功能型HRM标记Pita-G/T、Pik2-C/ T,已经在水稻种质基因筛选中得到了很好的应用。笔者拟利用与抗稻瘟病基因Pik和Pita紧密连锁的HRM分子标记Pita-G/T、Pik2-C/T[14],对黑龙江水稻种质资源进行基因分型,以期明确Pik和Pita基因在黑龙江水稻中的分布情况,为抗稻瘟病品种的合理利用及选育提供参考,为寒地水稻分子标记辅助选择育种(MAS)提供物质保障和技术支持。
1材料与方法
1.1材料
选取67份黑龙江省水稻主栽品种及优异种质资源为本研究供试水稻材料(见表1),由黑龙江省农业科学院佳木斯水稻研究所提供,于2014年种植于黑龙江省农业科学院佳木斯水稻研究所试验地中。
1.2方法
1.2.1水稻基因组DNA提取于水稻苗期对每份水稻材料分别随机选取3株的新鲜叶片,等量混合,按照文献[15]中使用的方法进行DNA的提取。
1.2.2用于HRM分析的多重巢式PCR扩增多重巢式PCR共包括以下两轮扩增反应:
第一轮PCR:2×Power Taq PCR Master Mix母液5μL,外引物F/R (10μmoL/L)各0.3μL,基因组DNA 0.5μL,总体积10μL,ddH2O补足。PCR程序:95℃预变性5 min,35个循环95℃变性20 s,57℃退火20 s,72℃延伸10 s;最后72℃延伸5 min。扩增程序在Gene-Amp9700型PCR仪(Applied Biosystems, USA)上進行。
反应结束后PCR管加200μL双蒸水,稀释第一轮产物。
第二轮PCR:2×Power Taq PCR Master Mix母液4μL,内引物F/R (10μmoL/L)各0.3μL,第一轮稀释PCR产物0.5μL,20×EvaGreen染料(Biotium, USA) 0.5μL,内外标样各1μL,最后ddH2O补足10μL。扩增程序在Gene- Amp9700型PCR仪(Applied Biosystems, USA)上进行。PCR程序:95℃预变性2 min,22个循环95℃变性20 s,59℃退火20 s,72℃延伸10 s;最后95℃延伸2 min。
1.2.3利用LightScanner96系统HRM分析第二轮
PCR产物中加入20μL矿物油(SIGMA公司),将PCR产物离心转移至HRM检测板中,2000 r/min离心2 min,检查无气泡,放入LightScanner96高分辨率熔解曲线分析系统(Idaho Technology Inc, USA)进行HRM检测,起点温度为60℃,终点温度为95℃,平均15 min一板PCR反应。反应完成后通过配套的LightScanner Call-IT软件进行基因型分析,选用Small Amplicon模式进行自动化分析;利用LightScanner分析系统进行HRM检测。
用于PCR扩增反应的引物名称、序列、位置及片段大小等详细信息见表2。
2结果与分析
本研究即利用这2对功能型标记,对黑龙江水稻种质资源进行基因分型。通过两轮的PCR反应,扩增终产物由于DNA双链上突变位点的退火温度(Tm值)不同,产生不同的峰值,从而区分不同基因型。
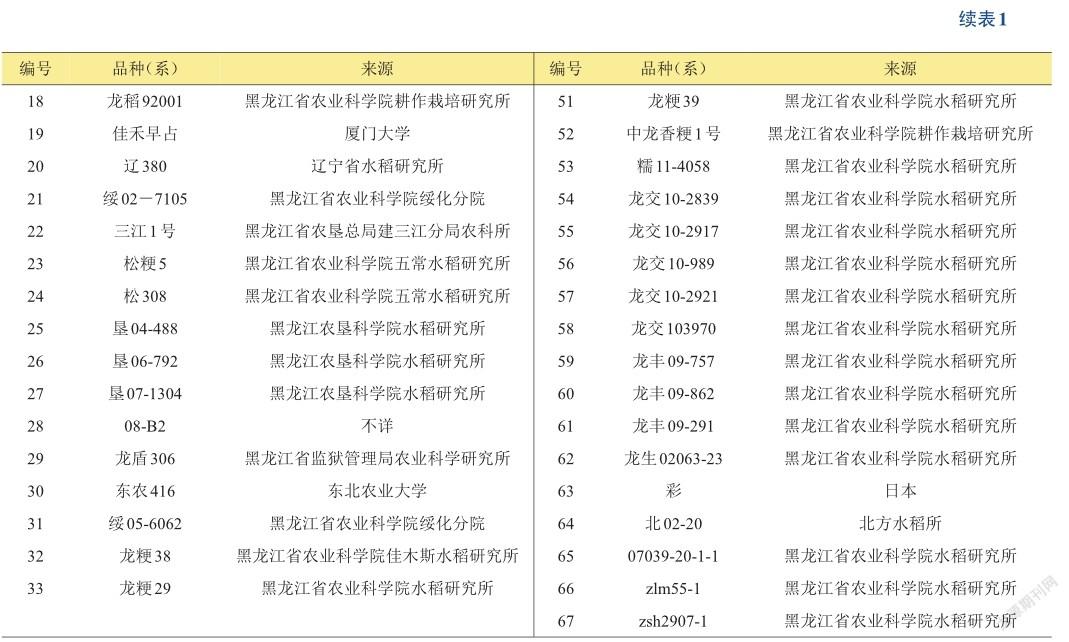
2.1 Pita在黑龙江水稻中的分布情况
Pita基因检测结果如图1所示,灰色曲线为碱基为G的稻瘟病感性基因pita,而红色曲线为碱基为T的稻瘟病抗性基因Pita。可以看出,Pita因T碱基氢键多于pita的G碱基,其退火温度(Tm值)高于pita的,从而产生不同的峰值,区分不同基因型。根据不同材料出现的先后顺序,确定出共有27份种质含有纯合的抗稻瘟病基因Pita,占参试种质的40.3%,分别为‘龙粳12’、‘龙粳18’、‘五稻8’、‘哈99352’、‘哈99774’、‘龙盾D904’、‘龙稻92001’、‘佳禾早占’、‘松粳5’、‘垦04-488’、‘龙盾306’、‘绥05-6062’、‘龙粳29’、‘北粳0907’、‘龙粳31’、‘龙粳32’、‘龙粳香1号’、‘龙联1号’、‘龙花07-211’、‘龙花04-350’、‘龙粳39’、‘龙交10-2917’、‘龙交10-989’、‘龙交103970’、‘龙丰09-291’、‘龙生02063-23’、‘zlm55-1’。其余40份为纯合的pita基因。可见,Pita抗稻瘟病基因在黑龙江省有着较为广泛的分布,对黑龙江省抗稻瘟病育种有着重要的作用。
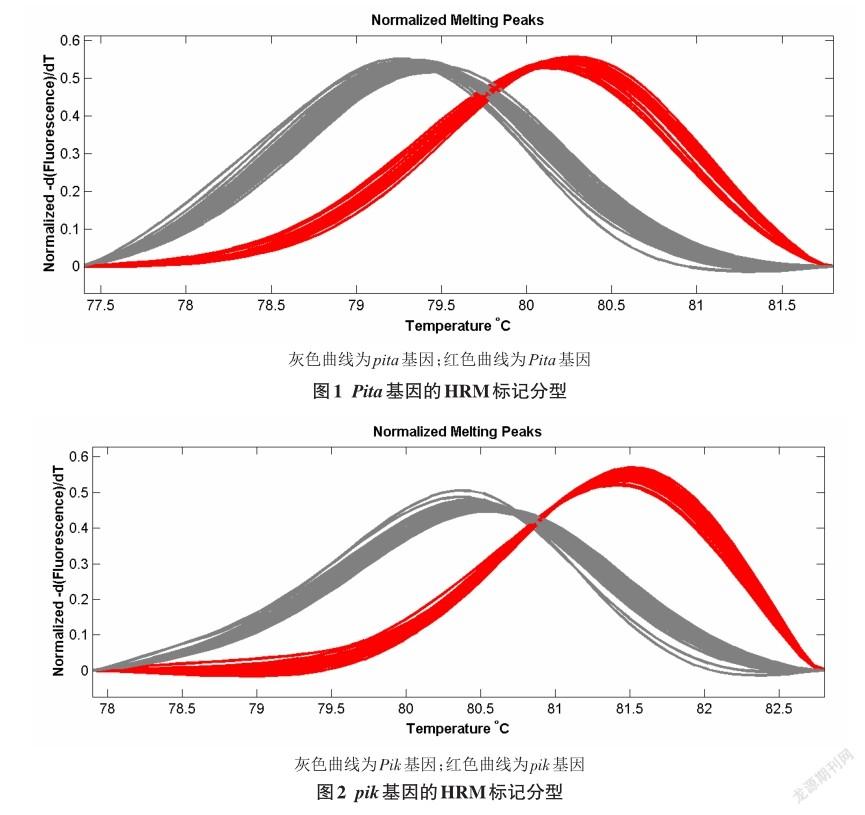
2.2 pik在黑龙江水稻中的分布情况
灰色曲线表示稻瘟病感性基因Pik的DNA熔解曲线,红色曲线为稻瘟病抗性基因pik的DNA熔解曲线。Pik由于含有C碱基位点,而pik含有T碱基,所以Pik的Tm值低于pik。结果表明,67份种质中,22份种质含有纯合的抗稻瘟病基因pik,比例为32.9%,其余45份含纯合的Pik基因。此22份种质分别为,‘龙粳18’、‘垦94437’、‘五稻8’、‘哈99352’、‘哈99774’、‘五优稻’、‘佳禾早占’、‘辽380’、‘松粳5’、‘绥05-6062’、‘龙粳38’、‘龙粳29’、‘空育139’、‘龙粳香1号’、‘龙花07-211’、‘中龙香粳1号’、‘糯11-4058’、‘龙交10-989’、‘龙交10-2921’、‘龙丰09-757’、‘龙丰09-291’、‘zsh2907-1’。可见,pik抗稻瘟病基因在黑龙江省分布也较为广泛,同样可以很好地应用于稻瘟病抗性育种中。

2.3 Pita及pik在黑龙江水稻中共同的分布情况
通过上述分析可以得出,‘龙粳18’、‘五稻8’、‘哈99352’、‘哈99774’、‘佳禾早占’、‘松粳5’、‘绥05-6062’、‘龙粳29’、‘龙粳香1号’、‘龙花07-211’、‘龙交10-989’及‘龙丰09-291’共12份种质同时含有Pita及pik两种稻瘟病抗性基因,占参试种质的17.9%。
3结论与讨论
基于黑龙江省稻瘟病害频发,且危害性大等特点,水稻抗稻瘟病育种一直以来都是研究的热点。而目前多数的分子标记辅助育种多集中在常规PCR手段上,而常规PCR技术需要通过凝胶电泳等步骤,实验步骤多,费时费力,效率偏低。而且辅助选择主要依赖于与目标基因连锁的标记进行,存在假阳性的概率较大。随着分子标记辅助选择在育种中的广泛应用,各类分子标记不断出现,而最为准确的是基于抗性基因本身的序列差异开发的分子标记,称为功能标记(FMs),能够直接反映目标性状的表型,有效避免由于重组引起的遗传信息的丢失,对FMs的筛选即能对性状进行筛选[16-18]。HRM技术与常规PCR技术区别主要在于不再需要凝胶电泳步骤,直接对PCR产物在管中进行分析,减少实验步骤,大大缩短实验时间,提高实验效率,同时降低了实验可能存在的污染率。利用该技术开发的功能标记已在玉米[11]、小麦[19]、大麦[12]及水稻[13]等农作物中都已成功应用。因此,本研究所用到的Pita-G/ T及Pik2-C/T标记是针对基因本身内部SNP位点,利用HRM技术开发得到的,一方面作为功能基因标记,可以准确的发现种质间存在的基因差异,同时,基于HRM技术,又实现了高效快速的优点,可以进行大规模的基因分型及筛查,大大提高了基因检测及标记辅助选择的效率。
针对水稻品种稻瘟病抗性而言,单个基因的抗性可能会随着病原菌生理小种的变化在几年内逐渐丧失,因此通过聚合育种的方法将多个稻瘟病抗性基因导入同一品种中,即可抵抗多种生理小种的侵害,从而达到广谱抗性的目的。本研究中‘龙粳29’、‘龙粳香1号’、‘龙花07-211’等12份种质同时聚合Pita及pik抗性基因,对稻瘟病各生理小种有着较为广泛的抗性,为抗稻瘟病育种研究提供了有利的种质资源。而本研究中所用的资源材料相对仍偏少,为了更好的鉴定分析Pita及pik在黑龙江水稻中的分布情况,需扩大鉴定材料的范围,更加广泛的搜集黑龙江水稻种质资源材料,丰富和完善Pita及pik在黑龙江水稻中的分布情况。
目前已定位的主效抗稻瘟病基因中,一半以上是以基因簇的形式存在于水稻各染色体上。Pita基因作为已克隆的显性单拷贝基因,与Pita2等许多其他抗病基因紧密连锁,使得该基因具有广谱抗性,在水稻育种研究及生产上有着较高的实用价值[20]。而目前已知的在Pik基因位点上有5个等位基因,广泛分布在水稻品种中,具有良好的广谱抗性。而本研究中通过2个功能性分子标记Pita-G/T及Pik2-C/T对黑龙江部分水稻主栽品种及种质资源展开基因分型,虽然明确了2个基因在这些材料中的分布情况,但仍需明确细化各基因的等位基因的具体分布情况。此外,在基因分型的同时,与之相对应的稻瘟病抗性的表型也需要调查,以此与基因型进行相互印证。
黑龙江省作为北方稻区水稻第一大省,稻瘟病一直以来是严重影响水稻产量的第一大病害。对部分黑龙江主栽品种及优异种质资源的Pita及pik基因的分型鉴定,结果表明,67份种质中,含纯合Pita有27份,占参试种质的40.3%;纯合pik有22份,占参试种质的32.9%,同时含有两基因的为12份,分别为龙粳18’、‘五稻8’、‘哈99352’、‘哈99774’、‘佳禾早占’、‘松粳5’、‘绥05-6062’、‘龙粳29’、‘龙粳香1号’、‘龙花07-211’、‘龙交10- 989’及‘龙丰09- 291’,占参试材料的17.9%。可见,Pita及pik基因在黑龍江分布较广,对抗稻瘟病品种的选育及抗病基因的合理利用有着重要的参考价值。
参考文献
[1]刘占领,雷财林,程治军,等.水稻稻瘟病抗性基因定位与克隆研究进展[J].作物杂志,2007(3):16-19.
[2]Couch B C. Hohn L M. Amultilocus gene genealogy concordant with host preference indicates segregation of a new species[J]. Magnaporthe oryzae. Mycol-ogia, 2002,94:683-693.
[3]Hittalmani S, Parco A, Mew T V, et al. Fine mapping and DNA marker- assisted pyramiding of the three major genes for blast resistance in rice[J]. Theoretical and Applied Genetics, 2000,100(7): 1121-1128.
[4]Jia Y, Mc Adams S A, Bryan G T, et al. Direct interaction of resistance gene and a virulence gene products confers rice blast resistance[J].The EMBO Journal, 2000,19(15):4004-4014.
[5]Stefano Costanzo, Jia Y L. Sequence variation at the rice blast resistance gene Pikm locus: Implications for the development of allele specific markers [J]. Plant Science, 2010,178:523-530.
[6]Ashikawa I, Hayashi N, Yamane H, et al. Two adjacent nucleotidebinding site Leucine-rich repeat class genes are required to confer Pikm specific rice blast resistance [J]. Genetics, 2008,180:2267-2276.
[7]Yuan B, Zhai C, Wang W J, et al. The Pikp resistance to Magnaporthe oryzae in rice is mediated by a pair of closely linked CC-NBS-LRR genes[J].Theoretical and Applied Genetics, 2011,122(5):1017-1028.
[8]Zhai C, Lin F, Dong Z Q, et al. The isolation and characterization of Pik, a rice blast resistance gene which emerged after rice domestication[J]. New Phytologist, 2011,189(1):321-334.
[9]De Koeyer D, Douglass K, Murphy A, et al. Application of highresolution DNA melting for genotyping and variant scanning of diploid and autotetraploid potato [J]. Molecular Breeding, 2010,25(1):67-90.
[10]Vossen R, Aten E, Roos A, et al. High-Resolution melting analysis(HRMA)- More than just sequence variant screening [J]. Human Mutation, 2009, 30(6): 860-866.
[11]Li Y D, Chu Z Z, Liu X G, et al. A cost-effective high-resolution melting approach using the EvaGreen dye for DNA polymorphism detecting and genotyping in plants [J]. Journal of Intergrative Plant Biology, 2010,52(12):1036-1042.
[12]Hofinger B J, Jing H C, Hammond- Kosack K E, et al. Highresolution melting analysis of cDNA-derived PCR amplicons for rapid and cost-effective identification of novel alleles in barley [J]. Theoretical andApplied Genetics, 2009,119(5):851-865.
[13]赵均良,张少红,刘斌.应用高分辨率熔解曲线技术分析水稻分子标记基因型[J].中国农业科学,2011,44(18):3701-3708.
[14]Luo W L, Guo T, Yang Q Y, et al. Stacking of five favorable alleles for amylase content, fragrance and disease resistance into elite lines in rice (Oryza sativa) by using four HRM- based markers and a linked gel-based marker[J]. Molecular Breeding, 2014,34:805-815.
[15]McCouch S R, Kochert G, Yu Z H, et al. Molecular mapping of rice chromosomes[J]. Theoretical and Applied Genetics, 1988,76:815-820.
[16]Bagge M, Xia X C, Lübberstedt T. Functional markers in wheat [J]. Current Opinion in Plant Biology, 2007,10:211-216.
[17]王豐,李金华,柳武革,等.一种水稻香味基因功能标记的开发[J].中国水稻科学, 2008,22(4):347-352.
[18]Iyer- Pascuzzi A S, McCouch S R. Functional markers for xa5-mediated resistance in rice (Oryza sativa L.) [J]. Molecular Breeding, 2007,19:291-296.
[19]Rakszegi M, Kisgyorgy B N, Tearall K, et al. Diversity of agronomic and morphological traits in a mutant population of bread wheat studied in the Health grain program [J]. Euphytica, 2010,174(3):409-421.
[20]时克,雷财林,程志军,等.稻瘟病抗性基因Pita和Pib在我国水稻主栽品种中的分布[J].植物遗传资源学报,2009,10(1):21-26.

