葡萄叶猕猴桃(猕猴桃科)叶绿体全基因组测序
滕晓梅 廖川江 刘畅 徐雨生
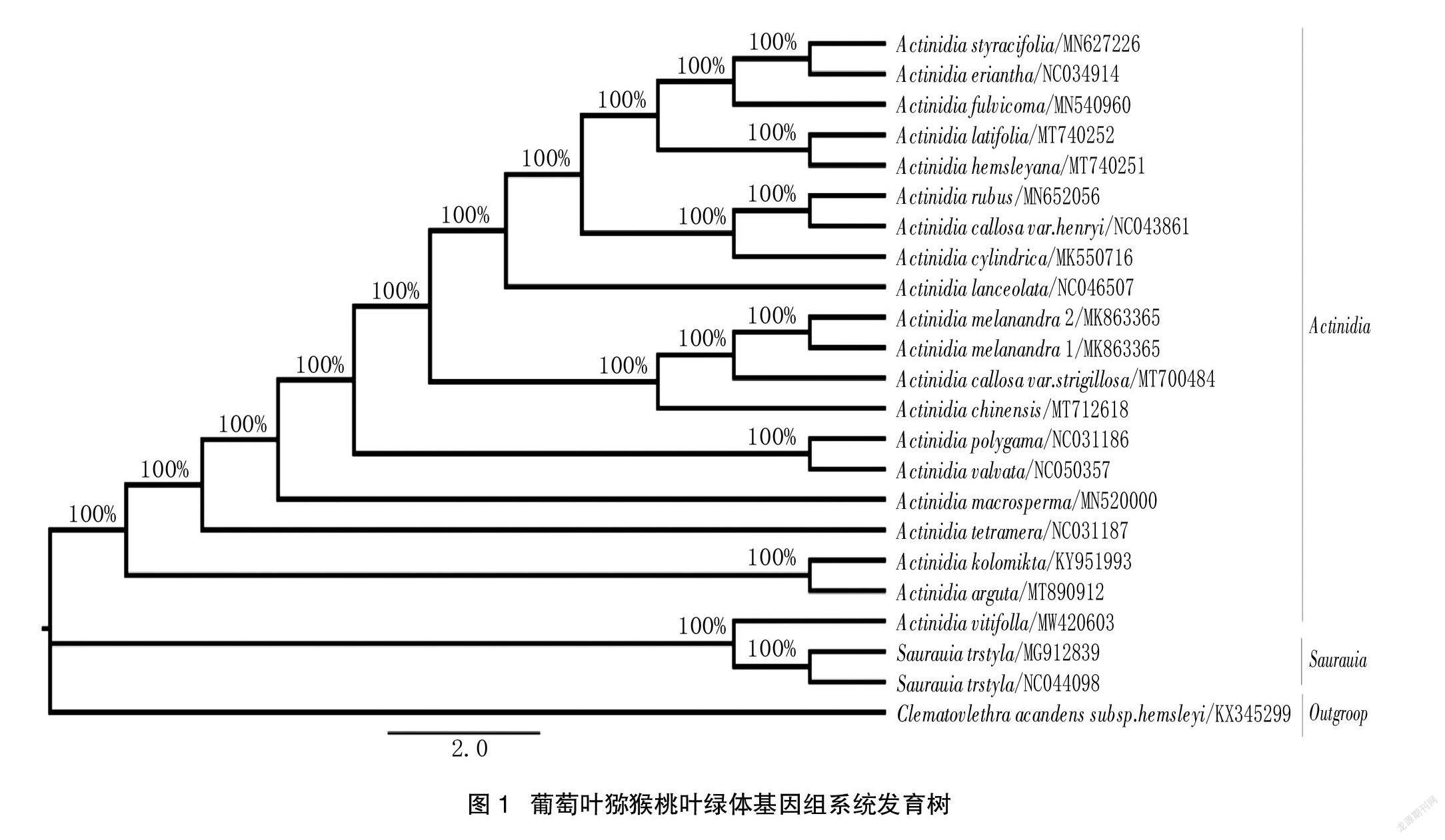
摘 要:葡萄叶猕猴桃(猕猴桃科)分布狭窄且现已濒临灭绝,本研究完成了对葡萄叶猕猴桃叶绿体全基因组的排序。结果显示,葡萄叶猕猴桃叶绿体全基因组长度为150 878 bp,其基因组包括130个基因;系统发育树结果表明,葡萄叶猕猴桃与水东哥为姊妹类群。
关键词:葡萄叶猕猴桃;濒危物种;叶绿体全基因组测序;系统发育树
文章编号:1005-2690(2021)21-0023-03 中国图书分类号:Q943.2;S663.4 文献标志码:B
随着现代分子生物学技术的不断进步,在植物分子系统学领域中,叶绿体基因组(Chloroplast DNA,
cpDNA)测序的方法已成为植物系统发育研究的最重要方法之一[1],在物种进化及鉴定中具有重要使用价值。绝大部分叶绿体基因组大小为120~250 kb,呈共价闭合环状的双链结构且由4个区域组成:一个大单拷贝序列区(Large Single-Copy region,LSC)、一个小单拷贝序列区(Small Single-Copy region,SSC)及一对反向重复序列区(Inverted Repeat region,IR)[2]。与核基因组比较,叶绿体基因组序列具有分子量小、结构稳定且高度保守等优点,在胞质遗传、植物系统发育和遗传多样性等方面发挥着重要作用[3]。
葡萄叶猕猴桃(Actinidia vitifolia)是猕猴桃科、猕猴桃属大型落叶藤本,主要分布于中国四川马边、雷波峨边、峨边和云南彝良等地,生长于海拔1 600 m[4]。葡萄叶猕猴桃现已属濒危物种,果实的风味甚佳;含有苯丙氨酸、亮氨酸、异亮氨酸等10多种氨基酸以及丰富的矿物质(钙、磷、铁),对保持人体健康具有重要的作用;含有较多的果酸,能够显著抑制黑色素沉淀及角质细胞内聚力,具有消除或淡化黑斑、改善干性或油性肌肤等功效,被称为“美容圣果”[5-6]。
猕猴桃属先被分别置于山茶科(Theacea)或五桠果科(Dilleniaceae),Dunn(1911)[7]首次提出,应将称猕猴桃属(Actinidia Lindl)、水东哥属(Saurauia Willd)、藤山柳属(Clematoclethra Maxim)分别从五桠果科或山茶科分出放在一起的设想。Gilg(1925)[8]建立了包括这3个近缘属的猕猴桃科。本研究对葡萄叶猕猴桃叶绿体全基因组进行排序,挑选获得猕猴桃属19种、水东哥属1种,同时挑选刚毛藤山柳作为外类群建立系统发育树,为更好地开发利用及保护葡萄叶猕猴桃提供了参考依据,同时为遗传学和系统发育学研究奠定了试验基础。
1 材料与方法
1.1 材料
葡萄叶猕猴桃样本叶采自中国云南省太平镇(28°24′40.76″ E,104°7′50.86″ N),其凭证标本保存于中国科学院昆明植物研究所标本馆。
1.2 仪器与试剂
移液枪(济南欧莱博技术有限公司,型号为F3系列);水浴锅(上海左乐仪器有限公司),型号为HH-W420;超微量紫外分光光度计(Nano Drop 2000)(上海旦鼎国际贸易有限公司),型号为Anodrop one-c;高通量组织研磨仪(上海达洛科学仪器有限公司),型号为Daxluot-24;冰箱(济南童鑫生物科技有限公司),型号为BDF-25H110;干燥器,型号为2892;2×CTAB缓冲液(上海尚宝生物科技有限公司),型号为R21954-500;氯仿(分析级),异戊醇(分析级)。
1.3 试验方法
1.3.1 DNA提取
在2 mL离心管(加入一颗钢珠)中装入约0.2 g待提材料叶片,将离心管转移至离心管速冻金属架并放入液氮中速冻2~3 min后,在高通量组织研磨仪中以45 Hz震荡破碎50 s。分別加入600 μL预热的2×CTAB缓冲液(65 ℃的水浴锅中预热),颠倒混匀后,将离心管放入65 ℃的水浴锅中温浴30 min。在离心管中加入600 μL的试剂氯仿和异戊醇(体积比为24∶1),颠倒混匀后, 12 000 rpm离心10 min。在通风橱中,将400 μL上清液转移至1.5 mL灭菌离心管中后加入冰冷的异丙醇400 μL,作好标记,放入-20 ℃冰箱中冰置30 min沉淀DNA。然后,12 000 rpm离心10 min,倒掉上清液。将离心管倒扣在吸水纸上,放置4 h使残余的异丙醇挥发后,在离心管中加入200 μL ddH2O,室温溶解4 h后,随机选择10个DNA样品利用Nano Drop 2000检测DNA样品的浓度和纯度,将检测合格的DNA样品储存于-20 ℃备用(样品DNA的OD260/OD280值在1.8左右)。
1.3.2 测序
将用硅胶干燥的叶片(合格样品)送至诺和生物进行测序。采用Illumina测序方法,该公司会返回原始数据(Raw data)与进行质控过滤后的数据(二代测序数据,Clean data),其后基于Clean data对其进行叶绿体基因组的组装。
1.3.3 数据采集
在iPlant植物智挑选获得猕猴桃属19种、水东哥属1种,再通过NCBI下载其完整叶绿体基因组序列(FASTA文件),同时挑选刚毛藤山柳作为外类群。
1.3.4 叶绿体基因组的组装和注释
采用叶绿体基因组组装软件(NOVOPlasty v3.1)[9]组装完整的叶绿体基因组。所得的单个文件碎片整理程序(contigs)被鉴定并定位到参考基因组中华猕猴桃(Actinidia chinensis)(NC026690)进行进一步核查。通过CpGAVAS2[10]和GeSeq[11]对组装的叶绿体基因组进行注释。使用生物信息学软件(Geneious v.9.0.2)[12]对起始和终止密码子进行手动校正。将正确无误的葡萄叶猕猴桃的完整叶绿体基因组序列提交至GenBank(MW420603)。
1.3.5 系统发育分析方法
基于每个基因组的基因位置获得基因顺序数据,利用RAxML8.0构建最大似然系统发育树(Maximum Likelihood estimator,ML),计算支持率时,将物种数据集计算1 000次重复[13]。
2 试验结果
2.1 葡萄叶猕猴桃叶绿体基因组特征
葡萄叶猕猴桃叶绿体基因是一个典型的四方体结构,完整叶绿体基因组长度为150 878 bp,其中大单拷贝(LSC)区长度为82 020 bp,小单拷贝(SSC)区长度为17 563 bp,一对反向重复区(IRs)长度为2 547 bp,总GC含量为38.5%。葡萄叶猕猴桃的叶绿体基因组包括130个基因,其中包括83个蛋白质编码基因、8个核糖体RNA(rRNA)基因和39个转运RNA(tRNA)基因。
2.2 系统发育结果
为了确定葡萄叶猕猴桃的系统发育关系,采用猕猴桃属完整叶绿体基因组(19个序列)、水东哥属的水东哥(2个序列)和作为外类群的藤山柳属刚毛藤山柳(1个序列)。系统发育树结果如图1,葡萄叶猕猴桃与水东哥为姊妹类群。
3 讨论
叶绿体为光合作用必不可少而存在于所有藻类微生物和绿色植物细胞中,且具有基因组结构较稳定、遗传物质相对独立等较多优势[14],因此叶绿体基因组非常适合进化学和系统学研究。cpDNA在植物总DNA中的含量相当丰富,且具有多拷贝、分子量小和结构简单的特点,易于提取和分析。植物叶绿体基因组全序列检测结果可以为其提供广泛且具体的分子信息,有利于特定基因的分离、鉴定、测定及构建物理图谱[15-16]。
中国葡萄叶猕猴桃的分布区域非常狭窄,该物种已濒临灭绝,个体数量已不足1 000株。本研究采用Illumina测序及NOVOPlasty v3.1对葡萄叶猕猴桃的叶绿体全基因组进行测序,并以发表的中华猕猴桃序列為参考组装出葡萄叶猕猴桃叶绿体基因组。与传统方法相比,该方法操作较为简单,不需要单独分离叶绿体和cpDNA,只需提取植物叶片全基因组DNA进行测序。通过与参考基因组比较后,将得到的序列结果与参考基因组Blast对比,即可轻松找出有效的reads,再用相对应的软件进行组装,并对组装结果进行补洞从而获得完整的全叶绿体基因组。该方法不仅操作简单、费用较低,而且提高了试验的可行性。通过对葡萄叶猕猴桃叶绿体基因组进行测序并对其进行特征分析,共注释得到130个基因,其中包括83个蛋白质编码基因、8个rRNA基因和39个tRNA基因;蛋白编码基因对应的密码子偏好使用A/T碱基。
试验结果为后期葡萄叶猕猴桃叶绿体分子标记的开发提供了充分的试验资料和参考价值。对葡萄叶猕猴桃叶绿体基因组的研究,有助于更好地研究猕猴桃科的系统发育和生物地理学,同时有助于进一步开展猕猴桃科植物杂交种质鉴定和基于叶绿体DNA数据的群体遗传学研究。
参考文献:
[1]李作洲.猕猴桃属植物的分子系统学研究[D].北京:中国科学院研究生院(武汉植物园),2006.
[2]Jansen R K,Ruhlman T A.Plastid genomes of seed plants[M].Dordrecht:Springer Netherlands,2012:103.
[3]付涛,王志龙,钱萍仙,等.高等植物DNA条形码最新研究进展及其应用[J].核农学报,2016,30(5):887-896.
[4]中国科学院中国植物志编辑委员会.中国植物志(第49卷)第一分册[M].北京:科学出版社,1999.
[5]李伟,严友兵,张永康,等.猕猴桃籽油护肤品的开发利用[J].中国油脂,2020,45(4):119-122.
[6]刘清清.猕猴桃籽油的体外抗氧化活性及其对高脂诱导小鼠肥胖的缓解作用研究[D].西安:西北大学,2018.
[7]Dunn S T.A revision of the genus Actinidia Lindl[J].Journal of the Linnean society of London.Botany,1911(39):390-410.
[8]Gilg E,Werdermann E. InEngler A,Prantl K.,et al.Die naturlichen Pflanzenfamilen 2nd reved Lepzig:Verlag Wilhelm Engelma[J].Actinidiaceae,1925(21):36-47.
[9]Dierckxsens N M P,Smits G.De novo assembly of organelle genomes from whole genome data[J].Nucleic Acids Res,2017,
45(4):18.
[10]Shi L C H,Jiang M,Wang L,et al.An integrated plastome sequence annotator and analyzer[J].Nucleic Acids Res,2019(47):65-73.
[11]Tillich M L P,Pellizzer T,Ulbricht-Jones Es,et al.Versatile and accurate annotation of organelle genomes[J].Nucleic Acids Res,2017(45):6-11.
[12]Kearse M M R,Stones-Havas S,Cheung M,et al.An integrated and extendable desktop software platform for the organization and analysis of sequence data[J].Bioinformatics,2012,28(12):1647-1649.
[13]A S.A tool for phylogenetic analysis and post-analysis of large phylogenies[J].Bioinformatics,2014(30):1312-1313.
[14]程慧,葛春峰,张红,等.果树叶绿体基因组测序及系统发育研究进展[J].核农学报,2018,32(1):58-69.
[15]李健仔,李思光,罗玉萍,等.叶绿体DNA分析技术及其在植物系统学研究中的应用[J].江西科学,2002(3):183-189.
[16]Lewis L A,McCourt R M.Green algae and the origin of land plants[J].American Journal of Botany,2004(91):1535-1556.

