植物细胞器基因编辑研究进展
武志强 周家伟

摘 要:基因编辑(gene editing),又称基因组编辑或基因组工程,是一种以插入、删除或碱基替换的形式引起DNA序列突变的技术。基因编辑技术有多种类型,如锌指核酸酶技术(zinc-finger nucleases,ZFNs)、转录激活因子样效应物核酸酶技术(transcription activator-like effector nucleases,TALENs),以及近几年迅速发展起来的规律成簇短回文重复序列及其核酸酶9技术(clustered regularly interspaced short palindromic repeats/CRISPR-associated nuclease 9,CRISPR/Cas9)。基因編辑技术的出现加速了植物功能基因组学的发展,并将在作物精准育种方面具有巨大的开发潜力。植物细胞器基因编辑主要是指对植物线粒体和叶绿体基因组的编辑。植物线粒体和叶绿体分别被称为细胞的“动力工厂”和“生产车间”,对于细胞及整个植株的生长发育及各种生命活动具有非常重要的作用。对线粒体和叶绿体基因组进行编辑将有利于了解其遗传规律从而开发它们在作物改良和工业生产上的应用。目前,细胞器基因编辑技术已经开始崭露头角,具有非常广阔的应用前景。该文将从基因编辑技术的发展、植物细胞器基因组的结构和特点、线粒体基因编辑和叶绿体遗传转化等方面进行总结,并对植物细胞器基因编辑的研究前景进行展望。
关键词:基因编辑,TALENs,细胞器基因组,线粒体基因编辑,叶绿体遗传转化
中图分类号:Q943
文献标识码:A
文章编号:1000-3142(2021)10-1654-11
Abstract:Gene editing,also known as genome editing or genome engineering,is a technique that introduces mutations in DNA sequences in the form of insertion,deletion,or base substitution. There are many types of gene editing techniques,such as zinc-finger nucleases (ZFNs),transcription activator-like effector nucleases (TALENs) and clustered regularly interspaced short palindromic repeats/CRISPR-associated nuclease 9(CRISPR/Cas9). CRISPR/Cas9 has developed rapidly in recent years. The emergence of gene editing techniques has accelerated the development of plant functional genomics and has great potential in precision crop breeding. Plant organelle gene editing mainly refers to editing plant mitochondrial and chloroplast genomes. Plant mitochondrion and chloroplast are often referred to as the “power house” and “production workshop”,respectively,due to their importance in central metabolic functions. Editing mitochondrial and chloroplast genomes will improve the understanding of the genetic function of these genomes and develop their applications in crop improvement and industrial production. At present,organelle gene editing techniques have emerged and have a very broad application prospect. In this review,we summarized the development of gene editing techniques,structures and characteristics of plant organelle genomes,mitochondrial gene editing,and chloroplast genetic transformation,and finally we proposed the future research directions and prospects of organelle gene editing.
Key words:gene editing,TALENs,organelle genome,mitochondrial gene editing,chloroplast genetic transformation
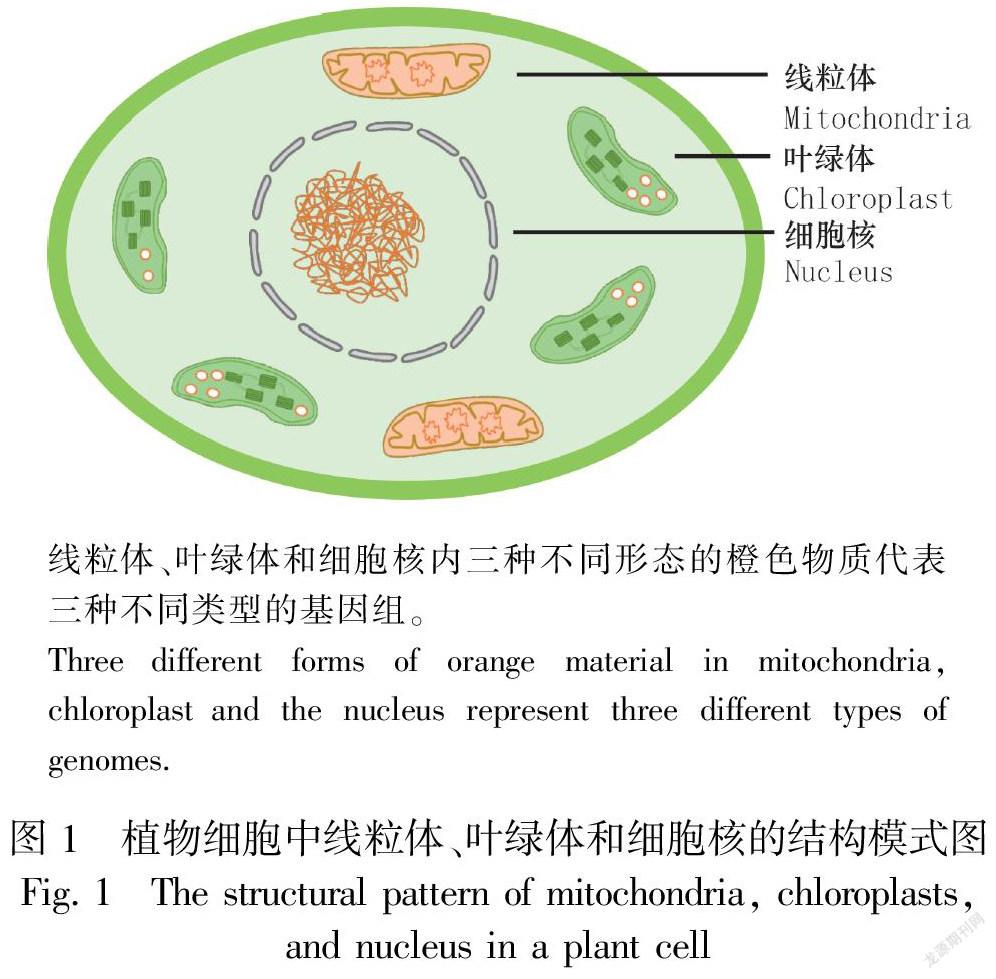
植物细胞内拥有三套独立的基因组——细胞核基因组、线粒体基因组和叶绿体基因组(图1)。除去拥有巨大遗传信息的细胞核之外,线粒体和叶绿体是植物细胞中具有双膜系统的细胞器,它们的发育和增殖是受细胞核基因组及其自身基因组两套遗传系统的控制,称为半自主性细胞器。细胞核与线粒体和叶绿体之间存在着密切的、精准的和严格调控的生物学机制。线粒体和叶绿体分别作为细胞中有氧呼吸和光合作用的场所,对细胞生命活动的正常进行至关重要。因此,了解它们的基因功能和生命过程机制,如复制、遗传、基因表达和基因组维持等对于进一步探索植物生命活动的奥秘和作物遗传改良等研究意义重大(Kazama et al.,2019)。近年来,利用基因编辑技术对植物细胞核基因组的修饰取得了显著的进展,已经成功应用在拟南芥、烟草、水稻、玉米、棉花、小麦、油菜、大豆、高粱、西瓜、葡萄、苹果、香蕉、橙子、西红柿、牵牛花、莴苣、木薯等众多植物中,并取得了相应的研究成果(Manghwar et al.,2019)。而植物细胞器基因编辑方面的研究却相对缓慢,目前仅在水稻、油菜、拟南芥、烟草、莴苣等植物中有相关报道。细胞器基因编辑技术的发展虽然面临许多严峻的挑战,但是在近几年取得了一些技术上的突破。本文将对植物细胞器基因编辑研究进展进行全面的综述。
1 基因编辑技术的发展
1.1 ZFNs基因编辑技术
ZFNs是第一代基因编辑技术,是由锌指蛋白的锌指DNA结合结构域与FokI内切酶的剪切结构域融合而成的限制性内切酶(图2:A)(Carroll,2011)。锌指(zinc-finger,ZF)是一种DNA结合基序,在许多识别DNA真核转录因子的DNA结合域发现它的存在(Khalil,2020)。FokI是源于细菌的ⅡS限制性内切酶(Li et al.,1992),其剪切结构域并没有明显的序列特异性(Carroll,2011)。当DNA结合域和DNA剪切域融合在一起时,就会形成一对高度特异性的“分子剪刀”而造成DNA序列的双链断裂(double strand break,DSB)。而造成的DSB 通常是通过同源介导的修复(homology-directed repair,HDR)和非同源末端连接(non-homologous end joining,NHEJ)的方式进行修复,由于修复过程容易发生错误进而导致基因突变的发生。
最常用的锌指结构是一组Cys2His2锌指,该结构中每30个左右的氨基酸结合一个锌原子,锌指DNA结合域通常包含3个独立的锌指结构,每个锌指结构能识别3个碱基,即一个锌指结构域能识别9 bp长度的DNA特异性序列(Carroll,2011)。由于FokI只有形成二聚体时才能行使剪切作用(Smith et al.,2000),所以一对ZFNs包括6个锌指结构,可以识别18 bp长的DNA特异性序列(Carroll,2011)。有研究表明,增加锌指的数量可以提高ZFNs的特异性和效率并改善靶向性,对锌指的定制组装可以靶向不同的DNA序列(Khalil,2020)。在农作物中,ZFNs诱导内源基因突变的第一个例子是在玉米细胞中,通过表达工程化的ZFNs靶向插入破坏玉米的IPK1基因,产生具有除草剂抗性和在种子发育过程中具有预期肌醇磷酸盐分布变化的基因编辑玉米植株(Shukla et al.,2009)。此外,对大豆(Curtin et al.,2011)、油菜、水稻、番茄、苹果和无花果(Peer et al.,2015)
及杂合杨树(Lu et al.,2016)等多个重要经济植物的细胞核基因也实现了定向突变。Bonawitz et al.(2019)利用ZFNs编辑技术通过NHEJ介导的多基因靶向插入获得了可育的转基因大豆植株。
1.2 TALENs基因编辑技术
TALENs是继ZFNs之后的第二代基因编辑技术,与ZFNs有许多相似之处:(1)TALENs也是一种人工限制性内切酶;(2)DNA结合域同样由多个模块组成;(3)也包含有FokI限制性内切酶。典型的TALENs系统是由N端的核定位信号(nuclear localization signal,NLS)、中间可识别并结合特异性DNA序列的TALE串联重复,以及C端FokI核酸组成(图2:B)(Khalil,2020)。TALE是由一种植物病原菌——黄单胞杆菌(Xanthomonas spp.)分泌产生的天然蛋白质(Gaj et al.,2013)。TALE的DNA识别结合结构域是一段长的串联重复序列,可以特异地识别并结合特异DNA序列。每个重复序列含有33~35个氨基酸残基,其中的第12位和第13位的氨基酸残基是识别特异DNA碱基的靶向关键位点,该位点随DNA序列的变化而变化,称作重复可变双残基(repeat variant diresidue,RVD),其他位置的氨基酸残基相对固定不变(Khalil,2020)。每个RVD都与一个特定的核苷酸结合:HD = C,NG = T,NI = A和NN = G(Khalil,2020)。重复单元经串联组装以后,就可以特异地识别一段DNA序列。根据这个原理,可以利用TALENs技术对植物基因进行定向特异性编辑。如Shinoyama et al.(2020)利用TALENs介导的基因编辑技术敲除菊花DMC1基因获得雌雄不育的菊花植株,这种产生完全不育植株的策略可广泛应用于防止转基因植株向野生近缘植物流动。TALENs比ZFNs对靶基因更具有序列特异性,更易于产生DSB和更小的细胞毒性,因此应用也更加广泛(Huo et al.,2019)。
1.3 CRISPR/Cas9基因编辑技术
CRISPR/Cas9技术是一种RNA引导的基因编辑技术,能对目标基因进行精确性的敲除、敲入、替换等,以实现探究基因功能、修复致病基因等目的(Gao,2021)。該技术凭借操作简便、价格低廉、可对基因位点进行编辑、可拓展性强等优势,在近几年迅速发展完善,成为继ZFNs和TALENs之后迅速发展起来的第三大基因编辑技术。瑞典皇家科学院将“2020年的诺贝尔化学奖”颁发给了开发出CRISPR/Cas9基因编辑技术的两位女科学家Emmanuelle Charpentier和Jennifer A. Doudna,可见CRISPR/Cas9技术的出现对于科学研究的巨大意义。
CRISPR/Cas9同样是一种人工核酸内切酶,其系统的主要组成部分包括DNA切割酶Cas9,由反式激活的crRNA(tracrRNA)和CRISPR RNA(crRNA)合成的向导RNA(sgRNA),以及PAM序列(protospacer adjacent motif)(NGG)(图2:C)(Jinek et al.,2012)。sgRNA向导序列(20 nt)靶向目的基因片段,招募Cas9蛋白的结合,在PAM上游的目标序列三个核苷酸处生成位点特异性DSB(Penewit et al.,2018)。这些DSB导致宿主细胞中DNA修复系统的激活,通常通过NHEJ途径进行修复,由于修复路径容易出错,因此在修复过程中将引入小的缺失或插入,从而产生突变(Voytas,2013)。CRISPR/Cas9技术的一些创造性和独特的CRISPR设计使该系统成为许多研究者用来操作基因的工具,已被广泛应用于水稻、棉花、高粱、玉米、小麦等植物的位点特异性诱变。例如最近的一项研究:中国农业科学院作物科学研究所国家植物转基因技术研究中心首次通过CRISPR/Cas9系统获得谷子单倍体诱导系(Cheng et al.,2021),开启谷子育种的新方向。
2 植物细胞器基因组的结构及特点
线粒体和叶绿体是植物细胞中仅有的两个具有独立遗传基因组的细胞器,它们的结构和功能受到细胞核基因组及自身基因组两套遗传系统的控制,称为半自主性细胞器。
2.1 线粒体基因组的结构和特点
线粒体是生物进行呼吸代谢,为生命活动提供能量的细胞器。不同生物的不同组织中线粒体数量的差异是巨大的。一般来说,细胞中线粒体数量取决于该细胞的代谢水平,代谢活动越旺盛的细胞中线粒体含量越多。一个细胞内有许多个线粒体,每个线粒体有几十至上百份基因组拷贝,所以一个细胞内有多个线粒体基因组。不同物种的线粒体基因组相差悬殊(Sloan et al.,2012;Gualberto & Newton,2017)。大部分植物线粒体基因组由不同大小、不同比例的环状DNA分子和线性DNA分子组成(Gualberto & Newton,2017)。陆地植物的线粒体基因组大小(208 kb~11 Mb)不一,远远大于动物的线粒体基因组(15~17 kb)(Sloan et al.,2012;Gualberto & Newton,2017)。线粒体基因组存在大量的重复序列,根据长度大小通常分为长重复序列(>500 bp)、中间重复序列(50~500 bp)和短重复序列(<50 bp)三种类型(Gualberto & Newton,2017)。大量重复序列的存在容易造成基因组重组或重排,导致线粒体基因组结构的变化。尽管植物线粒体基因组的大小和基因顺序有很大的差异,但是线粒体基因的数目相对保守(Small et al.,2013)。在陆生植物如被子植物、裸子植物(Sloan et al.,2012)、蕨类(Grewe et al.,2014)、石松类(Park et al.,2020)、角苔类(Gualberto & Newton,2017)、苔类(Guo et al.,2017)、藓类中,已知有30~70个基因被发现(Small et al.,2020)。这些基因编码rRNAs,tRNAs,核糖体蛋白,呼吸复合体Ⅰ、Ⅲ、Ⅳ的各个亚基,ATP合酶的亚基和细胞色素C发生因子(CCM)等成分(Small et al.,2020)。一些线粒体基因可能来自叶绿体或者细胞核,也可能在进化过程中转移到细胞核,这种现象称之为基因水平转移(horizontal gene transfer,HGT)或基因横向转移(lateral gene transfer,LGT)(Gao et al.,2014; Wu et al.,2017)。这种现象是编码基因数量在不同植物物种中差异的主要原因。例如,无油樟的线粒体含有超过250个编码基因,这可能就是进化过程中基因水平转移所导致的(Rice et al.,2013),以及被子植物中叶绿体基因转移到线粒体基因组中(Sloan & Wu,2014)。此外,线粒体基因组也可以转移到细胞核基因组中,如拟南芥的细胞核基因组中存在线粒体假基因(nuclear-encoded mitochondrial DNA sequences,NUMTs)的现象(Arimura et al.,2020)。
2.2 叶绿体基因组的结构和特点
叶绿体是绿色植物进行光合作用的场所。细胞中叶绿体基因组有多种构型,最常见的结构是双链环状结构,包括一个小的单拷贝区(SSC)和一个大的单拷贝区(LSC),这两个区域被一对反向重复区域(IRa,IRb)分开,形成典型的四分体结构,其基因组大小变化范围为120~160 kb(Hong et al.,2020)。与线粒体或细胞核基因组相比,植物叶绿体基因组在结构、基因数量和基因组成上具有更高的保守性,进化速度相对适中,介于核基因组和线粒体基因组之间,其自身缺少重组,有较小的基因组大小和高拷贝数。鉴于之前已有很多关于叶绿体基因组的综述报道,这里就不做详细的赘述。
3 线粒体基因编辑研究进展
在陆生植物中,尽管细胞核基因组的编辑修饰已经取得了很大的进展,但是线粒体基因组的遗传转化仍然进展缓慢(Kazama et al.,2019)。从结构上来说,植物线粒体基因组比动物线粒体基因组更大、更复杂(Small,2013;Gualberto & Newton,2017)。特别是在维管植物中,由于重复序列之间的活跃重组,即使在同一种植物中线粒体基因的顺序也会发生变化。尽管人们对陆地植物线粒体基因组序列了解甚多(Kubo & Newton,2008;Knoop,2013),但是由于缺乏稳定的线粒体转化方法,对植物线粒体基因组详细和直接的分子分析一直受到限制。同时,不能将gRNA递送到线粒体,阻碍了线粒体DNA(mtDNA)的编辑。因此,迄今为止,对mtDNA的操作仅限于通过设计核酸酶对线粒体基因的靶向突变。
3.1 mitoTALENs与mito-cTALEN介导的线粒体基因编辑
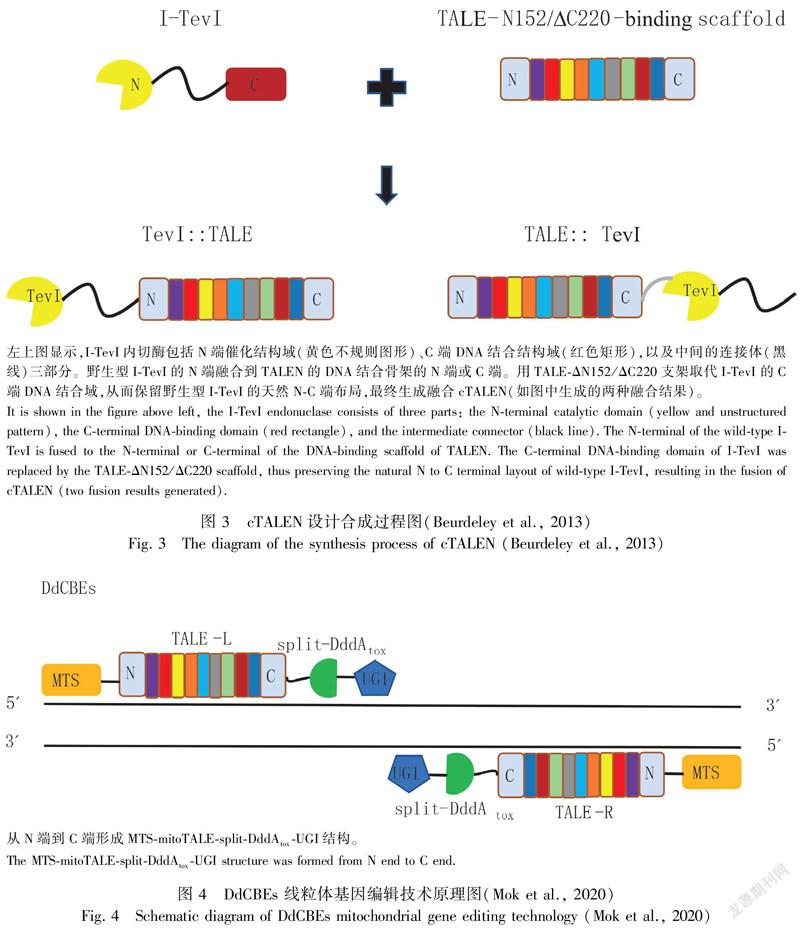
mitoTALENs线粒体基因编辑技术是直接在TALENs的N端加上一个线粒体定位信号(mitochondrial targeting signal,MTS),用来对线粒体基因组进行基因编辑(Kazama et al.,2019)。基于TALENs基因编辑的方法通常需要两个蛋白质,但有研究者对TALENs技术进行改进使得使用单一蛋白质成为可能,称为“compact TALEN”(cTALEN)(Kazama et al.,2019)。在cTALEN的N端加了一个线粒体定位信号得到mito-cTALEN,可实现对线粒体DNA的编辑。I-TevI(Mueller et al.,1995;Edgell et al.,2004)是GIY-YIG家族的歸巢内切酶成员,呈现出三部分蛋白布局。I-TevI的C端区域负责DNA结合的特异性以及大部分蛋白质与DNA的相互作用亲和力,一种连接体连接并调节N端催化结构域(Dean et al.,2002)。野生型I-TevI的N端融合到TALE的DNA结合骨架的N端或C端(Beurdeley et al.,2013)。用TALE-ΔN152/ΔC220支架取代I-TevI 的C端DNA结合域,从而保留野生型I-TevI的天然N-C端布局,最终生成融合cTALEN(图3)(Beurdeley et al.,2013)。
通过对靶基因靶点的选择设计合成TALE阵列,进而构建表达载体。功能元件在细胞核中转录、细胞质中翻译出MTS-TALE-FokI蛋白复合体,在线粒体导肽的作用下定位进入到线粒体中,TALE 中的DNA结合结构域靶向目的基因,FokI核酸酶形成二聚体后对目的基因进行切割,主要通过同源重组修复,修复过程中造成基因大片段缺失,最终导致基因破坏失去功能,达到基因敲除的目的。
在植物中,一种被称为细胞质雄性不育(cytoplasmic male sterility,CMS)的雄性不育系通常表现为花粉败育和结实率为零,在农业上经常被用于生产F1杂交种子(Kazama et al.,2019)。一般来说,CMS被认为是由线粒体基因引起的(Hanson & Bentolila,2004)。敲除典型线粒体基因可能是致命的或引起不利的表型变化,但敲除CMS植物中的一个CMS相关基因有望在不引起其他表型变化的情况下恢复育性(Kazama et al.,2019)。2019年,日本东京大学的一个科研团队同时以水稻BoroTaichung 型CMS材料(BTA)和油菜Kosena型CMS材料(SW18)作为研究对象,分别利用mito-cTALEN和mitoTALENs线粒体基因编辑技术对BTA中的线粒体基因orf79和SW18中的线粒体基因orf125进行基因编辑(Kazama et al.,2019)。该研究结果显示,被成功敲除CMS基因的株系花粉恢复了育性——自交产生了种子(Kazama et al.,2019)。这是对植物线粒体基因组稳定和可遗传靶向基因修饰的首次报道,是植物线粒体基因组编辑的重要突破性研究进展,具有重要意义(Arimura et al.,2020)。随后,该团队又在2020年利用mitoTALENs技术分别对拟南芥线粒体基因组中ATP合成酶亚基6基因—atp6-1和atp6-2进行了基因编辑,这两个基因分别被成功删除,验证了mitoTALENs技术在植物线粒体基因编辑中应用的广泛性。作者同时也比较了mitoTALENs和mito-cTALEN两种技术对线粒体基因的编辑效果,研究表明,mitoTALENs的编辑效率更好(Arimura et al.,2020)。
3.2 DdCBEs介导的线粒体基因编辑
上述两种编辑技术都能够进入线粒体发挥基因编辑的作用,但不能引起mtDNA中特定核苷酸的变化,这些系统是通过将携带的突变mtDNA直接降解来消除突变,会使得mtDNA拷贝数降低,而线粒体拷贝数的降低可能会在实际疾病治疗中带来更多的风险。2020年,哈佛大学David R. Liu团队利用一种细菌胞嘧啶脱氨酶毒素——DddA分开的两部分与TALE阵列蛋白和尿嘧啶糖基化酶抑制剂(UGI)融合产生了无RNA(无CRISPR)的DddA衍生的胞嘧啶碱基编辑器(DddA-derived cytosine base editor,DdCBE),实现了催化人mtDNA中C·G-T·A的转换,具有高的靶标特异性和产物纯度(Mok et al.,2020)。研究者使用DdCBEs来模拟人类细胞中与疾病相关的mtDNA突變,导致呼吸速率和氧化磷酸化的变化(Mok et al.,2020)。无CRISPR的DdCBEs可以精确地操纵mtDNA,而不是通过靶向核酸酶切割mtDNA来消除mtDNA拷贝,这对线粒体疾病的研究和潜在的治疗具有广泛的意义。
DddA是SCP1.201-like家族的一种细菌脱氨酶毒素,优先作用于dsDNA,因此被命名为双链DNA脱氨酶毒素A,或DddA(Mok et al.,2020)。DddA含有一个毒性结构域DddAtox,完整地表达DddA蛋白对人体细胞有毒害作用,为了避免这种毒性,研究者将蛋白质分成两个不活跃的部分,这两部分只有在相邻的目标DNA上组装时才能重新形成脱氨活性,类似于在ZFNs和TALENs中组装FokI单体来重新形成dsDNA核酸酶活性(Mok et al.,2020)。为了利用DddA的脱氨酶作用实现线粒体基因组的精准编辑,研究者将分开的DddA与TALE融合进行线粒体碱基编辑,形成N端到C端的“MTS-mitoTALE-split-DddAtox”连接结构,实现mtDNA上胞嘧啶C脱氨基变成尿嘧啶U(Mok et al.,2020)。然而,由于U是组成RNA的碱基,一旦DNA中出现了U,其就会在尿嘧啶糖基化酶的作用下重新转变成C,因此就相当于该编辑器对线粒体中DNA单碱基的替换无意义。为了防止这一现象的发生,研究者巧妙地在上述构建的碱基编辑器的C端连接了UGI(Nilsen et al.,1997;Komor et al.,2017;Fuentes et al.,2018),最终形成“MTS-mitoTALE-split-DddAtox-UGI”结构(图4)。这样就可以保护U免受糖基化酶的干扰,一直以尿嘧啶的形式存在直到下一轮DNA复制或修复发生为止,此时互补链的鸟嘌呤G(在编辑前与C配对)被腺嘌呤A取代,实现C·G-T·A的转换(Mok et al.,2020)。UGI的加入使编辑效率提高了3~10倍,从而最终获得了可实现mtDNA中碱基C-T高效特异性转换的胞苷单碱基编辑器DdCBEs(Mok et al.,2020)。为了探究DdCBEs在mtDNA编辑中的普遍性,研究者靶向了5个线粒体基因:MT-ND1、MT-ND2、MT-ND4、MT-ND5和MT-ATP8,证实了在HEK293T细胞中mtDNA编辑的持久性超过18 d,典型效率在5%~50%之间(Mok et al.,2020)。mtDNA编辑不会降低细胞活力,不会产生大量mtDNA删除,也不会干扰mtDNA拷贝数(Mok et al.,2020)。该研究成果是开发针对mtDNA疾病的基因疗法的重要进展。此外,通过使用该工具实验性地改变线粒体基因组,可以更好地了解mtDNA突变与复杂疾病、癌症和年龄相关的细胞功能障碍的相关性。
最近,Arimura团队利用DdCBEs碱基编辑技术靶向拟南芥质体基因组中16S rRNA、rpoC1和psbA三个基因,特异性地在拟南芥质体基因组的靶向窗口中引入同源C-T突变且突变能够稳定遗传给后代种子(Nakazato et al.,2021)。Kang et al.(2021)建立了Golden Gate克隆系统,共使用424个TALE子阵列质粒和16个表达质粒组装DdCBE编码的质粒进行植物细胞器碱基编辑。他们利用设计的DdCBEs分别先后对莴苣和油菜的叶绿体基因16S rRNA、psbA 和 psbB以及莴苣线粒体基因atp6和油菜线粒体基因rps14进行了碱基编辑,在莴苣和油菜原生质体中实现了高频率的C-T转换(Kang et al.,2021)。其DdCBEs在生菜或油菜愈伤组织中诱导碱基编辑频率高达25%(线粒体)和38%(叶绿体),并且植物细胞器的编辑在细胞分裂和植物发育过程中得以维持(Kang et al.,2021)。Li et al.(2021)将DdCBEs应用于水稻叶绿体基因编辑,选用一个编码光合I系统的保守叶绿素基因psaA实现了水稻中C·G-T·A的转换。
4 叶绿体的遗传转化研究进展
叶绿体的遗传转化是指将目的基因导入植物细胞叶绿体基因组的过程。相对于线粒体,叶绿体的转化技术较为成熟。与植物基因工程常用的常规核转化技术相比,叶绿体转化具有许多的优势(Jin & Daniell,2015;Fuentes et al.,2018)。在大多数高等植物中,由于叶绿体基因组的母系遗传特性,使得叶绿体转化在一定程度上成为可以遏制基因逃逸的基因技术,很少导致转基因的异交(Daniell et al.,2021)。又因叶绿体基因组是以高拷贝的形式存在,叶绿体转化可以通过在每个植物细胞中引入数千个外源基因的拷贝而导致异常高水平的蛋白质生产(DeCosa et al.,2001),提高基因转化效率。
如今,叶绿体基因组已被改造用于表达来自细菌、真菌、原生动物或人类基因组的外源基因,以进行各种生物技术应用(Daniell et al.,2021)。利用细菌调控序列表达了苏云金芽孢杆菌的细菌操纵子,达到了已知的最高水平的Bt蛋白(DeCosa et al.,2001)。小的人类基因——胰岛素,以很高的水平表达(可占叶片总蛋白的70%)(Ruhlman et al.,2010;Boyhan & Daniell,2011)。对CTB-FVIII-LC基因的密码子进行优化,使得最大的人血蛋白(FVIII,185 kDa单体)在叶绿体中成功表达(Kwon et al.,2018),目前已推进商业化生产和临床试验。密码子优化的ACE2现已在临床上经过测试,可治疗COVID-19患者(Daniell,2020)。PhylloZyme最近推出了几种在叶中制成并在叶绿体中表达的酶产品,以取代价格昂贵的微生物酶工艺(Daniell et al.,2019a,b; Kumari et al.,2019)。大多数叶片酶在较宽的pH和温度范围内作为粗叶提取物起作用而不需要纯化,叶片酶可以在环境温度下作为冻干植物细胞储存几个月甚至几年,而不丧失酶活性(Daniell et al.,2021)。当前,商业酶在生产过程中会产生大量二氧化碳排放而導致温室效应,用叶酶代替发酵制成的酶,除了防止释放二氧化碳外,还可以捕获二氧化碳,每产生1 kg酶或蛋白质,净捕获680 kg二氧化碳,从而减少人为温室气体排放(Daniell et al.,2021)。莴苣叶绿体中表达的蛋白药物(protein drugs,PDs)的口服给药已被开发用于治疗传染性或遗传性疾病,目前已进入临床阶段(Daniell et al.,2021)。最近的进展包括通过去除抗生素抗性基因在无标记叶绿体基因组中表达PDs(Daniell,2020; Park et al.,2020),在cGMP设备中生产莴苣中表达的PDs(Daniell et al.,2019a; Daniell,2020; Park et al.,2020),通过毒理学和药代动力学研究评估药物(Daniell ,2020),以及最近FDA批准通过口服花生细胞产生的PDs治疗过敏(Tilles & Petroni,2018; Vickery et al.,2018)。在开发SARS-CoV-2疫苗或治疗COVID-19患者的急性/致死性肺/心力衰竭下有所应用(Daniell et al.,2021)。接下来,对叶绿体遗传转化的几种方式进行介绍。
4.1 基因枪介导的叶绿体转化
叶绿体转化或者叫叶绿体基因工程,这一概念是由宾夕法尼亚大学的Daniell教授所开创的。Daniell通过康奈尔大学的John Sanford教授设计的一种生物微粒传递系统——基因枪,用叶绿体载体演示了第一个外来基因在叶绿体中的表达(Daniell et al.,1990),从而创造了最早的叶绿体基因工程专利。基因枪技术的基本原理就是采用一种微粒加速装置,使裹着外源基因的微米级的金或钨颗粒获得足够的动量打入靶细胞或组织。到目前为止,基因枪转化仍然是叶绿体转化领域应用最多、最有效的方法(刘佳音等,2020)。
Goldschmidt-Clermont(1991)引入了第一个叶绿体选择标记基因——aadA基因,随后将其用于转化烟草叶绿体基因组和大多数其他农作物。而绝大多数转叶绿体研究使用的是Daniell在1986年构建的第一个叶绿体DNA载体中引入的psbA调控序列(Daniell et al.,2016a,b,2021),基于植物叶绿体进行稳定的遗传转化对于基础研究和应用研究都有着很大的便利。然而,在主要作物和模式植物中应用叶绿体转基因技术还很困难,目前大多数的植物物种都不能进行叶绿体转基因,因此该技术的应用大大受到了局限。2019年,德国马普分子植物生理研究所开发了一个高效的拟南芥叶绿体转基因技术,该技术通过基因枪转化方法对来自于根的微愈伤组织进行遗传转化,结合CRISPR/Cas9技术敲除一个核等位基因,增强植株对于筛选药剂的敏感性以用于分离转基因植株,该方法能够高频生产可育的叶绿体遗传转化植株(Ruf et al.,2019)。
4.2 PEG介导的叶绿体转化
聚乙二醇(PEG)介导的叶绿体转化比基因枪方法要便宜很多。1993年首次报道了利用PEG法实现在烟草叶绿体中的稳定转化(Díaz & Koop,2014)。PEG介导叶绿体转化的原理:除去细胞壁的植物细胞原生质体在 PEG 的作用下细胞膜结构发生重构,原生质体缩水,在细胞膜结构发生不可逆破坏前去除 PEG,则细胞膜结构将恢复原来的状态,外源DNA 在这一过程中有机会进入植物细胞以及叶绿体中,实现转化(刘佳音等,2020)。PEG介导的叶绿体转化方法的成功依赖于良好的原生质体再生方案。由于叶绿体的稳定遗传转化是建立在同源重组的基础上的,因此转基因的两翼必须有叶绿体序列,以确保转基因整合到叶绿体的特定区域(刘佳音等,2020)。PEG介导叶绿体转化在烟草(O’Neill et al.,1993)、番茄(Nugent et al.,2005a)、花椰菜(Nugent et al.,2005b)和莴苣(Lelivelt et al.,2005)中均已实现。
4.3 纳米材料介导的叶绿体转化
麻省理工学院化学工程教授 Michael Strano 领衔的研究团队开发了一种利用纳米材料进行叶绿体转化的新工具,利用单壁碳纳米管选择性地将质粒DNA递送至叶绿体内,而无需额外设备或化学试剂(Kwak et al.,2019)。研究人员创造了由包裹着壳聚糖的碳纳米管所组成的纳米颗粒(chitosan-complexed single-walled carbon nanotubes,CS-SWNTs),带负电荷的 DNA 可与带正电荷的碳纳米管松散地结合(Kwak et al.,2019)。将带有DNA的纳米颗粒注入植株叶片,一旦通过叶片气孔进入叶片内部,纳米颗粒就会穿过细胞壁、细胞膜,之后穿过叶绿体的双层膜(Kwak et al.,2019)。进入叶绿体后,叶绿体的弱酸性环境就会促使 DNA 从纳米颗粒中释放出来。纳米穿透能力主要由纳米颗粒大小和表面电荷决定(Kwak et al.,2019)。为便于观察,研究人员使用了黄色荧光蛋白YFP基因,大约 47% 的植物细胞产生了YFP(Kwak et al.,2019)。在成熟的芝麻菜、豆瓣菜、菠菜、烟草等植物和分离的拟南芥叶肉原生质体中均实现了叶绿体靶向基因的传递和表达(Kwak et al.,2019)。选择性纳米颗粒介导的叶绿体转基因平台操作简单、成本低廉,可应用于不同品种的成熟植物,且不需要专门、昂贵的设备(Kwak et al.,2019)。同时,该研究提出的方法或可结合ZFNs、TALENs和CRISPR/Cas9技术用于精确的叶绿体基因组编辑(Kwak et al.,2019)。通过优化该方法,可在作物物种中实现稳定的叶绿体转化,有助于作物改良和农艺应用。
5 总结与展望
随着基因编辑技术的快速发展,植物核基因组编辑取得了大量成果,在植物功能基因组学和作物遗传改良等研究领域做出了巨大贡献。而植物中的另外两个基因组(线粒体基因组和叶绿体基因组)在基因编辑方面的研究进展却相对缓慢。在科研人员的不懈努力之下,近几年在线粒体基因组编辑和叶绿体遗传转化方面都取得了一些新的突破。2019年,日本东京大学实验室分别利用mitoTALENs和mito-cTALEN编辑技术编辑水稻和油菜线粒体中CMS相关基因,首次实现了植物线粒体基因组稳定和可遗传靶向基因修饰(Kazama et al.,2019)。接着,该实验室又利用mitoTALENs技术靶向拟南芥线粒体基因成功实现线粒体基因编辑(Arimura et al.,2020)。2020年,David R. Liu团队开发出新的线粒体基因编辑工具——DdCBEs技术,该技术利用细菌毒素在HEK293T细胞的mtDNA中引入特定核苷酸,实现了线粒体基因的精准编辑,这对人类mtDNA 突变引起的线粒体遗传疾病的研究和治疗意义重大(Mok et al.,2020)。与mitoTALENs技术相比,基因编辑器 DdCBEs 不降低细胞活力,不产生 mtDNA 缺失,不改变 mtDNA 拷贝数,展示了优秀的安全性和巨大的应用前景。最近,科研工作者们已经成功将DdCBEs技术用于拟南芥、莴苣、油菜和水稻的线粒体和叶绿体的基因组编辑,有效实现了目标基因C·G-T·A的轉换。尽管这些创新性的研究在细胞器编辑方面已取得了重要进展,但是仍有许多挑战需要克服,如需要有更高的编辑效率和种质选择能力以适应多种植物的技术(Lee et al.,2021)。同时,ABEs碱基编辑技术在线粒体基因组的应用以及如何突破CRISPR/Cas系统转化线粒体基因组的瓶颈抑或是开发出其他新的转化技术等,都需要研究者们进行更多的思考与尝试(Lee et al.,2021)。
相比于线粒体转化,叶绿体转化早在20世纪80年代就实现了,但技术上一直没有很大的突破。目前,基因枪仍然是较为常用的转化方法,但价格昂贵且效率不高;PEG介导的原生质体转化耗时耗力又有物种和组织限制性,最近的突破就是利用纳米材料进行的叶绿体转化。这个方法的主要优点就是受物种影响小,研究人员在菠菜、西洋菜、烟草、芝麻菜和拟南芥中都进行了测试。同时,这项技术不只限于碳纳米管,还有可能扩展到其他类型的纳米材料。另外,由于该方法仅对叶绿体进行操作,叶绿体是母系遗传的,可以传给后代,却不会转移到其他植物物种,这也是一大优势。同时,该研究提出的方法或可结合核酸酶介导的基因编辑技术,用于叶绿体基因编辑。由于很多植物再生的外植体不是绿色组织,比如下胚轴、子叶柄、幼胚等,只含有前质体或有色体,这些质体和叶绿体在体积上,基因表达调控上都有很大差异,因此基因枪介导的转化,很难将质粒递送到这些外植体中去,即使递送进去了,基因也很难正常表达。而目前主要的农作物,例如水稻(成熟胚)、玉米、小麦(幼胚)、棉花(下胚轴)、油菜(下胚轴)做转化时,外植体都不是绿色的。烟草、番茄、生菜这三个叶绿体转化很成功的作物,都是用绿色叶片作为外植体进行转化,之后通过器官发生途径获得再生植株。因此,如何在不同植物中建立以叶片为外植体的再生系统是进行叶绿体转化的重要前提;同时开发在非绿色组织中高效表达的调控元件也是实现叶绿体转化的重要基础。
雖然对细胞器基因编辑的研究困难重重,进度缓慢,且研究者较少,但是事实上,与核基因组相比,线粒体和叶绿体基因组在进行基因编辑时具有许多的优势。如叶绿体基因组作为插入基因载体的优点:(1)叶绿体是母系遗传,可以减少外源基因扩散的风险;(2)细胞中叶绿体拷贝数较高可使目的基因高水平表达获得大量蛋白;(3)叶绿体基因结构较简单,可以实现目的基因较精确的定位在叶绿体基因组上等优点。而线粒体参与植物生长过程中的许多重要环节,与植株育性等农艺性状有关。CMS相关基因存在于线粒体DNA上,对相关基因进行编辑可以恢复植株育性用于作物育种。线粒体与核之间也具有紧密的联系,研究线粒体基因可进一步了解细胞内及整个植株的生命活动机制。细胞器基因编辑具有很多的优点,有巨大的研究价值和研究潜力。无论线粒体还是叶绿体,对其基因组的研究都需要更加完善的基因编辑技术,相信在不断前行的步伐下,一定会取得更大的突破。
参考文献:
ARIMURA SI,AYABE H,SUGAYA H,et al.,2020. Targeted gene disruption of ATP synthases 6-1 and 6-2 in the mitochondrial genome of Arabidopsis thaliana by mitoTALENs [J]. Plant J,104(6):1459-1471.
BEURDELEY M,BIETZ F,LI J,et al.,2013. Compact designer TALENs for efficient genome engineering [J]. Nat Commun,4(1):1762. DOI:10.1038/ncomms2782.
BONAWITZ ND,AINLEY WM,ITAYA A,et al.,2019. Zinc finger nuclease-mediated targeting of multiple transgenes to an endogenous soybean genomic locus via non-homologous end joining [J]. Plant Biotechnol J,17(4):750-761.
BOYHAN D,DANIELL H,2011. Low-cost production of proinsulin in tobacco and lettuce chloroplasts for injectable or oral delivery of functional insulin and C-peptide [J]. Plant Biotechnol J,9(5):585-598.
CARROLL D,2011. Genome engineering with zinc-finger nucleases [J]. Genetics,188(4):773-782.
CHAR SN,YANG B,2020. Genome editing in grass plants [J].aBIOTECH,1(5):41-57.
CHENG ZX,SUN Y,YANG SH,et al.,2021. Establishing in planta haploid inducer line by edited SiMTL in foxtail millet (Setaria italica) [J]. Plant Biotechnol J,19(6):1089-1091.
CURTIN SJ,ZHANG F,SANDER JD,et al.,2011. Targeted mutagenesis of duplicated genes in soybean with zinc-finger nucleases [J]. Plant Physiol,156(2):466-473.
DANIELL H,2020. From conception to COVID-19:an arduous journey of tribulations of racism and triumphs [J]. Plant Biotechnol J,18(11):2147-2154.
DANIELL H,CHAN HT,PASORECK EK,2016a. Vaccination via chloroplast genetics:affordable protein drugs for the prevention and treatment of inherited or infectious human diseases [J]. Ann Rev Genet,50:595-618.
DANIELL H,JIN SX,ZHU XG,et al.,2021. Green giant—a tiny chloroplast genome with mighty power to produce high-value proteins:history and phylogeny [J]. Plant Biotechnol J,19(3):430-447.
DANIELL H,KULIS M,HERZOG RW,2019a. Plant cell-made protein antigens for induction of Oral tolerance [J]. Biotechnol Adv,37(7):107413-107426.
DANIELL H,LIN CS,YU M,et al.,2016b. Chloroplast genomes:diversity,evolution,and applications in genetic engineering [J]. Genome Biol,17(1):134. DOI:10.1186/s13059-016-1004-2.
DANIELL H,RIBEIRO T,LIN SN,et al.,2019b. Validation of leaf and microbial pectinases:commercial launching of a new platform technology [J]. Plant Biotechnol J,17(6):1154-1166.
DANIELL H,VIVEKANANDA J,NIELSEN BL,et al.,1990. Transient foreign gene expression in chloroplasts of cultured tobacco cells after biolistic delivery of chloroplast vectors [J]. Proc Natl Acad Sci USA,87(1):88-92.
DEAN AB,STANGER MJ,DANSEREAU JT,et al.,2002. Zinc finger as distance determinant in the flexible linker of intron endonuclease I-TevI [J]. Proc Natl Acad Sci USA,99(13):8554-8561.
DECOSA B,MOAR W,LEE SB,et al.,2001. Overexpression of the Bt cry2Aa2 operon in chloroplasts leads to formation of insecticidal crystals [J]. Nat Biotechnol,19(1):71-74.
DAZ AH,KOOP HU,2014. Nicotiana tabacum:PEG-mediated plastid transformation [J]. Method Mol Biol,1132:165-175.
EDGELL DR,STANGER MJ,BELFORT M,2004. Coincidence of cleavage sites of intron endonuclease I-TevI and critical sequences of the host thymidylate synthase gene [J]. J Mol Biol,343(5):1231-1241.
FUENTES P,ARMAREGO-MARRIOTT T,BOCK R,2018. Plastid transformation and its application in metabolic engineering [J]. Curr Opin Biotechnol,49:10-15.
GAJ T,GERSBACH CA,BARBAS CF,2013. ZFN,TALEN,and CRISPR/Cas-based methods for genome engineering [J]. Trend Biotechnol,31(7):397-405.
GAMMAGE PA,MORAES CT,MINCZUK M,2018. Mitochondrial genome engineering:the revolution may not be CRISPR-Ized [J]. Trend Genet,34(2):101-110.
GAO CH,REN XD,MASON AS,2014. Horizontal gene transfer in plants [J]. Funct Integr Genomics,14(1):23-29.
GAO CX,2021. Genome engineering for crop improvement and future agriculture [J]. Cell,184(6):1621-1635.
GOLDSCHMIDT-CLERMONT M,1991. Transgenic expression of aminoglycoside adenine transferase in the chloroplast:A selectable marker for site-directed transformation of chlamydomonas [J]. Nucl Acid Res,19(15):4083-4089.
GREWE F,EDGER PP,KEREN I,et al.,2014. Comparative analysis of 11 Brassicales mitochondrial genomes and the mitochondrial transcriptome of Brassica oleracea [J]. Mitochondrion,19:135-143.
GUALBERTO JM,NEWTON KJ,2017. Plant mitochondrial genomes:dynamics and mechanisms of mutation [J]. Ann Rev Plant Biol,68:225-252.
GUO WH,ZHU AD,FAN WS,et al.,2017. Complete mitochondrial genomes from the ferns Ophioglossum californicum and Psilotum nudum are highly repetitive with the largest organellar introns [J]. New Phytol,213(1):391-403.
HANSON MR,BENTOLILA S,2004. Interactions of mitochondrial and nuclear genes that affect male gametophyte development [J]. Plant Cell,16 (Suppl.):S154-S169.
HONG Z,WU ZQ,ZHAO KK,et al.,2020. Genomic architectural variation of plant mitochondria—A review of multichromosomal structuring [J]. J Syst Evol. DOI:10.1111/jse.12655.
HUO ZJ,TU J,XU A,et al.,2019. Generation of a heterozygous p53 R249S mutant human embryonic stem cell line by TALEN-mediated genome editing [J]. Stem Cell Res,34:101360. DOI:10.1016/j.scr.2018.101360. Epub 2018 Nov 30.
JIN SX,DANIELL H,2015. The engineered chloroplast genome just got smarter [J]. Trend Plant Sci,20(10):622-640.
JINEK M,CHYLINSKI K,FONFARA I,et al.,2012. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity [J]. Science,337(6096):816-821.
KANG BC,BAE SJ,LEE S,et al.,2021. Chloroplast and mitochondrial DNA editing in plants [J]. Nat Plants,7(7):899-905.
KAZAMA T,OKUNO M,WATARI Y,et al.,2019. Curing cytoplasmic male sterility via TALEN-mediated mitochondrial genome editing [J]. Nat Plant,5(7):722-730.
KHALIL AM,2020.The genome editing revolution:review [J]. J Genet Eng Biotechnol,18(1):68.
KNOOP V,2013. Plant mitochondrial genome peculiarities evolving in the earliest vascular plant lineages [J].J Syst Evol,51(1):1-12.
KOMOR AC,ZHAO KT,PACKER MS,et al.,2017. Improved base excision repair inhibition and bacteriophage Mu Gam protein yields C∶G-to-T∶A base editors with higher efficiency and product purity [J]. Sci Adv,3(8):eaao4774.
KUBO T ,NEWTON KJ,2008. Angiosperm mitochondrial genomes and mutations [J]. Mitochondrion,8(1):5-14.
KUMARI U,SINGH R,RAY T,et al.,2019. Validation of leaf enzymes in the detergent and textile industries:launching of a new platform technology [J]. Plant Biotechnol J,17(6):1167-1182.
KWAK SY,LEW TTS,SWEENEY CJ,et al.,2019. Chloroplast-selective gene delivery and expression in planta using chitosan-complexed single-walled carbon nanotube carriers [J]. Nat Nanotechnol,14(5):447-455.
KWON KC,SHERMAN A,CHANG WJ,et al.,2018. Expression and assembly of largest foreign protein in chloroplasts:oral delivery of human FVIII made in lettuce chloroplasts robustly suppresses inhibitor formation in haemophilia A mice [J]. Plant Biotechnol J,16(6):1148-1160.
LEE H,HONG C,HWANG J,et al.,2021. Go green with plant organelle genome editing [J]. Mol Plant. DOI:https://doi.org/10.1016/ j.molp.2021.07.012.
LELIVELT CLC,MCCABE MS,NEWELL CA,et al.,2005. Stable plastid transformation in lettuce (Lactuca sativa L.) [J]. Plant Mol Biol,58(6):763-774.
LI L,WU LP,CHANDRASEGARAN S,1992. Functional domains in Fok I restriction endonuclease [J]. Proc Natl Acad Sci USA,89(10):4275-4279.
LI RQ,CHAR SN,LIU B,et al.,2021. High-efficiency plastome base editing in rice with TAL cytosine deaminase [J]. Mol Plant,14:1-3.
LIU JY,LI RJ,QI SH,et al.,2020. Chloroplast genetic transformation system and its application progress [J]. J Anhui Agric Sci,48(6):16-19. [劉佳音,李儒剑,齐双慧,等,2020. 叶绿体遗传转化系统及其应用进展 [J]. 安徽农业科学,48(6):16-19. ]
LU HW,KLOCKO AL,DOW M,et al.,2016. Low frequency of zinc-finger nuclease-induced mutagenesis in Populus [J]. Mol Breed,36(9):121-133.
MANGHWAR H,LINDSEY K,ZHANG XL,et al.,2019. CRISPR/Cas system:recent advances and future prospects for genome editing [J]. Trend Plant Sci,24(12):1102-1125.
MOK BY,DE MORAES MH,ZENG J,et al.,2020. A bacterial cytidine deaminase toxin enables CRISPR-free mitochondrial base editing [J]. Nature,583(7817):631-637.
MUELLER JE,SMITH D,BRYK M,et al.,1995.Intron-encoded endonuclease I-TevI binds as a monomer to effect sequential cleavage via conformational changes in the td homing site [J]. EMBO J,14(22):5724-5735.
NAKAZATO I,OKUNO M,YAMAMOTO H,et al.,2021. Targeted base editing in the plastid genome of Arabidopsis thaliana [J]. Nat Plant,7(7):906-913.
NILSEN H,OTTERLEI M,HAUG T,et al.,1997. Nuclear and mitochondrial uracil-DNA glycosylases are generated by alternative splicing and transcription from different positions in the UNG gene [J]. Nucl Acid Res,25(4):750-755.
NUGENT GD,COYNE S,NGUYEN TH,et al.,2005b. Nuclear and plastid transformation of Brassica oleracea var. botrytis (cauliflower) using PEG-mediated uptake of DNA into protoplasts [J]. Plant Sci,170(1):135-142.
NUGENT GD,TEN HAVE M,VAN DER GULIK A,et al.,2005a. Plastid transformants of tomato selected using mutations affecting ribosome structure [J]. Plant Cell Rep,24(6):341-349.
O’NEILL C,HORTH GV,HORVTH E,et al.,1993. Chloroplast transformation in plants:polyethylene glycol (PEG) treatment of protoplasts is an alternative to biolistic delivery systems [J]. Plant J,3(5):729-738.
PARK J,YAN G,KWON KC,et al.,2020. Oral delivery of novel human IGF-1 bioencapsulated in lettuce cells promotes musculoskeletal cell proliferation,differentiation and diabetic fracture healing [J]. Biomaterials,233:119591.
PEER R,RIVLIN G,GOLOBOVITCH S,et al.,2015. Targeted mutagenesis using zinc-finger nucleases in perennial fruit trees [J]. Planta,241(4):941-951.
PENEWIT K,HOLMES EA,MCLEANK,et al.,2018. Efficient and scalable precision genome editing in Staphylococcus aureus through conditional recombineering and CRISPR/Cas9-mediated counterselection [J]. mBio,9(1):e00067-18.
RICE DW,ALVERSON AJ,RICHARDSON AO,et al.,2013. Horizontal transfer of entire genomes via mitochondrial fusion in the angiosperm Amborella [J]. Science,342(6165):1468-1473.
RUF S,FORNER J,HASSE C,et al.,2019. High-efficiency generation of fertile transplastomic Arabidopsis plants [J]. Nat Plants,5(3):282-289.
RUHLMAN T,VERMA D,SAMSON N,et al.,2010. The role of heterologous chloroplast sequence elements in transgene integration and expression [J]. Plant Physiol,152(4):2088-2104.
SHINOYAMA H,ICHIKAWA H,NISHIZAWA-YOKOI A,et al.,2020. Simultaneous TALEN-mediated knockout of chrysanthemum DMC1 genes confers male and female sterility [J]. Sci Rep,10(1):16165.
SHUKLA VK,DOYON Y,MILLER JC,et al.,2009. Precise genome modification in the crop species Zea mays using zinc-finger nucleases [J]. Nature,459(7245):437-441.
SLOAN DB,ALVERSON AJ,CHUCKALOVCAK JP,et al.,2012. Rapid evolution of enormous,multichromosomal genomes in flowering plant mitochondria with exceptionally high mutation rates [J]. PLoS Biol,10(1):e1001241.
SLOAN DB,WU ZQ,2014. History of plastid DNA insertions reveals weak deletion and at mutation biases in angiosperm mitochondrial genomes [J]. Genome Biol Evol,6(12):3210-3221.
SMALL I,2013. Mitochondrial genomes as living ‘fossils’ [J]. BMC Biol,11(1):30.
SMALL ID,RACKHAM O,FILIPOVSKA A,2013. Organelle transcriptomes:products of a deconstructed genome [J]. Curr Opin Microbiol,16(5):652-658.
SMALL ID,SCHALLENBERG-RDINGER M,TAKENAKA M,et al.,2020. Plant organellar RNA editing:what 30 years of research has revealed [J]. Plant J,101(5):1040-1056.
SMITH J,BIBIKOVA M,WHITBY FG,2000. Requirements for double-strand cleavage bychimeric restriction enzymes with zinc finger DNA-recognition domains [J]. Nucl Acid Res,28(17):3361-3369.
TILLES SA,PETRONI D,2018. FDA-approved peanut allergy treatment:the first wave is about to crest [J]. Ann Allergy Asthma Immunol,121(2):145-149.
VICKERY CR,CARDENAS J,BOWMAN ME,et al.,2018. A coupled in vitro/in vivo approach for engineering a heterologous type Ⅲ PKS to enhance polyketide biosynthesis in Saccharomyces cerevisiae [J]. Biotechnol Bioeng,115(6):1394-1402.
VOYTAS DF,2013. Plant genome engineering with sequence-specific nucleases [J]. Annu Rev Plant Biol,64(1):327-350.
WU ZQ,SLOAN DB,BROWN CW,et al.,2017. Mitochondrial retroprocessing promoted functional transfers of rpl5 to the nucleus in grasses [J]. Mol Biol Evol,34(9):2340-2354.
(責任编辑 何永艳)

