植物耐盐性:演化和盐基因组学
徐涵 郭容芳 张玉苗 李蓉 肖学宸 曹子谊 王姗姗 张可轩 陈晓慧 陈晓东 陈裕坤 叶炜 叶开温 林玉玲 赖钟雄

摘 要:由于社会的发展需要使用盐地产出植物,因此植物耐盐性的研究变得日益重要。植物在长期演化中主要形成嗜盐植物和非嗜盐植物两类,在细胞乃至群体处理盐的机制方面发生了广泛和深度的遗传变异。鉴于发掘植物耐盐、使植物耐盐和改变植物盐环境的各领域都取得了重大进展,结合植物的演化扩展对植物耐盐的认识,从细胞机制、个体机制扩展到群体机制乃至多基因组参与的生态互作,进行以盐基因组学为视角的研究就显得非常重要。本文根据该领域的新近进展,对植物耐盐性进行演化和组学视角的回顾和分析,以期对今后的植物耐盐研究提供参考。
关键词:植物耐盐;盐基因组;宏基因组;共生总基因组;生物演化
中图分类号:S332.6 文献标识码:A
Abstract: Plant salt-tolerance (halotolerance) has become increasingly important as the social development needs to use saline land to grow plants. In the long-term evolution, there are mainly two types of plants: halophilic plants and glycophytes, which have undergone extensive and deep genetic variations in the salt-processing mechanisms by cells and populations. In view of the great progress made in the exploration of plant salt tolerance, the salt tolerance improvement and the desalination of salt environment for plants, there is a need to expand the horizon from the perspective of cellular and individual mechanisms to populational mechanism and even ecological interactions with multi-genome involved by means of salt-genomics (halogenomics) and metagenomics. Here, according to the recent progress in this field, the evolution and omics analysis of plant salt-tolerance were made in the present review in order to provide a genomic reference for further research and exploration of plant halotolerance.
Keywords: plant salt-tolerance; halogenome; metagenomics; hologenome; evolution
DOI: 10.3969/j.issn.1000-2561.2020.10.005
1 植物耐鹽反应的架构
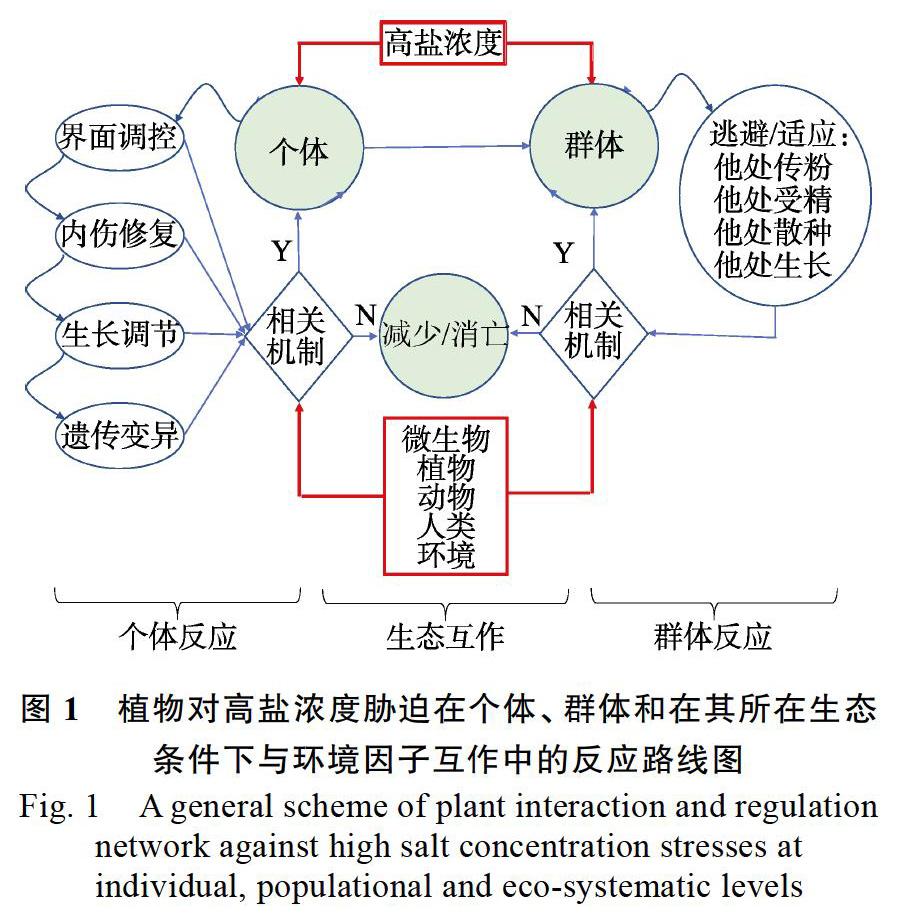
植物对高盐浓度胁迫的耐受与否及其反应,理论上包括个体、群体和环境3个层面的动态过程,具体涉及6个方面,包括个体的:(1)界面调控,(2)内伤修复,(3)生长调节,(4)遗传变异;群体的:(5)生态选择;生态的:(6)互作(图1)。个体和群体以及与生态互作的3个方面效应的叠加提供了植物在生态环境中耐盐演化的驱动力。这种植物与盐胁迫相关的基因及其调控网络的总和被笼统地称作盐基因组(halogenome),其基因组学研究相应地称作盐基因组学(halogenomics)。如图1所示,研究植物的耐盐性通过无机环境的改变和环境中的其他生命体的参与形成了生态对植物的个体和群体以及家族演化的选择压力,因此,全面地考察植物盐基因组需要连带研究植物个体与其他生物个体间的互作(宏基因组学或元基因组学,metag eno mics),乃至植物与所有相关生物的基因组学——共生总基因组学(hologenomics)[1-7],包括未来人工导入的基因组以及相关的生物演化中的古生物基因组。
本文所述的盐一般是指NaCl,所述的耐盐(halotolerance)广义地包括拒盐植物对盐的屏蔽和阻挡(resistance),泌盐植物对盐的吸收转运(secretion),储盐植物对盐的储存(storage),嗜盐植物(halophyte)对盐的依赖(halophilication)和稀释(dilution)及其在超过其嗜盐阈值时的胁迫反应,植物和菌根、内生菌及环境生物联合对盐进行反应的联纵反应(coalition),浮游植物、具游动精子类的个体植物和以个体死亡及选择性传粉为主要形式的植物群体对高盐环境的避盐逃避(avoidance)等方面。本文对植物个体和群体改良的进展按照上述耐盐反应的各层面主要对新的进展和关键问题进行评述[2, 7]。植物耐盐领域的生态改良方面主要包括例如盐碱乃至重金属污染土地的修复工程、内生菌和环境有益菌的配置、人类?动物?植物?微生物?生态链的总体研究和构建等方面。这些领域超出了共生总基因组学所涵盖的范围,并在近期都取得了很大的进展[8-13],限于篇幅,耐盐植物群体行为和生态改良领域的进展将不在本文论述。
2 植物盐基因组的演化
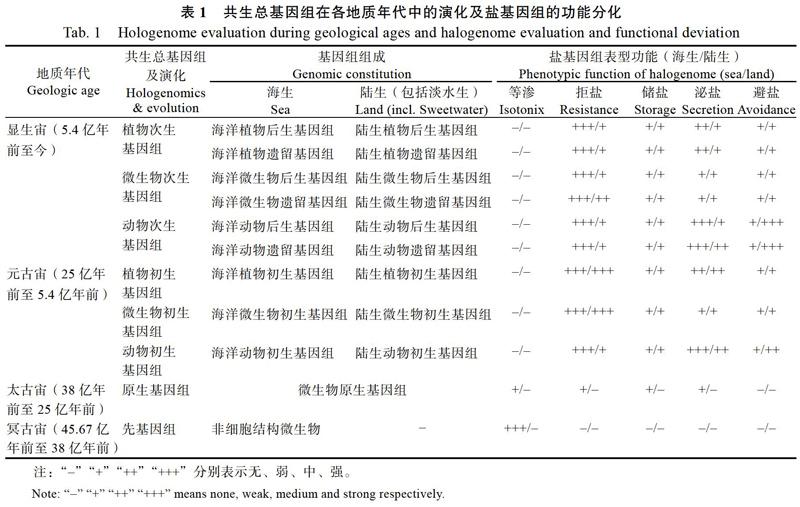
有机分子和无机盐在45亿年前的地球星云形成时就已产生并相伴[14]。从生物演化方面看(表1),45亿年前地球形成,在持续大约6亿多年的冥古宙(Hadean Eon,45.67亿年前至38亿年前)中,生物演化最重要的事件是在地球形成后的1.5亿年就形成了原始海洋。那时没有表面覆土的陆地,海洋形成时的含盐量是目前海水的大约2倍,pH约5.5[15],温度从100 ℃降至大约目前海洋的温度。至太古宙(Archean Eon,38亿年前至25亿年前),膜包被的生物大分子出现,估计只是单层脂类分子形成的膜对以RNA为遗传载体的先基因组(pregenome)的一种隔离和简单包裹,形成非典型细胞结构的细胞雏形。生物演化在地球形成后10亿年的时间里都是在原始海洋中的高浓度盐混合液中实现的。在最早出现的原始细胞中,细胞没有细胞器和细胞核,只含有RNA或RNA-DNA的原基因组(protegenome)核区。由于细胞膜结构简单,盐可以顺利通过膜进入细胞内达到盐的平衡或称稳态(homeostat)(有时也涉及综合渗透压),细胞内外所有物质都面临高盐浓度的胁迫。
3 植物盐基因组的功能
3.1 界面调控
植物细胞对高盐浓度的反应虽然可以归类为图1所示的层次和类别,但是实际发生的事件是多维的网络式多重反馈调控和影响的。反应的起始首先是界面调控。拒盐植物对盐的屏蔽和阻挡(resistance)包括角质层等表面的不渗透能力,膜系统的反转运能力,以及细胞质的高渗溶液拮抗能力。在陆生微管植物类群中,根的出现,包括根冠和根毛的分化,在细胞?组织?器官水平上对盐的胁迫进行有利于植物耐盐的反应,同时许多根的细胞也参与召集根际微生物进行盐的界面调控。高等植物根中的凯氏带,也具有盐阻挡作用。植物适应盐胁迫的各种形态结构的研究为盐基因组的进一步研究奠定了基础[6, 43]。相比动物细胞而言,在植物细胞里首先是要有信号传出以保证细胞壁的完整从而保护细胞膜及整个细胞[44-45]。进入细胞内的高盐浓度也被细胞内膜系统做进一步的界面调控,包括液泡等细胞器和细胞核,离子通道和转运蛋白也在这些膜上分布。胁迫会导致细胞器及其功能的失调,例如光合作用、能量代谢等。在细胞膜结构方面,膜和膜蛋白及其在细胞膜和细胞内膜系统上的表面的电性和其分布都是动态的[46]。膜外侧积聚表面结合密集的Na+的蛋白后,对盐形成梯度屏蔽。其中反转运蛋白在植物的营养和信号转导中起着重要的作用[32]。包括甘油在内的有机分子参与盐胁迫的耐盐性,一般认为是通过渗透压拮抗高盐浓度,但最近发现,其具有分子特异性和参与特定信号传导[47-48]。
3.2 内伤修复
高盐浓度在细胞内形成后会影响细胞结构和生理生化过程。这种过程一般会打破原先的生理生化和生长代谢等一系列平衡,甚至超出其调控的极限而产生负面的损害(例如自由基)。从耐盐的角度看,植物调控的结果,实际上是要看其调节的值域和超出值域时其修复损伤的能力,即内伤修复。在植物细胞膜及细胞壁在受到高盐胁迫后通过体积和构象变化、膨压、成分的改变和损坏等导致的信号传导,以及激素的反应进行适应性生长和修复。这些修复反应重要的一方面是通过渗透压机制补充游离态水进行稀释或补充其他无机或有机分子进行拮抗。细胞还会以细胞膜的质壁分离、细胞壁的变形调整共质体的体积,同时细胞器和内膜系统也会进行游离态水和膜所包被的囊泡体积的调整,首先是保证细胞壁-质膜复合体的完整性,启动相应生长修补程序[44-45]。当这一调控超出临界范围之后,高盐浓度导致的损伤,都不仅仅是物理变型,多是与化学变性相关,对细胞质的电导率、电位和各种盐的平衡及生物有机分子表面的电荷都会产生影响。超出蛋白质结构弹性变量范围的变性和对其酶活及其他功能的阻碍会引发结构的破坏、代谢的紊乱和自由基的产生,并进一步导致膜系统和蛋白质、核酸等其他生物大分子的恶性变性。相关细胞器的功能也受到损害。细胞质K+、Ca2+和甘油等有机分子的招募可以对Na+盐进行拮抗,相应的离子通道蛋白活性提高、有机分子浓度提高,这在一定程度上减轻盐胁迫的加重[49]。同时,抗氧化机制的启动是修复自由基的损失所必须的。自由基的消除,依赖于植物还原性物质含量和运输(招募)及合成能力,并与启动自由基消除代谢能力紧密相关。这种自由基消除途径,往往不是专门针对盐胁迫的,而是与温度、干旱等胁迫乃至形态建成和生理生化过程关联[50]。
3.3 生长调节
生长调节包含多个方面,同时又与盐胁迫的其他反应相关联。生长调节首先是信号的产生和传导及接收,包括内伤修复过程和结果所产生的信号。研究比较深入的信号途径有4大类,包括主要负责盐离子稳态调控的SOS途径(SOS regulatory pathway)[51],主要负责渗透压调控的蛋白激酶途径(protein kinase pathways)[52],还有磷脂信号途径(phospholipid signaling pathway)以及植物激素信号途径[3]。
3.3.1 SOS信号通路 在拟南芥中的盐过度敏感(SOS)基因,由SOS3和SCAPP8、SOS2和SOS1组成的SOS信号通路在调节Na+/K+离子稳态中起着关键作用,是重要的离子胁迫信号通路。SOS3和SCAPP8将盐诱导的Ca2+信号传递给SOS2激酶。SOS3激活并且将SOS2招募到质膜上。在正常情况下,PKS5通过促进SOS2与14-3-3和GIGANTEA(GI)蛋白的相互作用来抑制SOS2的活性;盐胁迫产生的Ca2+信号诱导PKS5和14-3-3的相互作用,导致SOS2-GI/14-3-3复合物释放SOS2,因此SOS2被SOS3激活。另外,SOS2的激酶活性也由蛋白磷酸酶2C ABI2调节。SOS1是一种质膜Na+/H+反转运蛋白,是将多余的Na+排出细胞从而缓解离子胁迫所必需的。在正常条件下,SOS1是自抑制的,在盐胁迫下,其C-末端结构域的Ser1044被SOS2磷酸化,SOS1活性被激活。值得注意的是,sos突变体仅在高盐胁迫下表现出超敏表型,而在甘露醇或聚乙二醇(PEG)的渗透胁迫下正常生长,表明SOS信号通路特别参与了对离子胁迫的反应[3, 7, 51-53]。
3.3.2 磷脂和蛋白激酶信号转导途径 在盐胁迫期间,膜磷脂的修饰起到信号传递的作用。在盐胁迫和渗透胁迫下几分钟内,会产生多种磷脂信号,包括多磷肌醇和磷脂酸(PA)。PA的形成是盐胁迫下钙信号传导的另一个下游反应。PA本身可以结合并影响ABA信号和生长素转运中的各种下游蛋白,这两种激素信号通路在盐胁迫信号传导中起主要作用。PA还影响了通过MITOGEN- ACTIVATED PROTEIN KINASE6的鈉转运,其下游靶点是SOS1/NHX7。总之,PA在盐胁迫和渗透胁迫期间影响蛋白质的定位和活化,从而影响激素信号和钠转运[54]。
3.3.3 植物对盐胁迫反应中ROS信号转导 盐胁迫与植物细胞快速产生活性氧有关。ROS水平升高作为一种信号,由ROS传感器/受体感知,并被转导以调节植物在盐胁迫下的反应、生长和发育。MAPKKK-MAPKK-MAPK(丝裂原活化蛋白激酶)级联,作为重要的ROS信号转导途径,参与ROS稳态、离子稳态和植物生长发育的调控。盐胁迫诱导ROS的积累,ROS对植物具有氧化胁迫诱导的毒性作用。除了毒性作用外,ROS还作为信号分子对环境刺激做出反应。活性氧必须存在于植物细胞中的适当水平。解毒信号通路参与控制细胞在各种应激条件下ROS水平的动态平衡。一旦细胞内稳态重建,应激损伤的机会就会降低[54]。
3.3.4 植物激素信号途径 植物应对盐胁迫的激素调控涉及目前发现的几乎所有植物内源激素及其相互间的整合和协调,例如包括脱落酸(ABA)、茉莉酸(JA)、生长素(IAA)、赤霉素(GA)、乙烯(ET)和水杨酸(SA)。在这些激素中,ABA对非生物胁迫的响应最为重要。渗透胁迫导致根和叶组织中ABA浓度迅速(几分钟内)大量的增加,同时ABA是导致气孔关闭的关键信号分子之一[55]。
茉莉酸及其衍生物统称为茉莉素,是植物体内一类重要的脂质激素。茉莉素作为抗性激素在植物抵抗生物胁迫和非生物胁迫过程中发挥重要作用。盐胁迫下许多JA生物合成基因上调,JA信号通路参与盐胁迫响应基因的调控[56],例如在小麦中TaAOC1和TaOPR1分别通过JA和ABA信号通路调控耐逆关键转录因子MYC2,从而提高小麦的耐盐能力[57]。
生长素和赤霉素作为调节植物生长发育的重要激素,也参与非生物胁迫下的生长调节。赤霉素信号转导过程中的抑制子DELLA蛋白通过限制高盐下的生长来促进植物的存活[58-59]。
乙烯参与调节植物生长发育以及对干旱、高盐、病原菌侵染等一系列胁迫的抗性反应。研究表明乙烯前体1-氨基环丙烷-1-羧酸(ACC)可提高盐胁迫耐受性;而乙烯信号通路相关基因(如ETR1、EIN4、EIN2和EIN3)的突变会导致植物对高盐的敏感反应[56, 60]。
水杨酸是一种酚类激素,作为植物应对生物胁迫及非生物胁迫反应的重要信号分子,能诱导植物对这些胁迫产生抗性反应。SA在盐胁迫耐受中的作用表现为其能够提高渗透保护剂(如脯氨酸、多胺)的积累,并在高盐度下增强抗氧化酶活性。用SA对植物进行预处理也减少了NaCl诱导的K+外流和H+内流,因此促进了植物对高盐度的适应[61]。
由于盐胁迫是多维的,植物也在不同结构尺度、不同生长发育时期和不同环境中进行反应,因此各种植物激素实际上都参与了盐胁迫相关的信号传导,其作用在不同植物和不同器官中所导致的植物个体水平的耐盐效果各有不同,同时在嗜盐植物和非嗜盐植物中,依据其耐盐策略和耐盐阈值外的内伤修复内容,相同的激素会产生相反的效果。例如生长类激素一般是促进生长而达到稀释盐的作用,或负调控时减少生长以集中能量用于胁迫的修复和耐性。
植物个体在可以移动的情况下,例如浮游植物、具游动精子类的个体植物和以个体死亡及选择性传粉为主要形式的植物群体对高盐环境的避盐逃避(avoidance),是盐基因组在个体和群体与环境的互作和最终生境的选择层面上的功能。这方面的研究目前还是空白。植物的群体行为多是生态学的统计内容,显示物种在不同时空及生态型中的分布、迁移和消长,目前还很少进行组学的机制研究。
3.4 遗传变异
植物對盐胁迫而产生的遗传变异有耐盐和不耐盐两个方向,其遵从一般的遗传变异规律,在植物中的研究已经很多,成为教科书的经典内容[6, 62]。在生物演化中,陆生植物,特别是甜土植物离开海生和高盐土壤后产生的次生基因组,由于失去盐胁迫选择压力,其中大部分植物失去耐盐特性,现存的陆生植物,虽然每个大分类群里都有嗜盐植物,但是绝大多数都成为了非耐盐的所谓甜土植物。特别是部分栽培作物,通过产量等的定向育种,加速失去了耐盐基因或耐盐调控机制,例如番茄的SOS1和HAK 20基因[42, 63]。
另一方面,耐盐的遗传变异也广泛存在。新的耐盐基因也在挖掘中[50, 64-65]。例如植物感受盐胁迫的受体[66]。这方面的研究受到人类育种的巨大影响,对植物的遗传变异产生了数量级级别的加速改变。
4 表观遗传学研究
从地球和分子演化的角度分析,在原始海洋的高盐度水环境中,先基因组在大量的RNA环境中产生。随着海水盐浓度的降低,RNA酶的增多和活性的升高导致核心遗传信息载体由RNA交接给更稳定的DNA,但RNA仍然保持为遗传变异最活跃的分子,并以较短的多样性序列担负遗传信息的储存、调取、使用、维护和转换的几乎所有调控功能,即表观遗传(epigenetics)。
近年来表观遗传学和表观遗传在植物抗性方面的研究取得重要进展[67-68]。自从miRNA被发现[69],大量的研究揭示了miRNA是一类经过一系列核酸酶的剪切加工而产生的长度约为21~24个核苷酸(nt)并能够转录后调节基因表达的内源性非编码的小RNA。植物miRNA通过调节靶基因的表达进而参与调控植物的多种生理代谢活动,参与植物对生物和包括盐胁迫的非生物胁迫反应并在植物盐胁迫中的作用已得到转基因验证[64, 70]。
植物耐盐性是个受多基因、多途径调控的复杂网络,许多与盐胁迫有关的miRNAs被预测和鉴定出来,其靶基因的实验验证和其调控其靶基因的表达进而参与植物对抗盐胁迫的机制及其应用,是未来研究的重要方向。从生物演化的角度看,RNA很可能在DNA出现以后进行了长期的生长发育的调控特化并形成目前一系列例如mRNA、tRNA、rRNA、siRNA、miRNA和大量其他RNA的品种和调控网络[71]。陆地植物在奥陶纪前后产生并大量在陆地上演化[20],同时miRNA恰恰也在此时出现大规模的分歧演化[72],可以推测miRNA多靶点和多形式的网络化调控的可塑性很强,可挖掘耐盐调控的潜力很大。从宏基因组和共生总基因组角度看,内生菌和根际微生物对植物耐盐性的提高也可能由水平基因转移和小RNA传递进行调控和变异实现[27, 72]。
表观遗传的耐盐性可能与其他胁迫抗性类似,能够遗传后代,这使历史上进化论的获得性遗传理论得到部分复活。在植物耐高温等方面已经发现了这种遗传现象,例如将高温记忆传递给下一代,形成一个由组蛋白去甲基化酶、染色质重塑因子、转录因子、泛素连接酶和小RNA共同组成的表观调控网络,维持植物对高温记忆传代的机制[73]。但是有性生殖对部分类型的表观遗传的回零作用对其代传设立了门槛[74]。表观遗传学的代传机制和稳定性尚有待进一步研究[67]。
5 组学及耐盐育种
植物盐基因组学离不开植物内外环境。宏基因组(metagenome)研究揭示,植物和其内生菌、菌根、根际微生物及其他环境生物也会参与植物耐盐的调控,对盐进行联纵反应(coalition)。在菌根提高植物耐盐性方面已经取得大量研究成果[8]。在盐胁迫地区的植株根部土壤发现大量增强植株耐盐性和促进生长的菌类,以及包含芽孢杆菌、假单胞菌、固氮杆菌和肠杆菌等植株抗盐的多种内生菌,诱导渗透压调节物质和抗氧化化合物的积累,提高抗氧化系统以及提供激素调节和植物营养,从多方面增强植物的抗盐性[26-27, 39, 75]。大多数耐盐细菌对IAA和ACC脱氨酶的产生呈阳性反应,是耐盐菌促进植物生长的重要性状[76]。这种植物和微生物互作形成对盐的联纵反应在过去研究较少,各种耐盐基因在组学水平上的评价尚未展开。
从信号传导的角度看,信号传导的一个重要问题是细胞由谁感知并启动最初的信号传导的。大量的研究是受动物研究的启发进行Ca2+及其相关蛋白的研究,但迄今没有发现在植物里找到与动物细胞高同源性的电压依赖型阳离子通道及其功能模型,植物的钙信号途径中只是钙离子充当第二信使。目前发现的植物盐胁迫特异性的启动信号是在细胞膜和内质网膜上分布的MOCA1(monocation induced Ca2+ increases 1),其编码肌醇磷酸化神经酰胺葡萄糖醛酸转移酶(IPUT1)催化合成鞘脂糖基肌醇磷酰神经酰胺(GIPC),后者与Na+结合,但同时可能引起膜极性的变化,从而打开膜上的Ca2+通道,导致细胞质Ca2+浓度增加,继而激活SOS通路泵出Na+盐,以适应盐胁迫环境。这一途径与K+、Li+、ABA及渗透压胁迫等无关,是目前唯一发现的植物盐胁迫特异性起始信号,而且是脂类分子作为第一信使[66]。
信号传导的另一个重要问题是细胞是如何感知生长并进而调控生长的。在酵母中,细胞周期的启动由细胞生长到足够尺度而开始。激酶Gin4和Hsl1能够感知出芽生长,当这两个激酶受抑制时则出现长时间的有丝分裂延迟和过度的出芽生长。进一步研究发现,当出芽生长时,这两个激酶呈相关的逐步的高磷酸化,并且其中Gin4逐步的高磷酸化需要与不断释放到出芽生长处的阴离子磷脂相结合,因此细胞感知生长的机制很可能是释放到出芽生长处的脂质产生了生长依赖性的信号,这个信号被细胞通过激酶感知了生长[52]。在拟南芥中,盐胁迫会削弱细胞壁,这种细胞壁的损伤可通过激活阻止细胞爆裂的FER信号通路来感知,并也会触发短暂的Ca2+增加以及自身的膜内移。其他胁迫和信号途径,以及膜和细胞壁的组分,都可能与之相关联,并启动细胞及植物体的生理生化过程[44]。盐胁迫的调控信号途径包括水通道蛋白、非选择性阳离子通道以及更综合的非生物胁迫及信号传导与生长调节[45, 54]。
泌盐植物对盐的吸收转运(secretion)反转运蛋白SOS1调控作用不仅在甜土植物中发挥作用,在泌盐型耐盐植物(recretohalophyte)中也发挥重要作用。菊科植物花花柴(Karelinia caspia)的长距离泌盐中SOS1在Na+的转运和分泌、K+的吸收、整株植物的盐稳态中都发挥作用[51-52]。进行嗜盐植物(halophyte)对盐的依赖(haloph ilic ation)和稀释(dilution)、储存(storage)及其在超过其嗜盐阈值时胁迫反应以及其与生态因子的互作的比较研究对陆生甜土植物的耐盐性理解和研发具有重要作用[1, 6]。
多数甜土植物没有能够从古菌和原绿藻的共同祖先的原基因组中继承下来耐盐基因组,即使传承下来的盐基因组成分也由于次生基因组的产生而被稀释或对新演化出的生长发育代谢失去靶针对性。目前所发现的大量miRNA对基因表达、转录和翻译过程的调控,可能反映了原基因组来源于RNA[71],而后来只有核基因组被更稳定的DNA取代,负责核心遗传信息储存,包括信号传导等其他功能仍主要由RNA负责执行(线粒体和质体基因组类似)。
无机环境的改变和环境中的其他生命体的参与,形成了生态对植物的个体和群体以及家族演化的作用力,因此,全面研究植物的耐盐性,必然从过去的植物形态结构生理及植物个体基因组学的研究,发展到植物个体与其他生物个体间的互作(宏基因组学或元基因组学,metage no mics)的研究,乃至植物与所有相关生物的基因组学——共生总基因组学(hologenomics)[1]研究,其中还应包括未来人工导入的基因组以及相关的生物演化中的古生物基因组(图2)。耐盐反应在个体、群体和生态环境不同层面的复杂性和其进化时空的复杂性,要求植物耐盐的机制研究应与更广泛的、多维的视角对网络化的调控进行整体的思考,以便在形态建成、生理生化、生长发育、遗传变异、繁殖、生物量、群落配置,以及生物工程和农业与环境工程的高效性等方面得到更高的受益。本文扩展的共生总基因组学,在原有的基础上包揽了必要的人工合成基因组和古基因组,即把生物演化和合成生物学的内容纳入系统生物学研究。
组学研究结合耐盐育种在许多重要植物中都取得了进展[77-80]。传统耐盐筛选和育种是商业公司的产业化行为,分子育种的实验验证为日常的产业化育种提供了有力参考。耐盐基因和修复基因及其调控网络的挖掘、刚刚开始的表观遗传学研究、盐基因组学和宏基因组学及扩展的共生总基因组学(图1、图2)将成为植物耐盐育种的有力指导工具。在非耐盐植物中发现耐盐基因,这听起来是个悖论。例如文心兰和拟南芥都是不耐盐植物,而其抗坏血酸过氧化物酶(ascorbate peroxidase, APX)被發现具有耐盐作用,并在文心兰中比其在拟南芥中参与更多的催化[50]。但从植物基因组演化的角度分析,植物初生基因组适应海洋高盐环境的耐盐基因在植物进入陆生环境中逐渐被次生基因组丢失或与新生的形态建成和生理生化途径脱钩而沉默或转为他用。同时,与海洋几乎恒定的盐浓度不同,陆地盐浓度波动大,不同种类的盐浓度波动更大,这种新型的变幅盐胁迫是催生陆生植物盐基因组演化的新型驱动力。
以宏基因组的视角进行耐盐育种和发掘耐盐资源近年来取得很大进展[1, 6, 8-12]。许多例如盐单胞菌(Halomonas)、芽孢杆菌(Bacillus)、链霉菌(Streptomyces)、海洋细菌(Oceanobacillus)和假单胞菌(Pseudomonas)等属的耐盐微生物,可通过信号传导、激素调节等途径提高植物的耐盐性[27, 29, 77],这显示了宏基因组学和共生总基因组学的强大作用。自从表观遗传的传代被提出后,表观遗传机制对后代的影响及对演化的贡献被广泛接受。表观遗传的机制是否能进行植物与内生菌和外生菌即其他环境微生物间的互作和稳定的遗传搭配尚需研究,这可能为耐盐育种提供新的线索。在今后的盐基因组“复壮”中,使陆生植物继承和衔接初生基因组的耐盐基因,使植物次生基因组发展出新的耐盐机制,以及从生境中配置耐盐的协同者,可能是育种成功的大概率事件。
6 展望
综上所述,盐基因组指自生物演化以来生物体对盐感受、信号转导、拒绝、转运、储存、分泌及相关的调节和互作等反应的全部基因组结构及功能。植物盐基因组涉及个体、群体、生态以及进化和包括表观遗传在内的多种遗传、变异、生长、发育、代谢和应激机制,并与基因编辑和未来的人工合成基因以及与农业耕作和生态修复工程息息相关。以多维的视角和系统的方法研究植物对高盐浓度胁迫的反应,以盐基因组为线索把古生物、包括内生菌和外生菌的微生物、动物和人类活动有机地联系起来进行动态的系统研究,可望为植物耐盐育种带来新的思路和途径。
参考文献
罗森伯格, 齐尔博-罗. 共生总基因组——人类、动物、植物及其微生物区系[M]. 孟 和, 译. 北京: 科学出版社, 2019.
Munns R, Teste M. Mechanisms of salinity tolerance[J]. Annual Review of Plant Biology, 2008, 59: 651-681.
Zhu J K. Salt and drought stress signal transduction in plants[J]. Annual Review of Plant Biology, 2002, 53: 247- 273.
李鳳霞. 盐碱地土壤微生物多样性特征研究[M]. 北京: 阳光出版社, 2015.
刘润进, 王 琳. 生物共生学[M]. 北京: 科学出版社, 2018.
赵可夫, 范 海. 盐生植物及其对盐渍生境的适应生理[M]. 北京: 科学出版社, 2005.
Zhu J K. Plant salt tolerance[J]. Trends in Plant Science, 2001, 6(2): 66-71.
唐 明. 菌根真菌提高植物耐盐性[M]. 北京: 科学出版社, 2010.
陈文新, 汪恩涛. 中国根瘤菌[M]. 北京: 科学出版社, 2011.
斯沃伊斯. 污染场地处置——从理论到实践应用[M]. 朱勇兵, 赵三平, 刘晓东, 译. 北京: 国防工业出版社, 2017.
李 隆. 间套作体系豆科作物固氮生态学原理与应用[M]. 北京: 中国农业大学出版社, 2013.
罗胜联, 刘承斌, 罗旭彪. 植物内生菌修复重金属污染理论与方法[M]. 北京: 科学出版社, 2013.
郝 闯, 唐 兵, 唐晓峰. 嗜盐微生物的工业应用研究及进展[J]. 生物资源, 2019, 41(4): 281-288.
Spezia R, Jeanvoine Y, Scuderi D. Ion–molecule reactions as a possible synthetic route for the formation of prebiotic molecules in space[M]//Pontarotti P. Origin and evolution of biodiversity, Cham: Springer, 2018.
Hazen R M. The story of earth: The first 4.5 billion years, from stardust to living planet[M]. New York: Penguin Group, 2012: 89-92.
Wacey D, Kilburn M R, Saunders M, et al. Microfossils of sulphur-metabolizing cells in 3,4-billion-year-old rocks of Western Australia[J]. Nature Geoscience, 2011, 4(10): 698-702.
Quayle J R, Ferenci T. Evolutionary aspects of autotrophy[J]. Microbiological Reviews, 1978, 42(2): 251-273.
刘浩东, 闫荣曼, 张传伦. 海洋浮游古菌MGII的研究进展[J/OL]. 微生物学报, 2020. [2020-08-17]. https://doi.org/ 10.13343/j.cnki.wsxb.20200105.
Jiao C, S?rensen I, Sun X, et al. The Penium margaritaceum genome: Hallmarks of the origins of land plants[J]. Cell, 2020, 18(5): 1097-1111.
Morris J L, Puttick M N, Clark J W, et al. The timescale of early land plant evolution[J]. Proceedings of the National Academy of Sciences, 2018, 115(10): 2274-2283.
赖晓娟, 陈海敏, 杨 锐, 等. 藻类基因组研究进展[J]. 遗传, 2013, 35(6): 735-744.
李星学, 周志炎, 郭双兴. 植物界的发展和演化[M]. 北京: 科学出版社, 1981.
Pellicer J, Hidalgo O, Dodsworth S, et al. Genome size diversity and its impact on the evolution of land plants[J]. Genes, 2018, 9(2): 88.
Panchy N, Lehti-Shiu M D, Shiu S H. Evolution of gene duplication in plants[J]. Plant Physiology, 2016, 171(4): 2294-2316.
Bowles A M C, Bechtold U, Paps J. The origin of land plants is rooted in two bursts of genomic novelt[J]. Current Biology, 2020, 30: 530-536.
Keeling P J, Palmer J D. Horizontal gene transfer in eukaryotic evolution[J]. Nature Reviews Genetics, 2008, 9(8): 605-618.
Tiwari P,Bae H. Horizontal gene transfer and endophytes: An implication for acquisition of novel traits[J]. Plants, 2020(9): 305-316.
Zhong Y, Cheng Z M. A unique RPW8-encoding class of genes that originated in early land plants and evolved through domain fission, fusion, and duplication[J]. Scientific Reports, 2016(6): 32 923.
Zerulla K, Soppa J. Polyploidy in haloarchaea: Advantages for growth and survival[J]. Frontiers in Microbiology, 2014(5): 274.
Xiao Y, Xu P, Fan H, et al. The genome draft of coconut (Cocos nucifera)[J]. Giga Science, 2017, 6(11): 1-11.
李 丹, 陳晓慧, 赖钟雄. 热带植物基因组研究进展[J]. 热带作物学报, 2019, 40(10): 1875-1888.
Apse M P. Salt tolerance conferred by overexpression of a vacuolar Na+/H+ antiport in Arabidopsis[J]. Science, 1999, 285: 1256-1258.
王伏雄, 陈祖铿. 银杏胚胎发育的研究——兼论银杏目的亲缘关系[J]. 植物学报, 1983, 25(3): 199-207, 295-298.
Xuhan X, Lammeren A A M Van. Structural analysis of embryogenesis and endosperm formation in celery-leafed buttercup (Ranunculus sceleratus L.)[J]. Acta Botanica Neerlandica, 1997, 46(3): 291-301.
Schel J H N, Kieft H, Lammeren A A M Van. Interactions between embryo and endosperm during early developmental stage of maize caryopses (Zea mays)[J]. Canadian Journal of Botany, 1984, 62(12): 2842-2853.
XuHan X, Christian Brière, Nathalie Vallée, et al. In vivo labeling of sunflower embryonic tissues by fluorescently labeled phenylalkylamine[J]. Protoplasma, 1999, 210(1-2): 52-58.
胡适宜. 被子植物胚胎学[M]. 北京: 人民教育出版社, 1983.
阎秀峰, 庞秋颖. 盐生植物盐芥的组学分析[M]. 北京: 科学出版社, 2012.
Lyu H, He Z, Wu C I, et al. Convergent adaptive evolution in marginal environments: unloading transposable elements as a common strategy among mangrove genomes[J]. New Phytologist, 2018, 217(1): 428-438.
Nguyen H T, Stanton D E, Schmitz N, et al. Growth responses of the mangrove Avicennia marina to salinity: Development and function of shoot hydraulic systems require saline conditions[J]. Annals of Botany, 2015, 115: 397-407.
杨传平. 柽柳耐盐抗旱分子基础研究[M]. 北京: 科学出版社, 2015.
Wang Z, Hong Y, Li Y, et al. Natural variations in SlSOS1 contribute to the loss of salt tolerance during tomato domestication[J/OL]. Plant Biotechnology Journal, 2020. [2020-08-17]. https://doi.org/10.1111/pbi.13443.
谷 俊, 耿 贵, 李冬雪, 等. 盐胁迫对植物各营养器官形态结构影响的研究进展[J]. 中国农学通报, 2017, 33(24): 62-67.
Feng W, Kita D, Peaucelle A, et al. The FERONIA receptor kinase maintains cell-wall integrity during salt stress through Ca2+ signaling[J]. Current Biology, 2018, 28: 666-675.
Ismail A, El-Sharkawy I, Sherif S. Salt stress signals on demand: Cellular events in the right context[J]. International Journal of Molecular Sciences, 2020, 21(11): 3918.
Yeung T, Gilbert G E, Shi J, et al. Membrane phosphatidylserine regulates surface charge and protein localization[J]. Science, 2008, 319(5860): 210-213.
Bahieldin A, Sabir J S M, Ramadan A, et al. Control of glycerol biosynthesis under high salt stress in Arabidopsis[J]. Functional Plant Biology, 2014, 41(1): 87-95.
Xu, L, Yeu Q, Bian F, et al. Melatonin enhances phenolics accumulation partially via ethylene signaling and resulted in high antioxidant capacity in grape berries[J]. Frontiers in Plant Science, 2017(8): 1426-1437.
趙振杰, 张海龙, 王明晶, 等. 植物耐盐性相关细胞内pH和离子稳态的调控机制[J]. 植物生理学报, 2020, 56(3): 337-344.
Chin D C, Kumar R S, Suen C S, et al. Plant cytosolic ascorbate peroxidase with dual catalytic activity modulates abiotic stress tolerances[J]. Science, 2019, 16: 31-49.
Guo Q, Meng L, Han J, et al. SOS1 is a key systemic regulator of salt secretion and K+/Na+ homeostasis in the recretohalophyte Karelinia caspia[J]. Environmental and Experimental Botany, 2020, 177: 104 098.
Jasani A, Huynh T, Kellogg D R. Growth-dependent activation of protein kinases suggests a mechanism for measuring cell growth[J]. Genetics, 2020, 215(3): 729-746.
Yang Z, Wang C, Xue Y, et al. Calcium-activated 14-3-3 proteins as a molecular switch in salt stress tolerance[J]. Nature Communication, 2019(10): 1199.
Zelm E V, Zhang Y X, Testerink C. Salt tolerance mechanisms of plants[J]. Annual Review of Plant Biology, 2020, 71: 24.1-24.31.
Osakabe Y, Yamaguchi-Shinozaki K, Shinozaki K, et al. ABA control of plant macroelement membrane transport systems in response to water deficit and high salinity[J]. New Phytologist, 2014, 202(1): 35-49.
Kazan K. Diverse roles of jasmonates and ethylene in abiotic stress tolerance[J]. Trends in Plant Science, 2015, 20(4): 219-229.
Zhao Y, Dong W, Zhang N, et al. A wheat allene oxide cyclase gene enhances salinity tolerance via jasmonate signa ling[J]. Plant Physiologist, 2014, 164: 1068-1076.
Colebrook E H, Thomas S G, Phillips A L, et al. The role of gibberellin signalling in plant responses to abiotic stress[J]. Journal of Experimental Biology, 2014, 217: 67-75.
Achard P, Cheng H, Grauwe D L, et al. Integration of plant responses to environmentally activated phytohormonal signals[J]. Science, 2006, 311(5757): 91-94.
Cao W H, Liu J, He X J, et al. Modulation of ethylene responses affects plant salt-stress responses[J]. Plant Physiologist, 2007, 143: 707-719.
Jayakannan M, Bose J, Babourina O, et al. Salicylic acid improves salinity tolerance in Arabidopsis by restoring membrane potential and preventing salt induced K+ loss via a GORK channel[J]. Journal of Experimental Botany, 2013, 64(8): 2255-2268.
趙可夫,李法曾,张福锁. 中国盐生植物[M]. 2版. 北京: 科学出版社, 2013,
Wang Z, Hong Y, Zhu G, et al. Loss of salt tolerance during tomato domestication conferred by variation in a Na+/K+ transporter[J]. The EMBO Journal, 2020, 39(10): e103256.
Li B, Duan H, Li J. et al. Global identification of miRNAs and targets in Populus euphratica under salt stress[J]. Plant Molecular Biology, 2013, 81(6): 525-539.
Xiang Y, Jiménez-Gómez J M. SlHAK 20: a new player in plant salt tolerance[J]. The EMBO Journal, 2020, 39(10): e104997.
Jiang Z, Tao M, Zhou X, et al. Plant cell-surface GIPC sphingolipids sense salt to trigger Ca2+ influx[J]. Nature, 2019, 572(7769): 341-346.
Li J, Yang D L, Huang H, et al. Epigenetic memory marks determine epiallele stability at loci targeted by de novo DNA methylation[J]. Nature Plants, 2020(6): 661-674.
刘小云, 吴邦庭, 邱 璐. 植物逆境响应的表观遗传调控研究进展[J]. 江汉大学学报(自然科学版), 2018, 46(2): 180-187.
Reinhart B J, Slack F J, Basson M, et al. The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans[J]. Nature, 2000, 403(6772): 901-906.
Ding D, Zhang L, Wang H, et al. Differential expression of miRNAs in response to salt stress in maize roots[J]. Annals of Botany, 2009, 103(1): 29-38.
Atkins J F, Gesteland R F, Cech T R. RNA worlds: From lifes origins to diversity in gene regulation[M]. New York: Cold Spring Harbor Laboratory Press, 2010.
Voinnet O. Origin, biogenesis, and activity of plant microRNAs[J]. Cell, 2009, 136(4): 669-687.
Ratajczak M Z, Ratajczak J. Horizontal transfer of RNA and proteins between cells by extracellular microvesicles: 14 years later[J]. Clinical and Translational Medicine, 2016(5): 7.
Lang-Mladek C, Popova O, Kiok K, et al. Transgenerational inheritance and resetting of stress-induced loss of epigenetic gene silencing in Arabidopsis[J]. Molecular Plant, 2010, 3(3): 594-602.
Crevillen P, Yang H, Cui X, et al. Epigenetic reprogramming that prevents transgenerational inheritance of the vernalized state[J]. Nature, 2014, 515(7528): 587-590.
Vaishnav A, Shukla A K, Sharma A, et al. Endophytic bacteria in plant salt stress tolerance: Current and future prospects[J]. Journal of Plant Growth Regulation, 2019, 38(2): 650-668.
Etesami H, Glick B R. Halotolerant plant growth-promoting bacteria: Prospects for alleviating salinity stress in plants[J]. Environmental and Experimental Botany, 2020, 178: 104 124.
劉艳丽, 许海霞, 刘桂珍, 等. 小麦耐盐性研究进展[J]. 中国农学通报, 2008(11): 202-207.
连 勇, 刘富中, 田时炳, 等.“十二五”我国茄子遗传育种研究进展[J]. 中国蔬菜, 2017(2): 14-22.
田丽丽, 黄建安, 刘仲华. 茶树抗盐碱研究进展[J]. 植物生理学报, 2017, 53(2): 167-173.

