综合生物信息学分析预测胃癌预后关键基因的研究
杨 倩,齐明明,董卫国
1.武汉大学人民医院消化内科, 湖北 武汉 430060; 2.消化系统疾病湖北省重点实验室; 3.贵州省人民医院消化内科
目前胃癌在全球癌症排名第五位(占所有癌症病例的5.7%),是癌症死亡(占所有癌症死亡的8.2%)的第三大原因[1]。胃癌发病机制复杂,研究表明幽门螺杆菌(H.pylori)感染是胃癌的危险因素之一。胃癌发病隐匿,难于在早期阶段发现。胃十二指肠镜检查是目前筛查早期胃癌的一种敏感手段,但由于内镜检查不耐受、心理恐惧等原因,使胃镜检查的价值未得到充分发挥。鉴于胃癌的高发病率和死亡率,利用全球癌症基因组数据库公共资源筛选出有利于胃癌早期诊断的特异性分子生物标志物及探究其潜在的分子机制具有一定的临床意义。
利用基因芯片和基因检测技术,可以发现新的疾病亚型,识别新的肿瘤生物标志物以预测预后。随着基因芯片的广泛应用,在线公共数据库可以提供大量的基因组数据为分子细胞学研究提供有价值的信息。然而由于样本和单队列研究的异质性所得结果往往有限或不一致,因此,在胃癌中暂无一致可靠的生物学标记。本研究分别从GEO及TCGA数据库下载多个胃癌的基因表达谱数据,筛选出差异表达基因,通过利用生物信息学方法对差异基因的生物功能学及相关信号通路分析以研究胃癌致病机理,所得到的相关基因和通路可能成为胃癌预后判断因子及潜在治疗靶标。
1 材料与方法
1.1 数据下载及数据处理从NCBI-GEO[2](http://www.ncbi.nlm.nih.gov/geo)数据库中获得胃癌及其配对癌旁组织基因表达谱数据集GSE19826(12个胃癌和癌旁配对样本)、GSE79973(10个胃癌及癌旁配对组织)[3]。基于GPL570[HG-U133_Plus_2]Affymetrix Human Genome U133 Plus 2.0 Array 平台,根据相应平台的注释信息,将探针转化为相应的基因ID。然后利用Perl对芯片数据合并后用R软件(版本3.6.1)sva包对数据进行批次矫正。从TCGA(https://portal.gdc.cancer.gov/)下载胃癌患者相关临床资料。
1.2 差异表达基因筛选利用R软件中Limma包(Version 3.40.2)进一步筛选出胃癌与癌旁组织中差异表达基因,筛选条件:|log2FC|>1.5,矫正后P值(adj.P)<0.05。
1.3 构建蛋白互作网络与筛选核心基因利用STRING[4](http://string-db.org)在线检索工具对筛选出的差异基因进行检索,预测互作网络。设置信评分>0.7。 利用Cytoscape软件绘制蛋白互作网络,并用其中的分子复合体检测(MCODE)(版本1.4.2)及cytoHubba插件提取复杂网络,筛选标准如下:“MCODE 评分 >10”,Hubba 节点:“Top 15连接度最高节点”,可视化条件:“显示最短路径”。
1.4 GO分析和KEGG富集分析利用 DAVID[5](http://david.ncifcrf.gov)(版本 6.7)数据库对获取差异基因进行GO分析[6]和KEGG富集分析[7],并用ImageGP (http://www.ehbio.com/imageGP/)进行可视化。
1.5 关键基因表达验证
1.5.1 关键基因转录水平表达与总体生存分析:利用cBioPortal(http://www.cbioportal.org)在线平台对中心基因及其总体生存情况进行分析,数据来源于TCGA RNA-SeqV2胃癌患者样本(412个)。GEPIA[8](http://gepia.cancer-pku.cn/)数据库验证LUM、SERPINH1、TIMP1等基因表达,并通过使用箱线图来可视化基因在胃癌和癌旁胃组织中表达差异及其与临床分期关系。筛选条件设置:(1)“基因表达分析:()”;(2)“箱式图”;(3)“基因:LUM/SERPINH1/TIMP1”;(4)“|log2FC|差异倍数截断值:2,Pvalue截断值:0.001”;(5)“肿瘤类型:胃腺癌(STAD)”;(6)“对应TCGA正常组及GTEx数据”,设置信评分>0.7。
1.5.2 关键基因蛋白水平表达分析:利用HPA[9](The Human Protein Atlas, https://www.proteinatlas.org/)获取胃癌患者和非癌患者的免疫组化数据,进一步验证LUM蛋白的表达。
1.5.3 关键基因表达与临床病理相关性分析:采用Cox回归分析TCGA患者的临床病理特征与总生存期的关系;采用多因素Cox分析比较LUM表达与其他临床特征(年龄、性别、分期、分级、肿瘤原发灶、淋巴结状态、远处转移状态)对生存的影响。LUM表达的截断值为其中位值。统计分析利用R软件完成。
1.5.4 关键基因通路富集分析:采用基因富集分析(Gene Set Enrichment Analysis,GSEA)(http://software.broadinstitute.org/gsea/register.jsp)识别胃癌中LUM基因差异激活的信号通路,以LUM的表达中位值为截断值将其分为高、低两组,在两组数据集之间进行基因富集分析。以LUM的表达水平作为表型标签,利用名义P值和归一化富集评分(NES)对各表型中富集的途径进行排序,每个分析进行1 000次基因组排列。
2 结果
2.1 差异表达基因的筛选将GSE19826、GSE79973数据集微阵列结果合并去除批次效应后得到287个差异表达基因,其中胃癌组织与非癌组织间下调基因133个,上调基因154个(见图1)。
2.2 差异基因互作蛋白分子网络构建及分析利用STRING构建差异基因的蛋白质互作网络(见图2A),并列出该网络中排名前20紧密互作基因,如图2B中显示,在该网络中与FN1临近基因有25个。接着利用Cytoscape中MCODE插件提取Mcode 1 Scores 12.522(见图2C),再用CytoHubba插件获得Mcode 1中“shortest path”互作最短路径,如图2D所示,在最短路径网络中获得的基因与各种类型胶原蛋白相互作用密切,按照互作等级评分依次为FN1、FBN1、COL1A1、COL1A2、COL5A1、COL3A1、COL5A2、COL8A1、COL11A1、SERPINH1、COL6A3、LUM、COL2A1、TIMP1、COL10A1。于是我们下一步将对该组网络基因进行GO分析和KEGG富集分析,并对其中FN1、FBN1、LUM、SERPINH1、TIMP1等5个与胶原蛋白互作的基因进一步验证。
2.3 中心基因生物功能及生存分析为了进一步分析互作最短路径基因的生物功能,我们使用DAVID对其进行了GO分析和KEGG富集分析。结果表明,这些基因主要富集于胶原蛋白合成代谢、细胞外基质、细胞黏附(见图3A)。GO分析结果显示,这些基因主要富集在胶原蛋白组织和细胞外基质的生物代谢过程,分子功能(MF)的变化主要富集于生长因子结合及细胞外基质结合分子活性。细胞成分(CC)的变化主要集中在胶原蛋白及蛋白质细胞外基质形成。KEGG通路分析发现,基因富集于黏着斑及ECM受体相互作用。 然后利用cBioPortal在线平台对15个最短路径基因进行总体生存分析。结果显示,最短路径网络基因表达差异的胃癌患者表现出更差的总体生存率(见图3B,P=0.0187)及更低的疾病无进展率(见图3C,P=0.0204)。通过上述分析我们发现,FN1、FBN1、SERPINH1、LUM、TIMP1在网络中是与胶原蛋白相互作用关系密切的基因,并且可能与胃癌患者的生存相关。
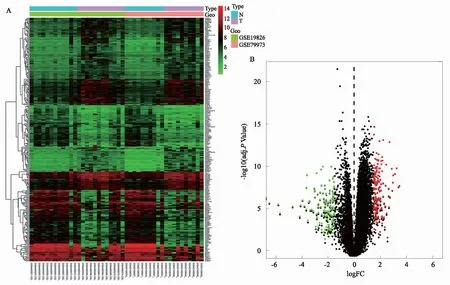
注:红色表示上调的差异基因(adj.P<0.05和log2FC >1.5),绿色表示下调的差异基因(adj.P<0.05和log2FC<-1.5),黑色表示无差异基因。
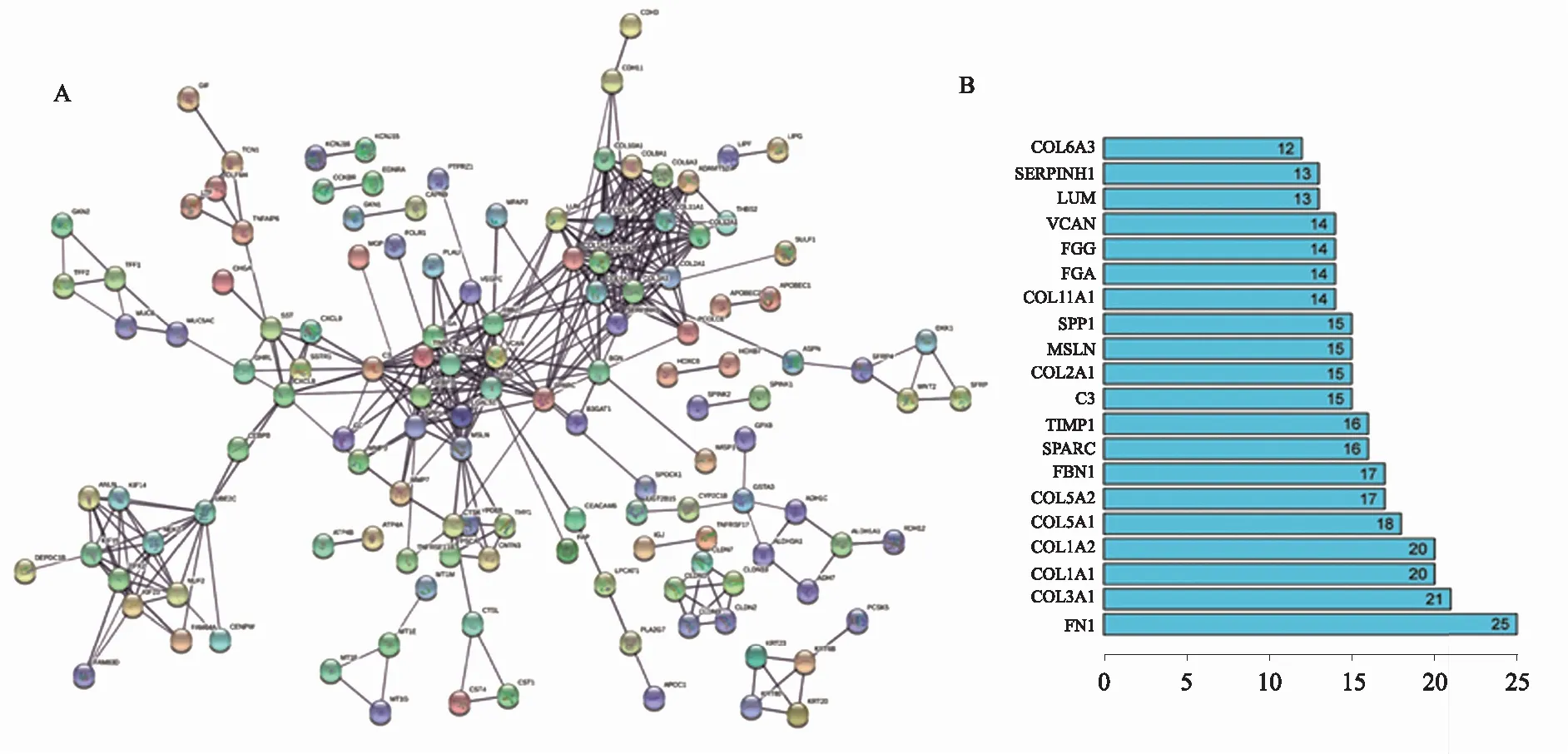
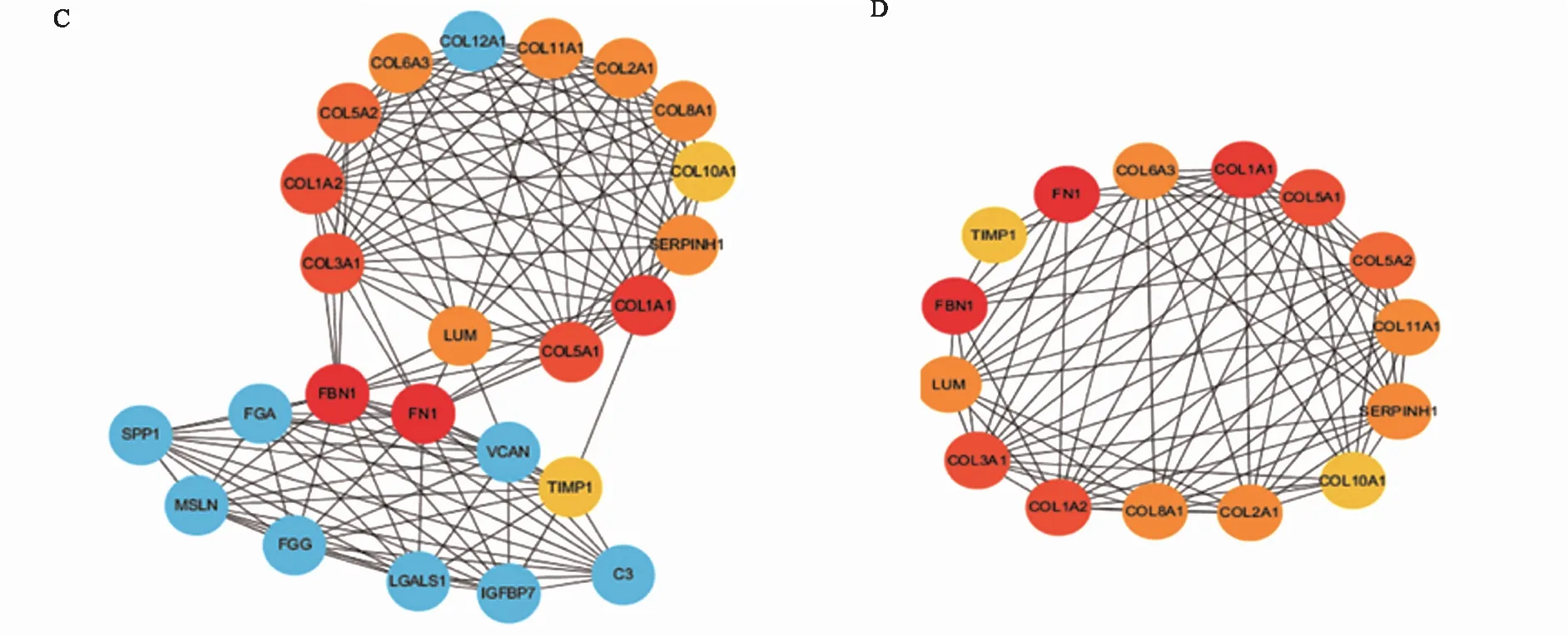
注:红色表示等级越高,黄色表示等级越低。
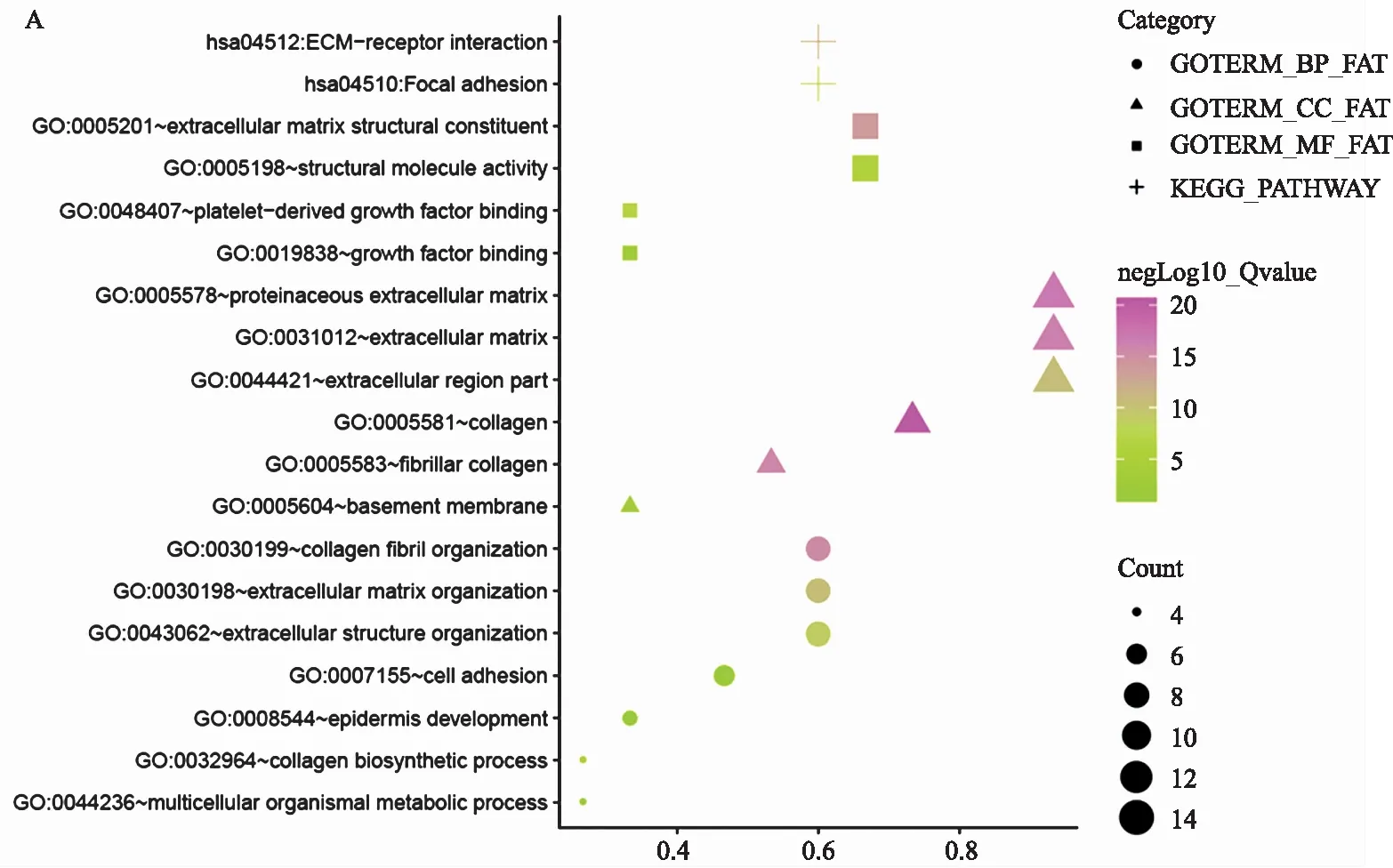
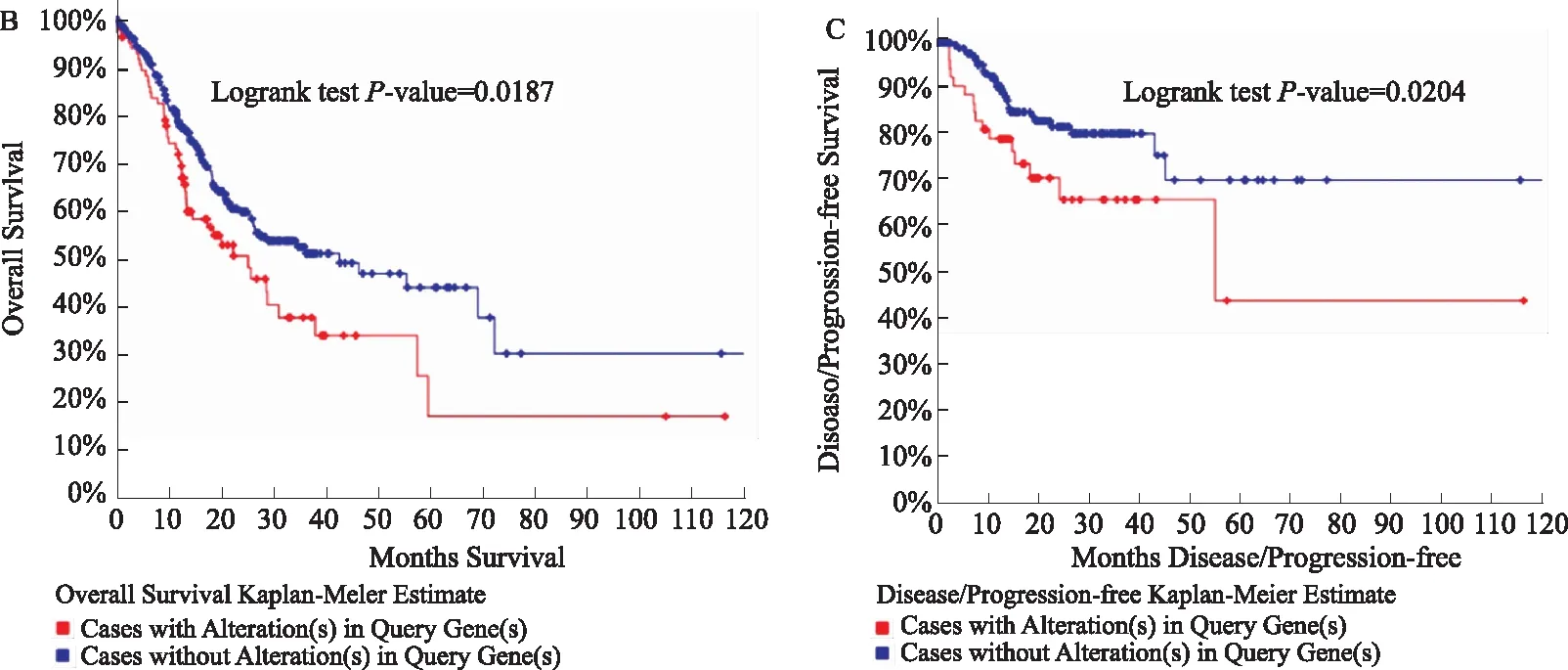
图3 互作最短路径基因GO及KEGG 富集分析(A);利用cBioPortal对最短路径基因进行胃癌总体生存率分析(B)和无进展生存率分析(C)
2.4 关键基因验证分析GEPIA外部数据库验证相较于正常胃组织,仅LUM、SERPINH1、TIMP1在胃癌中显著高表达[STAD T(肿瘤)=408例,N(正常)=211例;P<0.05,见图4A],且LUM、TIMP1基因表达与胃癌患者分期相关(见图4B),随着分期增加,LUM和TIMP表达上升,然后在Ⅲ和Ⅳ期进入稳定表达期,较前无增加[LUMF=9.03,Pr(>F)=8.69E-06;TIMP1F=6.43,Pr(>F)=0.000297],SERPINH1表达与分期无相关性(P>0.05)。结果表明,LUM和TIMP1表达水平可被用来判断Ⅰ、Ⅱ期的胃癌,但可能无法评估进展期(Ⅲ期和Ⅳ期)胃癌。此外利用TCGA数据库来源的胃癌数据可得到192例LUM、SERPINH1、TIMP1高、低表达患者的总生存期曲线(见图4C),研究发现,在胃癌中LUM基因高表达的总生存率显著低于低表达患者(P=0.041),风险比HR(high)=1.4,P(HR)=0.042。而SERPINH1及TIMP1表达均与生存无相关性(P>0.05)。鉴于上述分析结果提示,LUM可能是胃癌预后生存相关基因,LUM在胃癌组织中蛋白表达上调(见图4D),进一步Oncomine数据库Meta分析验证了LUM基因同样在多个亚型胃癌研究中均表达上调(Median rank=77.0,P=5.95E-6)(见图4E),且在H.pylori阳性胃癌患者中LUM表达显著升高(见图4F)。
另外,通过TCGA下载的胃癌患者临床资料,经删除临床信息不完整数据后建立基线资料表(见表1),进一步用Cox回归分析LUM表达与胃癌生存预后关系(见表2):单因素分析显示,LUM表达高与较差的总生存期显著相关(HR=1.001,95%CI:1.000~1.003,P=0.024),其他与不良生存相关的临床病理因素包括年龄、临床分期、T分级、淋巴结浸润、远处转移。在多因素分析中显示除年龄因素外,仅有LUM表达与患者生存预后相关(HR=1.002,95%CI:1.00~1.03,P=0.011),表明LUM表达可作为胃癌患者独立预后判断因子。
为了识别胃癌中LUM表达上调差异激活的信号通路,我们在低、高LUM表达数据集之间进行了GSEA分析。GSEA显示了在MSigDB Collection(C2.cp.kegg.v5.2.symbols.gmt.)中有显著性差异(FDRq<0.05, NOMP<0.05)富集通路。我们根据其归一化富集分数(NES)选择了最显著富集的信号通路(见图4G): “KEGG_FOCAL_ADHESION”、“KEGG_ECM_RECEPTOR_INTERACTION”、“KEGG_TGF_BETA_SIGNALING_PATHWAY” 、“KEGG_MAPK_SIGNALING_PATHWAY”、“KEGG_PATHWAYS_IN_CANCER”在LUM高表达表型中差异富集。
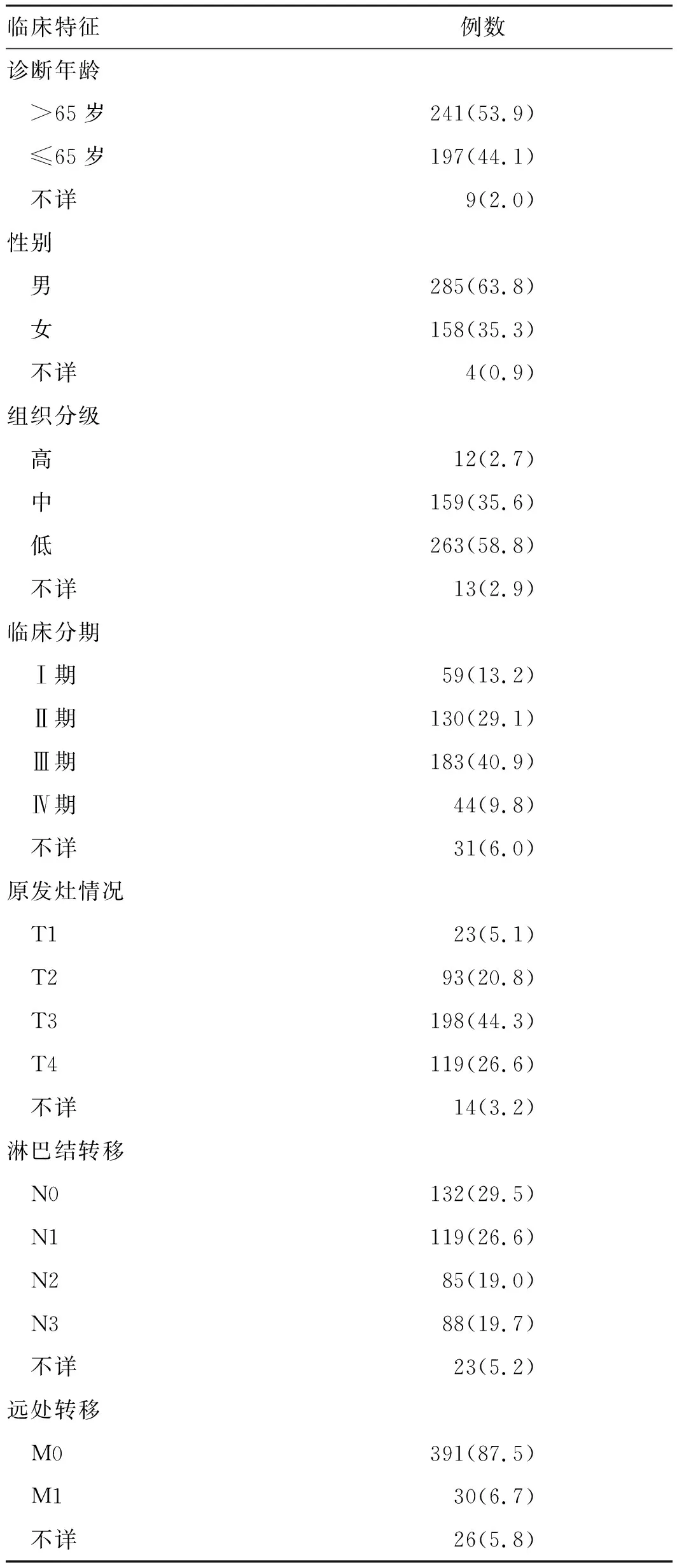
表1 TCGA 胃癌患者基线资料[例数(%)]Tab 1 Baseline data of gastric cancer patients of TCGA[n(%)]
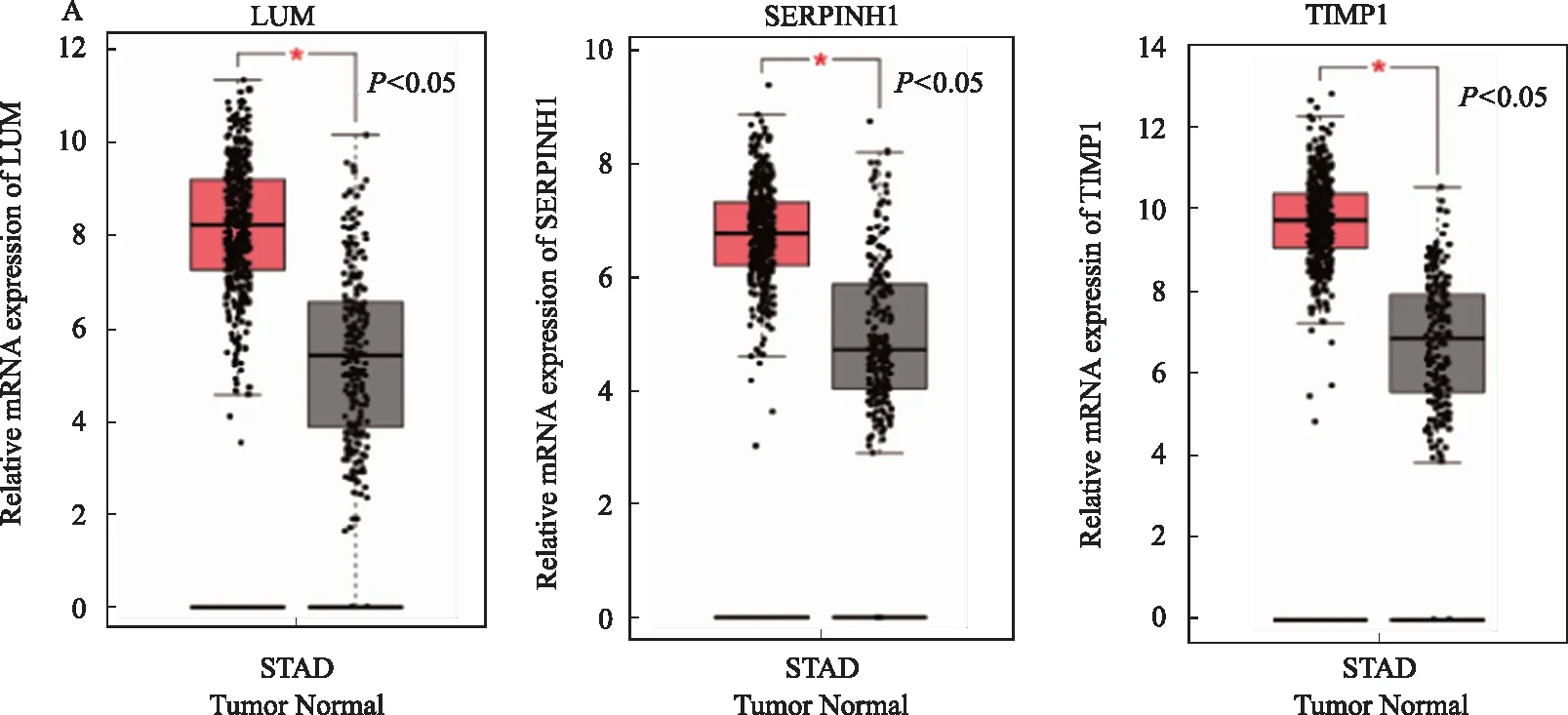
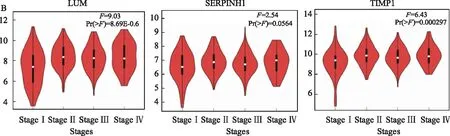
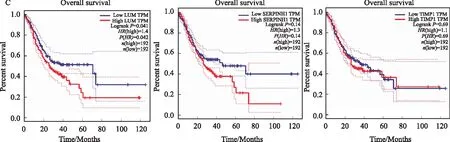

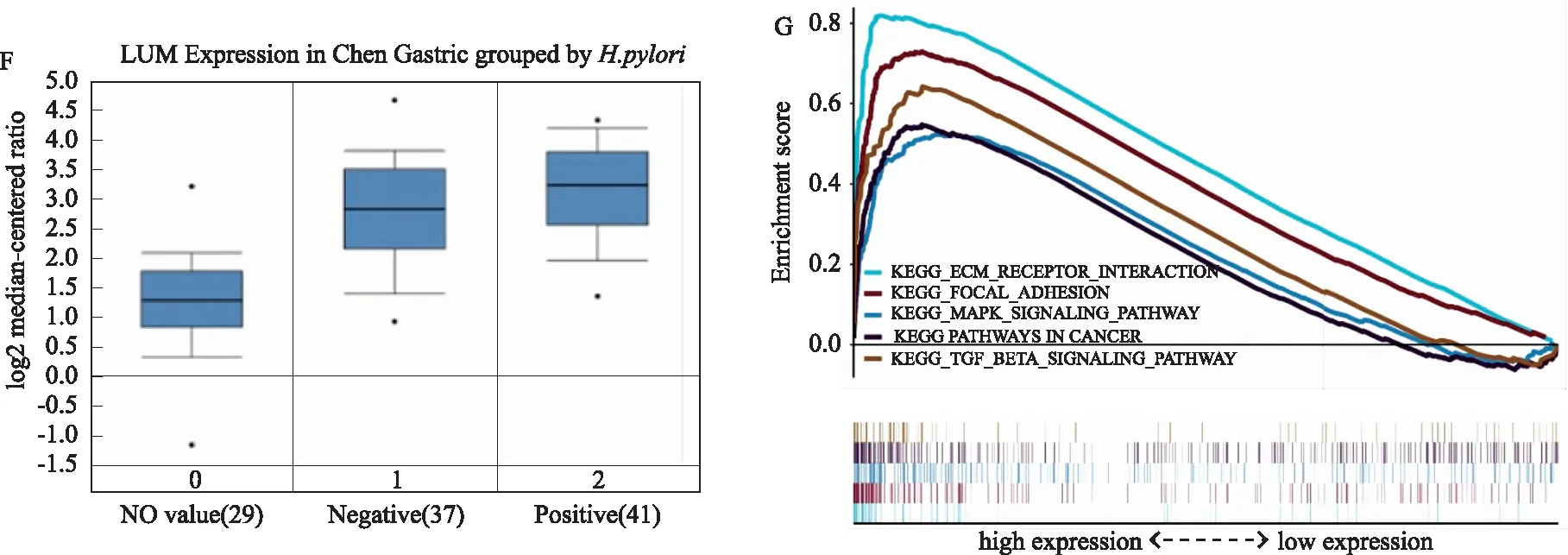
图4 A:LUM、SERPINH1、TIMP1在胃癌组织和正常胃组织中mRNA的表达水平;B:LUM、SERPINH1、TIMP1 与胃癌患者分期关系;C:Kaplan-Meier曲线法分析LUM、SERPINH1和TIMP1表达水平与胃癌患者总生存期的关系;D:基于人蛋白图谱数据库,与正常胃组织相比,STAD组织中LUM蛋白表达明显上调[正常胃组织为女性,年龄65岁(患者编号:2326;染色:阴性);胃癌组织来自男性,63岁(患者编号:5394;染色:中等;强度:中等;数量:75%;定位;胞质/膜)]; E:Oncomine数据库验证LUM在各种亚型胃癌中表达;F:LUM表达与胃癌患者H.pylori感染关系;G:ECM受体互作、黏着斑、癌症通路、MAPK及TGF-β等通路在LUM上调表型中存在差异富集
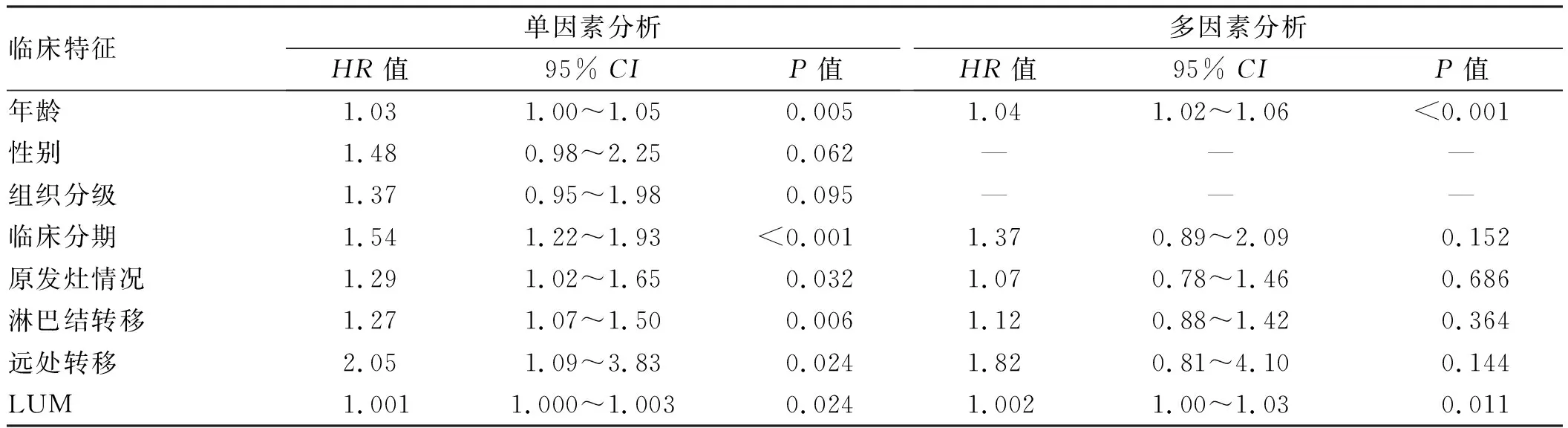
表2 基于TCGA的胃癌患者总生存期的单因素和多因素Cox回归分析Tab 2 Univariate and multivariate Cox regression analysis for overall survival in gastric cancer patients based on TCGA
3 讨论
胃癌的发生、发展是一个非常复杂的过程,内窥镜检查仍是早期胃癌筛查的主要手段,但因其为有创性检查,多数患者/健康体检者对于胃镜检查的不耐受及心理恐惧等原因使得胃镜检查暂未能列入常规早癌筛查的项目。目前临床上针对胃癌早期筛查的生物标志物主要有糖类抗原724(CA724)、癌胚抗原(CEA)、糖类抗原19-9(CA19-9)及糖类抗原242(CA242),但由于环境污染、食品安全及饮食结构等因素的改变,以往的血清学标志物不能满足临床需求。因此,研究者们不断地寻找更多更有敏感性及特异性的分子标志物以提高早期胃癌筛查的准确性。
在大数据发展的时代,资源共享进一步加快了科研发展的脚步,通过对全球癌症数据资源再分析节约经济成本的同时也减小了因样本、地域及种族等因素给研究带来的偏倚。
基于基因芯片的快速发展及测序平台在疾病研究中的应用,为我们研究胃癌的发病机制提供了良好的技术手段。本研究中首先从GEO中得到不同团队提供的胃癌患者表达谱数据分析,找出了共同差异表达基因,减少了数据偏差。综合利用生物信息学方法从多维度筛选出核心基因并在TCGA、Oncomine两个肿瘤资源大数据库中进行验证分析,最终确定了LUM在各种亚型胃癌中表达均显著上调,H.pylori感染阳性患者LUM表达增加,这是以往研究暂未发现的,未来我们将在临床上进行深入研究。LUM可能是1个潜在、独立的胃癌预后判断因子,为进一步临床研究提供了可靠的理论依据。
细胞外基质可阻止肿瘤细胞的侵袭,并有下调促癌细胞增殖信号传递的功能。本研究筛选的FN1、FNB1、LUM、SERPINH1、TIMP1均是与胶原蛋白作用密切、广泛表达于细胞外基质的基因,经与影响细胞外基质及细胞黏附过程参与了肿瘤的增殖、迁移及侵袭。纤丝蛋白-1(Fibronectin 1,FN1)是整合素受体家族众多成员的配体,参与细胞黏附和迁移过程,已有研究证实,其在多种肿瘤包括胃癌中高表达[10],体外敲低FN1可抑制胃癌细胞迁移和侵袭[11],这与我们预测结果相符。FBN1(Fibrillin 1)是一种细胞外基质糖蛋白,过表达促进肿瘤增殖转移[12-14]。SERPINH1,又称Hsp47,是一种重要的伴侣蛋白,它能正确折叠和分泌胶原蛋白,其表达水平与肿瘤的发生密切相关。研究发现,其在肾透明细胞癌[15]和胶质瘤[16]中高表达,并与肿瘤迁移和侵袭相关。金属蛋白酶抑制剂1(tissue inhibitor of metalloproteinases 1,TIMP1),其作用是与目标金属蛋白酶(如胶原酶)形成一对一的复合物,作用于多种基质金属酶。有研究发现,TIMP1对胃癌的促增殖作用可被TFF1抑制[17];TIMP1在溃疡性结肠炎相关的结直肠癌的起始和发展过程中持续过表达,可能是结直肠癌预后较差的潜在的生物靶标[18]。 LUM定位于12q21.3-q22,该基因编码一个小的富含亮氨酸的蛋白多糖(SLRP)家族成员,包括装饰蛋白、纤维调节蛋白、角化蛋白和骨黏蛋白等。LUM是角膜中主要的硫酸角蛋白多糖,但也分布于全身的间质胶原基质中。LUM可以调节胶原纤维组织和周向生长、角膜透明度、上皮细胞迁移和组织修复。小的富含亮氨酸的蛋白多糖是组织基质结构中普遍存在的细胞外基质成分,因此可调节癌细胞增殖、血管生成和迁移。有研究表明,LUM高表达可通过整合素β1-FAK信号通路促进胃癌进展[19],与结直肠腺瘤向肠癌进展相关[20],还可通过自分泌调节机制促进肺癌骨转移[21],促进膀胱癌的增殖和迁移[22],可能作为乳腺癌的上皮-间质转化和侵袭性标志物[23]。基于生物信息学方法,本研究从转录水平及蛋白水平确定了LUM在胃癌组织(尤其是合并H.pylori感染)中显著上调,且可能作为胃癌预后判断因子,该基因及其相关通路可能是胃癌治疗的潜在生物靶点。本研究方法能够为研究者提供一些研究思路,后续仍需进一步分子细胞学实验进行功能验证。
本研究通过综合生物信息学方法筛选确定了LUM、SERPINH1、TIMP1在胃癌中显著高表达,其中H.pylori感染胃癌患者LUM表达增加,LUM高表达提示胃癌患者预后不良。我们希望本分析能为胃癌后续分子机制的研究提供精准方向和强有力的理论基础,为发现新的诊断生物标志物和治疗策略提供线索。

