转基因大豆MON89788 芯片式数字PCR 定量方法的 建立
杨镇州 刘刚 梁文
(上海市计量测试技术研究院化学与电离辐射所生物计量实验室,上海 201203)
据国际农业生物技术应用服务组织(ISAAA)公布的数据表明,伴随着生物技术及相关产业的飞速发展,截至2018 年底,全球转基因作物种植面积已达1.92 亿hm2,大约为中国大陆面积的2 倍[1];与此同时,美国在2009 年已批准了8 种抗虫害转基因新型玉米的商业化许可;加拿大等国也开始批量开发生产转基因大豆等作物,我国在2009 年11 月也为转基因抗虫水稻和转植酸酶基因玉米颁发了生物安全证书。由此可见,转基因作物的相关研究和应用将会越来越广泛。因此各国家和地区开始对转基因产物进行系统管理,实施标识制度,如欧盟规定合法转基因成分标识阈值为0.9%,巴西、新西兰标识阈值为3%,俄罗斯、日本标识阈值为5%等[2]。这一制度的采用,将转基因安全管理变得更科学合理。与此同时,也对我们传统转基因检测方法的精确定量提出了更高要求。
转基因检测目前主要分为核酸检测与蛋白检测两种[3]。相比于蛋白,核酸在生物细胞内含量更稳定、不易被破坏,且核酸检测的操作简易、灵敏度更高,因此成为转基因检测的主流方法[4]。传统的转基因成分定量检测基本采用实时荧光定量PCR 法(Realtime fluorescence quantitative PCR,qPCR)[5], 但qPCR 只能实现相对定量,并且只能够分辨约两倍的浓度差异(如5 000 拷贝与10 000 拷贝的差异),且对低浓度的拷贝数差异分辨能力更低。因此大多数的qPCR 在转基因检测方法的应用还停留在定性阶段。数字PCR(digital PCR,dPCR)是一种对PCR反应体系进行有限分割,在不同反应微单元中进行扩增,最后根据泊松分布原理和阳性单元的个数得出模板DNA 的起始拷贝数的技术。相较于传统PCR技术,dPCR 有着高灵敏度、高精确度、绝对定量、反应单元不易受干扰等优势[6]。因此,dPCR 技术在近年来发展飞速,已多次被报道用于转基因成分的定量检测研究中。
转基因品系大豆MON89788 是抗虫耐除草剂大豆的新品种,目前已在欧盟、韩国、墨西哥等国家被批准用于商业化种植或食用。在我国,MON89788大豆于2008 年首次获得农业部颁发的进口安全证书,仅限原料加工。目前国内该品系转基因大豆的定量检测方法主要使用普通PCR 法进行定性检测[7]。而国际上主要采用qPCR 进行定量检测[2],尚未见采用dPCR 进行精确定量检测的报道。本实验以芯片式dPCR 为检测平台对转基因MON89788 品系大豆进行定量检测方法研究,从提取方法、基因组DNA浓度检测方法开始进行类比分析,对dPCR 的反应体系进行优化,并对方法的重复性和定量限进行评估,对其他转基因定量检测技术有着重要的借鉴 作用。
1 材料与方法
1.1 材料
植物基因组DNA 提取试剂盒(天根DP305,北京);引物、探针(佰力格,上海);3D 数字PCR系统(QuantStudioTM3D,ABI 公司);核酸定量仪(Nanodrop2000、Qubit3.0,thermo 公 司);5%、1%和0.1%的转基因MON89788 大豆粉末,由中国农业科学院生物技术研究所制备。
1.2 方法
1.2.1 试剂盒法抽提转基因大豆基因组 DNA 用植物基因组DNA 提取试剂盒进行转基因大豆基因组DNA 的抽提。在抽提过程中需要用到酚氯仿,以有效去除大豆材料中的多糖和多酚成分。
1.2.2 CTAB 法抽提转基因大豆基因组DNA 参考GB/T 19495.3-2004 附 录C 《CTAB-1 法 提 取DNA》进行转基因大豆基因组DNA 的抽提。
1.2.3 反应体系和反应条件 从国内通用的大豆 MON89788 定量检测标准中筛选品系特异序列和内源基因的引物探针,并建立各自的PCR 反应体系和反应条件。转基因大豆品系的芯片dPCR反应体系(总体积为20 μL)中,内外源基因的正反向引物终浓度均为500 nmol/L,探针终浓度为250 nmol/L。Master mix 体 积 为10 μL,H2O 体 积 为5.5 μL。3D 芯 片dPCR 的反应条件为:酶激活和预变性阶段:96℃,10 min;循环扩增阶段:98℃/30 s,60℃/2 min,共40 个循环。升温速率为0.8℃/s,降温速率为1.2℃/s。
1.2.4 检测方法的重复性 随机抽取10 瓶转基因含量为5%的大豆MON89788 样品,每瓶独立提取3 次,根据1.2.1 和1.2.2 的方法进行DNA 抽提,随后根据
1.2.3 的反应体系及条件进行dPCR。
1.2.5 方法的定量检测限 提取转基因含量分别为5%、1%和0.1%大豆粉末,控制基因组DNA 的浓度在100 ng/μL 左右,根据1.2.3 的反应体系及条件进行dPCR,每个扩增实验重复3 次,并计算测得平行数据的RSD 值,以RSD<25%作为有效定量数据的判断标准。
2 结果
2.1 提取方法的优化
本实验分别选择了传统的CTAB 法和较通用的植物基因组抽提层析柱试剂盒法进行对比。结果见表1。核酸溶液在紫外260 nm 处有特征吸收峰,若A260/A280在1.8-2.0 之间,说明核酸的纯度较好。A260/A280<1.8,说明溶液中可能含有机试剂或蛋白杂质。实验结果表明,层析柱法提取大豆基因组DNA的纯度显著优于CTAB-1 法,CTAB-1 提取的核酸A260/A280=1.68,提示可能有有机溶剂的污染。同样的样品量(约100 mg),层析柱法提取的浓度是传统CTAB-1 法的2 倍。因此后续试验均采用层析柱法进行样品基因组提取。
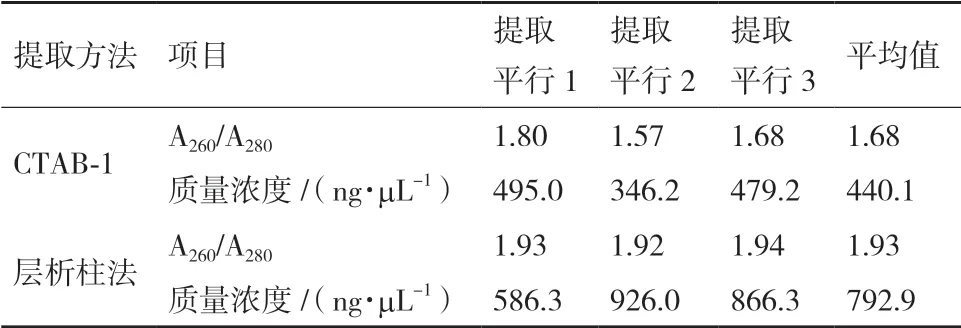
表1 采用不同方法提取的转基因MON89788 品系大豆基因组DNA
2.2 DNA模板浓度的控制
数字PCR 法的模板DNA 浓度应该在47 000-99 723 拷贝范围内,从而避免低转基因含量样本出现转基因片段漏取样的情况,也避免DNA 拷贝数过高,阳性过载使数据精确度下降的情况。为了准确调控模板的浓度范围,必须对模板浓度进行预估。本实验设计使用微量紫外分光光度计法及荧光检测法分别检测提取转基因大豆基因组DNA 的浓度,并与数字PCR 结果进行对比。实验结果见图1,采用紫外分光检测的浓度结果高于荧光检测的浓度,可能因为紫外分光法受部分蛋白或有机溶剂的干扰使浓度值偏高,而荧光检测法能特异性的与双链DNA结合。但荧光检测法定量依靠标准曲线的建立,而标准物质和样品与荧光基团的结合能力不同导致浓度定量的偏差,有时表现出浓度偏低的现象。
紫外分光法测定浓度和数字PCR 实际测定浓度的线性相关系数为0.94,而荧光检测法的线性相关系数为0.69,说明紫外分光法检测浓度与数字PCR法检测浓度相关性更好。
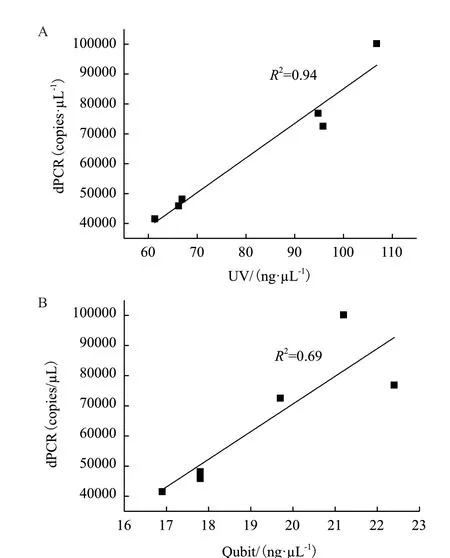
图1 使用不同检测方法测定DNA 的浓度与数字PCR 定值的线性关系图
确定紫外分光法作为初步浓度预估方法后,可以根据质量浓度和拷贝数浓度的转换公式,计算核酸溶液的拷贝数浓度,见公式1:

其中C 为体积为1 μL 体积DNA 模板的拷贝数,m为1 μL 体积DNA 模板的质量,M 为大豆的基因组DNA 的分子量(约为6.6×1011),NA 为阿伏伽德罗常数。
最终,控制模板DNA 浓度,使20 μL 反应体系中模板DNA 拷贝数总量落在47 000-99 723 个拷贝的范围内。
2.3 反应条件的优化
本实验将实时荧光PCR 检测标准方法中的引物探针应用到数字PCR 上,并对数字PCR 反应条件进行优化。标准方法中的引物探针序列见表2。数字PCR 采用终点法判定PCR 微反应单元为阴性或阳性,阳性信号的荧光值是否足够高十分关键。本文使用Quanstudio 3D 芯片数字PCR 平台进行实验优化。转基因大豆MON89788 外源基因用FAM 荧光基团标记,大豆内源基因1-lectin 采用FAM 荧光基团标记,2-lectin 采用VIC 荧光基团标记,结果见图2。实验结果表明,同样来自GB 19495.5-2018 标准中的大豆内源基因lectin 的引物探针,采用适合芯片数字PCR 的扩增条件,在数字PCR 上的扩增荧光信号的区分效果有很大差异。1-lectin 引物探针对的区分效果很差,阴性和阳性信号有大量的重叠现象。而2-lectin 引物探针对由于阳性孔中PCR 扩增效果好,PCR 扩增孔的荧光信号很高,足以和阴性信号有效区分。
2.4 方法的重复性
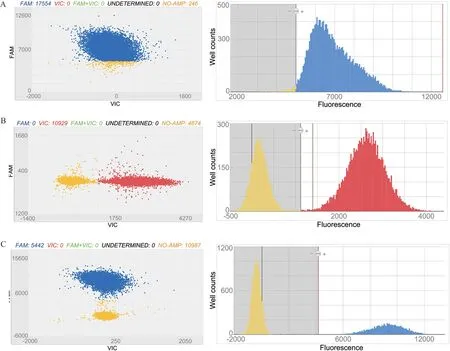
图2 转基因大豆MON89788 的芯片dPCR 扩增图

表2 靶基因引物探针序列
随机抽取10 瓶含量为5%的MON89788 转基因大豆样品,每组独立取3 次样品,并独立进行内源基因和外源基因dPCR 实验,计算并统计每组的转基因含量,结果见表3。实验结果表明,将本方法应用于样品检测中,在考虑了提取效率对转基因定量结果的影响下,10 组样品中检测重复RSD 最大的9.97%,最小的仅1.17%,均满足转基因成分定量检测行业公认的检测平行数据RSD 可接受误差25%的要求[8],说明本方法重复性优异。该样品的转基因标准值为5%,检测结果的示值误差仅0.05%。
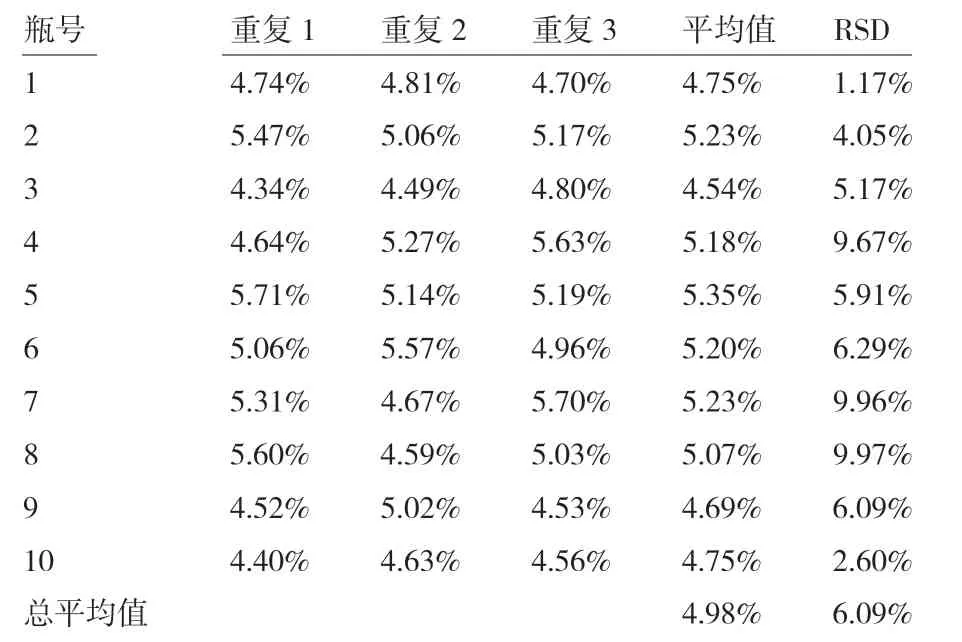
表3 5%转基因大豆MON89788 检测的重复性
2.5 方法的定量检测限
提取转基因含量为5%、1%、0.1%的MON89788 转基因大豆基因组DNA,并进行dPCR 检测,结果 (表4)分别为5.20%、0.94%和0.11%,相对偏差分别为0.3%、-0.06%和0.01%,RSD 分别为6.2%、3.6%和15.2%。检测结果的相对偏差和RSD 均远远小于转基因检测行业规定的偏差小于25%,因此该方法可以用于转基因的定量检测[9]。
3 讨论
本文研究了一种芯片数字PCR 定量检测大豆MON89788 的方法,研究内容涵盖基因组DNA 提取方法及浓度检测方法、引物探针的序列选择设计、引物探针的最佳浓度、PCR 反应过程的时间、温度 等。运用该方法进行实际样品检测,10 组5%的转基因大豆样品的检测重复性RSD 在1.17%-9.97%之间。0.1%的转基因大豆样品的定量示值误差为0.01%,定量重复性RSD 为15.2%,定量限满足欧盟的0.9%转基因定量标识限要求。
在样品前处理阶段,本方法采用试剂盒提取大豆基因组DNA,其效果优于CTAB 法,可能的原因是:试剂盒中吸附柱更容易甩脱杂质,而CTAB 提取方法中异丙醇与水层颜色区分不明显、吸取DNA 不准确,易带来有机溶剂杂质。在基因组DNA 浓度估算中,相比于荧光检测Qubit 法,紫外分光法定量检测提取得到的基因组DNA 浓度与数字PCR 结果一致性更高。当基因组DNA 纯度满足A260/280在1.8-2.0之间时,用微量紫外分光光度计估算核酸的拷贝数浓度更可靠。
在PCR 反应阶段,实时荧光定量PCR 的反应体系不能照搬到数字PCR 的反应体系中,而阴性和阳性信号的区分是数字PCR 定量结果准确性和重复性的可靠保证。如果荧光信号阈值的拖动会造成阳性反应单元数有5%以上的变动,定量结果重复性会更差,此时需注意优化PCR 方法。优化数字PCR方法,优先考虑更换引物探针序列;其次是优化引物探针浓度,优化PCR 反应的延伸时间,退火温度等反应过程。
在转基因定量检测中,相比传统的qPCR,dPCR 不需要制作标准曲线[10],也避免了昂贵的标准物质的使用,以及PCR 扩增效率差异对实验结果的影响[11]。将该方法用于转基因大豆的定量检测,能为规范我国转基因监管工作的实施提供强有力的技术支撑和方法依靠。
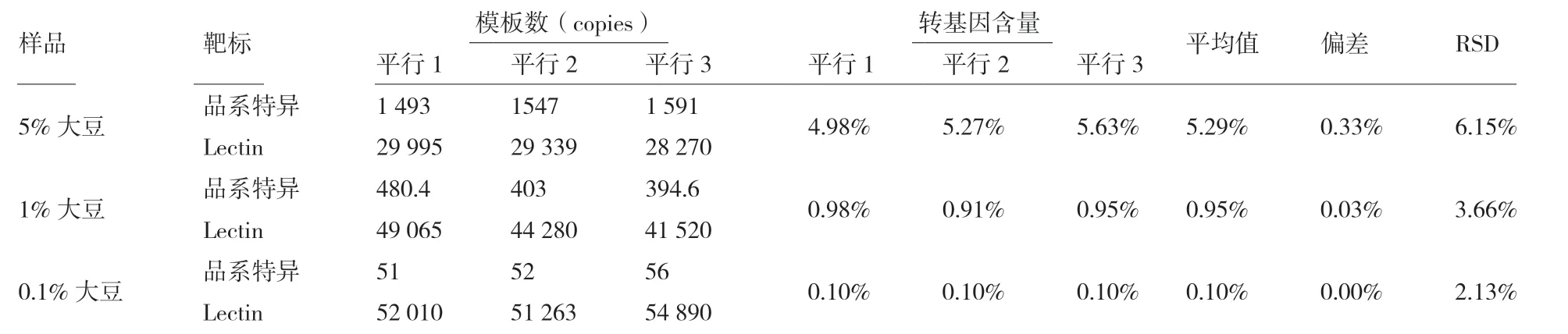
表4 转基因大豆MON89788 dPCR 定量检测限实验结果

