基于TaqMan实时荧光PCR检测植物中转基因成分
刘国强 海小 罗建兴


摘 要:转基因植物的安全性一直备受关注,转基因成分检测是保障食品安全、维护消费者知情权的重要手段。本研究构建并鉴定了pCry3A、pCry1A(c)、pCP4-EPSPS和pCTP2-CP4-EPSPS 4种阳性质粒,并在转基因棉花、转基因大豆、转基因小麦、转基因玉米、非转基因玉米中分别检测玉米内源基因(adh1)、抗虫基因(Cry3A、Cry1A(c))和抗除草剂基因(CP4-EPSPS、CTP2-CP4-EPSPS),最后将转基因棉花和转基因大豆DNA模板原液进行梯度稀释确定Cry1A(c)和CP4-EPSPS的检出限,通过检出限检测建立标准曲线确定此方法的转基因成分定量检测能力。结果表明:4种阳性质粒鉴定结果良好,可以作为转基因检测的阳性对照;转基因棉花为转入Cry1A(c)基因的抗虫棉;转基因大豆为转入CP4-EPSPS基因的抗除草剂大豆;转基因小麦为转入Cry1A(c)基因的抗虫小麦;转基因玉米为转入Cry1A(c)、CP4-EPSPS、CTP2-CP4-EPSPS基因的混合转基因玉米;Cry1A(c)和CP4-EPSPS的引物和探针的检出限均可达0.1ng;建立的2种标准曲线的R2≥0.95,说明引物和探针可以满足定量检测要求。本研究对TaqMan实时荧光PCR法检测植物中的转基因成分提供一些借鉴,同时为保障食品安全、维护消费者知情权提供方法支持。
关键词:转基因玉米;阳性质粒;检出限;定量
中图分类号:S-3 文獻标识码:A
DOI:10.19754/j.nyyjs.20200530002
转基因植物是指把从微生物、植物和动物获得的重要功能基因,利用生物化学方法将功能基因和调控基因表达的DNA序列组合成1个表达盒,之后通过各种转基因技术将完整的表达盒转移到植物基因组中进行表达,使植物获得抗虫、抗除草剂、抗病、优质等新的农艺性状[1]。2016年全球种植转基因作物面积达1.851亿hm2,大豆、玉米是种植面积最大的2种转基因作物,转基因大豆和转基因玉米也是我国主要进口的转基因粮食[2]。随着转基因技术的突飞猛进,我国已经培养出多种具有优良性状的转基因植物,如抗虫(转Cry1Ab基因)[3]、抗病(转NibT基因)[4]、抗旱(转SAMS基因)[5]、抗除草剂(转2mG2-epsps基因)[6]等。
由于基因工程和生物技术的进步,消费市场被越来越多的转基因农产品充斥着[7],社会各界也越来越关注转基因植物及其产品的安全性问题[8-11]。考虑到转基因食品的安全、对消费者的保护以及对转基因产品的管理,越来越多的国家对转基因食品进行立法[12]。为了与立法相一致,许多国家的监管机构要求提供检测方法作为登记包含的一部分。目前,最广泛使用同时又符合立法的检测方法是PCR(Polymerase Chain Reaction, PCR),因为其简单、特异、灵敏[13]。其中的实时荧光PCR通过对每个循环的扩增产物进行实时监测,实现对目标DNA模板的定性和定量检测。由于SYBR Green荧光染料PCR的缺点是包括引物二聚体在内的任何双链DNA都会在嵌入染料产生荧光,会出现假阳性[14]。而TaqMan探针提高了实时荧光PCR的扩增效率和特异性,能够快速并准确地得到检测结果[15]。相比之下,TaqMan实时PCR方法是转基因检测的最佳方案,最关键的是这种方法与法律相一致[16]。综上所述,研发出科学并合理的特异性检测转基因植物TaqMan实时荧光PCR方法是非常必要的。
苏云金芽孢杆菌d-内毒素Cry3A基因[17]和Cry1A(c)基因[18]是2种比较常见的抗虫基因,被广泛运用于转基因马铃薯和棉花中。5-烯醇丙酮莽草酸-3-磷酸合成酶CP4-EPSPS基因[19]和含有叶绿体转运肽的6-烯醇丙酮莽草酸-3-磷酸合成酶基因的构建体CP2-CP4-EPSPS基因[20-22]是2种常见的抗除草剂基因,多数运用在大豆上。因此本研究选择这4种基因作为转基因成分的判定。乙醇脱氢酶adh1基因[23]作为玉米的内源基因,用来判断体系是否正常。因此,本研究参照标准SN/T 1204-2016[24]建立了1种特异性检测转Cry3A、Cry1A(c)、CP4-EPSPS和CP2-CP4-EPSPS基因植物的TaqMan实时荧光PCR方法。
1 材料与方法
1.1 样品准备
转基因棉花、转基因大豆、转基因小麦、转基因玉米、非转基因玉米均由从事转基因基础研究的实验室提供。将样品进行研磨,置于1.5mL离心管中,并保存于-4℃。
1.2 试剂
TransStart Probe qPCR SuperMix(北京全式金生物技术有限公司);实时荧光定量PCR引物和探针合成(北京睿博兴科公司)。
1.3 仪器
Eppendorf 5418R Centrifuge高速台式离心机(德国Eppendorf AG公司);Nanodrop 2000c核酸蛋白测定仪(美国Thermo Fisher公司);7300plus实时荧光PCR扩增仪(美国ABI公司)。
1.4 方法
1.4.1 引物和探针
引物与探针序列如下表1。
1.4.2 DNA提取和质量控制
取1.1准备好的植物样品,利用溴化十六烷三甲基铵(Cetyltrimethylammonium bromide,CTAB)法[27]提取其DNA,方法如下。称取研磨好的样品约50mg,置于1.5mL离心管中,加入DNA裂解液800μL,旋涡混匀,65℃金属浴30min,期间不时振荡混匀;13000rpm离心5min,转移上清液于洁净离心管中,加入等体积三氯甲烷/异戊醇(24/1)混合液,充分混匀,13000rpm离心5min,取上清液于新的离心管中加入等体积的异丙醇,充分混匀,13000rpm离心5min,弃去上清液,75%乙醇洗涤1次,晾干,加入适量的灭菌ddH2O溶解,利用Nanodrop 2000c核酸蛋白分析仪测量其浓度,将母液稀释至100ng/μL左右,保存于-20℃。
1.4.3 反应体系与反应条件
反应体系(总体积20μL):TransStart Probe qPCR SuperMix 10 μL,上下游引物各1μL,探针1μL,模板DNA1μL,补水至20μL。
反应条件:94℃30s,1个循环;94℃5s,60℃34s,40个循环,在每次循环退火时收集荧光信号。
1.4.4 阳性质粒的构建
通过人工合成的方法合成抗虫基因(Cry3A、Cry1A(c))和抗除草剂基因(CP4-EPSPS、CTP2-CP4-EPSPS)的序列,连接到载体上,转化进感受态细胞,经过PCR检测、酶切、测序对阳性质粒上插入的外源序列进行验证。
1.4.5 阳性质粒的鉴定
将1.4.4提取的大肠杆菌DNA稀释液进行实时荧光定量PCR检测,进行阳性质粒的鉴定实验(每组2个平行)。
1.4.6 植物的鉴定
将提取好的转基因棉花、转基因大豆、转基因小麦、转基因玉米、非转基因玉米DNA(每组2个平行)进行玉米内源基因(adh1)、抗虫基因(Cry3A、Cry1A(c))和抗除草剂(CP4-EPSPS、CTP2-CP4-EPSPS)的检测。
1.4.7 检出限分析
从100ng/μL的转基因棉花和转基因大豆DNA稀释液加灭菌ddH2O开始稀释,每1/10稀释1次,共稀释7次。分别为100ng/μL、10ng/μL、1ng/μL、0.1ng/μL、0.01ng/μL、0.001ng/μL、0.0001ng/μL、0.00001ng/μL(每组3个平行),进行检出限检测实验。
1.4.8 定量分析
根据1.4.7的检出限分析确定棉花和大豆的定量曲线,同时分析引物和探针的定量能力。
1.4.9 数据处理
利用7300 plus实时荧光PCR扩增仪自带分析软件对结果进行扩增曲线和循环(Cycle threshold,Ct)的分析。每次实验结果都以平行实验的平均值±标准差表示。
2 结果与分析
2.1 阳性质粒的构建与鉴定
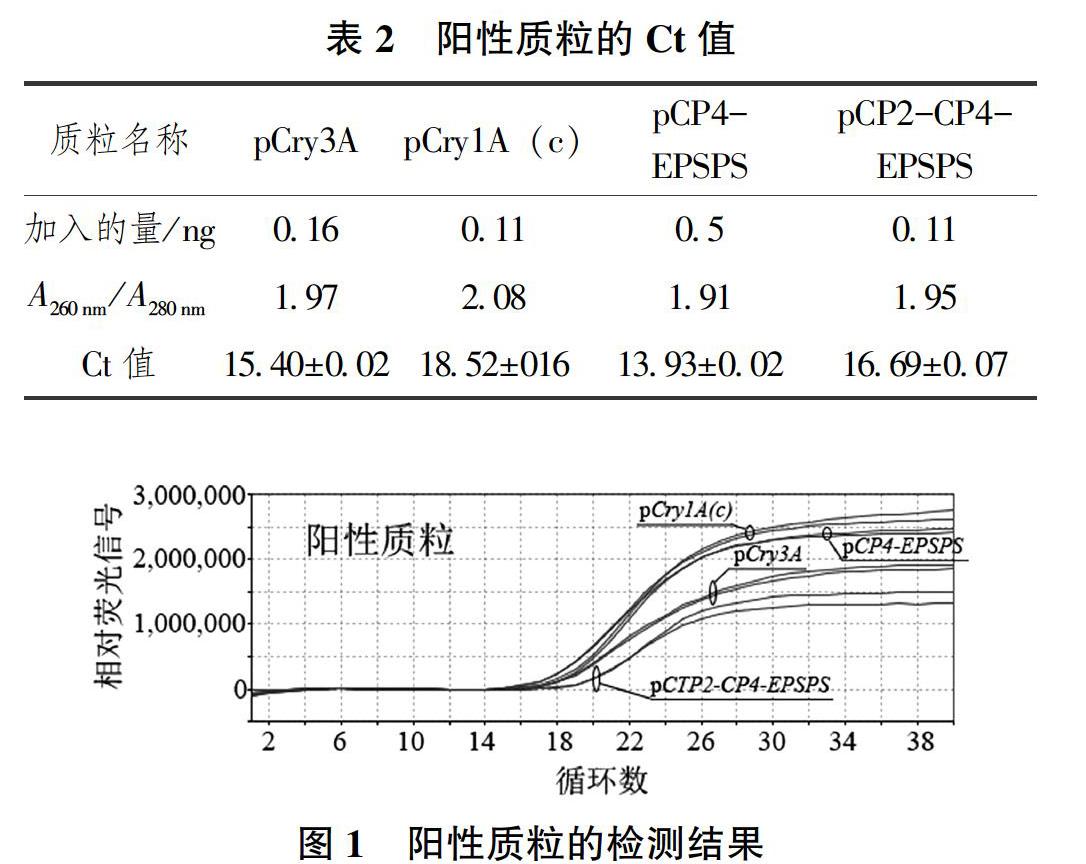
将提取好的大肠杆菌质粒稀释液分别利用各自相对的抗虫(Cry3A、Cry1A(c))和抗除草剂(CP4-EPSPS、CTP2-CP4-EPSPS)的引物和探针进行实时荧光定量PCR实验,阳性质粒的检测结果如表2所示,扩增结果如图1所示。由表2和图1可以看出,阳性质粒能够匹配抗性基因的引物和探针,且扩增曲线良好。扩增结果的Ct值为15.40±0.02(pCry3A),18.52±016(pCry1A(c)),13.93±0.02(pCP4-EPSPS),16.69±0.07(pCTP2-CP4-EPSPS)。综上所述,阳性质粒的鉴定结果良好,可以作为转基因检测的阳性对照。
2.2 植物的鉴定
将转基因棉花、转基因大豆、转基因小麦、转基因玉米和非转基因玉米的DNA,分别利用玉米内源(adh1)、抗虫(Cry3A、Cry1A(c))和抗除草剂(CP4-EPSPS、CTP2-CP4-EPSPS)的引物和探针进行实时荧光定量PCR实验,扩增结果如表3和图2所示,Ct值≤35判定为检出。由表3和图2可知,adh1基因在转基因玉米和非转基因玉米中均有检出(图2A),Ct值分别是33.89±0.04和30.01±0.15;Cry3A基因在所选植物样品中均没有检出;Cry1A(c)基因在转基因棉花、转基因小麦和转基因玉米中均有检出(图2B),Ct值分别是25.27±0.03、33.08±0.09和33.95±0.08;CP4-EPSPS基因在转基因大豆和转基因玉米中均有检出,Ct值分别是32.29±0.11和33.87±0.69(图2C);CTP2-CP4-EPSPS只在转基因玉米中有检出,Ct值为33.10±0.25(图2C)。综上所述,转基因棉花为转入Cry1A(c)基因的抗虫棉;转基因大豆为转入CP4-EPSPS基因的抗除草剂大豆;转基因小麦为转入Cry1A(c)基因的抗虫小麦;转基因玉米为转入Cry1A(c)、CP4-EPSPS、CTP2-CP4-EPSPS基因的混合转基因玉米。
2.3 检出限分析
通过Cry1A(c)和CP4-EPSPS的引物和探针对100ng/μL、10ng/μL、1ng/μL、0.1ng/μL、0.01ng/μL、0.001ng/μL、0.0001ng/μL、0.00001ng/μL等8个不同浓度梯度的转基因棉花样品DNA和转基因大豆样品DNA进行荧光定量PCR扩增实验,结果如图3所示,具体数值如表4所示。由图3和表4可知,稀释至0.1ng/μL的转基因棉花样品DNA(图3A)和转基因大豆样品DNA(图3B)也出现典型扩增曲线,且Ct值分别为34.59±0.34和34.51±0.08,以扩增结果Ct值≤35时判定为确认检出,所以上述结果表明Cry1A(c)和CP4-EPSPS的引物和探针已达到检出限度。综上所述,Cry1A(c)和CP4-EPSPS的引物和探针的检出限均可达0.1ng。
2.4 定量分析
利用棉花DNA和大豆DNA做标准曲线定量检测转基因棉花中Cry1A(c)和大豆中CP4-EPSPS的含量。根據2.3的实验结果制作棉花(Cry1A(c))和大豆(CP4-EPSPS)定量检测标准曲线,如图4所示。所得棉花(Cry1A(c))的标准曲线为y=-2.428x+47.007,R2=0.984;大豆(CP4-EPSPS)的标准曲线为y=-1.246x+40.669,R2=0.96556。因为R2≥0.95,所以这2种引物和探针可以达到定量要求,即可根据此标准曲线定量检测植物中转基因成分的含量。可通过将检测植物的相应成分的Ct值带入标准曲线中的公式可以得到植物种转基因成分的定量检测结果。以上所述都说明以上2种抗性基因的引物和探针具有较好的定量检测能力。
3 討论
目前最被人们接受的转基因成分的检测方法是基于DNA分子的,因为DNA分子经过极端条件处理稳定性也很高[28,29]。迄今为止已经报道单一测定反应中能够检测多达5个转基因成分的多重传统PCR系统[30-32]。这些测定中获得的扩增片段,可以在琼脂糖凝胶中进行鉴定。尽管该方法非常适用于定性分析,但其检出限较差,可通过使用更灵敏的检测技术(实时荧光PCR技术)来改善。实时荧光PCR技术中的TaqMan实时荧光定量PCR因为拥有不同荧光染料来标记多个独立的探针,其特异性大大加强而且适用于定量检测使其脱颖而出[33],同时检测过程中处于封闭体系中进行扩增,降低了电泳过程中PCR产物被污染的可能,有效地减少了检测时间,特别是检测大批量样品时其优势尤为明显[34]。
本研究的创新点在于构建阳性质粒和在检出限的基础上通过建立标准曲线来评价方法的定量检测能力。通过NCBI数据库得到4种抗性基因的序列,构建阳性质粒。通过鉴定,4种阳性质粒(pCry3A、pCry1A(c)、pCP4-EPSPS和pCTP2-CP4-EPSPS)结果良好,可以作为转基因成分检测的阳性对照。对转基因棉花、转基因大豆、转基因小麦、转基因玉米和非转基因玉米进行鉴定,得到转基因棉花为转入Cry1A(c)基因的抗虫棉;转基因大豆为转入CP4-EPSPS基因的抗除草剂大豆;转基因小麦为转入Cry1A(c)基因的抗虫小麦;转基因玉米为转入Cry1A(c)、CP4-EPSPS、CTP2-CP4-EPSPS基因的混合转基因玉米。对转基因棉花和转基因大豆DNA模板原液进行梯度稀释确定Cry1A(c)和CP4-EPSPS的检出限为0.1ng。根据检出限结果建立的2种抗性基因的定量曲线R2≥0.95,根据此标准曲线可以定量检测植物中转基因成分的含量。
4 结论
综上所述,上述方法可以有效鉴定转基因植物和对转基因植物进行定量分析。随着生物技术作物的快速发展,人们越来越关注生物技术作物衍生食品和饲料产品的潜在风险。为了保护消费者的知情权和实施转基因标签政策的先决条件时可以使用可靠且高效的检测技术准确测量食品或饲料产品中的转基因成分[35];国际贸易也需要可靠的转基因生物分析,以便对产品中转基因成分的可比较进行测量[36]。这些因素都促使转基因检测技术朝着高效、快速的方向发展[37],所以多重实时荧光定量的多通道检测[20,22,38]、环介导等温技术的快速检测[39]以及数字液滴PCR的高通量检测[40,41]将会成为未来转基因检测的研究方向。
参考文献
[1] 孟志刚,崔艳,张锐.转基因——农业生产新助力[J].生命世界,2018(5):14-17.
[2]祁潇哲.加强粮食收储环节转基因质量安全监管探讨[J].粮油食品科技,2018,26(5):58-62.
[3]杜娟,胡汉桥,余云舟,等.用基因枪法将Bt杀虫基因导入玉米自交系的研究[J].吉林农业大学学报,2003,25(3):260-262.
[4]杜建中,孙毅,王景雪,等.转水稻NibT基因玉米植株的获得及抗病性研究[J].西北植物学报,2011,31(5):893-901.
[5]杨政伟,胡银岗,王琨,等.转SAMS基因玉米自交系获得及抗旱性分析[J].西北植物学报,2011,31(6):1084-1089.
[6]余桂容,杜文平,宋军,等.基因枪介导抗除草剂基因2mG2-epsps转化玉米的初步研究[J].分子植物育种,2010,8(5):885-890.
[7]陈永红,李哲敏,许世卫.农业转基因生物安全性分析[J].中国食物与营养,2003(9):4-5.
[8]姜先荣,杨杰,周衍茂.转基因技术应用及转基因产品安全性研究进展[J].合肥师范学院学报,2008,26(3):99-103.
[9]KNIG A. A framework for designing transgenic crops—science, safety and citizen s concerns[J]. Nature Biotechnology, 2003, 21(11):1274-1279.
[10]STEWART C N, JR, HALFHILL M D, WARWICK S I. Transgene introgression from genetically modified crops to their wild relatives[J]. Nature Review Genetics, 2003, 4(10): 806-817.
[11]王旭静, 贾士荣. 国内外转基因作物产业化的比较[J]. 生物工程学报, 2008, 24(4):541-546.
[12]BAEUMLER S, WULFF D R, TAGLIANI L, et al. A Real-Time Quantitative PCR Detection Method Specific to Widestrike Transgenic Cotton (Event 281-24-236_3006-210-23)[J]. Journal of Agricultural and Food Chemistry, 2006, 54(18): 6527-6534.
[13]LIPP M, SHILLITO R, GIROUX R, et al. Polymerase Chain Reaction Technology as Analytical Tool in Agricultural Biotechnology[J]. Food Compsition and Additives, 2005, 88(1):136-155.
[14]DREO T, PIRC M, RAVNIKAR M. Real-time PCR, a method fit for detection and quantification of Erwinia amylovora[J]. Trees, 2012, 26(1):165-178.
[15]陳颖, 徐宝梁, 苏宁, 等. 实时荧光定量PCR技术在转基因玉米检测中的应用研究[J]. 作物学报, 2004, 30(6): 602-607.
[16]MIRAGLIA M, BERDAL K G, BRERA C, et al. Detection and traceability of genetically modified organisms in the food production chain[J]. Food and Chemical Toxicology, 2004, 42(7):1157-1180.
[17]FERRY N, MULLIGAN E A, MAJERUS M E, et al. Bitrophic and tritrophic effects of Bt Cry3A transgenic potato on beneficial, non-target, beetles[J]. Transgenic Research, 2007, 16(6):795-812.
[18]GUNNING R V, DANG H T, KEMP F C, et al. New resistance mechanism in Helicoverpa armigera threatens transgenic crops expressing Bacillus thuringiensis Cry1Ac toxin[J]. Applied and Environmental Microbiology, 2005, 71(5):2558-2563.
[19]CHHAPEKAR S, RAGHAVENDRARAO S, PAVAN G, et al. Transgenic rice expressing a codon-modified synthetic CP4-EPSPS confers tolerance to broad-spectrum herbicide, glyphosate[J]. Plant Cell Reports, 2015, 34(5):721-731.
[20]DOBNIK D, SPILSBERG B, BOGOZALEC KOSIR A, et al. Multiplex quantification of 12 European Union authorized genetically modified maize lines with droplet digital polymerase chain reaction[J]. Analytical Chemistry, 2015, 87(16):8218-8226.
[21]GERDES L, BUSCH U, PECORARO S. GMOfinder—A GMO Screening Database[J]. Food Analytical Methods, 2012, 5(6):1368-1376.
[22]HUBER I, BLOCK A, SEBAH D, et al. Development and validation of duplex, triplex, and pentaplex real-time PCR screening assays for the detection of genetically modified organisms in food and feed[J]. Journal of Agricultural and Food Chemistry, 2013, 61(43):10293-10301.
[23]GERLACH W L, PRYOR A J, DENNIS E S, et al. cDNA cloning and induction of the alcohol dehydrogenase gene[J]. Proceedings of the National Academy of Sciences, 1982, 79(9):2981-2985.
[24] SN/T 1204-2016,植物及其加工产品中转基因成分实时荧光PCR定性检验方法 [S]. 北京:中国质检出版社, 2016.
[25]ARYA M, SHERGILL I S, WILLIAMSON M, et al. Basic principles of real-time quantitative PCR[J]. Expert Review of Molecular Diagnostics, 2005, 5(2):209-219.
[26]LEE J, MOON S U, LEE Y S, et al. Quantum dot-based molecular beacon to monitor intracellular microRNAs[J]. Sensors (Basel), 2015, 15(6):12872-12883.
[27]ROGERS S O, BENDICH A J. Extraction of DNA from milligram amounts of fresh, herbarium and mummified plant[J]. Plant Molecular Biology, 1985, 5(2):69-76.
[28]HBNER P, WAIBLINGER H-U, PIETSCH K, et al. Validation of PCR methods for quantitation of genetically modified plants in food[J]. Journal of AOAC International, 2001, 84(6):1855-1863.
[29]WISE A, BIRRELL N M. Design and analysis of food frequency questionnaires - review and novel method[J]. International Journal of Food Sciences and Nutrition, 2009, 53(3):273-279.
[30]MATSUOKA T, KURIBARA H, AKIYAMA H, et al. A Multiplex PCR Method of Detecting Recombinant DNAs from Five Lines of Genetically Modified Maize[J]. Journal of the Food Hygienic Society of Japan (Shokuhin Eiseigaku Zasshi), 2001, 42(1):24-32.
[31]R.ERMINGEAT H, REGGIARDO M N I, VALLEJOS R H. Detection and Quantification of Transgenes in Grains by Multiplex and Real-Time PCR[J]. Journal of Agricultural and Food Chemistry, 2002(50):4431-4435.
[32]ALARY R, SERIN A, MAURY D, et al. Comparison of simplex and duplex real-time PCR for the quantification of GMO in maize and soybean[J]. Food Control, 2002(13):235-244.
[33]HERN′ANDEZ M, PLA M, ESTEVE T, et al. A specific real-time quantitative PCR detection system for event MON810 in maize YieldGard based on the 3'-transgene integration sequence[J]. Transgenic Research, 2003(12):179-189.
[34]NAGESWARA-RAO1 M, KWIT C, AGARWAL S, et al. Sensitivity of a real-time PCR method for the detection of transgenes in a mixture of transgenic and non-transgenic seeds of papaya (Carica papaya L.)[J]. BMC Biotechnology, 2013, 13(1):69.
[35]LAUWAARS M, ANKLAM E. Method validation and reference materials[J]. Accreditation and Quality Assurance, 2004, 9(4-5):253-258.
[36]TRAPMANN S,EMONS H. Reliable GMO analysis[J]. Analytical and Bioanalytical Chemistry, 2005, 381(1):72-74.
[37]WU Y, LI J, LI X, et al. Development and strategy of reference materials for the DNA-based detection of genetically modified organisms[J]. Analytical and Bioanalytical Chemistry, 2019, 411(9):1729-1744.
[38]WANG F, FENG J, YE S, et al. Development of a multiplex fluorescence quantitative PCR for detection of genetically modified organisms[J]. Biologia, 2018, 73(1):21-29.
[39]TAKABATAKE R, KAGIYA Y, MINEGISHI Y, et al. Rapid Screening Detection of Genetically Modified Crops by Loop-Mediated Isothermal Amplification with a Lateral Flow Dipstick[J]. Journal of Agricultural and Food Chemistry, 2018, 66(29):7839-7845.
[40]JACCHIA S, KAGKLI D M, LIEVENS A, et al. Identification of single target taxon-specific reference assays for the most commonly genetically transformed crops using digital droplet PCR[J]. Food Control, 2018(93):191-200.
[41]DEMEKE T, DOBNIK D. Critical assessment of digital PCR for the detection and quantification of genetically modified organisms[J]. Analytical and Bioanalytical Chemistry, 2018, 410(17):4039-4050.
(責任编辑 常阳阳)

