染色质结构与肾脏疾病遗传风险解析
邢月仙 张 静 王 琪 综述 杨敬平 校审
随着全基因组关联分析(GWAS)的不断开展,大量与肾脏疾病发病风险相关的遗传变异位点被有效识别,极大地丰富了我们对肾脏疾病遗传背景的认识。虽然GWAS可找到与疾病相关的变异位点,但目前并不清楚这些位点是如何发挥作用的。随着表观遗传学现广泛应用于各个领域,基于染色质结构的生物学信息为明确GWAS遗传变异位点的位置及调控机制提供了有效途径。因此,通过染色质结构解析GWAS对阐述肾脏疾病的发病风险及发生机制具有重要意义。
GWAS在肾脏疾病的应用
发展现状人类个体间的基因序列99.9%是相同的,而0.1%的差异则与复杂疾病的易感性及临床表现多样性密切相关。单核苷酸多态性(SNPs)是指基因组中单个碱基变异引起的DNA序列多态性,约占基因序列差异的90%以上。GWAS可在全基因组范围内找出变异位点,并从中筛选出与疾病发病风险相关的SNPs。自2009年Kottgen等[1]发现与肾小球滤过率显著相关的遗传变异位点后,大量的GWAS相继展开。截至2019年,已有超过1 000 000样本的GWAS Meta分析,研究共揭示了将近300个与慢性肾脏病(标志性状为肾小球滤过率)显著相关的遗传变异位点[2-3]。此外,GWAS还在IgA肾病中发现了18个遗传变异位点[4-5],在膜性肾病中发现了2个遗传变异位点[4]。尽管GWAS已经成为寻找疾病相关遗传变异位点的有利武器,但这些位点是如何发挥作用的并不清楚。因此,寻找GWAS SNPs的靶基因成为了阐述疾病发病风险的必经之路。
寻找肾脏疾病的因果基因基因是控制生物性状的基本遗传单位。因此,阐述SNPs与基因表达的关系有望揭示SNPs在疾病中的作用机制。然而,GWAS发现的与疾病相关的SNPs约有93%位于基因组的非编码区[6]。这意味着大多数与疾病相关的SNPs是通过顺式或反式作用调控靶基因表达而作用于表型。因此,阐述SNPs对基因表达的调控作用是理解疾病的关键。
寻找因果基因是将SNPs与疾病表型进行关联的桥梁。通过不同方法预测得到的SNPs的靶基因皆被称作其候选因果基因。明确SNPs对基因功能的影响及候选因果基因与疾病表型之间的因果关系后,该候选因果基因即可称为疾病的因果基因。以往的研究中,GWAS常通过与基因的线性距离来标记SNPs的候选因果基因。但位于非编码区的SNPs可通过多种方式参与表型决定。例如,与肥胖强相关且位于FTO第一个内含子区的SNPrs9930506,其作为增强子调控距离其几十万碱基之外的基因IRX3和IRX5的表达,而不影响距离最近的FTO的表达[7]。因此,通过与基因距离远近来标记候选因果基因是不可靠的。
为了更好地理解GWAS并确定疾病的因果基因,表达数量性状基因座(e_QTL)被有效运用。e_QTL通过对个体基因型与基因表达进行关联分析,鉴定出与基因表达相关的遗传变异位点,即e_QTL位点。而该基因则被认为是疾病的候选因果基因[8-9]。虽然转录组数据可用于检测组织中的基因表达水平并与样本基因型进行关联性分析,但用于e_QTL分析的转录组测序需要较大样本量,使得来源有限的生物样本分析受到了极大限制。为了解决这一难题,全转录组关联研究(TWAS)应用而生。TWAS提供了一种对复杂性状的候选因果基因进行优先级排序的方法,利用小规模的基因型和基因表达数据作为训练集,拟合基因型与基因表达量之间的关系模型,然后利用该模型估计GWAS人群中的基因表达值,最后对大样本人群的性状表型和预测的基因表达值进行关联分析,实现GWAS数据库的基因-性状关联[10]。
虽然TWAS的应用可以将预测的基因表达与疾病的性状关联起来,但它并不能确保疾病与基因表达之间的因果关系,原因有三:其一,GWAS很少识别单个变异-性状关联,而是在连锁不平衡中识别相关变体的模块;其二,基因间的关联表达、预测表达相关性以及共有的GWAS变异位点均可导致变异位点与基因表达信息的错误匹配;其三,具有来自非性状关联组织的表达谱偏倚性[11]。
随着表观遗传学的快速发展,表观研究已广泛应用于各个领域[12-14]。其中,运用表观遗传学技术揭示染色质结构在解析肾脏疾病的发病机制中有了突破性进展。例如,以染色质一维结构为基础,研究者发现与肾脏病GWAS和e_QTL相关的遗传变异位点常富集在DNA的调控元件区[15]。而对GWAS数据进行精准解读的过程中,相较于以往的关联性分析,染色质三维结构数据可更直观、全面地呈现染色质的空间调控网络。因此,染色质结构所提供的注释信息以及基因表达调控信息在GWAS的精准解读,寻找疾病的因果基因、肾脏病发生机制的精准阐述以及肾脏疾病的精准治疗中具有重要意义[6,13,15]。
染色质一维结构解析疾病发病风险
染色质一维结构的特征染色质一维结构特征常包括DNA甲基化、组蛋白修饰、染色质可及性和非编码RNA调控等。在揭示染色质构象及功能、解读疾病的发病风险中,染色质可及性和组蛋白修饰可提供更具参考价值的生物学信息。染色质的高度折叠、压缩结构仅保留了具有生物活性的基因区域,这些“可接近”的染色质特性称为染色质可及性,可用于识别不同类型的调控元件[16-17]。组蛋白是核小体的重要组成部分,其翻译后修饰主要包括甲基化、乙酰化、磷酸化、泛素化等。基因组不同功能区域具有不同的组蛋白修饰特征。在常见的组蛋白标记中,组蛋白H3赖氨酸4(H3K4)甲基化标记基因具有转录活性:其中,H3K4的一甲基化(H3K4me1)通常标记增强子区,H3K4的三甲基化(H3K4me3)通常标记启动子区;组蛋白H3赖氨酸27的乙酰化(H3K27ac)标记位点的活化状态[18-19]。因此,根据位点的组蛋白修饰特征,可注释基因位点的调节功能。
染色质一维结构解析疾病发病风险的优势染色质可及性特征是由于转录因子结合能引起染色质结构的局部改变,使周围的DNA在物理上变得更容易接近。染色质可及性可用于判断基因位点是否具有转录因子结合并发挥功能的潜质。而组蛋白修饰特征可对基因位点的调节功能进行注释。因此,在与GWAS的联合分析中,染色质一维结构的应用优势众多。
其一,可显示位于非编码区的SNPs是否为功能性调控元件[20-21]。在冠状动脉疾病/缺血性中风的一项研究中,Krause等[21]识别到与疾病性状相关的SNP rs17114036位于基因PLPP3第五个内含子的增强子样调控元件中。研究通过转座酶研究染色质可进入性的高通量测序(ATAC-seq)与染色质免疫共沉淀测序(CHIP-seq)的联合分析发现,该位点为染色质可及性位点,且同时有H3K27ac和H3K4的二甲基化(H3K4me2)修饰,提示该位点为功能性调控元件。该作者在人主动脉内皮细胞中利用CRISPR-Cas9系统选择性地敲除rs17114036所在的~66bp的基因组区域,结果显示接受基因编辑的内皮细胞PLPP3表达显著降低,从而验证该位点确实为功能性调控元件。
其二,可揭示功能性SNPs的作用机制。改变染色质可及性和转录因子结合是遗传变异导致基因表达差异的主要机制[22],提示通过染色质可及性数量性状基因座(ca_QTLs)寻找改变染色质可及性和转录因子结合的SNPs可能在揭示变异位点的作用机制中扮演重要作用。SNP rs488797位于CELF4内含子中的超级增强子区,该位点的C等位基因与该区域的染色质可及性减弱密切相关,为ca_QTL位点。作者通过HaploReg预测SNP rs488797 的C等位基因可导致转录因子FOXA2的结合遭到破坏。经ChIP-seq实验证实,CC纯合子个体中rs488797位点确实不存在FOXA2的结合信号,提示在SNP rs488797位点C等位基因可能通过破坏FOXA2的结合影响增强子活性及基因的表达调控[23]。
其三,可揭示SNPs的组织特异性。例如,多发性硬化相关的SNPs其开放信号主要富集在B淋巴细胞与T淋巴细胞中,而精神分裂症相关的SNPs则主要富集在抑制性神经元等神经组织中[16]。提示SNPs只在某些特定的细胞类型中发挥作用。
综上所述,一维染色质结构可通过判断调控元件的功能、阐述其作用机制以及明确其组织特异性的作用方式,为GWAS SNPs向分子生物学机制转化提供了有利途径(图1)。
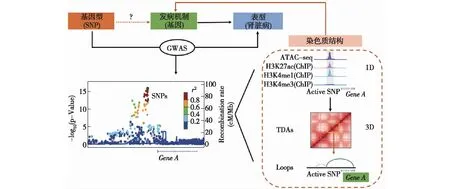
图1 染色质结构在解析肾脏疾病遗传风险中的作用SNP:单核苷酸多态性;GWAS:全基因组关联分析;TADs:拓扑关联结构域;Loops:染色质环;1D:一维;3D:三维
染色质三维结构解析疾病发病风险
染色质三维结构的特征染色质三维结构常在不同的组织器官中表现出共同的特征结构,包括:染色体域、染色体区室、拓扑关联结构域(TADs)及染色质环。在真核生物的细胞核中,每个染色体都被限制在一个离散区域,该区域称为染色体域,其内部DNA呈有序排列,有助于调控元件以恰当的方式对特定的基因位点进行调控[24]。根据状态的不同,染色质可被分为A和B两个区室。其中,A区室富集大量基因,具有高转录活性;而B区室所含基因贫乏,且呈低转录活性[25]。区室中的TADs是基因调控的基本单元,是相对独立的基因区域。TADs的形成限定了内部调控元件的空间交互范围,并阻止不同TADs之间的染色质互作[26]。染色质环是基因表达调控的功能单元,其形成是调控元件与靶基因互作时的主要机制,该结构的形成使线性距离很远的基因位点可以在空间上密切接触[27-28]。
染色质三维结构在解析疾病发病风险中的优势染色质三维结构可反映线性距离很远的调控元件于空间上的相互接触,亦可反映调控发生的基本单元,常用于揭示染色质互作信息、定位调控元件的靶基因。Liguel-Escaladd等[18]在2型糖尿病的相关研究中,用启动子Capture Hi-C技术揭示了2型糖尿病的SNPrs11257655与远端基因OPTN的启动子区于空间上的高频接触,提示SNP rs11257655可能调控OPTN的表达。该作者通过基因编辑技术证实,敲除该SNP位点OPTN表达下调,验证了SNP rs11257655对基因OPTN的调控作用。因此,基于染色质三维结构可精准揭示线性距离很远的调控元件间的互作信息,可用于揭示GWAS中SNPs的远端靶基因。
除染色质环可直接显示调控元件间的空间互作外,TADs亦可作为解析GWAS的有利手段。例如,2型糖尿病中与性状胰岛素分泌相关的SNP rs10428126位于IGF2BP2基因区域。根据Hi-C结果显示,IGF2BP2是SNP rs10428126所在的TAD中唯一的基因,提示其可能是该位点的候选因果基因。经验证,SNP rs10428126的风险等位基因C可显著降低胰岛的染色质可及性、增强子活性以及基因IGF2BP2的表达[13]。
综上所述,在与GWAS的联合分析中,染色质三维结构可精确定位与疾病相关的遗传变异位点所调控的靶基因,有助于揭示疾病的发生发展机制(图1)。
染色质结构解析肾脏疾病发病风险
与肾癌易感性相关的SNP rs35252396位于致癌基因MYC(上游136 kb)和致癌性长链非编码RNAPVT1(下游14 kb)之间。Grampp等[29]前期的研究证实,MYC和PVT1是转录因子HIF的靶基因。在肾脏透明细胞癌中,HIF的结合信号几乎与SNP rs35252396于基因组中的位置重合。经FAIRE-seq和ChIP-seq研究证实,该位点位于染色质可及性区域,且存在H3K4me1和H3K27ac的结合信号,提示该位点为活性增强子区域,可能通过HIF的结合驱动MYC和PVT1转录在肾脏肿瘤的发生中起重要作用。为了验证这一猜想,作者运用4C技术观察到SNP rs35252396所在且有HIF结合的调控元件区域存在与基因MYC和PVT1启动子区的染色质互作。此外,该作者利用CRISPR/Cas9技术在体外细胞中证实干扰这一位点确实可出现MYC和PVT1表达水平的降低。为了进一步揭示SNP rs35252396是否通过HIF的结合介导上述调控过程,该作者在杂合的肾癌细胞系及原代近端肾小管上皮细胞中展开研究,通过ChIP-seq和FAIRE-seq证实,风险基因型样本中HIF结合增强且有染色质可及性增强。因此,该作者提出SNP rs35252396通过影响HIF的结合驱动MYC和PVT1转录并在肾脏肿瘤的发生中起重要作用。
为了验证肾脏GWAS SNPs与其潜在靶基因之间的物理关联,Sieber等[15]将人肾脏组织新鲜分离的肾小球进行了Hi-C实验,并因此确定了42个SNPs对应的46个靶基因。其中,与肾结石和骨密度相关的SNP rs219779/rs219780与基因SIM2存在远程物理互作,且二者的互作信息位于同一个TAD中,提示SIM2为SNP rs219779/rs219780的靶基因。在现有的慢性肾脏病的相关研究中,Brandt等[30]通过STARR-seq评估了遗传变异对DNA调控元件的影响,此过程共发现了39个功能性的遗传变异位点。为了鉴定DNA调控元件的靶基因,作者应用4C测序技术对肾小球内皮细胞和肾小管上皮细胞分别进行了测序分析。分析结果显示39个慢性肾脏病相关的遗传变异位点与304因果基因的转录起始位点存在相互作用。尽管上述两项案例缺少功能实验对结果进行验证,但染色质结构所提供的生物学信息极大地丰富了肾脏疾病发病风险的机制研究。
展 望
随着表观遗传学的不断发展,越来越多的研究基于染色质结构解析疾病的发病风险。染色质一维及三维结构的检测方法现已逐渐发展成熟,美国国立卫生研究院也已建立了迄今为止最大的人类表观基因组图谱,为运用染色质结构揭示肾脏疾病的发病风险和发病机制提供了丰富资源和有效手段。然而,肾脏细胞种类繁多,不同细胞发挥的作用又不尽相同。因此,建立和完善肾脏细胞种类特异性的染色质特征图谱对了解肾脏基因组功能意义重大。在与GWAS的联合分析中,有效揭示肾脏病相关SNPs于哪种细胞类型中发挥怎样的调控作用将为肾脏疾病的科学研究提供精准靶向。随着单细胞测序技术的不断进展,染色质结构捕获与单细胞测序的联合应用将逐渐拉开疾病发生发展的神秘面纱,也将为肾脏疾病的精准治疗带来深远影响。

