广西不同地区克氏原螯虾群体遗传多样性微卫星分析
黄小芳 唐章生 刘俊丹 张宏燕 钟一治 卢智发 侯树鉴 王大鹏 陆专灵

摘要:【目的】了解當前广西克氏原螯虾(Procambarus clarkii)群体的遗传背景,为其人工养殖及种质资源保护提供参考依据。【方法】选取广西5个地区(南宁、大塘、融水、柳州和来宾)的克氏原螯虾群体作为研究对象,利用8对微卫星引物对各克氏原螯虾群体基因组DNA进行PCR检测,然后通过PopGene32和NTSYSpc 2.1等在线软件分析广西克氏原螯虾群体的遗传多样性。【结果】南宁(NN)、大塘(DT)、融水(RS)、柳州(LZ)和来宾(LB)5个克氏原螯虾群体的平均有效等位基因数(Ne)为2.5142~3.0574,平均期望杂合度(He)为0.5708~0.6489,平均多态信息含量(PIC)为0.4899~0.5843,存在中度至高度的遗传多样性,其中南宁群体和大塘群体的遗传多样性略低于融水群体、柳州群体和来宾群体。5个克氏原螯虾群体的平衡偏离指数(D)均为负值,且发生一定程度的Hardy-Weinberg平衡偏离,杂合子缺失现象普遍存在。5个克氏原螯虾群体间的基因流(Nm)为2.3898~6.0284,遗传分化指数(Fst)为0.0398~0.0947,表明各群体间存在广泛的基因交流,仅存在低度至中度的遗传分化;各克氏原螯虾群体间的遗传相似系数和遗传距离分别为0.6861~0.8583和0.1528~0.3768,其中,遗传距离趋势与Fst趋势一致,而遗传相似系数与Nm趋势一致,均表明柳州群体与来宾群体具有高度的相似性,遗传距离较近。UPGMA聚类分析结果也显示,南宁群体和大塘群体聚类为一支,而柳州群体先与来宾群体聚类再与融水群体聚类成另一支。【结论】广西不同地区克氏原螯虾群体具有一定的遗传多样性,但其杂合子缺失现象普遍存在,应通过适当引种及加强不同地区群体间的遗传交流,保护好克氏原螯虾的优良种质资源。
关键词: 克氏原螯虾;遗传多样性;微卫星位点;杂合子缺失;广西
中图分类号: S917.4 文献标志码: A 文章编号:2095-1191(2020)02-0437-08
Genetic diversity microsatellite analysis of Procambarus clarkii populations in different regions of Guangxi
HUANG Xiao-fang1,2, TANG Zhang-sheng1, LIU Jun-dan2, ZHANG Hong-yan2,
ZHONG Yi-zhi1,3, LU Zhi-fa1, HOU Shu-jian1, WANG Da-peng1*, LU Zhuan-ling1*
(1Guangxi Academy of Fishery Sciences/Guangxi Key Laboratory of Aquatic Genetic Breeding and Healthy Aquaculture, Nanning 530021, China; 2College of Animal Science and Technology/State Key Laboratory for Conservation and Utilization of Subtropical Agro-bioresources, Guangxi University, Nanning 530004, China; 3Guangxi Nanning Yanleshang Biotechnology Co., Ltd., Nanning 530004, China)
Abstract:【Objective】To understand the genetic background of Procambarus clarkii populations in Guangxi, and provide reference for its artificial culture and germplasm conservation. 【Method】The P. clarkii populations in five regions (Nanning, Datang, Rongshui, Liuzhou and Laibin) of Guangxi were selected as research objects, and the genomic DNA of five populations was detected by PCR using eight pairs of microsatellite primers, then the genetic diversity of P. clarkii populations in Guangxi was analyzed using PopGene32 and NTSYSpc 2.1. 【Result】The results showed that the average effective number of alleles (Ne) in Nanning (NN), Datang (DT), Rongshui (RS), Liuzhou (LZ) and Laibin (LB) was 2.5142-3.0574, and the average expected heterozygosity (He) was 0.5708-0.6489, the average polymorphic information content (PIC) was 0.4899-0.5843, representing a moderate to high genetic diversity. Among them, the genetic diversity of NN and DT populations were slightly lower than those of RS, LZ and LB populations. The equilibrium indexes (D) of five populations were negative, indicating a certain degree of Hardy-Weinberg equilibrium deviation occurred, and he-terozygous deletion was ubiquitous. The gene flow(Nm) among different P. clarkii populations was 2.3898-6.0284, and the genetic differentiation index(Fst) was 0.0398-0.0947, demonstrating that there were extensive gene exchanges and only low or moderate differentiation between different populations. The genetic similarity coefficient and genetic distance between different populations were 0.6861-0.8583 and 0.1528-0.3768, respectively, among them, the trend of genetic distance was consistent with Fst, and the trend of genetic similarity coefficient was consistent with Nm, demonstrating that LZ and LB populations had high similarity and short genetic distance. UPGMA cluster analysis displayed that NN and DT populations belonged to the same branch; LZ and LB populations were grouped into one category and then merged with RS population into the other branch. 【Conclusion】The P. clarkii populations in different regions of Guangxi have a certain high genetic diversity, however, the heterozygous deletion is very common. Therefore, it is necessary to protect the good germplasm resources of P. clarkii by suitable introduction and strengthening the genetic communication between different populations.
Key words: Procambarus clarkia; genetic diversity; microsatellite loci; heterozygote deletion; Guangxi
Foundation item: Guangxi Innovation Driven Development Project(Guike AA17204094-6,Guike AA17204095-4); Independent Project of Guangxi Key Laboratory of Aquatic Genetics Breeding and Healthy Aquaculture (18-A-04-07)
0 引言
【研究意义】克氏原螯虾(Procambarus clarkii)又称淡水小龙虾,原产于北美洲,因其味道鲜美、营养丰富、价格相对低廉,深受消费者喜爱,并迅速扩散至夏威夷、日本、欧洲和尼罗河流域(Yi et al.,2018;徐滨等,2019)。克氏原螯虾于20世纪30年代末由日本引入我国南京(张爱军和沈继红,2005),因其适应能力强,在长江流域一带大量繁殖,现已广泛分布于我国南方地区的湖泊和沟渠,是一种重要的水产资源(宋光同等,2018)。尤其是戴爱云(1983)首次提倡利用鳌虾作为水产资源加以开发以来,克氏原螯虾养殖业得到快速发展,目前全国养殖面积已超过100万ha,且有进一步扩大的趋势(严维辉等,2019)。随着克氏原螯虾养殖地域和规模的不断扩大,其逆向选择及种苗自繁自育的养殖模式导致克氏原螯虾个头逐年变小、病害频发、种质退化严重,因此,迫切需要从基因水平上开展不同群体遗传变异及遗传关系研究,为其良品繁育提供种质资源,以促进克氏原螯虾养殖业的健康发展。【前人研究进展】微卫星(Microsatellite)又称简单序列重复(Simple sequence repeats,SSR),是一类在真核生物基因组中高度变异的简单重复DNA序列(Schl?tterer and Tautz,1992),符合孟德尔遗传规律,具有共显性和多态性,且稳定性较好,是简单快速的基因型检测对象(秦海峰等,2014),已广泛应用于生物群体间的遗传关系研究(Cruz et al.,2002;孙效文等,2008;熊良伟等,2018)。Belfiore和May(2000)、Zhu和Yue(2008)先后对克氏原螯虾基因组文库进行筛选,分别获得23和11对可良好扩增微卫星位点的引物,均可用于研究克氏原螯虾的物种入侵路线、遗传多样性和种群结构。王长忠等(2009)利用17对微卫星引物对长江下游地区4个克氏原螯虾群体遗传多样性进行分析,结果表明其遗传多样性处于中等水平,群体间基因流水平较高、遗传分化程度较小。曹玲亮等(2010)选用9对微卫星引物构建安徽三大水系的克氏原螯虾种群遗传格局,发现安徽地区的克氏原螯虾遗传多样性水平较高,尤其是杂合度较高,认为水系间的交流是种群扩散的主要途径。费腾等(2010)通过5个高多态性的微卫星分子标记研究封闭式小水体养殖对克氏原螯虾的影响,证实3~5年的封闭性养殖还不足以显著改变其遗传多样性。彭刚等(2010)基于7对微卫星引物对比分析3个克氏原螯虾野生群体和养殖群体间的遗传多样性,结果发现野生群体相对于养殖群体具有较高的遗传多样性。Li等(2012)通过线粒体基因序列和12对微卫星位点对克氏原螯虾进入我国的起始地点、传播方式、遗传多样性和遗传结构进行分析,结果证实南京是克氏原螯虾最初在我国传播的起始地点,且与日本来源的克氏原螯虾含有相似遗传成分,但至今并未经历明显的种群扩张。邢智珺等(2014)使用8个微卫星分子标记分析江苏省8个主要产区克氏原螯虾野生群体的遗传多样性,结果发现江苏主要产区克氏原螯虾野生群体的遗传多样性较高,且存在群体间中度分化现象。李喜莲等(2016)基于微卫星分子标记对桐庐、太湖、湖北、江西、洪泽湖、崇明、廣西和海盐等8个克氏原螯虾群体的遗传多样性和遗传结构进行研究,结果表明8个群体的平均观测杂合度(Ho)为0.5257~0.7400,平均期望杂合度(He)为0.6238~0.7434,其中以广西群体的遗传多样性最高。【本研究切入点】目前,国内已有较多关于克氏原螯虾遗传多样性分析的报道,但只有李喜莲等(2016)的研究涉及到广西群体,即广西地区的克氏原螯虾遗传多样性尚未得到系统研究。【拟解决的关键问题】选取8个微卫星分子标记对广西不同地区的克氏原螯虾群体进行遗传多样性分析,旨在了解当前广西克氏原螯虾群体的遗传背景,为其人工养殖及种质资源保护提供参考依据。
1 材料与方法
1. 1 试验材料
收集广西5个地区的克氏原螯虾群体样本,每个地区20~30尾,分别取自南宁(NN)、大塘(DT)、融水(RS)、柳州(LZ)和来宾(LB)的小龙虾养殖场,其虾苗均未经过人工选育。不同地区的克氏原螯虾虾苗来源、采集时间和数量信息详见表1。
1. 2 微卫星引物筛选与合成
从Belfiore和May(2000)已发表的克氏原螯虾微卫星位点中挑选出8对微卫星引物用于克氏原螯虾群体遗传多样性分析,并委托生工生物工程(上海)股份有限公司合成。微卫星引物信息见表2。
1. 3 基因组DNA抽提
根据天根生化科技(北京)有限公司的DNA抽提试剂盒(#DP304)说明抽提克氏原螯虾基因组DNA,具体步骤:取20 mg背部肌肉剪碎,加入200 μL缓冲液GA和20 μL Proteinase K溶液,混匀,56 ℃水浴锅消化3 h;加入200 μL缓冲液GB,充分混匀,70 ℃作用10 min后加入200 μL无水乙醇,充分振荡混匀15 s;将所得溶液加入吸附柱CB3中,12000 r/min离心1 min,弃废液;将吸附柱CB3放回收集管中,加入500 μL缓冲液GD,12000 r/min离心1 min,弃废液;将吸附柱CB3放回收集管中,再加入600 μL漂洗液PW,12000 r/min离心1 min,弃废液;将吸附柱CB3放回收集管中,12000 r/min空离2 min,室温静置5 min,向吸附柱CB3中央滴加80 μL灭菌ddH2O,12000 r/min离心2 min洗脱DNA;最后在NanoDrop 1000分光光度计(Thermo)上测定DNA浓度,选择OD260/280在1.8~2.2的样品,-80 ℃保存备用。
1. 4 PCR扩增
采用8对微卫星引物对待检测的克氏原螯虾样品进行PCR扩增,反应体系20.0 μL:10×Taq PCR MasterMix聚合酶[天根生化科技(北京)有限公司]10.0 μL,正、反向引物(10 μmol/L)各0.5 μL,DNA模板3.0 μL(400 ng),ddH2O 6.0 μL。扩增程序:95 ℃预变性5 min;95 ℃ 30 s,56 ℃ 30 s,72 ℃ 1 min,进行35个循环;72 ℃延伸10 min。取出PCR扩增产物,制备2.0%琼脂糖凝胶,并按样品编号顺序,以20和50 bp DNA Ladder(TaKaRa)为参考,每孔加样8.0 μL,90 V下电泳3~5 h(不同微卫星座位的电泳时间有所不同),待条带分离开后用GE Healthcare ImageQuant LAS500(Uppsala)成像仪进行成像,并采用ImageQuant TL进行条带分析。
1. 5 统计分析
经电泳获得各样品微卫星位点信息后,采用PopGene32和NTSYSpc 2.1等在线软件分析不同地区克氏原螯虾群体微卫星位点的等位基因数(Na)、有效等位基因数(Ne)、Shannon?s信息指数(I)、Ho、He、基因流(Nm)、遗传分化指数(Fst)、遗传相似性系数及遗传距离等参数,使用PHYLIP v3.5进行聚类分析。Hardy-Weinberg平衡偏離指数(D)是反映群体在某个微卫星位点偏离Hardy-Weinberg平衡程度的参数,计算公式为D=(Ho-He)/He;多态信息含量(PIC)反映微卫星DNA的变异程度,是衡量微卫星位点多样性的重要指标(Botstein et al.,1980)。
2 结果与分析
2. 1 PCR扩增电泳结果
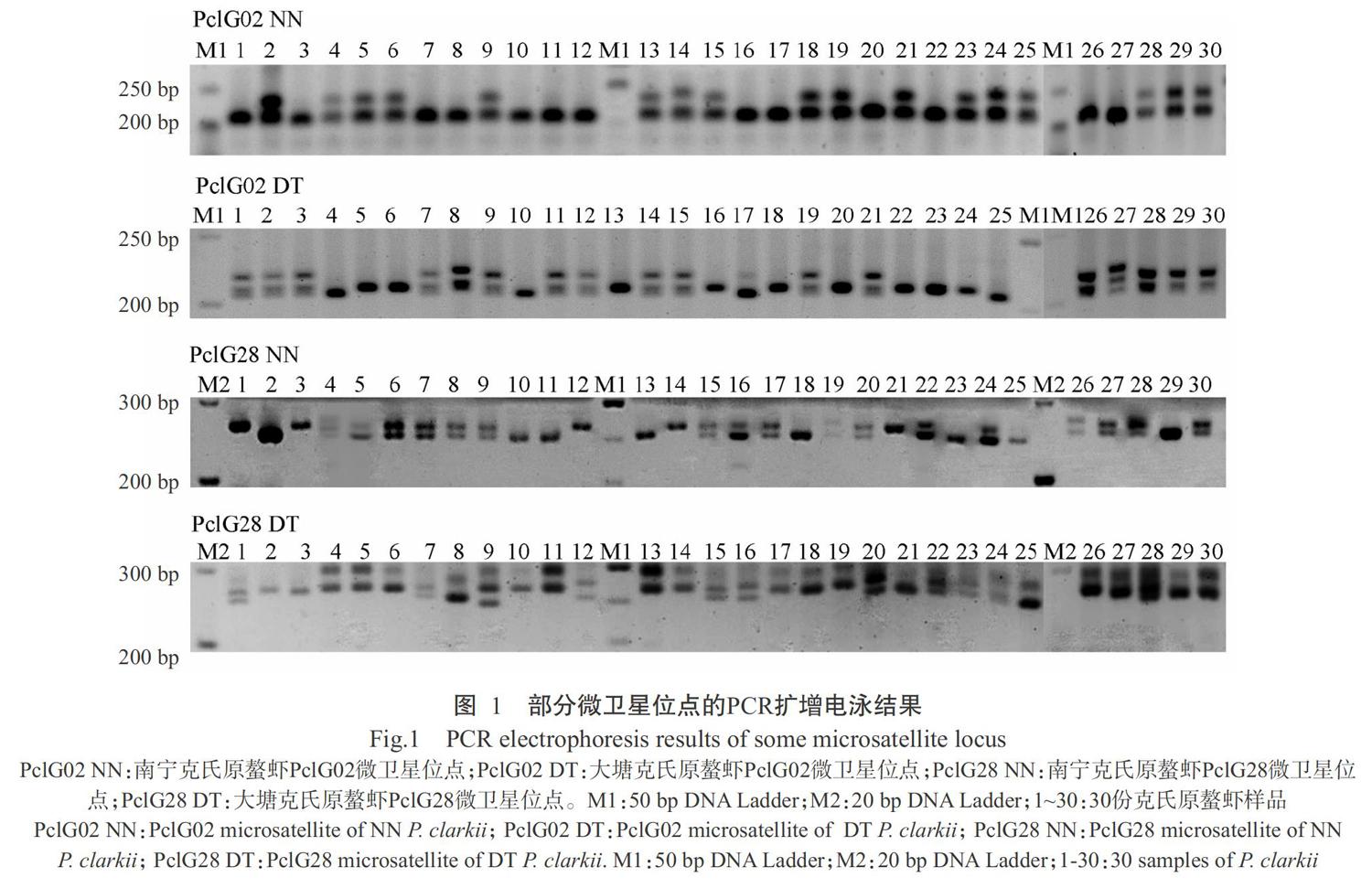
采用8对微卫星引物对广西不同地区克氏原螯虾群体共140份样品进行PCR扩增,经电泳凝胶成像均获得清晰的电泳条带,部分微卫星位点的PCR扩增电泳结果如图1所示。
2. 2 不同地区克氏原螯虾群体的等位基因分析结果
选取8个微卫星位点对广西不同地区克氏原螯虾群体的等位基因及有效等位基因进行分析,结果发现5个克氏原螯虾群体的8个微卫星位点共检测到37个等位基因,平均Na为3.925个,每个微卫星位点的Na在3~6个,其中,PclG02的Na最少(3个),PclG07的Na最多(6个),各地区克氏原螯虾群体的平均Na为3.750~4.250(表3),不存在明显差异。8个微卫星位点的平均Ne为1.5267~4.6997;各地区克氏原螯虾群体的平均Ne为2.5142~3.0574,其中柳州群体和融水群体的Ne较高(2.9328和3.0574),而南宁群体的Ne较低(2.5142)。
2. 3 不同地区克氏原螯虾群体内的遗传多样性分析结果
通过PopGene32对广西不同地区克氏原螯虾群体8个微卫星位点进行群体内遗传多样性分析,结果显示,南宁克氏原螯虾群体的平均 I 为0.9843,大塘、融水、柳州和来宾4个克氏原螯虾群体的平均 I 则在1.1000以上(表3)。从表4可看出,5个克氏原螯虾群体在8个微卫星位点中均存在不同程度的多态性。其中,5个群体的平均Ho在0.3563~0.5208,以南宁群体和柳州群体最高、来宾群体最低;但南宁群体的平均He最低(0.5708),而其他4个群体均在0.6200以上。南宁克氏原螯虾群体的平均PIC为0.4899,呈中度多态性(0.25
5个克氏原螯虾群体的平均D均为负值,表明广西克氏原螯虾群体总体上处于杂合子缺失状态。南宁群体中有4个微卫星位点的杂合子缺失,平均D最接近于0,相对于其他群体而言较平衡;其他4个群体的8个微卫星位点中有6~7个处于杂合子缺失状态,以来宾群体最明显,D为-0.4418,杂合子缺失及偏离Hardy-Weinberg平衡最严重(表4)。经卡方检验,发现南宁群体有4个微卫星位点处于极显著水平(P<0.01,下同)及1个微卫星位点处于显著水平(P<0.05,下同),其他4个群体则存在6~8个微卫星位点处于显著或极显著水平(表5),表明广西不同地区克氏原鳌虾已发生不同程度的Hardy-Weinberg平衡偏离。
2. 4 不同地区克氏原螯虾群体间的遗传分化分析结果
采用PopGene32对广西不同地区克氏原螯虾群体进行种群间的遗传分化分析,结果显示,5个群体间的Nm在2.3898~6.0284(表6),说明不同群体间存在一定程度的交流;其中,柳州群体与来宾群体的Nm最高(6.0284),其次是融水群体与柳州群体的Nm(5.2530)及南宁群体与柳州群体的Nm(4.2289),说明这些地区属于随机交配的群体;而其他群体间的Nm在2.0000~4.0000,属于高度交流的群体。群体间的Nm越大,则Fst越小。5个克氏原螯虾群体间的Fst在0.0398~0.0947,其中,柳州群体与来宾群体的Fst最小,其次是柳州群体与融水群体(0.0454),而其他群体间处于中度分化水平,其所有群体间尚未出现高度分化现象。
由表7可知,广西不同地区克氏原螯虾群体间的遗传相似系数在0.6861~0.8583,均具有较高的亲缘关系,对应的遗传距离为0.1528~0.3768。其中,柳州群体与来宾群体的遗传相似系数最高,遗传距离最近,表明这两个群体间的亲缘关系最近,遗传变异程度较小;大塘群体与来宾群体的遗传相似系数最低,遗传距离最远,表明这两个群体间在5个群体中的亲缘关系较远,变异程度相对较大。
2. 5 不同地区克氏原螯虾群体的遗传聚类分析结果
基于遗传距离通过PHYLIP v3.5对广西不同地区克氏原螯虾群体进行UPGMA聚类分析,结果显示5个克氏原螯虾群体可分为两大分支(图2),其中,南宁群体和大塘群体聚类为一支,而柳州群体先与来宾群体聚类再与融水群体聚类成另一支。由各克氏原螯虾群体间的遗传距离长度(在各自线条上方)也可知,柳州群体与来宾群体的遗传距离最近。
3 讨论
明确克氏原螯虾的生物多样性,可提高其育种筛选效率,进而增强克氏原螯虾的适应能力。广西克氏原螯虾的生物多样性至今尚未得到系统研究,仅有李喜莲等(2016)曾报道广西克氏原螯虾群体相对于其他省份的克氏原螯虾群体具有较高遗传多样性,但广西不同地区克氏原螯虾群体间并未进行系统比较。开展广西不同克氏原螯虾群体生物多样性分析除了有利于促进优势物种繁育外,对了解克氏原螯虾在华南地区的遗传多样性也具有重要意义。王长忠等(2009)研究表明,长江中下游地区克氏原螯虾群体的遗传多样性均处于中等水平,而南京地区是我国最早引进克氏原螯虾的地方,故其群体具有较高遗传多样性。彭刚等(2010)研究发现,长江流域4个克氏原螯虾群体均已发生一定程度的Hardy-Weinberg平衡偏离,群体内的基因型频繁发生变动,且野生群体的遗传多样性高于养殖群体。本研究选取广西5个地区(南宁、大塘、融水、柳州和来宾)的克氏原螯虾群体作为研究对象,利用8个微卫星位点进行生物多样性分析,结果发现5个克氏原螯虾群体的Na在30~34个,平均Ne在2.5142~3.0574,与王长忠等(2009)、费腾等(2010)、彭刚等(2010)的研究结果相似。广西不同地区克氏原螯虾群体的平均He均较高,即存在一定程度的遗传多样性;PIC在0.4899~0.5843,高于王长忠等(2009)、费腾等(2010)的研究结果,而与彭刚等(2010)的研究结果相似,说明广西克氏原螯虾群体的多态性高于封闭式养殖群体;5个克氏原螯虾群体的 I 在0.9843~1.1856,也说明广西克氏原螯虾群体具有较高的遗传多样性。此外,进一步分析发现广西不同地区克氏原螯虾群体的平均D均为负值,总体上处于杂合子缺失状态,与王长忠等(2009)的研究结果相似,究其原因可能是群体数量较少且瓶颈效应的出现导致稀有基因缺失。
Fst是衡量群体间遗传分化的重要参数,Fst在0.05~0.15属于中等分化水平,其与Nm呈负相关;Nm则反映群体间基因交流的程度,Nm越大基因交流越頻繁(刘倩倩等,2018)。本研究结果显示,柳州群体与来宾群体的Nm最大、Fst最小,说明两个群体间进行交流的程度最高,其次是柳州群体与融水群体,可能是由于地理距离较近及频繁人为引种所致;而来宾群体与南宁群体及大塘群体的基因交流相对较少,可能是这些群体是不同的种群,且具有各自独特的遗传多样性。遗传距离可反映群体间的变异情况和分化程度(Crawford and Littlejohn,1998),与其遗传相似系数呈对立关系,而遗传相似系数代表着群体间的亲缘关系。广西不同地区克氏原螯虾群体的遗传距离趋势与Fst趋势一致,而遗传相似系数与Nm趋势一致,均表明柳州群体与来宾群体具有高度的相似性,遗传距离较小,与这两个地区的地理距离最近相符。UPGMA聚类分析结果也显示,南宁群体和大塘群体聚类为另一支,而柳州群体先与来宾群体聚类再与融水群体聚类成一支,其聚类分析结果符合地理区域分布。广西5个地区的克氏原螯虾群体虽然聚类分成两支,但各群体间均有不同程度的基因交流,保持着中度至高度的遗传多态性。其中,南宁群体和大塘群体的遗传多样性略低于融水群体、柳州群体和来宾群体,可能与其引种有关,为提高克氏原螯虾遗传多样性,可适当从遗传多样性较高的融水、柳州或来宾等地引种进行杂交,选育出多样性丰富的群体进行繁殖,保证各地区克氏原螯虾群体的遗传多样性。广西不同地区克氏原螯虾群体均处于杂合子缺失状态,与南京克氏原螯虾群体杂合子缺失的结论(王长忠等,2009)相似,可能与养殖环境和养殖时间有关,为避免杂合子缺失,还建议适当从国外引进优良品种,并加强不同地区间克氏原螯虾群体的遗传交流。
4 结论
广西不同地区克氏原螯虾群体具有一定的遗传多样性,但其杂合子缺失现象普遍存在,应通过适当引种及加强不同地区群体间的遗传交流,保护好克氏原螯虾的优良种质资源。
参考文献:
曹玲亮,周立志,张保卫. 2010. 安徽三大水系入侵物种克氏原螯虾的种群遗传格局[J]. 生物多样性,18(4):398-407. [Cao L L,Zhou L Z,Zhang B W. 2010. Genetic patterns of an invasive Procambarus clarkii population in the three river basins of Anhui Province[J]. Biodiversity Science,18(4):398-407.]
戴爱云. 1983. 介绍一种水产资源——蝲蛄[J]. 动物学杂志,(3):48-50. [Dai A Y. 1983. Introducing an aquatic resource— Crayfish[J]. Chinese Journal of Zoology,(3):48-50.]
费腾,李佳佳,黄越峰,黄成,唐建清. 2010. 封闭小水体养殖对克氏原螯虾基因多样性的影响[J]. 江苏农业科学,(5):67-69. [Fei T,Li J J,Huang Y F,Huang C,Tang J Q. 2010. Effects of enclosed water body on genetic polymorphisms of Procambarus clarkii[J]. Jiangsu Agricultural Sciences,(5):67-69.]
李喜莲,李飞,朱俊杰,顾志敏. 2016. 基于SSR标记的克氏原螯虾种质资源遗传多样性研究[J]. 华中农业大学学报,35(2):63-68. [Li X L,Li F,Zhu J J,Gu Z M. 2016. Genetic diversity of the red swamp crayfish(Procambarus clarkii) resources based on SSR markers[J]. Journal of Huazhong Agricultural University,35(2):63-68.]
刘倩倩,叶浩婷,李放,王荣婷,田恩伟. 2018. 杭白芷种质资源遗传多样性的SSR分析[J]. 南方农业学报,49(3):418-423. [Liu Q Q,Ye H T,Li F,Wang R T,Tian E W. 2018. SSR analysis for genetic diversity of Angelica dahu-rica var. formosana[J]. Journal of Southern Agriculture,49(3):418-423.]
彭刚,刘伟杰,李佳佳,严维辉,唐建清. 2010. 长江流域3个克氏原螯虾野生群体遗传结构的微卫星分析[J]. 江苏农业学报,26(5):1115-1117. [Peng G,Liu W J,Li J J,Yan W H,Tang J Q. 2010. Microsatellite DNA analysis of genetic structure of three wild populations of Procambarus clarkii in the Yangtze River basin[J]. Jiangsu Journal of Agricultural Sciences,26(5):1115-1117.]
秦海峰,龙宁,吴建国,石春海. 2014. 甜叶菊微卫星富集文库的构建与多态性标记的筛选[J]. 作物学报,40(3):447-456. [Qin H F,Long N,Wu J G,Shi C H. 2014. Construction of microsatellite-enriched library and isolation of icrosatellite markers in Stevia rebaudiana[J]. Acta Agro-nomica Sinica,40(3):447-456.]
宋光同,何吉祥,吳本丽,陈静,黄龙,汪翔,武松. 2018. 克氏原螯虾雌性生殖系统发育及组织结构观察[J]. 江西农业学报,30(2):68-75. [Song G T,He J X,Wu B L,Chen J,Huang L,Wang X,Wu S. 2018. Study on development of female reproductive system and histological structure of Procambarus clarkii[J]. Acta Agriculturae Jiangxi,30(2):68-75.]
孙效文,张晓锋,赵莹莹,张研,贾智英,常玉梅,鲁翠云,梁利群. 2008. 水产生物微卫星标记技术研究进展及其应用[J]. 中国水产科学,15(4):689-703. [Sun X W,Zhang X F,Zhao Y Y,Zhang Y,Jia Z Y,Chang Y M,Lu C Y,Liang L Q. 2008. Development and application of microsa-tellite markers in aquatic species[J]. Journal of Fishery Sciences of China,15(4):689-703.]
王长忠,李忠,梁宏伟,呼光富,吴勤超,邹桂伟,罗相忠. 2009. 长江下游地区4个克氏原螯虾群体的遗传多样性分析[J]. 生物多样性,17(5):518-523. [Wang C Z,Li Z,Liang H W,Hu G F,Wu Q C,Zou G W,Luo X Z. 2009. Genetic diversity in four Procambarus clarkii populations in the lower reaches of the Yangtze River[J]. Biodiversity Science,17(5):518-523.]
邢智珺,姜虎成,陆伟,钱照君,于宏伟,李家乐. 2014. 江苏8个克氏原螯虾群体遗传多样性微卫星分析[J]. 上海海洋大学学报,23(5):656-662. [Xing Z J,Jiang H C,Lu W,Qian Z J,Yu H W,Li J L. 2014. Genetic diversity analysis of eight Procambarus clarkii stocks in Jiangsu Province based on microsatellites[J]. Journal of Shanghai Ocean University,23(5):656-662.]
熊良伟,王帅兵,岳丽佳,王建国,陶桂庆,徐亮,王权. 2018. 宽体金线蛭基因组SSR序列特征分析及其分子标记开发[J]. 南方农业学报,49(11):2298-2303. [Xiong L W,Wang S B,Yue L J,Wang J G,Tao G Q,Xu L,Wang Q. 2018. SSR sequence characters for genome of Whitmania pigra Whitman and development of molecular markers[J]. Journal of Southern Agriculture,49(11):2298-2303.]
徐滨,李忠,魏开金,马宝珊,朱祥云,徐进. 2019. 6个克氏原螯虾群体形态学分析[J]. 淡水渔业,49(6):27-32. [Xu B,Li Z,Wei K J,Ma B S,Zhu X Y,Xu J. 2019. Morphological variations among six populations of Procambarus clarkia[J]. Freshwater Fisheries,49(6):27-32.]
嚴维辉,唐建清,许志强,李佳佳. 2019. 克氏原螯虾大棚育苗试验总结[J]. 水产养殖,(12):8-9. [Yan W H,Tang J Q,Xu Z Q,Li J J. 2019. Summary of greenhouse seedling experiment of Procambarus clarkii[J]. Journal of Aquaculture,(12):8-9.]
张爱军,沈继红. 2005. 龙虾的综合加工利用[J]. 中国资源综合利用,(9):35-36. [Zhang A J,Shen J H. 2005. The comprehensive machining utilization of Chinese lobster[J]. China Resources Comprehensive Utilization,(9):35-36.]
Belfiore N,May B. 2000. Variable microsatellite loci in red swamp crayfish,Procambarus clarkii,and their characte-rization in other crayfish taxa[J]. Molecular Ecology,9(12):2231-2234.
Botstein D,White R L,Skolnick M H,Davis R W. 1980. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. The American Journal of Human Genetics,32(3):314-331.
Crawford A M,Littlejohn R P. 1998. The use of DNA mar-kers in deciding conservation priorities in sheep and other livestock[J]. Animal Genetic Resources Information,23:21-26.
Cruz P,Mejia-Ruiz C H,Perez-Enriquez R,Ibarra A M. 2002. Isolation and characterization of microsatellites in Pacific white shrimp Penaeus(Litopenaeus) vannamei[J]. Mole-cular Ecology Notes,2(3):239-241.
Li Y H,Guo X W,Cao X J,Deng W,Luo W,Wang W M. 2012. Population genetic structure and post-establishment dispersal patterns of the red swamp crayfish Procambarus clarkii in China[J]. PLoS One,7(7):e40652.
Schl?tterer C,Tautz D. 1992. Slippage synthesis of simple sequence DNA[J]. Nucleic Acids Research,20(2):211-215.
Yi S K,Li Y H,Shi L L,Zhang L,Li Q B,Chen J. 2018. Characterization of population genetic structure of red swamp crayfish,Procambarus clarkii,in China[J]. Scientific Reports,8(1):5586.
Zhu Z Y,Yue G H. 2008. Eleven polymorphic microsatellites isolated from red swamp crayfish,Procambarus clarkii[J]. Molecular Ecology Resources,8(4):796-798.

