大豆生育期E1、E2、E3和E4基因的研究与应用
吕世翔 王 萍 孙明明 夏正俊
(1.黑龙江省农业科学院 信息中心,哈尔滨 150086;2.中国科学院 东北地理与农业生态研究所/大豆分子设计育种重点实验室,哈尔滨 150081)
在特定生态环境下获得适应的成熟期是最大限度提高农作物产量的潜在前提条件[1]。作为一种典型短日作物,栽培大豆开花和成熟时间是其重要生态和农艺性状。不同品种的光周期敏感性使其具有不同开花和成熟时间,种植于长日照高纬度地区的大豆品种需要具有光不敏感性,以保证其在霜冻前成熟。因而大豆光周期敏感性遗传因素的研究对于大豆育种具有重要意义[2-3]。早在1927年,Owen[4]发现大豆中存在能够控制其生育期的遗传因子。1942年,王金陵[5]的《大豆之光期性》开启中国大豆光周期研究,拉开对大豆生育期遗传规律研究的序幕[6]。盖钧镒等[7]在前人关于中国大豆栽培区域划分的基础上,将中国大豆品种生态区划分为6大区,并在其中3个区内进一步分亚区。国内外学者相继对不同生育期大豆品种比对研究,结果表明大豆生育期由多个基因同时调控,目前发现的控制大豆开花和成熟的基因有E1[8]、E2[9]、E3[10]、E4[11]、E5[12]、E6[13]、E7[14]、E8[15]、E9[16]、E10[17]和J[18]基因,其中E1、E3、E4和E7基因是决定光周期敏感性的主要基因[19]。随着分子遗传学和分子生物学的发展,E1[20]、E2[21]、E3[22]、E4[23]、E9[16]、E10[17]和J[24]已经被定位或克隆。其中,E1~E4基因是研究最为深入、应用最为广泛的大豆生育期基因,研究表明E1~E4基因与大豆品种的生态类型密切相关,对大豆开花期变异贡献率高达62%~66%[23]。分子水平的研究显示E1~E4基因分别位于大豆基因组C2、O、L和I连锁群上[20-23]。
大豆生育期基因E1是对大豆开花期及成熟期影响最大的E系列基因,主要功能是抑制开花和延长生育期。E1基因是豆科作物特有的转录因子,在四季豆(Phaseolusvulgaris)及百脉根(Lotusjaponicus)等豆科作物中存在同源性高的基因序列,在水稻与拟南芥中没有相应同源序列[20, 25]。Zhang等[26]研究菜豆中E1同源基因Phvul.009G204600(PvE1L)和苜蓿中E1同源基因Medtr2g058520(MtE1L),证明PvE1L基因能够延迟大豆开花时间,而MtE1L基因对大豆的开花时间没有影响,但MtE1L基因能够促进苜蓿开花。推测豆科作物中E1基因家族在开花和植株生长调控作用中功能的保守性和变异性可能跟系谱和基因组复制有关。E1蛋白是一个具有转录抑制活性的转录因子,N端含一个双元核定位信号,C端含有与植物转录因子B3远缘相近的结构域[20]。E2基因与拟南芥GI(GIGANTEA)基因同源,对开花期的调控可能不受到光周期影响,而与生物钟节律相关,不同环境条件下E2基因对开花期的作用相对稳定[19]。E3和E4基因分别编码光敏色素A同源基因[22-23,27],其但对光质量的反应既重叠又互不相同[22, 25, 28]。E3基因对光质的差异并不敏感,导致种植在不同光质长日照条件下植株的开花期相近[29],E3与E4基因对E1基因的表达有调控作用[20]。
近年来各国学者广泛开展E1~E4基因不同变异类型与不同大豆品种生态适应性关系研究。但是对变异类型的总结、鉴定和应用前景方面鲜有报道,中国亟待形成适应我国地域特点的基于E1~E4基因变异类型的分子辅助育种模式。本研究就E1~E4基因的不同变异类型、变异类型鉴定方法及各国学者针对世界各地不同大豆品种E1~E4基因开展的相关研究进行综述,以期为大豆生育期遗传调控机理的全面深入研究提供参考,同时为适应不同生态区域大豆遗传育种工作提供依据,以期促进中国大豆生育期分子育种模式的形成。
1 大豆E1~E4基因的等位变异
1.1 E1基因类型变异
E1基因(Glyma06g23040.1)编码区全长864 bp,没有内含子。常见的E1基因变异类型包括e1-fs、e1-nl、e1-as和e1-b3a[20, 30]。e1-fs和e1-nl同为功能丧失型,均表现出极早的开花期和成熟期,e1-fs型实变体DNA序列49 bp处单碱基缺失导致移码突变;e1-nl为基因整体缺失。e1-as基因型为非同义突变,DNA序列44 bp处出现单碱基突变,使编码蛋白的定位发生变化,该基因型为早花型[20]。e1-as型开花及成熟期介于E1与功能丧失型(e1-fs及e1-nl)之间。e1-b3a型334~338 bp存在变异,在444 bp提前终止,蛋白质三级结构发生改变,定位于细胞核,该基因型也为早花型[29]。除此之外,e1-re基因型在e1基因起始位点上游148 bp处有1个3.1 kb 的LINE(long interspersed nuclear element)序列插入,该基因型的品种较基因型背景相近品种提前开花14 d[30]。e1-p基因型在E1基因5′侧翼序列存在差异[30]。
1.2 E2基因变异类型
E2基因(Glyma10g36600)编码区全长3 758 bp,包含14个外显子,编码1 170个氨基酸的蛋白。目前发现的E2基因变异类型有e2-ns、e2-in和e2-dl。e2-ns型最常见,编码区内4个单碱基位点变异致使编码序列提前终止。e2-ns基因型品种(Harosoy)较e2基因型品种(Clark)开花时间早[21]。e2-in和e2-dl型是E2基因的无义突变,分别为第8个内含子处36 bp插入和缺失,5′侧翼序列均存在序列变异[30]。
1.3 E3基因变异类型
E3基因(Glyma19g41210)编码区包含4个外显子,编码1 130个氨基酸的蛋白质。目前发现的E3基因的变异类型包括E3-Mi、e3-Ha、e3-T(e3-tr)、e3-Mo、e3-ns和e3-fs,其中e3-Mi、E3-Ha和e3-T型较常见。e3-Mi型编码正常光敏色素A,e3-Mo型第四内含子处插入1个2 633 bp反转录转座子且在第三外显子处碱基序列改变导致编码蛋白功能减弱,E3-Ha型包含1个与e3-Mo相似的反转录转座子,但其编码正常光敏色素A。e3-T型第三外显子之后出现13.33 kb缺失,使组氨酸结构域出现部分缺失,导致编码蛋白没有功能[22]。e3-ns和e3-fs型是E3基因的无义突变型。e3-ns型第三外显子单碱突变,e3-fs型第一外显子单碱基插入导致移码突变,序列提前终止[31]。
1.4 E4基因变异类型
E4基因(Glyma20g22160)编码区包含4个外显子,编码1 123个氨基酸的GmPhyA2蛋白质序列。目前发现的E4基因变异类型包括e4-SORE1、e4-kam、e4-kes、e4-oto和e4-tsu。e4(e4-SORE-1)型第一外显子存在6 238 bp的Ty1/copia-like反转座子插入,插入序列中的终止密码子使序列提前终止,产生功能缺失的蛋白序列。e4-kam和e4-kes型第二外显子单碱基缺失和插入导致编码蛋白的组氨酸激酶结构域缺失,而保留调控下游的PAS结构域。e4-oto和e4-tsu型分别为第一和第二外显子单碱基缺失,导致组氨酸激酶结构域和PAS2结构域均缺失[28, 32]。
2 大豆E1~E4基因变异类型的分子标记鉴定
E1~E4,4个基因等位变异型的分子标记鉴定方法主要包括CAPS、dCAPS、InDel和FLP,根据其基因序列和长度设计引物,采用合适的限制性内切酶对其加以区分,如表1。
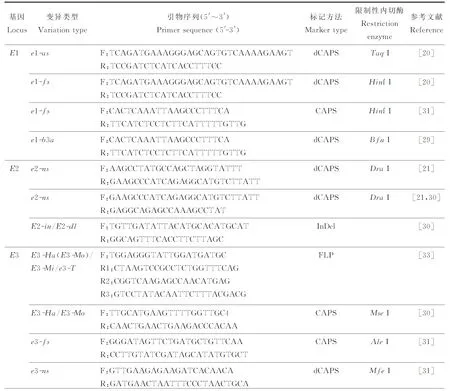
表1 E1~E4基因变异类型的鉴定Table 1 Identification of E1-E4 variation type

表1(续)
3 大豆E1~E4基因功能研究进展
3.1 E1~E4基因调控大豆开花时间
研究表明,大豆特异的调控开花的光周期通路PHYA(E3E4)-J-E1-FT,与拟南芥和水稻光周期反应的调控主通路CO-FT存在明显差异。E3和E4基因编码光反应通路必不可少的光敏色素,J基因编码蛋白能够结合在E1基因启动子上,调控E1基因的表达,E1基因编码豆科作物特有的和具有转录抑制功能的转录因子,负向调控大豆成花基因FT2a和FT5a的表达[20,24]。
E1~E4基因能够调控大豆开花时间,有功能E1基因的转录丰度与开花时间显著相关,基因表达量越高开花时间越晚,这一点在转基因大豆和普通大豆品种中均被证实[2, 20]。E2基因是拟南芥生物钟GI基因的同源基因,在长日照和短日照条件下均表现出节律性表达特性,纯和隐性基因型e2/e2通过诱导GmFT2a的表达使大豆开花提前,而在不同光照条件下E2基因对开花期的作用相对稳定[21]。E3和E4基因编码的光敏色素对不同光质的敏感性不同,不同纬度条件下,其对E1基因的表达量的影响随纬度变化而有所改变。E3和E4基因所编码的光敏色素蛋白对不同光质的敏感性不同,E3基因编码的GmPhyA3对光质的差异并不敏感,导致种植在不同纬度植株的开花期差异不大。而随着海拔的升高,R:FR逐渐变小,E4基因编码的GmPhyA2对光照的敏感性明显增强。隐性E3和E4基因同时存在才能使大豆品种表现出光照不敏感特征[10-11]。
3.2 E1~E4在大豆整个生长期的调控作用
E1基因正常表达能够延迟开花,但不能延长生殖期,E2和E3基因正常表达能够延长花期和开花后的时期,E4基因正常表达能够推迟开花和成熟[12]。不论是开花前还是开花后的光周期反应中,光敏色素E3和E4基因都是主要调控者,基因型为E3或E4品种的繁殖期和开花后的茎生长对光照变化很敏感,原因在于GmphyA2和GmphyA3能够调控生长习性Dt1基因表达,使茎尖分生组织的营养活动存在持续性[31]。隐性基因型e1、el-as、e2、e3和e4对于大豆品种在高寒地区的适应性具有重要意义,其可以不同程度地促进大豆开花和成熟,而且其隐性基因数量越多表现出的光周期不敏感性越强。研究表明绝大多数高寒地区大豆品种为e1/e2基因型[1,34]。对332个栽培大豆品种和85个野生大豆品种中e4-SORE-1基因型分布情况的研究结果表明,仅日本北部地区栽培大豆品种存在基因型为e4-SORE-1的品种,说明e4-SORE-1编码功能缺失的GmphyA2,导致大豆植株表现光不敏感特性,从而适应高纬度生存环境[30, 35]。
4 大豆E1~E4等位变异在生育期研究中的应用
目前世界上大豆种植范围已经超过50°N和40°S[36]。近年来各国学者广泛研究不同地理区域大豆品种的E1~E4基因型,并对不同基因型组合品种生态适应性展开了更为深入的分析。目前,中国、日本、俄罗斯、哈萨克斯坦、美洲和欧洲等地区关于大豆品种E1~E4基因型鉴定与分析工作已经开展多年。
4.1 中国大豆品种E1~E4等位变异与生育期关系研究
2014年Zhai等[3]鉴定180个具有代表性的中国大豆品种E1~E4基因型,在全国主要大豆生态区的6个试点(33°57′~45°70′N,117°17′~126°64′E)对大豆开花期与成熟期进行表型观察,结果表明E2基因的隐性基因型比率最高,高达84.45%。明确我国品种的基因型主要可分为8个类型,e1-nf、e1-asE4、E1-asE3E4、e1-asE2(E3)E4、E1、E1E4(E3)、E1E3E4和E1E2E3E4,其中E1E3E4为优势基因型,占26.1%。e1-nf型(E1功能缺失型)开花最早,而来自南部地区的E1E2E3E4基因型品种不能适应高纬度地区日照时间较长的环境,开花很晚或者在高纬度地区霜冻之前没有开花。研究发现不同品种中E1基因的表达量与大豆开花期呈极显著相关。进一步证明E1~E4尤其是E1显性基因对大豆光周期敏感性的抑制作用。e1-as具有一定抑制开花的功能,其对开花的抑制作用小于E1基因。
2017年Li等[37]研究299个中国大豆核心种质在中国4不同地区的成熟期,采用Sequenom MassARRAY iPLEX platform系统鉴定228个品种E1~E4基因型并分析其与开花期和成熟期的综合效应。结果表明E1/e2-ns/E3/E4和E1/E2/E3/E4是主要基因型,4个基因的隐性基因型不同程度促进早花,而其显性基因型在成熟期较长的品种中出现频率较高。研究结果表明中国核心种质资源在基因型和表型上存在较大遗传多样性,能够为中国大豆种植的生育区划分和地理适应性分析提供重要信息,同时为培育适应特殊气候环境的品种提供种质资源。
4.2 国外大豆品种E1~E4等位变异与生育期关系研究
国外大豆品种E1~E4基因型鉴定工作整体晚于中国,2014年Jiang等[35]检测85个大豆品种的E1~E4基因型,其中除46个品种为中国品种外,还包括来自南美MG 000~MGV III和MG X的13个生育期组的38个大豆品种,1个俄罗斯大豆品种(50°N),中国大豆品种中有4个为野生型大豆品种(23°N、39°N、46°N和50°N)。85个品种中共有10种基因型,其中E1/E2/E3/E4和E1/e2/E3/E4型是主要基因型。e1基因型大豆品种更适宜高纬度地区种植,e4等位变异对于大豆品种对高纬度地区的适应性具有重要意义,4个带有隐性e4等位基因的品种均适应高纬度地区,并表现出光周期不敏感性。带有等位基因e1、e1-as或e2的品种的出苗期~初花期天数变化范围较出苗期~成熟期和初花期~成熟期短。
2017年Kurasch等[38]研究75个欧洲大豆品种E1~E4基因型与成熟期相关性状。首次发现有两个品种是杂合基因型品种,Ruzica品种的E3基因为杂合位点,基因型是e3-tr/e3-fs,Carla_TD品种的E4基因为杂合位点,基因型是E4/e4-SORE-1。没有检测出E1基因型的品种,在MG 000 和MG 00生育区检测出4个e1-nl基因型品种,每个生育区中均有e1-as基因型品种。e1-nl基因型品种的开花和成熟时间最早,在所有22个种植地点都能成熟。与e1-as基因型品种相比,e1-nl基因型品种营养生长期在整个生长期的占比较长。该研究结果说明大豆光周期作用和E系列基因研究对欧洲栽培大豆品种生态适应性研究的重要意义。2017年Abugalieva等[39]鉴定了来自哈萨克斯坦农业科学院的120个品种(系)E1~E4基因型,这些品种是该地区育种工作的主要品种来源,研究材料来自西欧、东欧、北美、东亚和哈萨克斯坦5个地区12个国家。该研究共检测到17个不同的基因型组合,5个主要组合是e1-as/e2/E3/E4、e1-as/e2/e3/E4、e1-as/e2/e3/e4、e1-nl/e2/E3/E4和e1-nl/e2/E3/e4。该研究分析了产量表型与基因型的关系,e1-as/e2/E3/E4基因型的品种(系)数量最多,但在3个地区都不是产量最高的基因型,具有显性等位基因E3或E4基因的品种在这3个地区的产量均较高。5个主要组合的E2基因全部为隐性基因型。研究表明东南和东部地区形成一个整体环境,与需要种植成熟期较早的大豆品种的北部地区明显区分开来,这些研究结果将为哈萨克斯坦大豆种植区的基因和表型模式关系的研究与应用提供参考。
2017年Langewisch等[1]鉴定238个大豆品种(系)E1/e1-as、E2/e2和E3-Ha/E3-Mi/e3-tr基因型,238个品种中127个为美国地方品种,48个为中国栽培大豆品种,17个为北美祖先品种,46个为北美栽培大豆品种,其中,40%品种采用分子标记方法鉴定,对60%品种进行重测序,利用Zhou等[40]的302个大豆品种的重测序SNP数据库分析其基因型。结果表明,美国和加拿大现代大豆育种工作以有限亲本材料为基础,大部分亲本材料来自中国,本土品种E1~E3基因等位变异类型存在局限性。美国MG 0~MG IV生育区错误的全部使用了基因型为e1-as的大豆品种,美国至少2 400万hm2大豆产量受此限制。该研究形成基于大豆主要成熟期位点E1、E2和E3的美国大豆生育区分子育种模式,该模式能够使育种者更高效的培育出适应不同生态区、具有特定农艺性状的大豆品种。该方法也可以用于优化基因预测及筛选方案,从而整体提高美国和加拿大的大豆品种繁育效率。
不同大豆种植区的研究结果进一步说明尽管4个E基因不同变异的影响程度存在差异,但其不同基因型组合显著地决定品种的生态适应性,4个E位点等位基因组合和相应分子标记技术能够促进大豆分子繁育从而提高大豆产量。
5 展 望
E1变异基因型e1-b3a编码蛋白具正常核定位信号,但B3结构域发生变异。e1-b3a在高纬度哈尔滨地区种植的开花期较E1基因型提前30 d,在淮安地区约提前10 d,而e1-b3a蛋白与E1蛋白的亚细胞定位结果一致,而与e1-as不同,证明B3结构域对E1基因生物学功能具有重要作用。Tsubokura等[28]发现少数品种为e1-re、e1-p、E2-in和E2-dl变异类型。对这些变异类型的深入研究将有助于对E1和E2基因功能的深入揭示。
E1~E4对开花的综合贡献估计高达62%~66%,还有目前尚不为人所知的基因控制[28]。已知基因(如E3、E4和J)和其他未知因素,至少可以通过调控E1基因表达来发挥作用[2, 24]。大豆中很多农艺性状相关QTLs已经被定位但没有克隆出来,相关基因的克隆将有助于对大豆生育期基因调控网络特点的进一步认识[41],最终实现对大豆生育期调控网络的深入揭示。
重测序和Sequenom MassARRAY iPLEX platform系统等[37,40]更多新型、快速的鉴定方法将有助提高E1~E4基因型的鉴定效率,从而带来高效率群体鉴定与分析的可能,包括E1~E4基因型鉴定在内的更多大豆农艺性状相关基因的研究和鉴定将促进分子遗传学研究结果应用到大豆育种工作中。

