一种共生藻PCR模板DNA的快速制备法
龙 寒,禤金彩
(广西民族大学海洋与生物技术学院、广西海洋微生物资源产业化工程技术研究中心、广西多糖材料与改性重点实验室,广西 南宁530008)
共生藻(Symbiodinium)是共生于海洋无脊椎动物体内的单细胞藻类,依靠光合作用为宿主提供氨基酸、糖等生长必需的化合物[1]。异养营养尽管能为宿主供给部分代谢能量[2],但由于其自然来源的不稳定性等特征,只能作为宿主的次要能量来源。因此,在含藻共生体生活史上,共生藻具有不可替代的重要性。
目前国内外对共生藻的研究主要包括共生藻的分类鉴定[3]、共生藻的逆境生理[4]、共生藻与宿主的共生机制[5]等,其中利用基因组学和转录组学展开的基因表达调控机制是共生藻研究的前沿和热点,而作为PCR模板的DNA制备是对共生藻开展分子水平研究的基础。
对于共生藻DNA的提取,既有采用先与宿主分离再进行DNA提取的方法[6],也有采用与宿主混合采样的提取方法[7]。在实际操作中,许多共生体(如珊瑚)由于其产生大量粘多糖,共生藻往往难于分离,李秀保等(2009)提出用冲洗法对活体珊瑚共生藻进行分离[8];张露(2017)比较了冲洗法、液氮研磨法和酒精研磨法对共生藻的分离效果,发现酒精研磨法分离效率高、分离质量好[9];张跃环等(2018)提出了用毛细管法获得砗磲单克隆共生藻[10]。在共生藻DNA提取方面,目前常用的方法有TRIZOL提取法、CTAB提取法、改良SDS提取法等。SDS对于多糖的去除能力较差,提取效果不佳;CTAB法操作繁琐、耗时长、试剂具有毒性;TRIZOL抽提法尽管样品消耗量小操作较为简单,但苯酚、异硫氰酸胍等试剂毒性强,对人体及环境危害性大。
本研究采用Chelex-100树脂法,制备可应用于PCR的模板DNA,旨在探索一种便捷、高效、安全的共生藻DNA提取法。Chelex-100 是由苯乙烯、二乙烯苯共聚体组成的化学螯合树脂,含有成对的亚氨基二乙酸盐离子,可螯合多价离子,对高价金属离子有很高的亲和力和螯合作用。在低离子强度、碱性及煮沸的条件下,Chelex-100可以使细胞膜破裂、蛋白质变性,能有效去除非核酸有机物,保留DNA。Chelex-100树脂法由于其便捷性被广泛应用于法学鉴定如血液、血痕、组织和骨骼等DNA的提取中[11-13],在真菌[14]、细菌[15]、放线菌[16]等原核生物以及高等植物[17-18]DNA提取中也有使用,但目前未见用于藻类DNA提取的报道。
本研究首次将Chelex-100应用于藻类DNA提取,成功制备了共生状态及纯培养状态下的27份共生藻样品DNA,并通过叶绿体23S rDNA基因片段扩增验证了提取效果(叶绿体23S rDNA含有演化速度很高的V区域,在共生藻种属水平分类上是十分有效的研究工具[19]),以期为共生藻分子分类、功能基因研究等分子水平研究工作的效率提升提供一定的技术参考。
1 材料与方法
1.1 材料
1.1.1 供试生物 供试生物样品及其编号见表1, 纯培养共生藻GY-H50分离自东海舟山海域。

表1 供试生物样品及其编号Tab.1 Biological samples for experiment and numeration
1.1.2 试剂 Chelex-100的制备:将Chelex-100胶体颗粒浸泡于去离子水中,在121℃下灭菌15 min用于DNA提取;23S-M引物由北京奥科鼎盛公司合成提供,序列为23S-M/F(5’-CACGACGTTGTAAAACGACGGCTGTAACTATAACGGTCC-3’),23S-M/R(5’-GGATAACAATTTCACACAGGCCATCGTATTGAACCCAGC-3’)[19];新设计引物23S-N由上海生工合成提供,序列为23S-N/F(5’-CCTGCATGAAACATAGAACGATT-3’),23S-N/R(5’-CAGGCCATCGTATTGAACCC-3’);2×Taq PCR Master Mix、DL2000 DNA marker购自康为世纪。
1.1.3 培养基 共生藻GY-H50采用f/2海水培养基进行培养[20]。
f/2海水培养基(1 000 cm3):NaNO375 mg,NaH2PO4·H2O 5 mg,维生素B10.1 mg,维生素B120.000 5 mg,生物素 0.000 5 mg,Na2SiO3·9H2O 30 mg,微量元素母液 1.0 cm3。
微量元素母液(1 000 cm3):ZnSO4·7H2O 22 mg,EDTANa24.36 g,Na2MoO4·2H2O 6 mg,CuSO4·5H2O 10 mg,CoCl2·6H2O 10 mg,MnCl2·4H2O 180 mg,FeCl3·6H2O 3.15 g。
1.2 方法
1.2.1 实验取材 具有伸展组织的生物体在其自然伸展状态下迅速剪取小块组织约2 mm3(如葵珊瑚的触手、砗磲的外套膜等);脑珊瑚、海绵用眼科剪连骨剪取约3~4 mm3的组织。剪取的组织放入装有0.5 cm3无菌海水的EP管内,轻轻震荡后,将组织转移至另一只装有无菌海水的管内,如此进行3~5次清洗,最后将组织转移至装有0.2 cm3无菌蒸馏水的EP管内,以灭菌解剖针对其进行捣碎,捣碎时,可见管内液体变黄。吸取黄色样品液0.1 cm3放入PCR管内,再加入 Chelex-100胶体颗粒25 mm3。
在生物体(砗磲、海葵、葵珊瑚等)自然伸展状态下迅速剪取材料,可见生物体仍然保持伸展状态,几乎不受取样影响,而所有的生物体在取样后均能正常生长,可见,此取样方法对生物体基本不构成伤害,能确保生物体继续存活并可支持更多次实验。
取共生藻GY-H50的纯培养藻液1 cm3,于6 000 r/min下离心10 min收集藻体,沉淀用0.1 cm3无菌水重悬后转移至PCR管,加入Chelex-100胶体颗粒25 mm3。
1.2.2 DNA提取 样品经沸水浴(99~100℃)15 min后,6 000 r/min离心3 min,上清液即为DNA提取液。
1.2.3 PCR扩增及效果检验 PCR扩增反应25 mm3体系包括: 2×Taq PCR Master Mix 12.5 mm3、上游引物0.5 mm3、下游引物0.5 mm3、DNA提取液1 mm3、双蒸水10.5 mm3。
PCR程序为:95℃预变性7 min;95℃变性0.5 min,55℃退火1.5 min,74℃延伸1 min 30 s,循环30次;74℃延伸10 min。
PCR产物初步检验:采用2%琼脂糖凝胶进行水平电泳。电泳在120 V电压下进行20 min左右。以DL2000 为 marker,marker与样品上样量均为2 mm3。电泳结束后将凝胶置于0.5 ug/cm3EB染液中浸泡30 min显色,再于凝胶成像仪内260 nm紫外光下进行拍照。
PCR产物测序:将电泳检验中有条带的扩增样品送至广州天一辉远基因科技有限公司进行测序,所得序列在NCBI中进行Nucleotide BLAST查询比对。
1.2.4 引物设计 用序列比对软件AlignX分析最初获得的22条23S rDNA序列,判断序列的保守区。选择一条序列作为模板,载入引物设计软件Oligo7,根据保守区选择上游引物位置并设定预期扩增片段长度为650 bp,Search完成引物设计。设计完成的引物通过Analyse进行评价,确定序列。
2 结果与讨论
2.1 琼脂糖凝胶电泳结果
经过Chelex-100树脂热裂解后制备所得的27份DNA样品,取1 mm3为模板,以23S-M为引物进行PCR扩增,扩增产物经琼脂糖凝胶水平电泳检验,结果如图1所示,得到了22条清晰条带的电泳图,其中4、8、11、12、20号样品为双条带,判断原因为引物特异性不足造成;3、5、10、15、19、21号生物样品未扩增出条带。所有获得条带除23号样品(750 bp)外,大小基本一致,在700 bp左右;marker及样品上样量均为2 mm3,从亮度对比来看,所获得的扩增产物具有较高的浓度,符合测序要求。
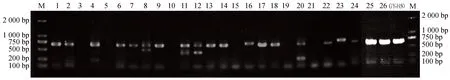
图1 供试生物样品共生藻及GY-H50 DNA提取物的23S-M PCR扩增结果Fig.1 23S-M PCR products by DNA extracts from experimental biological samples’ symbiodiniums and GY-H50M:DL2000 DNA marker;1~26、GY-H50:1~26号样品共生藻及GY-H50 DNA提取物的23S-M PCR扩增产物;其中,1~24对应左侧marker,25、26、GY-H50对应右侧marker。
针对未扩增出条带的6份样品,利用测序返回的22份序列,按照“1.2.4”的方法进行引物设计,得到新引物23S-N。PCR所使用的模板DNA仍然采用“1.2.2”制备的DNA提取液(-20℃保存),无需重新制备。扩增产物经琼脂糖凝胶水平电泳检验,结果如图2所示,除3号样品外,其余均扩增出了700 bp左右的条带,条带明亮、清晰,符合测序要求。3号样品未扩增成功的原因,可能是所设计的引物不匹配。
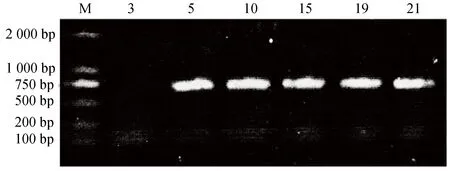
图2 部分供试生物样品共生藻DNA提取物的23S-N PCR扩增结果Fig.2 23S-N PCR products by DNA extracts from part experimental biological samples’ symbiodiniumsM:DL2000 DNA marker;3、5、10、15、19、21: 3、5、10、15、19、21号样品共生藻DNA提取物的23S-N PCR扩增产物。
2.2 扩增序列测序结果
扩增产物针对700 bp片段进行测序,共获得26个基因序列,将序列依次在NCBI 中进行Nucleotide BLAST查询比对,选择分值最高项且E值最小项编入表2(E值是分值score的可靠性评价,即随机情况下,其他序列与目标序列)。从表2可以看到,所有比对结果与扩增的目的基因一致,均为叶绿体23S rDNA,覆盖率最低为95%,一致性最低为90%,说明比对结果可信度高,序列扩增正确。该结果说明由Chelex-100树脂热裂解制备的模板DNA具有PCR可操作性。
从表2统计结果中可看到:在被测27份共生藻中,A类群共生藻仅见于帘形目即番红砗磲(Tridacnacrocea),B类群共生藻跨类珊瑚目和角海葵目两目,存在于气泡菇珊瑚(Ricordeaflorida)及角海葵(Cerianthussp.)中,C类群共生藻共生范围跨度最大,存在于石珊瑚目、软珊瑚目、海葵目、群体海葵目及韧海绵目中,东海舟山海域分离培养的共生藻GY-H50为E类群共生藻,为共生藻属中营自由生活的种类。本次实验为确保采样种类的多样性,使用了3门3纲9目20科23属26个物种作为生物样品。测序结果发现C类群共生藻是本次模拟调查范围中的优势种,该结果与Huang等(2006)对造礁石珊瑚共生藻遗传多样性调查结果一致[21]。实验结果还可为后续共生藻分子系统发育树的构建、共生藻的系统发生关系分析以及共生关系生态学研究提供参考数据。
表2 供试生物样品共生藻及GY-H50 的PCR扩增序列比对结果
Tab.2 BLAST results of PCR amplified sequences from experimental biological samples’ symbiodiniums and GY-H50
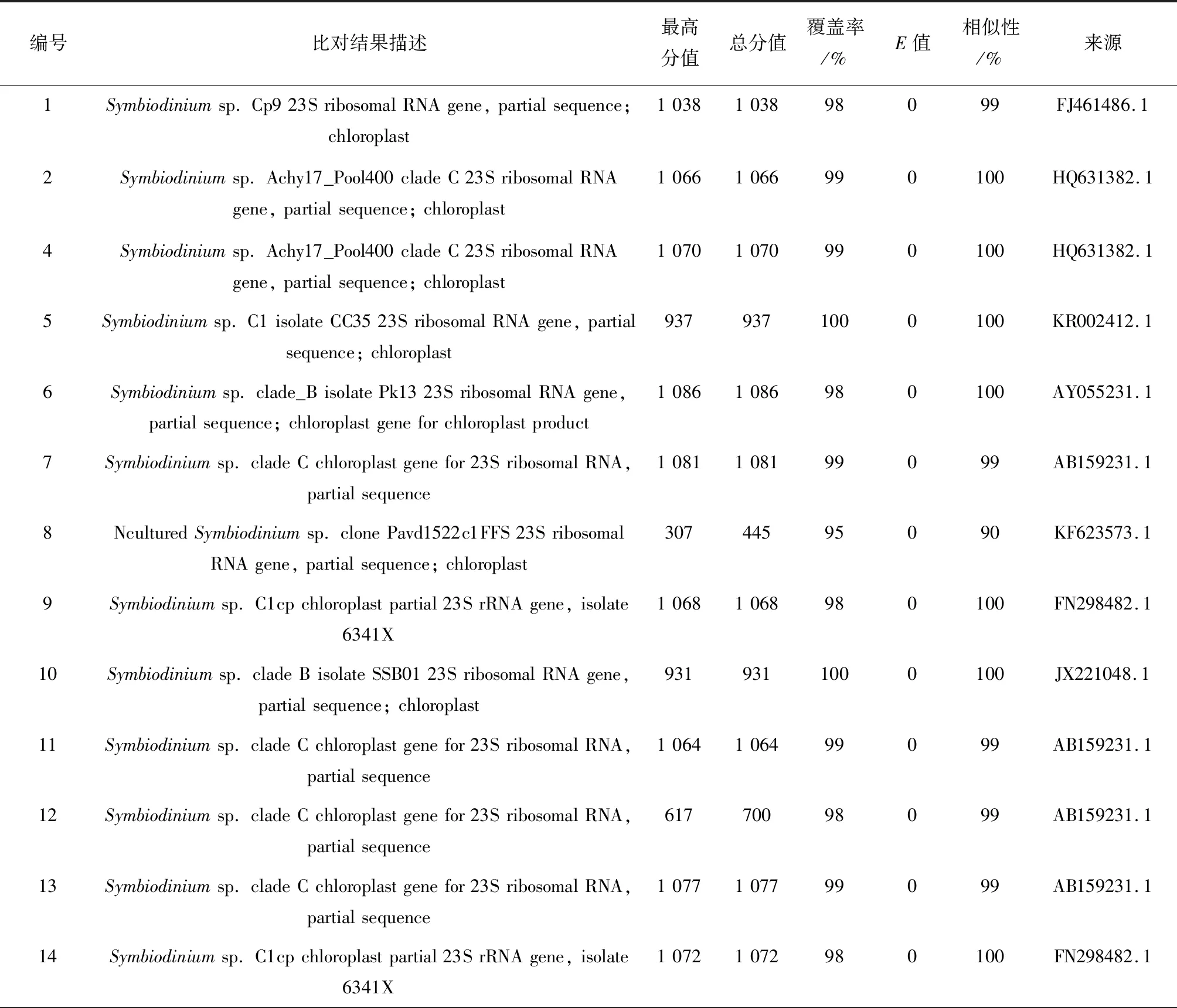
编号比对结果描述最高分值总分值覆盖率/%E值相似性/%来源1Symbiodiniumsp.Cp923SribosomalRNAgene,partialsequence;chloroplast1038103898099FJ461486.12Symbiodiniumsp.Achy17_Pool400cladeC23SribosomalRNAgene,partialsequence;chloroplast10661066990100HQ631382.14Symbiodiniumsp.Achy17_Pool400cladeC23SribosomalRNAgene,partialsequence;chloroplast10701070990100HQ631382.15Symbiodiniumsp.C1isolateCC3523SribosomalRNAgene,partialsequence;chloroplast9379371000100KR002412.16Symbiodiniumsp.clade_BisolatePk1323SribosomalRNAgene,partialsequence;chloroplastgeneforchloroplastproduct10861086980100AY055231.17Symbiodiniumsp.cladeCchloroplastgenefor23SribosomalRNA,partialsequence1081108199099AB159231.18NculturedSymbiodiniumsp.clonePavd1522c1FFS23SribosomalRNAgene,partialsequence;chloroplast30744595090KF623573.19Symbiodiniumsp.C1cpchloroplastpartial23SrRNAgene,isolate6341X10681068980100FN298482.110Symbiodiniumsp.cladeBisolateSSB0123SribosomalRNAgene,partialsequence;chloroplast9319311000100JX221048.111Symbiodiniumsp.cladeCchloroplastgenefor23SribosomalRNA,partialsequence1064106499099AB159231.112Symbiodiniumsp.cladeCchloroplastgenefor23SribosomalRNA,partialsequence61770098099AB159231.113Symbiodiniumsp.cladeCchloroplastgenefor23SribosomalRNA,partialsequence1077107799099AB159231.114Symbiodiniumsp.C1cpchloroplastpartial23SrRNAgene,isolate6341X10721072980100FN298482.1
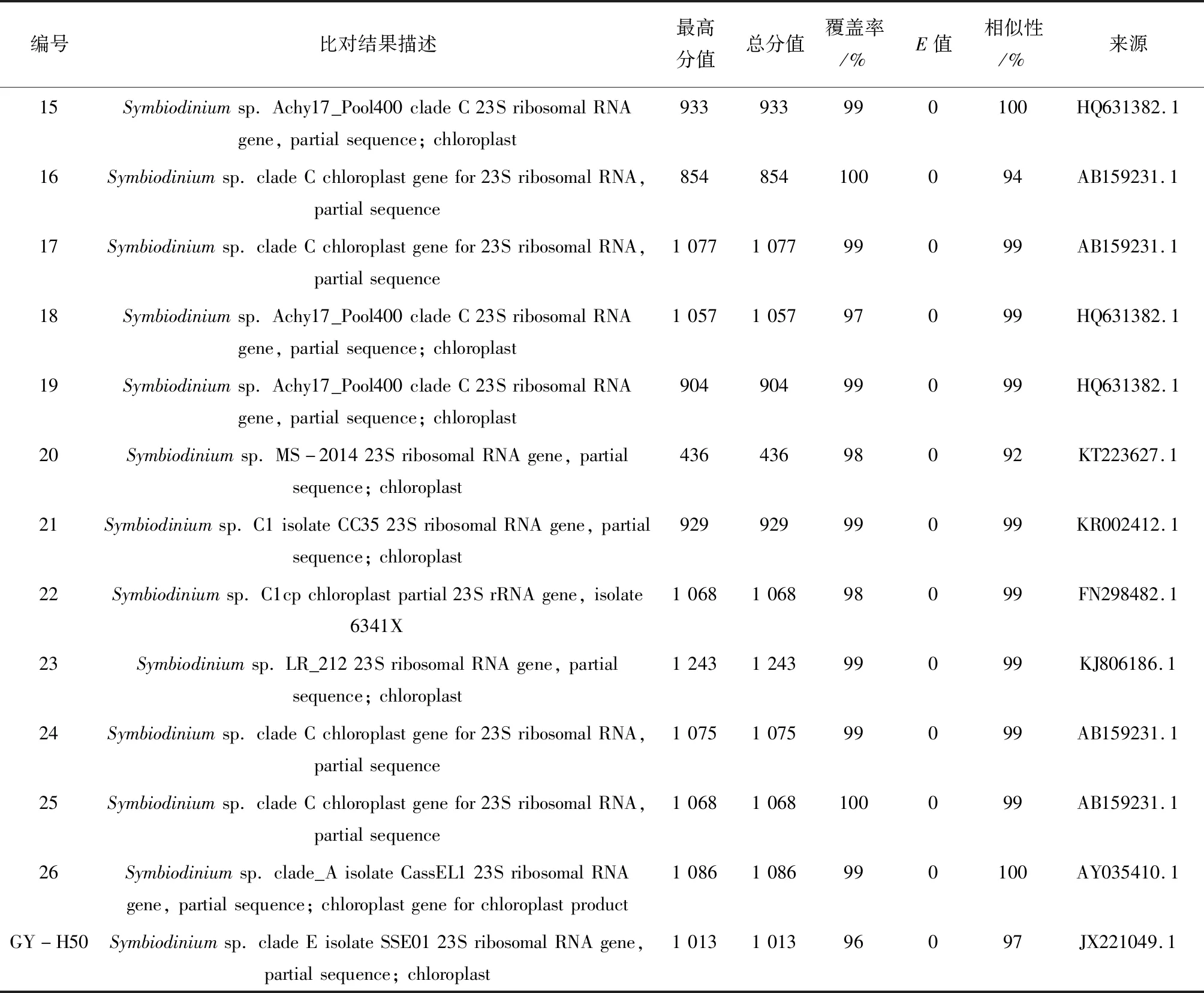
续表2
注:3号供试生物样品共生藻未扩出。
2.3 讨论
对于Chelex-100树脂法,戴文申等(2006)利用该法提取痕量DNA的研究中指出,56℃保温15~30 min、100℃保温15 min左右(不超过30 min)有利于细胞核的裂解,促进DNA的释放[22],但周双清等(2010)在对放线菌进行Chelex-100树脂热裂解时仅通过沸水浴10 min即获得了良好的PCR模板DNA[23]。共生藻在共生生物体内含量可高达3.95×106cells/cm2[24],即使是少量的材料(如本次实验采样量),在稍加搅拌或吹打的情况下,即可见棕黄色的藻液被释放出来,可见细胞含量充足,不属于痕量DNA提取,因此可以缩短实验步骤,不进行56℃保温;而与原核生物相比,藻类DNA提取又存在细胞壁破碎这一屏障,因此实验采用了Chelex-100树脂热裂解15 min提取的共生藻DNA样品。扩增产物电泳条带明亮清晰、能够测出有效序列,说明Chelex-100树脂法能够快速实现腔肠动物、海绵动物、软体动物的共生藻-宿主混合样品或者纯培养共生藻样品的DNA提取,且制备的模板DNA能够满足PCR扩增需求。
值得注意的是,许多含有共生藻的生物都属于珍稀物种[如石珊瑚、库氏砗磲(Tridacnagigas)等],进行科研实验时,在确保完成科研工作的基础上探索更合理的可持续实验模式是每一位科研工作者都应当充分考虑的问题,具体做法如尽可能减少样品的消耗量、采用不危及生物体生命的采样方式、采用人工养殖代替自然采集(如果是生态调查,应当在满足调查的同时尽可能减少取样量)、生态调查中所取的样品合理地放归原环境等[25]。
3 结论
采用Chelex-100进行了3门3纲9目20科23属26个物种的共生藻及1株纯培养共生藻的PCR模板DNA快速制备,并通过PCR扩增23S rDNA基因片段进行制备效果检验,结果表明,制备过程通过Chelex-100树脂热裂解15 min,即能为26份样品提供合格的PCR扩增DNA模板(有1份样品未能扩增成功判断原因为引物不匹配)。实验结果说明Chelex-100树脂法能够用于多种状态及类型共生藻的PCR模板DNA制备,且具有操作简单、耗时短、无污染、无设备要求、样品消耗量小等优点。

