基于GSA的厌氧发酵原料碳氮比NIRS快速检测
刘金明 程秋爽 甄 峰 许永花 李文哲,5 孙 勇,5
(1.东北农业大学工程学院, 哈尔滨 150030; 2.黑龙江八一农垦大学电气与信息学院, 大庆 163319;3.中国科学院广州能源研究所中国科学院可再生能源重点实验室, 广州 510640;4.东北农业大学电气与信息学院, 哈尔滨 150030;5.黑龙江省寒地农业可再生资源利用技术及装备重点实验室, 哈尔滨 150030)
0 引言
碳氮比(碳、氮元素质量的比值)对厌氧发酵微生物的生长繁殖和产物合成具有重要影响,测定厌氧发酵底物的碳氮比已成为优化厌氧发酵原料配比的重要环节[1-2]。在以玉米秸秆为主要原料进行厌氧发酵生产沼气时,通过预处理打破玉米秸秆自身紧密的木质纤维素结构,能够有效提高玉米秸秆的生物转化利用率[3-4]。同时,由于玉米秸秆碳氮比过高,常与碳氮比较低的畜禽粪便混合共同发酵,以提高厌氧发酵产沼气的效率和能力[5-6]。为了分析碳氮比对厌氧发酵的影响,进而对预处理后玉米秸秆及秸秆和粪便混合物的厌氧发酵过程进行有效调控,有必要对发酵原料的碳氮比进行快速、准确的测定,采用传统化学方法测定其碳氮比时存在测试速度慢、成本高的问题。
近红外光谱(Near infrared spectroscopy, NIRS)分析技术具有快速、低成本及多组分同步检测等优点[7-8],已广泛用于有机动植物废弃物碳氮含量的快速测定[9-10]。当使用NIRS对畜禽粪便中的碳、氮含量进行检测时,氮含量的检测精度明显优于碳含量[11-12]。在使用NIRS对植物中所含碳、氮成分进行检测时,能够获得较高的氮含量检测精度[13-14],但碳含量检测精度较低[15-16]。针对厌氧发酵过程中原料碳氮比的快速检测需求,以及NIRS在秸秆和粪便碳氮含量检测方面的优势与不足,本文提出使用NIRS对厌氧发酵原料直接进行碳氮比的快速检测。
针对以全谱建模时冗余波长点严重影响模型检测精度和效率的问题,相关学者提出应用遗传算法(Genetic algorithm,GA)[17-18]进行NIRS波长变量组合优化。GA因其具有较强的鲁棒性和全局搜索能力,在NIRS特征波长优选方面得到了广泛应用,其随机搜索能力能够有效解决光谱波长点之间的共线性问题[19],还可以与其他光谱谱区优化算法相结合进行特征波长点的优选[20-21]。但GA存在早熟问题,且进化后期搜索效率较低。遗传模拟退火算法(Genetic simulated annealing algorithm, GSA)[22]通过将GA与模拟退火算法(Simulated annealing algorithm, SA)相结合,引入温度参数对适应度函数进行改进设计,在充分发挥算法强大搜索能力的同时,有效解决了GA存在的不足。
本文提出基于GSA构建遗传模拟退火区间偏最小二乘算法(Genetic simulated annealing interval partial least squares algorithm, GSA-iPLS)和双重遗传模拟退火偏最小二乘算法(Double genetic simulated annealing partial least squares algorithm, DGSA-PLS)分别用于特征谱区优选和特征波长点优选,进而获取与碳氮比相关性高的有效特征波长变量,建立厌氧发酵原料碳氮比的NIRS快速检测模型。
1 材料与方法
1.1 样品采集与制备
实验用玉米秸秆取自东北农业大学校内实验田,猪粪取自哈尔滨市三元畜产实业公司,牛粪取自哈尔滨市宇峰奶牛养殖农民专业合作社,羊粪取自东北农业大学阿城实验实习基地。玉米秸秆、猪粪、牛粪和羊粪各采集1个样品,用于后续秸秆预处理和秸秆粪便混合样品制备。采集的玉米秸秆样品自然风干后装袋保存,采集的猪粪、牛粪和羊粪样品于-18℃冷冻保存。在厌氧发酵样品制备过程中,先将风干玉米秸秆切成10 mm长的秸秆段,再将秸秆段、猪粪、牛粪、羊粪干燥、粉碎,过40目筛后装袋备用。依据秸秆厌氧发酵过程中碱性预处理的高效性和生物预处理的环境友好性,分别采用地衣芽孢杆菌(生物方法)、NaOH溶液(碱性试剂)、猪粪沼液(富含微生物的弱碱性试剂)和NaOH沼液溶液(富含微生物的强碱性试剂)对玉米秸秆进行预处理实验。生物方法预处理秸秆样品取自本课题组进行的地衣芽孢杆菌秸秆降解实验[23]。按最优培养条件活化培养地衣芽孢杆菌后,将其液体菌种接种于秸秆粉末固体培养基上,进行为期10 d的降解实验,每2 d采样1次,共计采样5个。其它方法预处理实验过程中,将10 mm长的秸秆段浸泡于处理液中3 s后,捞出挤压排水并装入自封袋进行密封处理。定时采样并用蒸馏水充分洗涤样品5次后,将样品干燥、粉碎过40目筛后装袋密封保存,制备预处理秸秆样品45个。NaOH、沼液预处理秸秆实验方案如表1所示。
将粉碎玉米秸秆按比例与粉碎后的猪粪、牛粪、羊粪粉末进行混合,制备秸秆和粪便混合发酵原料样品36个。混合比例(质量比)为9∶1、8∶2、7∶3、6∶4、5∶5、4∶6、3∶7、2∶8、1∶9和3个随机比例。连同玉米秸秆、猪粪、羊粪、牛粪样品4个,共计采集与制备样品90个。
1.2 碳氮比的测定
样品碳、氮含量的测定按照干烧法的原理[24],采用EURO EA3000型元素分析仪测定,测试模式为碳氢氮模式,测试温度为980℃,运行时间320 s,样品杯为5 mm×9 mm锡囊,装样质量1~3 mg,氦气为载气,反应管型号为E13041。标样为琥乙红霉素标准品(C43H75NO16),其碳、氢、氮、氧质量分数分别为59.911%、8.769%、1.625%、29.695%,与玉米秸秆碳氮比接近,适于测试玉米秸秆碳氢氮含量。每个样品测试3次,取3次的平均值作为待测样品的碳、氮含量值,然后通过计算可得样品的碳氮比。
1.3 光谱数据采集
对发酵原料样品使用Bruker TANGO型近红外光谱仪进行积分球漫反射光谱扫描,光谱采集范围3 946~11 542 cm-1(波长866~2 534 nm),分辨率为8 cm-1,样品扫描32次,装样方式为50 mm样品杯旋转台扫描,装样质量约7 g,将所测吸光度进行保存。在光谱扫描时,使用镀金样品杯上盖压实样品粉末,并采用3次扫描的平均值作为样品的原始光谱。每个样品原始光谱的波长点数为1 845个。
1.4 波长优选方法
1.4.1GSA算法
GSA算法[25]融合了GA[26]的高效遗传操作和SA[27]的退温策略,采用二进制编码方案,以偏最小二乘(Partial least squares,PLS)回归模型的交叉验证均方根误差(Root mean squared error of cross-validation,RMSECV)为目标函数,通过结合温度参数设计适应度函数,基于Metropolis判别准则实现扰动解的选择复制,有效解决了GA算法存在的早熟收敛和进化后期搜索效率低的两点不足,又克服了SA算法进化速度慢的问题。
1.4.2GSA-iPLS算法
针对GSA以全谱波长点个数为码长进行二进制编码时,码长过长容易导致解空间发散的问题,将GSA与区间偏最小二乘法(Interval PLS,iPLS)[28]相结合构建GSA-iPLS算法,通过选取多个有效的光谱子区间参与建模,能够有效提高模型的性能。
GSA-iPLS基于iPLS的思想,将NIRS划分为n个等宽子区间,然后使用GSA优选出有效的特征谱区建模,以提高模型精度。GSA-iPLS采用二进制编码方式,以子区间个数为码长,进行GSA的种群初始化。“1”和“0”分别表示对应子区间所包含波长点对应的数据“是”、“否”选中参与运算。根据种群初始化结果计算各染色体的目标函数值,确定初始温度和降温操作,并计算各染色体的适应度函数值。然后依据适应度函数值对种群中的染色体依次执行带最优保留策略的赌轮选择、离散重组交叉、离散变异和Metropolis选择复制操作,完成一轮次的GSA种群进化过程。经过多个轮次的种群进化,满足设定的算法终止条件后,即完成NIRS特征谱区优选。按如上方法,执行多次特征谱区优选算法,求出不同子区间个数下碳氮比对应的多个备选特征子区间组合,通过综合评测模型性能后,基于RMSECV确定发酵原料碳氮比对应的最佳子区间个数和最佳特征谱区。
1.4.3DGSA-PLS算法
GSA-iPLS优选的光谱子区间内部仍然可能存在不相关的波长点和波长点之间的共线性问题。DGSA-PLS算法利用GSA对GSA-iPLS优选的光谱子区间进行二次优化,能够有效去除谱区内的不相关波长点,解决波长点之间的共线性问题。
DGSA-PLS以GSA-iPLS优选后特征谱区包含的特征波长点数为码长,进行二进制编码和种群初始化。“1”和“0”分别表示该波长点对应的数据“是”、“否”选中参与运算。在确定初始温度、降温操作,计算适应度函数值后,执行多个轮次的GSA选择、交叉、变异和Metropolis选择复制进化操作,完成NIRS特征波长点的优选。针对GSA优化结果的随机性问题,多次执行特征波长点优选算法,并选择多次重复选中的波长点为特征波长变量建立PLS回归模型,能够得到较高的回归模型性能。
1.5 回归模型建立及评价
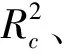
本文算法(包括光谱预处理、样品集划分、GSA-iPLS算法、DGSA-PLS算法及回归模型构建等)全部在Matlab R2012b软件平台中实现。
2 结果与分析
2.1 采集数据分析

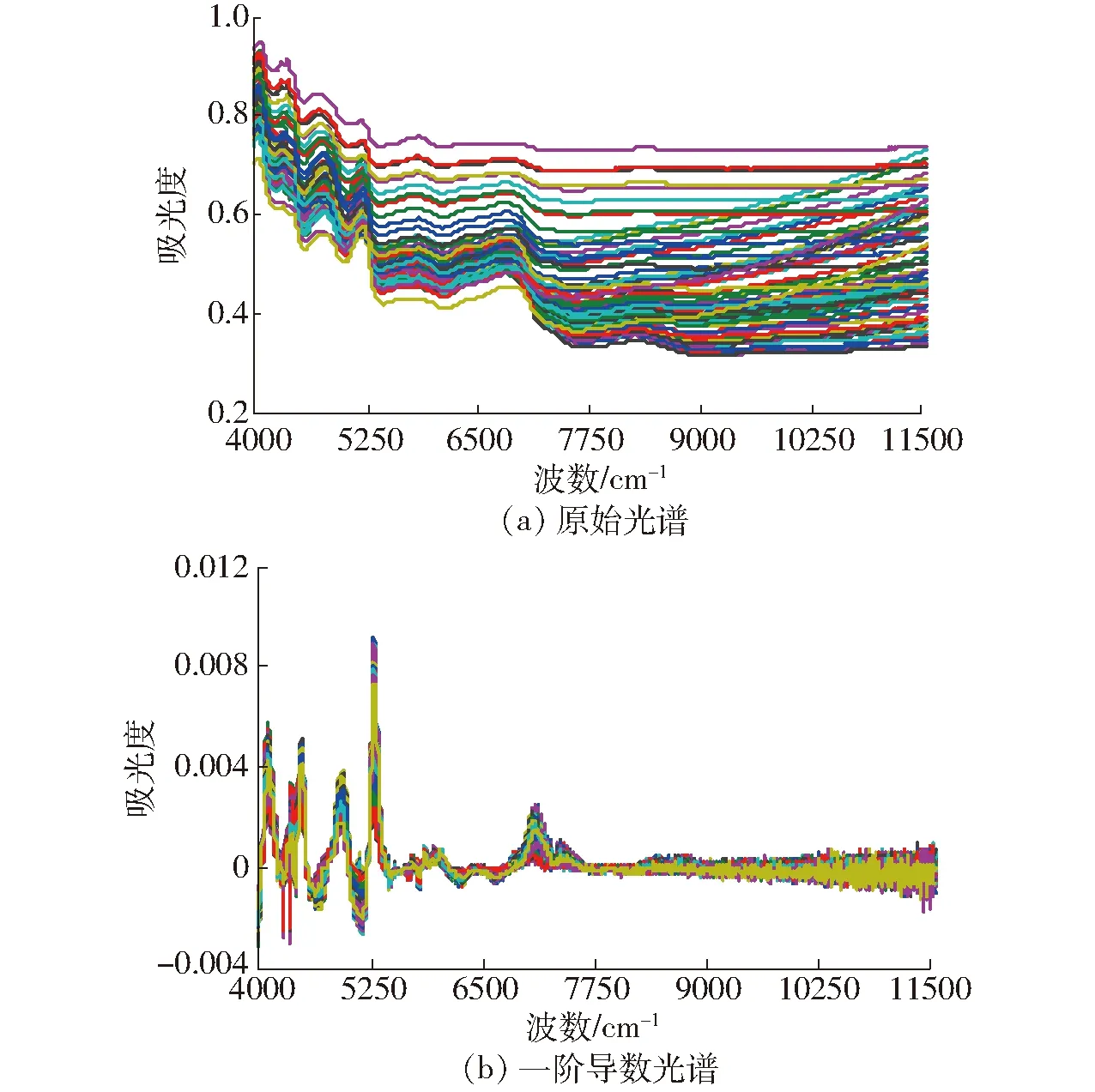
图1 样品光谱数据Fig.1 Spectroscopic data of samples
对90个样品的原始光谱经一阶导数预处理后,使用KS法按2∶1的比例进行样本划分,得到校正集样本60个、验证集样本30个,对应的碳氮比如表2所示。

表2 厌氧发酵原料碳氮比Tab.2 C/N ratio of anaerobic fermentation materials
对预处理后的NIRS进行主成分(Principal components, PCs)分析,第1、第2和第3主成分的贡献率分别为71.136%、8.596%和5.327%,前3个PCs的累积贡献率达85.059%。校正集和验证集的三维主成分空间分布情况如图2所示。在样本主成分空间分布图中,左侧为秸秆粪便混合物样本对应数据点,右侧为预处理秸秆样本对应数据点,产生如此清晰分类的结果与样品性状差异、原始光谱数据分布吻合。
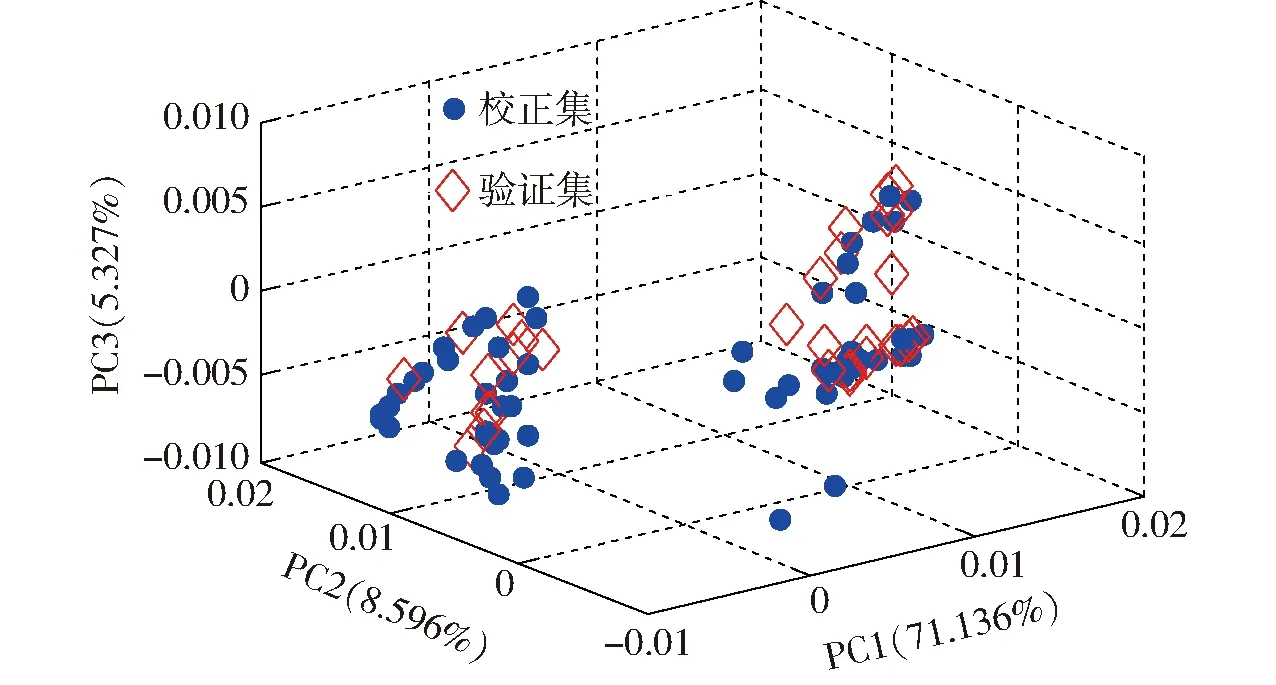
图2 样本主成分空间分布Fig.2 Distribution of samples in PCs space
由表2和图2可知,校正集样本碳氮比基本涵盖了验证集,且校正集和验证集样本在主成分空间上分布比较均匀,可以使用该样本划分方法进行NIRS分析。
2.2 特征波长优选
2.2.1GSA-iPLS特征谱区优选
GSA-iPLS先按照iPLS将全谱划分成多个均匀的子区间,再以子区间个数为码长、以RMSECV为目标函数运行GSA算法,优选特定子区间数下的特征谱区。为考察分割波长点个数对波长选择及模型预测性能的影响,分别按约30、40、50、60、80、100、120个波长点划分子区间,依次将预处理后的一阶导数光谱划分为61、46、37、31、23、18、15个子区间,依据RMSECV优选有效的子区间组合作为GSA-iPLS优选的特征谱区。为解决GSA优选结果的随机性问题,在每个子区间划分个数下,执行10次GSA-iPLS算法,并选定回归模型性能最佳的子区间组合作为该子区间数下的碳氮比特征谱区。在进行GSA-iPLS特征谱区优选时,种群规模设为100,初温确定系数取200,降温系数取0.950,进化代数取200,交叉概率取0.700,变异概率取0.010,邻域解扰动位数取码长的1/10向上取整。不同子区间数下优选的碳氮比特征谱区信息如表3所示。
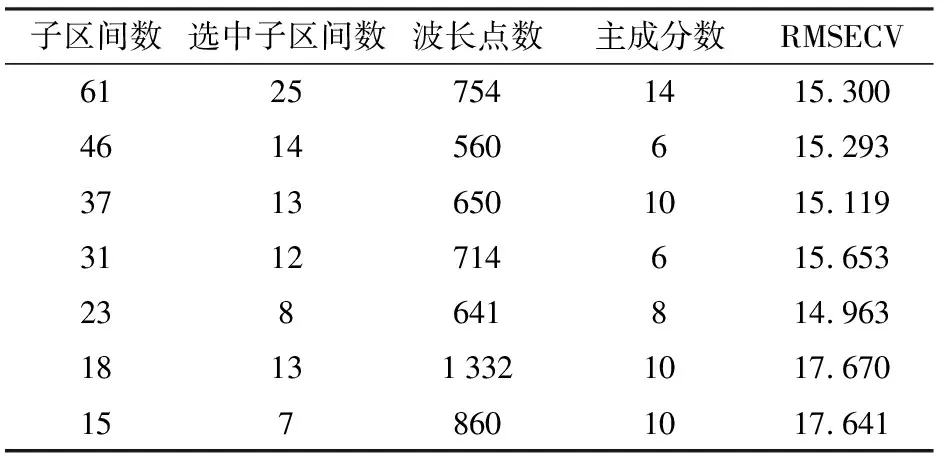
表3 GSA-iPLS优选结果Tab.3 Results optimized by GSA-iPLS
由表3可知,采用子区间划分个数为23,优选谱区的选中子区间数为8,波长点数为641时,回归模型的性能最佳。GSA-iPLS优选谱区如图3阴影部分所示。

图3 GSA-iPLS优选谱区Fig.3 Spectral intervals selected by GSA-iPLS
基于各含氢基团在近红外谱区中的分布特性可知,在选中的8个子区间中,3 950~4 935 cm-1(波长2 026~2 534 nm)对应着C—C、—CH、—CH2、—CH3和—NH2基团组合频,7 242~7 567 cm-1(波长1 322~1 381 nm)、7 901~8 226 cm-1(波长1 215~1 266 nm)和8 560~8 885 cm-1(波长1 125~1 168 nm)对应着—CH、—CH2和—CH3基团的二级倍频,9 219~9 544 cm-1(波长1 048~1 084 nm)对应着—CH、—NH2基团的三级倍频。
当特征波长点在整个谱区中分布比较集中时,GSA-iPLS谱区优选算法的性能优越,去除冗余波长点的效果较好。当特征波长点的分布比较分散时,GSA-iPLS以子区间为单位进行特征谱区优选,在去除冗余波长点时会连带去除部分有效波长点,进而影响回归模型的性能。此时,需要增大子区间个数,减小子区间内波长点的数量,防止GSA-iPLS算法去除过多的有效波长点。但子区间数过多时,编码码长过长,影响GSA算法搜索效率的同时还可能导致解空间的发散问题。因此,在进行问题求解时,需要结合实际情况,设置合理的算法参数,实现算法运行效率和求解精度的统一。
2.2.2DGSA-PLS特征波长优选
DGSA-PLS在进行特征波长点优选时,以GSA-iPLS优选的特征谱区波长点数为码长,随机生成160个码长为641的染色体构建初始种群,邻域解扰动位数取20,其它算法初始参数与GSA-iPLS一致。为消除GSA算法的随机性,执行算法50次对碳氮比特征波长点进行优选。多次执行时,每次都选中的波长点代表了染色体的优良基因,以这些特征波长点作为特征波长变量建立回归模型时,可以有效消除GSA算法的随机性,且能够得到较高的回归模型性能。DGSA-PLS波长优选结果与预处理后光谱的平均值对比如图4所示。

图4 DGSA-PLS优选波长变量Fig.4 Wavelength variables selected by DGSA-PLS
图4中,重复选中次数为1时,选中628个波长点;重复选中次数为50时,选中19个波长点。测试发现,校正集RMSECV和验证集RMSEP都随选中波长点数的增加呈先减小后增大的趋势,但两者的趋势存在较大差别。为了分析特征波长变量数目与模型性能的关系,绘制RMSECV、RMSEP与选中波长点数的关系图,如图5所示。
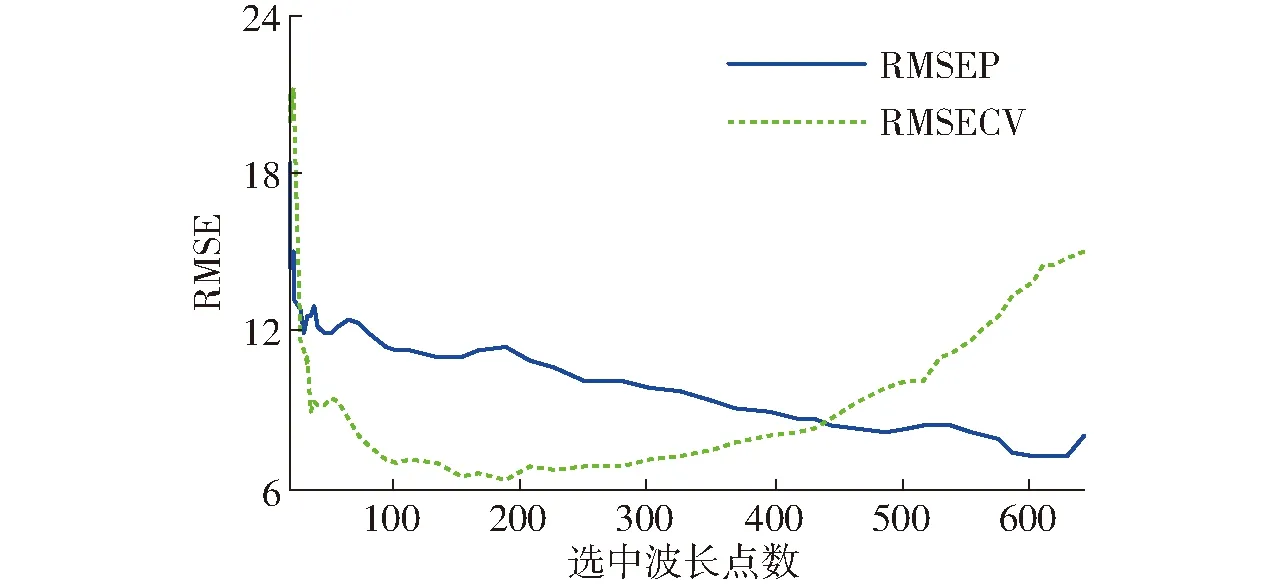
图5 RMSE与选中波长点数间的关系Fig.5 Relationship between RMSE and number of variables
由图5可知,RMSECV最小值早于RMSEP出现,当选中波长点数为189、重复选中次数为27次时,RMSECV最小。RMSEP最小时,对应的波长点数为628,重复选中次数为1次。RMSECV与RMSEP随选中波长点数变化趋势差异较大的主要原因在于GSA以校正集的RMSECV为依据进行特征波长优选,验证集性能拐点出现时表明校正集发生了过拟合。因此,选取RMSEP最小时对应的628个波长点作为DGSA-PLS优选的碳氮比特征波长变量。
2.3 回归模型评价与分析
为评测本文构建的两种波长优选算法的建模性能,以GSA-iPLS和DGSA-PLS优选后的特征波长变量作为PLS回归模型的输入,建立厌氧发酵原料碳氮比定量回归模型,并与全谱(Full-PLS)、协同区间偏最小二乘(Synergy iPLS,SiPLS)[30]和反向区间偏最小二乘(Backward iPLS,BiPLS)[31]优选特征波长的建模性能进行对比,结果如表4所示。

表4 不同回归模型评价指标Tab.4 Evaluation indicators of different regression models
由表4可知,SiPLS和BiPLS作为两种最典型iPLS算法,SiPLS的建模性能弱于全谱建模,而BiPLS的建模性能优于全谱建模。主要原因在于:SiPLS选取2~4个固定个数的子区间作为备选谱区,再通过比较RMSECV确定最佳谱区;而BiPLS通过剔除相关性较差的子区间,搜索RMSECV最小的子区间组合作为特征谱区;BiPLS比SiPLS更适于特征波长点分布比较分散问题的求解。而碳氮比对应着谱区中所有含碳和含氮基团的吸收峰,这些吸收峰在整个谱区中分布较广,适合采用BiPLS进行特征谱区优选。GSA-iPLS作为一种新型近红外光谱特征谱区优选算法,具有良好的随机搜索能力。在使用GSA-iPLS算法进行特征谱区优选时,与BiPLS相比扩展了搜索结果的随机性。通过多次搜索并选取建模性能最佳的搜索结果作为GSA-iPLS优选特征谱区,该方式能够有效提高算法的特征波长优选性能。


图6 碳氮比实测值与预测值分布Fig.6 Distribution of measured and predicted values for C/N ratio

3 结束语
探讨了采用NIRS技术结合化学计量学方法进行厌氧发酵原料碳氮比快速检测的可行性。为提高NIRS回归模型的检测精度和效率,基于GSA算法构建了GSA-iPLS和DGSA-PLS两种算法进行碳氮比特征波长的优选。GSA-iPLS将光谱数据划分成多个子区间后,以子区间个数为码长,搜索有效的特征波长子区间组合作为特征谱区,有效减少了建模变量个数,提高了碳氮比检测模型的精度和效率。DGSA-PLS在GSA-iPLS优选谱区的基础上,以波长点个数为码长进行特征波长变量优选,有效去除不相关的冗余波长点,得到628个特征波长点,建立的碳氮比检测模型RMSEP为7.178,RPD为3.805。与全谱建模相比,基于DGSA-PLS建立的回归模型有效波长点个数减少了65.94%,RMSEP减小了15.87%,有效地提高了模型的检测精度。

