本生烟响应蛋白激发子PevD1的差异表达基因鉴定与分析
梁颖博,李泽,邱德文,曾洪梅,李广悦,杨秀芬
本生烟响应蛋白激发子PevD1的差异表达基因鉴定与分析
梁颖博,李泽,邱德文,曾洪梅,李广悦,杨秀芬
(中国农业科学院植物保护研究所植物病虫害生物学国家重点实验室,北京 100081)
【】通过RNA-Seq筛选本生烟()响应大丽轮枝菌()蛋白激发子PevD1的差异表达基因(differentially expressed gene,DEG),分析PevD1诱导植物产生抗病性的潜在分子机制。用10 μmol·L-1的PevD1蛋白液渗入4周龄的本生烟叶片,分别在处理后6、12和24 h取样提取RNA,构建mRNA文库后采用BGISEQ-500平台进行测序。筛选各时间点的差异表达基因进行GO和KEGG分析;重点分析与诱导抗病相关的富含亮氨酸重复序列类受体蛋白激酶(leucine-rich repeats RLKs,LRR-RLKs)、转录因子(transcription factor,TF)以及病程相关蛋白(pathogenesis related protein,PR蛋白)家族差异表达基因;采用qRT-PCR对差异表达基因进行定量验证。GO功能富集以及KEGG通路富集分析表明,PevD1诱导6 h后的差异表达基因主要与细胞识别、光合作用、光收割等功能相关,显著富集在光合作用-天线蛋白通路、萜类化合物合成通路、黄酮和黄酮醇等次生代谢产物合成相关通路中;12 h和24 h的差异表达基因主要与细胞识别和胞内激酶等生物学功能相关,显著富集在植物-病原互作通路、倍半萜和三萜生物合成通路、黄酮和黄酮醇生物合成通路、亚麻酸代谢等次生代谢产物合成相关通路中。与光合作用相关的差异表达基因主要呈下调趋势,与萜类、黄酮类等抗病相关次生代谢产物合成通路相关的差异表达基因主要呈上调趋势。PevD1诱导后大量的LRR-RLKs、TF以及PR蛋白家族基因显著上调表达,这些基因与激发子识别、基因转录调控和抗病性相关。经qRT-PCR验证后,所检测基因的表达趋势与转录组测序结果一致。PevD1诱导本生烟中大量基因转录重排,大量LRR-RLKs、TF和PR蛋白家族基因上调表达,激活了植物免疫系统,使植物产生抗病性。研究结果可为今后深入探讨PevD1诱导植物免疫的机理提供依据。
本生烟;大丽轮枝菌;蛋白激发子PevD1;RNA-Seq;LRR-RLKs;转录因子;PR蛋白
0 引言
【研究意义】植物为了抵御环境中各种不良因素的危害形成了复杂有序的免疫调控机制。研究植物响应胁迫的分子机理,不仅能为农作物抗病育种提供有价值的基因资源,同时可为研究植物诱导性免疫反应的分子机制提供理论依据。采用模式植物本生烟()进行转录组测序,比较蛋白激发子PevD1处理前后差异表达基因(DEG)的转录表达情况,并对这些差异基因进行GO和KEGG富集分析,可为阐述PevD1激发植物免疫的分子机制打下基础。【前人研究进展】大丽轮枝菌()能引起多种植物的黄萎病,寄主植物超过400种,常年对棉花、马铃薯、茄子、番茄、辣椒、甘蓝、黄瓜和西瓜等造成严重的经济损失。目前在生产上主要依赖化学农药进行防治,缺乏有效的抗/耐病品种[1-3]。利用植物自身免疫系统减轻病害发生是未来植物病害绿色防控的重要途径之一[4]。PevD1是笔者实验室分离自大丽轮枝菌的蛋白激发子,能够诱导植物叶片产生超敏反应(HR),激发植物早期防御反应(NO、H2O2,细胞外培养基的碱化),引起胼胝质、酚类化合物及木质素的积累,提高植物叶片中的过氧化物酶(POD)、多酚氧化酶(PPO)和苯丙氨酸解氨酶(PAL)等防御相关酶的活性,诱导棉花和烟草系统抗病性[5-7]。近期研究表明,在烟草叶片中瞬时表达包含信号肽的PevD1重组蛋白能够引起强烈的细胞坏死反应,相比之下,切除信号肽的PevD1重组蛋白引起的细胞坏死反应则明显减弱[8],说明植物细胞对PevD1的识别过程可能主要位于细胞间质中,PevD1很可能作为真菌PAMP激发植物的PTI反应,但具体的作用机制尚不清楚。【本研究切入点】PevD1诱导植物抗病性的过程会引起大量抗病相关基因的转录调控,近年来飞速发展的转录组测序技术为全面研究植物免疫机制提供了丰富的数据基础。本研究以本生烟为材料,筛选PevD1诱导本生烟前后的差异表达基因,分析PevD1诱导本生烟抗病性的潜在机制。【拟解决的关键问题】通过RNA-Seq技术筛选本生烟中响应PevD1的差异表达基因并分析这些基因的主要富集途径,综合分析PevD1诱导本生烟的抗病机制。
1 材料与方法
试验于2018年5月至2019年3月在中国农业科学院植物保护研究所蛋白农药组完成。
1.1 材料
重组蛋白激发子PevD1表达与纯化的方法参照文献[8];本生烟种子由本实验室保存。
1.2 方法
1.2.1 样品准备 将纯化的激发子PevD1蛋白液稀释成10 μmol·L-1后,取4周龄的本生烟植株,分别用无针的注射器在本生烟叶片背部渗透注射,每个叶片注射20 µL,以等量的Tris-HCl(pH 8.0)为对照。分别在处理后6、12、24 h对叶片取样,液氮速冻后于-80℃保存备用。
1.2.2 BGISEQ-500平台测序 提取上述样品总RNA,质检合格后采用华大BGISEQ-500平台进行建库测序。
1.2.3 差异表达基因的筛选 使用DEGseq算法进行差异表达基因检测,DEGseq方法基于泊松分布[9]。为了提高准确性,将差异倍数为两倍以上(fold change,FC≥2)并且Q-value≤0.001(adjusted-value≤0.001)的基因筛选为显著差异表达基因。
1.2.4 差异表达基因GO及KEGG富集分析 根据官方Gene Ontology数据库分类(http://www.geneontology. org/),使用R软件中的phyper函数将差异表达基因进行GO功能富集分析。根据KEGG注释结果以及官方分类,使用R软件中的phyper函数将差异表达基因进行生物通路富集分析。
1.2.5 差异表达基因的qRT-PCR检测 参考茄科数据库(https://solgenomics.net/)的目的基因序列,使用Beacon Designer 8.0软件设计特异性引物(表1)。以上述样品提取的RNA反转录后的cDNA为模板,以为内参基因,采用荧光定量试剂盒(北京全式金)特异性扩增各个目的基因。参照2-ΔΔCt计算方法,分别对mRNA进行相对定量分析[10]。
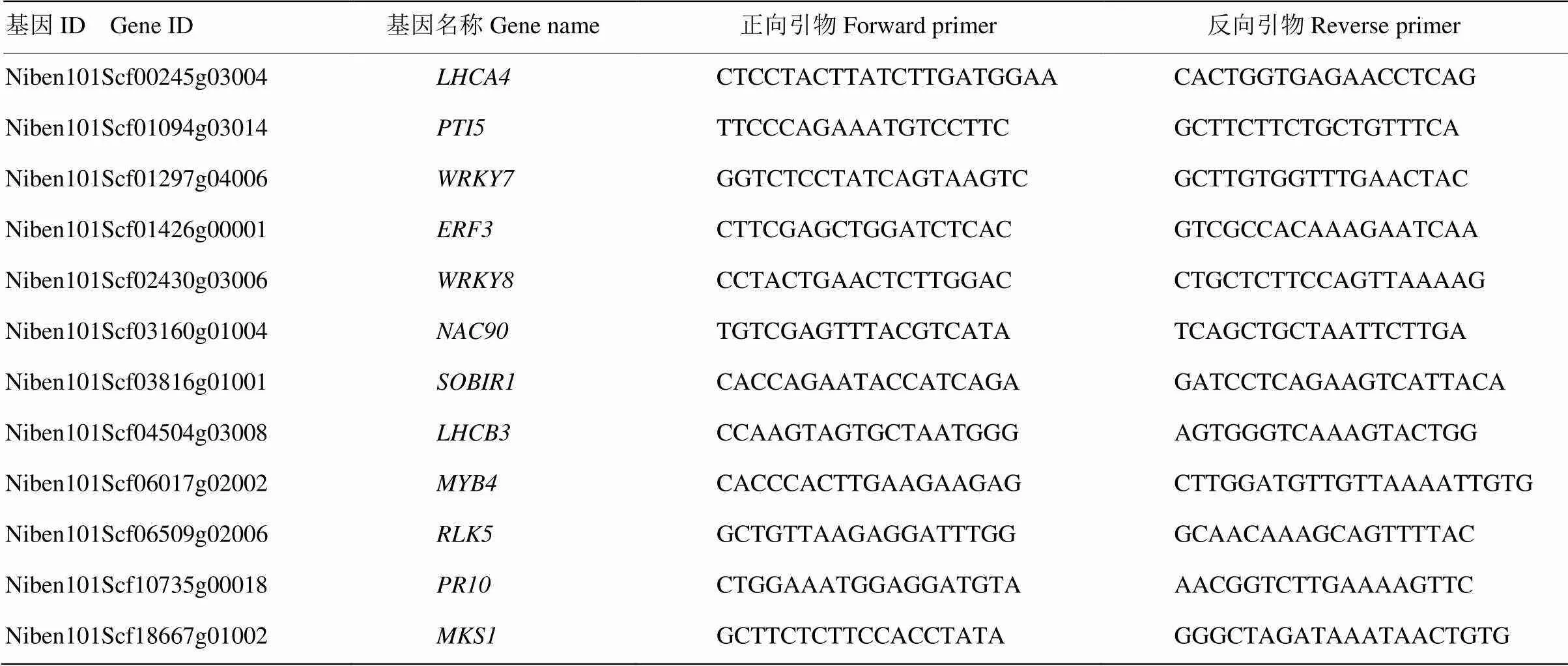
表1 待测差异表达基因的定量验证引物
2 结果
2.1 PevD1诱导本生烟的差异表达基因筛选
为了明确PevD1的诱导对本生烟基因转录的影响,在差异表达倍数FC≥2及Q-value≤0.001的条件下对PevD1处理组与对照组的差异表达基因进行了筛选。PevD1处理6 h时,共筛选到4 053个差异表达基因,其中2 911个上调,1 142个下调;PevD1处理12 h时,共筛选到5 180个差异表达基因,其中4 183个上调,997个下调;PevD1处理24 h时,共筛选到4 202个差异表达基因,其中3 082个上调,1 120个下调。这些差异表达基因反映了本生烟受PevD1诱导后产生的应答反应,有助于分析PevD1诱导本生烟产生抗性的机制。
2.2 差异表达基因的功能富集分析
对上述筛选到的差异表达基因进行GO功能富集分析。结果表明,PevD1诱导6—12 h后,差异表达基因主要与碳水化合物的结合活性、蛋白激酶活性、光合作用以及细胞间的识别作用相关(图1-A、1-B);PevD1诱导24 h后,差异表达基因主要与细胞识别及细胞的生物学过程相关(图1-C)。
KEGG通路富集分析显示,PevD1诱导6 h后,差异表达基因主要富集在植物-病原互作和光合作用-天线蛋白通路(图2-A);PevD1诱导12—24 h后,差异表达基因主要富集在植物-病原互作通路、MAPK信号通路、苯丙素生物合成和萜类化合物合成通路(图2-B、2-C)。在以上显著富集的通路中,光合作用-天线蛋白通路相关的差异表达基因主要呈下调趋势,而植物-病原互作、MAPK信号通路、苯丙素生物合成和萜类化合物合成通路相关的差异表达基因主要呈上调表达趋势。

A、B、C分别代表差异表达基因在6、12、24 h各时间点的GO富集结果,结果仅显示Q-value值最小的前20个富集条目。红圈标注的通路为Rich factor>0.2和Q-value<0.05的阈值下显著富集的条目
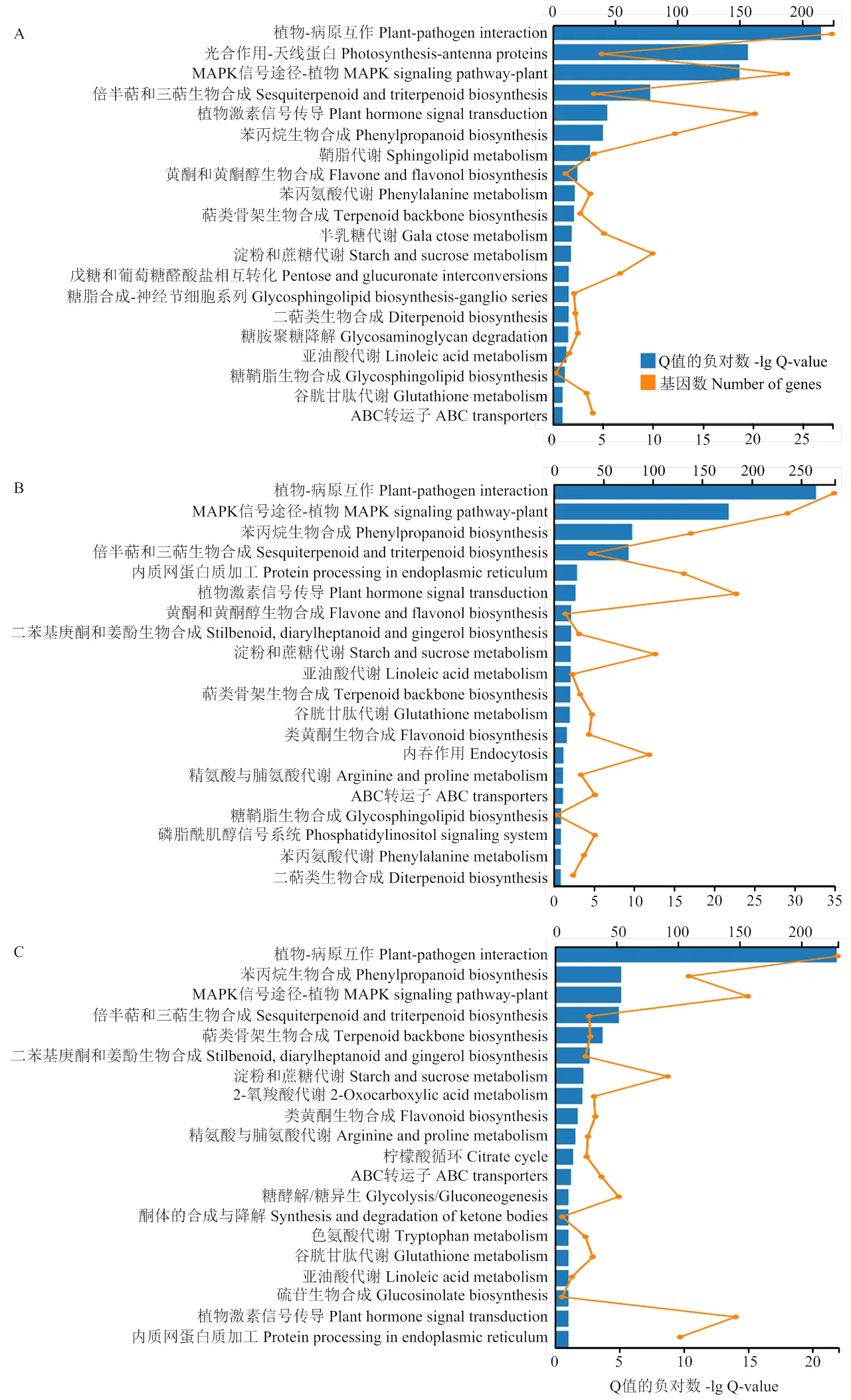
图2 各时间点差异表达基因的KEGG富集
2.3 LRR-RLKs分析
植物免疫识别系统是激活植物免疫反应的开关,通常包括位于细胞表面的膜识别受体PRRs(pattern- recognition receptors)和位于细胞质的胞内识别受体[11-12]。本研究根据PRG(plant resistance gene)数据库,通过序列比对预测的方法,对每组差异表达基因进行抗病基因分析,结果发现这些差异表达基因中有大量的RLP和RLK被诱导表达(图3),说明这些基因有可能参与了植物的早期识别反应。大量研究表明,LRR-RLKs(leucine-rich repeat receptor-like protein kinases)是植物受体激酶中研究较多的一类家族。进一步通过在线Blast的方法获取差异表达基因中的LRR-RLKs。结果表明,在所有的差异表达基因中共鉴定到109个LRR-RLKs,其中有27个在检测的时间点内有极显著变化(FC≥4)(表2)。鉴定这些基因能够更好地揭示植物对蛋白激发子PevD1的识别机制。
2.4 转录因子分析
转录因子在调控植物抗病性的过程中发挥着重要作用,它们通过与启动子上特定的DNA序列结合来激活或抑制基因的转录。本研究对差异表达基因所属的转录因子家族进行了分类统计。结果如图4所示,PevD1诱导后的各时间点均检测到大量差异表达的转录因子,其中以AP2-EREBP、WRKY、MYB和NAC为主,并且这些转录因子主要呈现上调表达的趋势。

表2 差异表达基因中LRR-RLKs的筛选

图3 PevD1处理后各时间点差异表达基因注释到PRG的数目
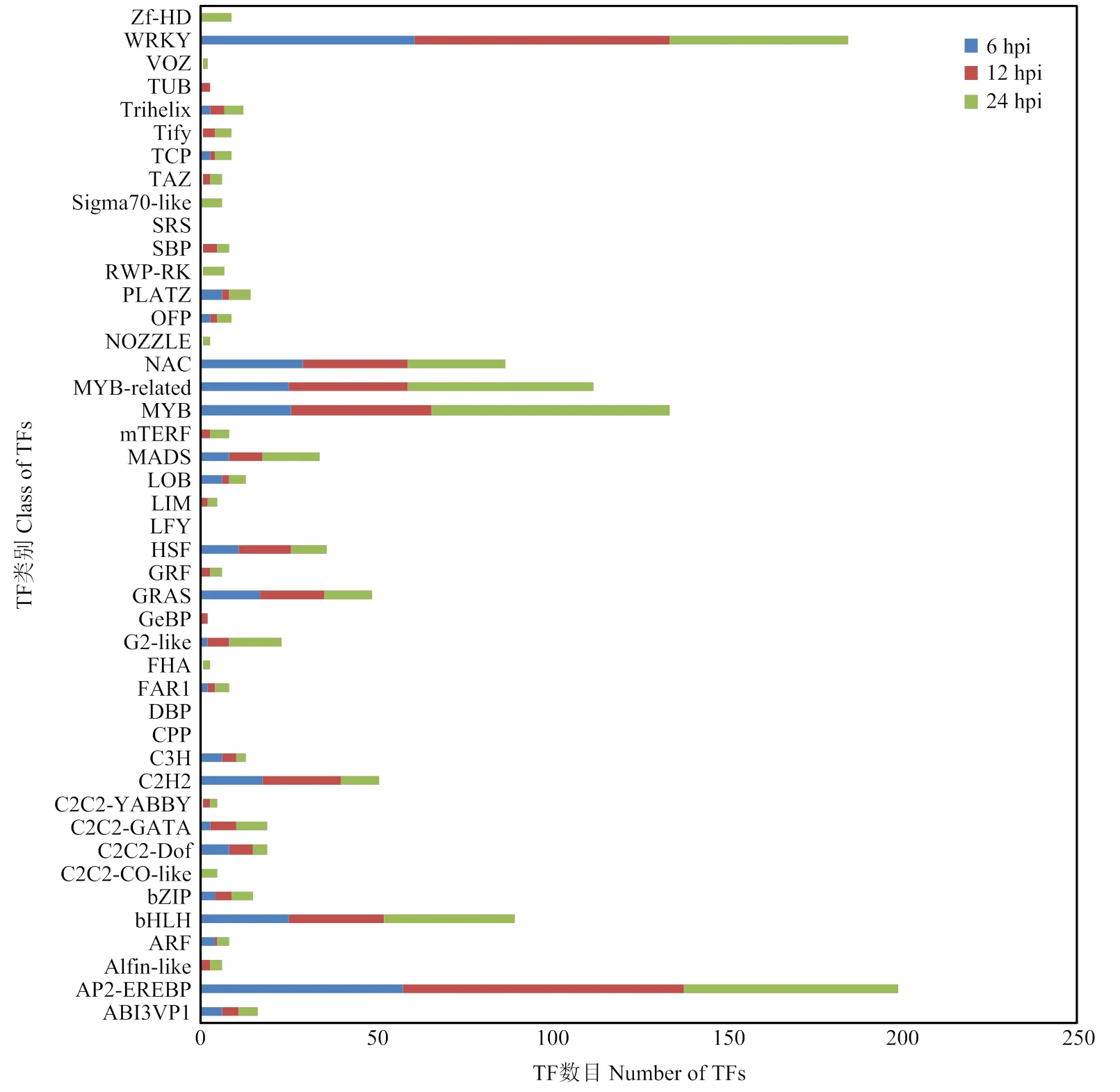
图4 转录因子分析
2.5 病程相关蛋白家族差异表达基因分析
病程相关蛋白(pathogenesis related protein,PR蛋白)的诱导和积累是植物产生防御反应的另一个重要特征,同时也是植物产生系统获得抗性(systemic acquired resistance,SAR)的重要组成部分。基于烟草中已有相关报道的PR家族基因序列,通过在线BLAST的方法从差异表达基因中共鉴定到了29个PR基因,包括、、、、、、、和,并且发现这些PR基因呈现显著性上调趋势(表3)。
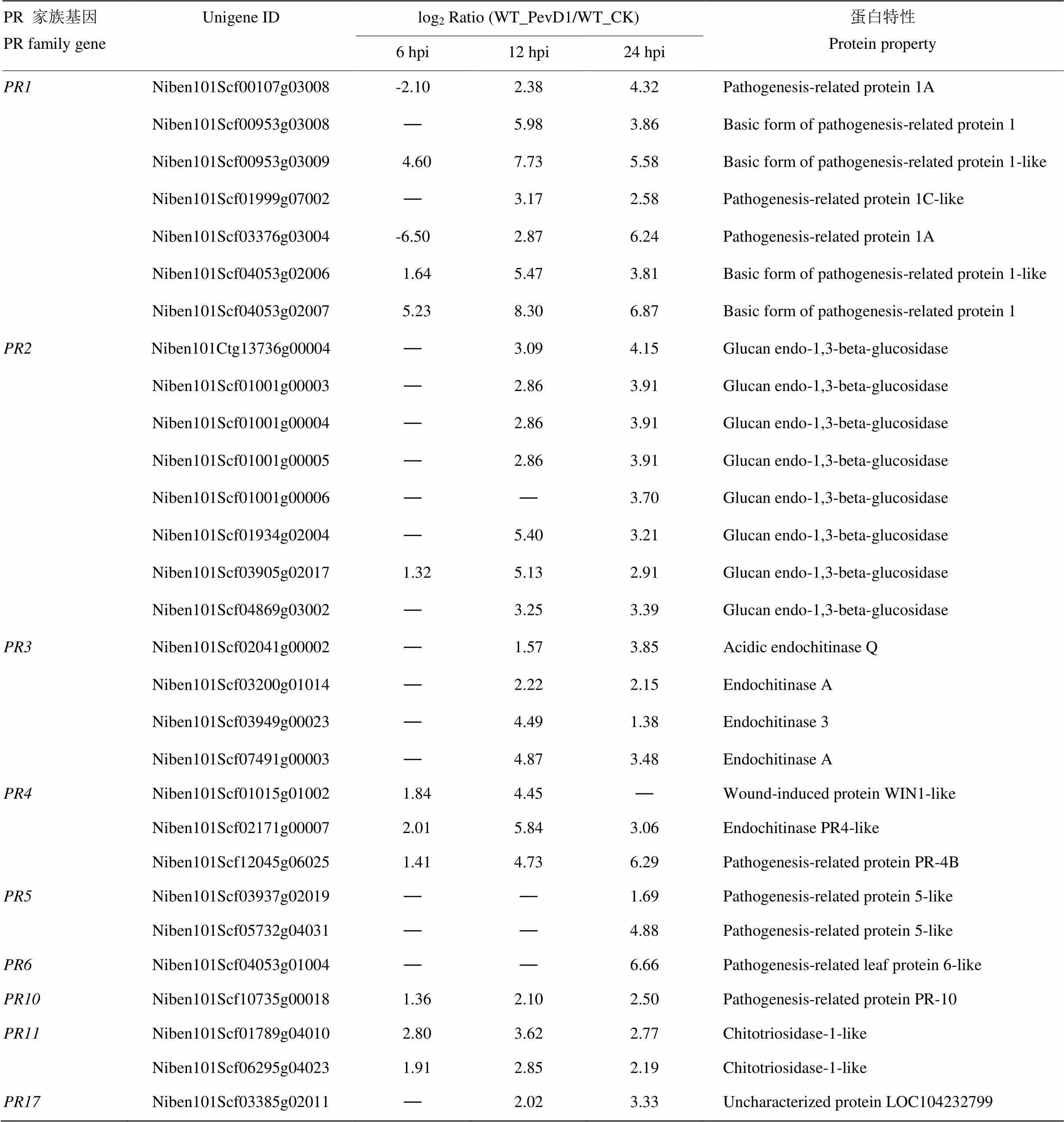
表3 PevD1诱导病程相关蛋白家族差异表达基因的筛选
2.6 差异表达基因的RT-qPCR验证
为了验证本研究中转录组测序数据的可靠性,选取12个与上述分析结果密切相关的基因用于qRT-PCR验证。结果表明这些基因表达模式与转录组测序结果一致(图5)。

图5 差异表达基因的qRT-PCR验证
3 讨论
PevD1是从大丽轮枝菌发酵液中分离得到的蛋白激发子。前期研究表明,重组表达的PevD1蛋白能够诱导烟草和棉花产生过敏反应,并能诱导NO和H2O2的积累、细胞外培养基的碱化、胼胝质的沉积、酚类化合物及木质素的合成等[5-7],但对于PevD1诱导植物产生免疫反应的作用机理仍有待探索。目前,RNA-Seq技术已被广泛应用于植物抗病机制的相关研究中,该技术可为研究提供大量的基因表达信息,从基因转录的差异入手,有助于更好地挖掘抗病相关的基因信息以及相关的代谢通路,从而更好地理解植物免疫反应的相关机理。
对本生烟响应PevD1诱导后的差异表达基因进行分析,其结果体现了本生烟受PevD1诱导后的免疫应答规律。本研究中,光合作用-天线蛋白通路相关的差异表达基因主要呈下调趋势,而植物-病原互作、MAPK信号通路、苯丙素生物合成和萜类化合物合成通路相关的差异表达基因主要呈上调趋势。由于植物免疫系统的启动涉及到大量基因的转录重编程过程,该过程需要植物耗费大量能量。已有研究表明,植物在产生免疫反应的过程中,能够通过MAPK级联反应主动抑制光合作用从而使免疫反应更为高效[13]。因此推测,在PevD1诱导初期,植物首先启动与细胞识别相关的基因,调整能量代谢,使得大量与光合作用相关的基因下调表达。随着植物免疫应答反应的持续,植物与病原互作相关通路的基因被逐步激活,进而引起抗病相关的次生代谢产物的积累,全面启动免疫应答反应。
LRR-RLKs在植物生长发育和胁迫应答的过程中起着重要的作用。拟南芥的受体类激酶FLS2能够识别细菌的鞭毛蛋白Flg22,进而与BAK1形成复合体共同激活下游的植物免疫信号[14-15];SOBIR1和BAK1都能够与受体类蛋白(LRR-RLPs)形成复合体,从而激活下游的植物免疫信号[16];拟南芥中的细菌延伸因子(EF-Tu)受体EFR能够特异性的识别EF-Tu,从而快速激活植物的PTI反应[17]。本研究中有27个LRR-RLKs基因被显著诱导表达,表明LRR-RLKs在PevD1对本生烟免疫系统的激活中起到了极其重要的作用。
植物中存在多个转录因子家族,相关报道表明AP2-EREBP(APETALA2/ethylene-responsive element binding protein)、WRKY、MYB、NAC家族的转录因子在植物抗病防御反应的调控中起着重要的作用[18]。AP2-EREBP家族的转录因子在调控植物生长发育以及胁迫响应中发挥着重要作用[19];拟南芥中的APD1作为AP2-EREBP家族的成员,能够正调节水杨酸(SA)的合成,在防御细菌性病原物的过程中起着重要作用[20];OsERF101是水稻生殖和营养阶段对干旱胁迫产生适应性反应的重要正调控因子[21];WRKY家族的转录因子能够与多种植物防御反应基因启动子序列中的W-box顺式作用元件相结合,从而启动相应抗病基因的转录表达[22];AtWRKY33能够正调控拟南芥对坏死型真菌的免疫反应[23];OsWRKY53对水稻的基础免疫起正调控作用,在水稻中过表达能够提高水稻对稻瘟病的抗性[24];MYB家族转录因子在苯丙烷类衍生物的合成代谢中发挥着重要的作用[25-26];在烟草中过表达能够检测到烟草叶片中黄酮含量增加,但总木质素水平降低,表明的过表达可能导致代谢通量从木质素合成途径定向到类黄酮合成途径[27];NAC转录因子在植物响应生物和非生物胁迫中充当调控网络的节点[28],油菜中的NAC87能够调控细胞中ROS的产生并介导细胞的坏死反应,同时伴随着细胞形态学水平的典型变化[29];在烟草中过表达番茄的能够促进干旱和盐胁迫下的根生长和发育,同时能够提高转基因烟草对细菌病原体的抗性[30]。本研究中,笔者筛选到了大量差异表达的AP2-EREBP、WRKY、MYB、NAC家族的转录因子,表明这些转录因子在一定程度上参与了PevD1诱导植物免疫的过程。
PR蛋白的产生和积累是植物产生免疫反应的一个重要特征,也是植物产生系统获得性抗性(SAR)的重要组成部分[31]。在目前的研究中,PR蛋白可分为17个家族,其中PR-1、PR-2、PR-3、PR-4、PR-5、PR-10、PR-11、PR-17被报道与植物的SAR相关[32-37]。本研究基于在线Blast以及序列比对的方法鉴定到了29个PR基因,它们的表达水平在PevD1诱导后被显著上调,推测这些PR蛋白可能在PevD1诱导本生烟的防御反应中发挥着重要作用。
4 结论
本生烟经PevD1诱导后,分别在6、12、24 h检测到了4 053、5 180和4 202个差异表达基因。对这些差异表达基因进行分析,发现光合作用-天线蛋白通路相关的差异表达基因主要呈下调趋势,而植物-病原互作通路、MAPK信号通路、苯丙素生物合成和萜类化合物合成通路相关的差异表达基因主要呈上调趋势,推测PevD1诱导本生烟免疫反应的初期可能通过降低光合作用以应对胁迫,随着诱导时间的延长,与植物免疫相关的大量基因上调表达,免疫系统激活。进一步对LRR-RLKs、转录因子和PR蛋白家族的基因进行分析,发现这些与免疫识别、信号传递和抗性相关的基因PevD1诱导后主要呈上调趋势,推测本生烟通过这些防御相关基因的转录激活来响应PevD1的刺激,最终产生系统抗性。
[1] BARBARA D J, CLEWES E. Plant pathogenicspecies: How many of them are there?, 2003, 4(4): 297-305.
[2] DURESSA D, ANCHIETA A, CHEN D, KLIMES A, GARCIA- PEDRAJAS M D, DOBINSON K F, KLOSTERMAN S J. RNA-seq analyses of gene expression in the microsclerotia of., 2013, 14: 607.
[3] CARROLL C L, CARTER C A, GOODHUE R E, LAWELL C c l, SUBBARAO K V. A review of control options and externalities forwilts.,2018, 108(2): 160-171.
[4] 邱德文. 我国植物病害生物防治的现状及发展策略. 植物保护,2010, 36(4): 15-18.
QIU D W. Current status and development strategy for biological control of plant diseases in China., 2010, 36(4): 15-18. (in Chinese)
[5] WANG B, YANG X, ZENG H, LIU H, ZHOU T, TAN B, YUAN J, GUO L, QIU D. The purification and characterization of a novel hypersensitive-like response-inducing elicitor fromthat induces resistance responses in tobacco.,2012, 93(1): 191-201.
[6] 王炳楠, 杨秀芬, 曾洪梅, 邱德文. 大丽轮枝菌分泌蛋白激发子的分离纯化及生物功能研究. 生物技术通报,2011(11): 166-171.
WANG B N, YANG X F, ZENG H M, QIU D W. Purification and its bioassay of secreted elicitor protein from., 2011(11): 166-171 (in Chinese)
[7] 卜冰武, 邱德文, 曾洪梅, 郭立华, 袁京京, 杨秀芬. 大丽轮枝菌蛋白激发子PevD1诱导棉花抗病性及作用机理. 植物病理学报,2014, 44(3): 254-264.
BU B W, QIU D W, ZENG H M, GUO L H, YUAN J J, YANG X F. Induced resistance and mechanism of protein elicitor PevD1 againstin cotton., 2014, 44(3): 254-264. (in Chinese)
[8] ZHANG Y, GAO Y, LIANG Y, DONG Y, YANG X, QIU D.PevD1, an Alt a 1-like protein, targets cotton PR5-like protein and promotes fungal infection., 2019, 70(2): 613-626.
[9] WANG L K, FENG Z X, WANG X, WANG X W, ZHANG X G. DEGseq: an R package for identifying differentially expressed genes from RNA-seq data.,2010, 26(1): 136-138.
[10] SCHMITTGEN T D, LEE E J, JIANG J. High-throughput real-time PCR.,2008, 429: 89-98.
[11] SAIJO Y, LOO E P, YASUDA S. Pattern recognition receptors and signaling in plant-microbe interactions.,2018, 93(4): 592-613.
[12] LIANG X, ZHOU J M. Receptor-like cytoplasmic kinases: Central players in plant receptor kinase-mediated signaling.,2018, 69: 267-299.
[13] SU J, YANG L, ZHU Q, WU H, HE Y, LIU Y, XU J, JIANG D, ZHANG S. Active photosynthetic inhibition mediated by MPK3/MPK6 is critical to effector-triggered immunity., 2018, 16(5): e2004122.
[14] Chinchilla D, Zipfel C, Robatzek S, Kemmerling B,Nürnberger T, Jones J D, Felix G, Boller T. A flagellin- induced complex of the receptor FLS2 and BAK1 initiates plant defence.,2007, 448(7152): 497-500.
[15] Schulze B, Mentzel T, Jehle A K, Mueller K, Beeler S, Boller T, Felix G, Chinchilla D. Rapid heteromerization and phosphorylation of ligand-activated plant transmembrane receptors and their associated kinase BAK1.,2010, 285(13): 9444-9451.
[16] LIEBRAND T W, VAN DEN BURG H A, JOOSTEN M H. Two for all: receptor-associated kinases SOBIR1 and BAK1.,2014, 19(2): 123-132.
[17] Zipfel C, Kunze G, Chinchilla D, Caniard A, Jones J D, Boller T, Felix G. Perception of the bacterial PAMP EF-Tu by the receptor EFR restricts-mediated transformation.,2006, 125(4): 749-760.
[18] NG D W K, ABEYSINGHE J K, KAMALI M. Regulating the regulators: The control of transcription factors in plant defense signaling.,2018, 19(12): 3737.
[19] DIETZ K J, VOGEL M O, VIEHHAUSER A. AP2/EREBP transcription factors are part of gene regulatory networks and integrate metabolic, hormonal and environmental signals in stress acclimation and retrograde signalling.,2010, 245(1/4): 3-14.
[20] GAUTAM J K, NANDI A K. APD1, the unique member ofAP2 family influences systemic acquired resistance and ethylene-jasmonic acid signaling.,2018, 133: 92-99.
[21] JIN Y, PAN W, ZHENG X, CHENG X, LIU M, MA H, GE X., an ERF family transcription factor, regulates drought stress response in reproductive tissues.,2018, 98(1/2): 51-65.
[22] PANDEY S P, SOMSSICH I E. The role of WRKY transcription factors in plant immunity.,2009, 150(4): 1648-1655.
[23] Zheng Z, Qamar S A, Chen Z, Mengiste T.WRKY33 transcription factor is required for resistance to necrotrophic fungal pathogens.,2006, 48(4): 592-605.
[24] CHUJO T, TAKAI R, AKIMOTO-TOMIYAMA C, ANDO S, MINAMIE, NAGAMURA Y, KAKU H, SHIBUYA N, YASUDA M, NAKASHITA H, Umemura K, Okada A, Okada K, Nojiri H, Yamane H. Involvement of the elicitor-induced gene,2007, 1769(7/8): 497-505.
[25] DUBOS C, STRACKE R, GROTEWOLD E, WEISSHAAR B, MARTIN C, LEPINIEC L. MYB transcription factors in.,2010, 15(10): 573-581.
[26] LIU J, OSBOURN A, MA P. MYB transcription factors as regulators of phenylpropanoid metabolism in plants.,2015, 8(5): 689-708.
[27] MA Q H, WANG C, ZHU H H.cloned from wheat regulates lignin biosynthesis through negatively controlling the transcripts of both cinnamyl alcohol dehydrogenase and cinnamoyl-CoA reductase genes.,2011, 93(7): 1179-1186.
[28] HUANG Y, LI T, XU Z S, WANG F, XIONG A S, HUANG Y, LI T, XU Z S, WANG F, XIONG A S. Six NAC transcription factors involved in response to TYLCV infection in resistant and susceptible tomato cultivars.,2017, 120: 61-74.
[29] YAN J, TONG T, LI X, CHEN Q, DAI M, NIU F, YANG M, DEYHOLOS M K, YANG B, JIANG Y Q. A novel NAC-type transcription factor, NAC87, from oilseed rape modulates reactive oxygen species accumulation and cell death.,2018, 59(2): 290-303.
[30] WANG G, ZHANG S, MA X, WANG Y, KONG F, MENG Q. A stress-associated NAC transcription factor (SlNAC35) from tomato plays a positive role in biotic and abiotic stresses.,2016, 158(1): 45-64.
[31] VAN LOON L C, REP M, PIETERSE C M J. Significance of inducible defense-related proteins in infected plants.,2006, 44: 135-162.
[32] ANTONIW J F, RITTER C E, PIERPONT W S, VAN LOON L C. Comparison of three pathogenesis-related proteins from plants of two cultivars of tobacco infected with TMV., 1980, 47(1): 79-87.
[33] VAN LOON L C. Regulation of changes in proteins and enzymes associated with active defence against virus infection//, 1982, 37: 247-273.
[34] LAGRIMINI L M, BURKHART W, MOYER M, ROTHSTEIN S. Molecular cloning of complementary DNA encoding the lignin- forming peroxidase from tobacco: Molecular analysis and tissue- specific expression.,1987, 84(21): 7542-7546.
[35] 李瑞博, 崔秀明, 刘玉忠, 吴志刚, 林淑芳, 申业, 黄璐琦. 三七病程相关蛋白1基因的克隆与表达分析. 药学学报,2014, 49(1): 124-130.
LI R B, CUI X M, LIU Y Z, WU Z G, LIN S F, SHEN Y, HUANG L Q. Cloning and expression analysis of pathogenesis-related protein 1 gene of.,2014, 49(1): 124-130. (in Chinese)
[36] MELCHERS L S, APOTHEKER-DE GROOT M, VAN DER KNAAP J A, PONSTEIN A S, SELA-BUURLAGE M B, BOL J F, CORNELISSEN B J, VAN DEN ELZEN P J, LINTHORST H J. A new class of tobacco chitinases homologous to bacterial exo-chitinases displays antifungal activity., 1994, 5(4): 469-480.
[37] OKUSHIMA Y, KOIZUMI N, KUSANO T, SANO H. Secreted proteins of tobacco cultured BY2 cells: identification of a new member of pathogenesis-related proteins.,2000, 42(3): 479-488.
Identification and Analysis of Differentially Expressed Genes Induced by Protein Elicitor PevD1 in
Liang Yingbo, Li Ze, Qiu Dewen, Zeng Hongmei, Li Guangyue, Yang Xiufen
(State Key Laboratory for Biology of Plant Disease and Insect Pests, Institute of Plant Protection, Chinese Academy of Agricultural Sciences, Beijing 100081)
【】The objective of this study is to screen the differentially expressed genes (DEGs) induced by protein elicitor PevD1 ofinby RNA-Seq, and to analyzethe potential mechanism of PevD1-induced disease resistance. 【】Leaves of 4-week-oldwere infiltrated with 10 μmol·L-1PevD1 solution, and samples were taken at 6, 12 and 24 h after PevD1 treatment, then RNA was extracted. The mRNAlibraries were constructed and sequenced by BGISEQ-500 platform. The DEGs at each time point were screened for GO and KEGG analysis. The leucine-rich repeat receptor-like kinases (LRR-RLKs), transcription factors (TFs) and pathogenesis-related (PR) proteins family in DEGs were analyzed, and qRT-PCR was used to verify the expression levels of relevant DEGs.【】GO functional enrichment and KEGG pathway enrichment analysis indicated that the DEGs at 6 h post infiltration (hpi) were mainlyrelated to cell recognition, photosynthesis, light-harvesting, and were significantlyenriched in photosynthesis-antenna protein pathways, terpenoid synthesis pathway, flavonoid and flavonol, and other secondary metabolite synthesis pathways. The DEGs at 12 hpi and 24 hpi were mainly associated with cell recognition and biological functions such as intracellular kinase activity, and were significantly enriched in plant-pathogen interaction pathway, sesquiterpene and triterpenoid biosynthetic pathway, flavonoid and flavonol biosynthetic pathway, linolenic acid metabolism. The unigenes in photosynthesis-antenna proteins, photosynthesis and porphyrin, chlorophyll metabolism pathway were mainly down-regulated, and the unigenes in sesquiterpenoid and triterpenoid biosynthesis, flavone and flavonol biosynthesis, plant-pathogen interaction, phenylpropanoid biosynthesis and terpenoid backbone biosynthesis pathway were mainly up-regulated. after PevD1 induction, a large number of LRR-RLKs,TFsand PR proteins family genes weresignificantly up-regulated, which were related to elicitor recognition, gene transcriptional regulation and disease resistance. qRT-PCR analysis showed that the expression pattern of the detected DEGswas consistent with the RNA-Seq results.【】PevD1 induced a large number of gene transcriptional rearrangements in. Lots of LRR-RLKs, TFs and PR proteins family genes were up-regulated, which activated the plant immune system and conferred to plants disease resistant.These results can provide a basis for further study on the mechanism of PevD1-induced immunity in the future.
;; protein elicitor PevD1; RNA-Seq; LRR-RLKs; transcription factor; PR protein
10.3864/j.issn.0578-1752.2019.21.008
2019-05-22;
2019-06-28
国家重点研发计划(2017YFD0201101)、国家自然科学基金(31772151)
梁颖博,E-mail:Lyingbo16@163.com。通信作者杨秀芬,E-mail:yangxiufen@caas.cn
(责任编辑 岳梅)

