大肠杆菌胞内代谢物组学分析样品前处理条件优化
李阳 王继彤 刘晓露 田静 贾铮 肖志明 樊霞
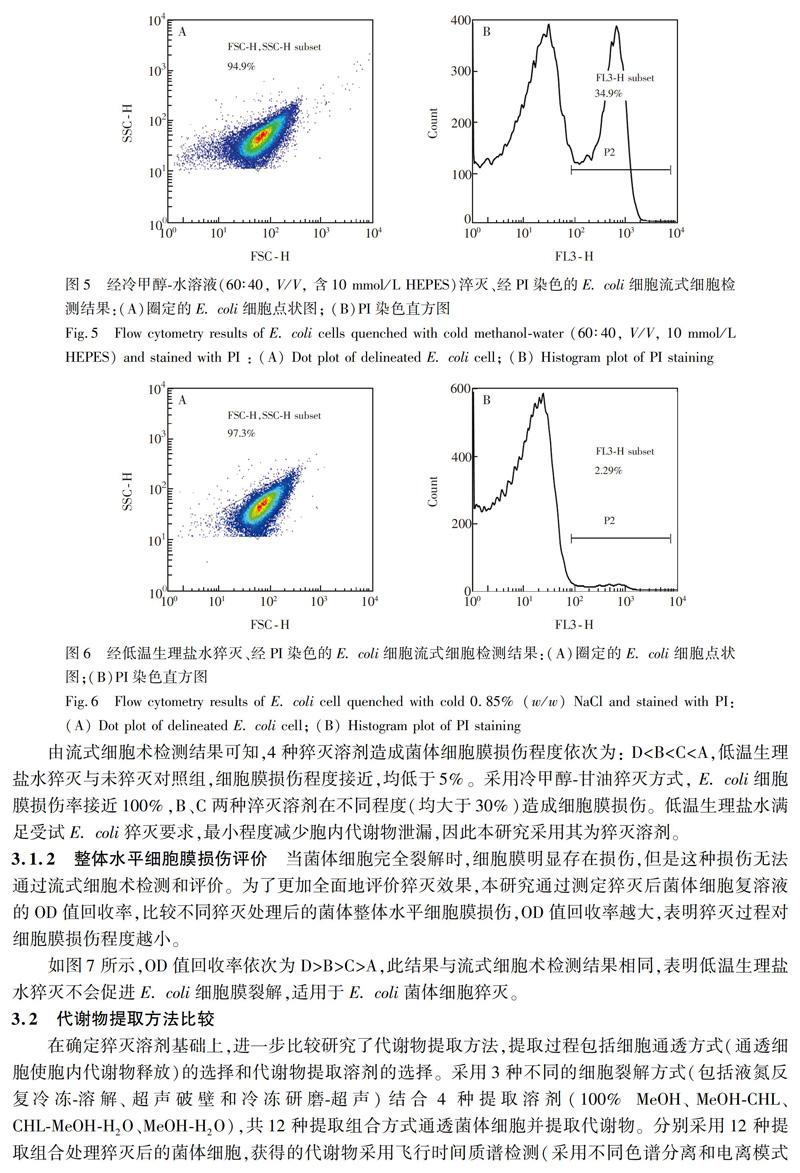
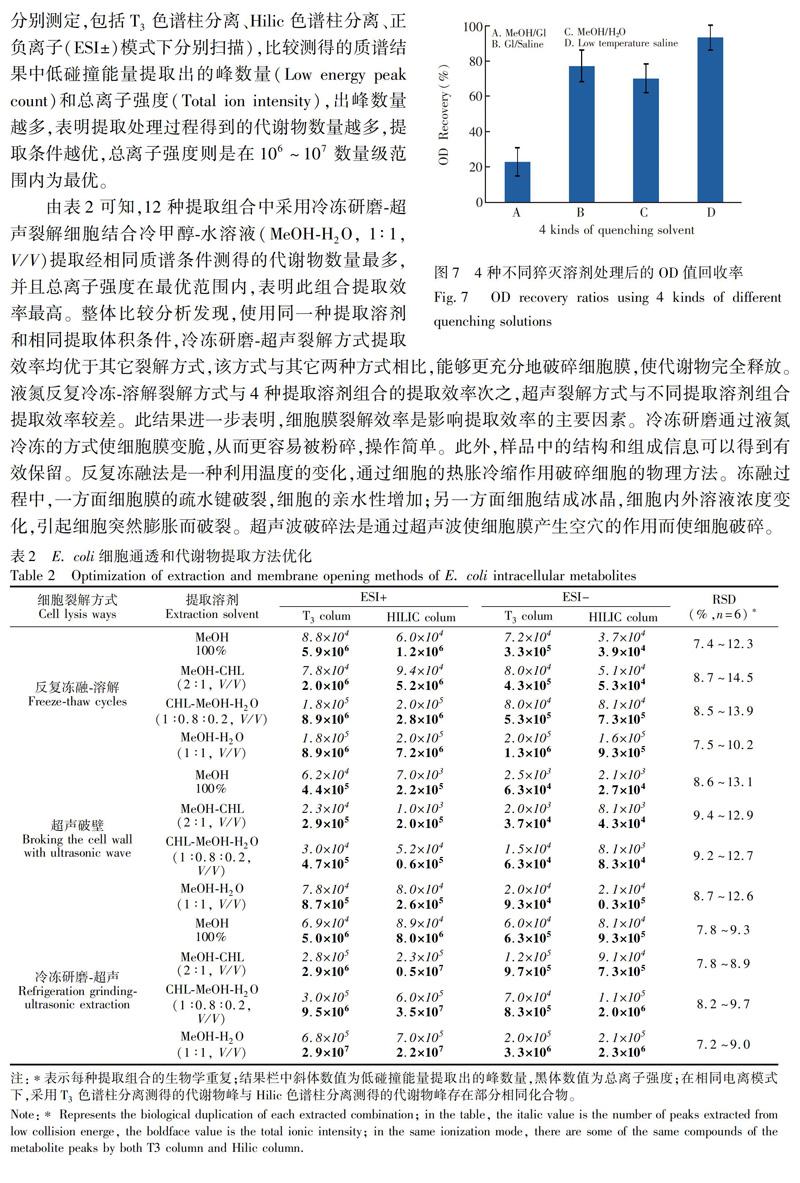
摘 要 :建立了适用于大肠杆菌胞內代谢物组学分析的样品前处理条件。大肠杆菌菌体细胞采用冷冻盐水(0.85% NaCl,80℃预冷15 min)进行猝灭,猝灭后的菌体细胞经过真空冷冻干燥,再通过液氮冷冻研磨结合超声裂解的方式通透细胞膜,最后采用冷甲醇水(MeOHH2O,1∶1,V/V,4℃)提取胞内代谢物。分别采用流式细胞术检测和OD值回收率从单细胞水平和整体水平评价猝灭溶剂对大肠杆菌细胞膜损伤影响。结果表明,采用冷冻盐水猝灭大肠杆菌细胞损伤率小于5%。采用液相色谱飞行时间质谱(T3和Hilic 2种色谱分离结合ESI±扫描模式)检测结果中低碰撞能量提取出的峰数量和总离子强度评价了3种细胞通透方式和4种提取溶剂组合的胞内代谢物提取效果。结果表明,冷冻研磨结合超声裂解通透细胞膜、冷甲醇水提取代谢物测得的代谢物数量最多(≥105),并且总离子强度在最优范围(106~107)内。本研究采用冷冻干燥、冷冻研磨和超声萃取相结合的代谢物提取方式,有效促进了细胞膜裂解,提高了代谢物提取效率。本研究建立的前处理条件适用于大肠杆菌胞内代谢物组学分析。
关键词 :大肠杆菌; 胞内代谢物; 组学分析; 样品前处理
1 引 言
随着分离、分析技术和计算机信息学的不断发展,代谢组学技术日趋成熟和进步,已被应用到多个领域。由于微生物在生物体系中具有重要地位,开展微生物代谢组学研究受到广泛关注,并形成具有独特性和系统性的研究策略[~3]。利用代谢组学技术对微生物细胞内所有低分子质量的代谢物(包括代谢中间产物、激素、信号分子和次生代谢产物)同时进行定性和定量分析,获得高通量代谢物结果信息,再利用化学计量学建模和预测,进而了解相关代谢途径调控和变化的关键信息,为深入了解微生物代谢调控提供了更直接的技术手段[4,5]。微生物代谢组学实验主要内容是样品前处理、数据采集、数据分析和生物学活性和功能分析归属,其中样品前处理是实验的第一步,也是关键的一步[6]。微生物代谢组学样品前处理包括受试菌的复苏和培养、菌体细胞猝灭、细胞膜通透以及代谢物提取[7]等。由于不同受试微生物生物活性和生理结构不完全相同,所采用的前处理条件各异。目前尚无通用的标准方法能够适用于所有微生物样品前处理。菌体细胞猝灭是样品前处理过程中至关重要的实验步骤,已报道的猝灭方式包括有机试剂法[8]、极端pH法[9]、快速过滤法[0]和冷溶剂猝灭法[1]。其中高浓度有机试剂或极端pH条件容易造成微生物细胞膜破裂,进而导致胞内代谢物泄漏。快速过滤方式操作不便,如果滤纸孔径太大会影响菌体收集数量,滤纸孔径过小导致滤膜堵塞,过滤时间延长。冷溶剂猝灭是温和、高效的猝灭方式,能够瞬间抑制细胞内代谢酶活性,将细胞代谢“冻结”在某一时间点[2]。需要注意的是, 使用低温溶剂同样需要考虑菌体细胞膜破裂导致胞内代谢物泄漏。细胞通透的目的是将细胞膜打开,使代谢物全部释放,以便于后续提取。目前,应用较多的通透方式为反复冻溶和超声处理[3,14]。代谢产物提取则应根据关注的代谢物溶解特性选择最适提取试剂,最大限度将代谢物提取出来。同时,选择提取溶剂时还应考虑代谢物稳定性、提取溶剂是否容易去除, 以及提取溶剂与仪器分析系统的兼容性。
大肠杆菌(Escherichia coli,E. coli)遗传背景清楚且培养条件简单,是各种生物化学、生物技术研究的重要模式生物[5]。开展E. coli代谢组学研究可为更深入地阐明E. coli代谢调控机理提供新的解决途径。本研究采用流式细胞术检测[5]和OD值回收率[6]评价了不同低温猝灭溶剂对E. coli细胞损伤的影响,利用飞行时间质谱检测结果中低碰撞能量下的出峰数量和总离子强度评价了不同细胞通透方式和提取溶剂组合的代谢物提取效果。通过比较优化,确定了适用于E. coli胞内代谢物组学分析的细胞猝灭溶剂和代谢物提取溶剂,提出了猝灭细胞冷冻干燥后冷冻研磨结合超声萃取的代谢物提取方式,在细胞猝灭过程中最大程度避免细胞膜损伤,提取过程中有效促进细胞壁破裂,提高了胞内代谢物提取效率, 此条件能够满足E. coli代谢组学分析要求。
2 实验部分
2.1 仪器与试剂





References
1 Aldridge B B, Rhee K Y. Curr. Opin. Microbiol., 2014, 19: 90-96
2 ZHOU HongWei, TAN FengYi, ZHONG Yin, LUAN TianGang. Chinese J. Anal. Chem., 2007, 35(2): 309-314
周宏伟, 谭凤仪, 钟 音, 栾天罡. 分析化学, 2007, 35(2): 309-314
3 YU Wei, GAO JiaoQi, ZHOU YongJin. Chinese Journal of Chromatography, 2019, 37(8): 798-805
禹 伟, 高教琪, 周雍进. 色谱, 2019, 37(8): 798-805
4 Gulik W M V, Andre B C, Seifar R M, Heijnen J J. Metabolomics in Practice. Weinheim: WileyVCH Verlag GmbH & Co KGaA, 2013: 1-19
5 Heberling C, Dhurjati P. Int. J. Mol. Sci., 2015, 16: 8949-8967
6 Wang X, Xie Y, Gao P, Zhang S, Tan H, Yang F, Lian R, Tian J, Xu G. Anal. Biochem., 2014, 451: 48-55
7 Patejko M, Jacyna J, Markuszewski M J. J. Chromatogr., 2017, 1043: 150-157
8 Tian J, Sang P, Gao P, Fu, R, Yang D, Zhang L, Zhou J, Wu S, Lu X, Li Y, Xu G. J. Sep. Sci., 2009, 32(13): 2281-2288
9 Li X, Long D, Ji J, Yang W, Zeng Z, Guo S, Ji Z, Qi G, Chen S. J. Microbiol. Meth., 2013, 94: 61-67
10 Kim S, Lee D Y, Wohlgemuth G, Park H S, Fiehn O, Kim K H. Anal. Chem., 2013, 85(4): 2169-2176
11 TaymazNikerel H, de Mey M, Ras C, ten Pierick A, Seifar R M, Van Dam J C, Heijnen J J, Van Glilik W M. Anal. Biochem., 2009, 386: 9-19
12 Baran R, Bowen B P, Northen T R. Mol. Biosyst., 2011, 7(12): 3200-3206
13 LI JingJing, JI GuoZhen, GAO AiWu, CAO Ping, YANG JinLi, WANG HaiRong. China Animal Husbandry & Veterinary Medicine, 2017, 44(3): 651-658
李晶晶, 冀國珍, 高爱武, 曹 平, 杨金丽, 王海荣. 中国畜牧兽医, 2017, 44(3): 651-658
14 Ohashi Y, Hirayama A, Ishikawa T, Nakamura S, Shimizu K, Ueno Y, Tomita M, Soga T. Mol. Biosyst., 2008, 4(2): 135-147
15 YE YangFang, ZHANG LiMin, AN YanPeng, HAO FuHua, TANG HuiRu. Chinese J. Anal. Chem., 2011, 39(8): 1186-1194
叶央芳, 张利民, 安艳捧, 豪富华, 唐惠儒. 分析化学, 2011, 39(8): 1186-1194
16 FAN XuJia, BIAN YanHui, YIN HaiSong, QIAO ChangSheng. Biotechnology Bulletin, 2016, 32(6): 54-59
范栩嘉, 边艳慧, 殷海松, 乔长晟. 生物技术通报, 2016, 32(6): 54-59

