牛及其他各物种SLC11A1基因的结构多态与疾病相关性的研究进展
王利平 苑梦雅 刘相莹
摘要:SLC11A1基因与许多种胞内病原菌如沙门氏杆菌、结核分枝杆菌和利什曼原虫等病原菌的抗性和易感性有关,作为牛综合抗病的优良候选基因之一,它对于疾病的抵抗性是属于非病原特异性的。综述SLC11A1基因的结构特点、作用机制、表达调控,重点综述牛和其他不同物种SLC11A1基因结构多态与疾病的相关性,有望对牛的抗病育种提出理论依据。
关键词:SLC11A1基因;结构特征;作用机制;疾病抗性;易感性
中图分类号: S858.23 文献标志码: A 文章编号:1002-1302(2019)07-0001-04
抗病或感病是指动物在生长发育过程中受到各种病原物侵袭时的表现。随着分子生物学技术的发展,通过筛选鉴定抗病相关基因及其分子标记的方式,培育出抗病力较强的畜禽品种,从而达到提高当下畜牧生产水平的目的。溶质转运蛋白家族是一类膜转运蛋白,也是一类pH值依赖性二价阳离子反向转运蛋白,鉴于其亚细胞位于巨噬细胞静止晚期细胞内腔,因此会伴随着巨噬细胞吞噬病原体之后转移到吞噬体膜[1-3]。该家族包括SLC11A1和SLC11A2共2类[4]。SLC11A1基因比较保守,主要在免疫系统组织器的巨噬细胞、血液外周白细胞、嗜中性粒细胞以及脾脏、肺脏中表达,影响动物的天然免疫。最近,西北农林科技大学的张涌教授团队应用基因打靶编辑技术把SLC11A1基因插入牛的胎儿成纤维细胞,通过体细胞转移,最终得到对结核杆菌病具有较强抵抗力的转基因牛,又一次证明了SLC11A1基因在抗病育种中是一个良好的候选基因[5-6]。
1 溶质载体转运蛋白家族基因SLC11A1的发现
溶质载体转运蛋白家族11成员1(Solute carrier family 11 member 1:SLC11A1),又名天然抗性相关巨噬细胞蛋白1基因(natural resistance associated macrophage protein 1,NRAMP1),最初在小鼠中发现并被克隆。Ity、Lsh和Bcg[7]也是该基因的名称,来源于该基因能够控制早期感染的鼠伤寒沙门氏菌(Salmonella typhimurium)、杜氏利氏曼菌(Leishmaniadonovani)以及与之相关的各种分枝杆菌包括牛分枝杆菌(Mycobacterium bovis)、麻风分枝杆菌(Mycobacterium lepraemurium)。
2 溶质载体转运蛋白家族基因SLC11A1的功能
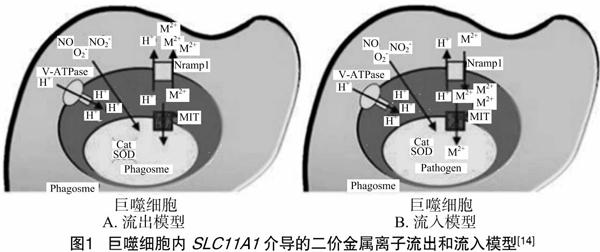
尽管关于SLC11A1基因作用机制的研究尚未有一致结论,但以下5个方面已经基本达成共识(图1):(1)SLC11A1基因把溶酶体的金属离子耗尽之后发挥作用,导致被吞噬的胞内病原体缺乏繁殖所必需的镁、铁和其他金属离子[8];(2)巨噬细胞中的“多向性效应”可在吞噬体单向转运二价金属离子时被激活,包括抗原递呈、蛋白激酶C产生、NO产生等功能的上调,其中脂多糖增多、IFN-γ(γ-干扰素)、TNF-α(肿瘤坏死因子-α)、IL-1β(白细胞介素1β)表达增强,皆是引起上调作用的刺激因子[9-10]。(3)细菌被吞噬细胞内吞后,Fe2+等金属离子作为辅助因子能够合成自身的防御酶系来抗衡内吞小体产生的活性氧或氮的中间产物NO和NO2等,这些金属离子可以被SLC11A1基因运出内吞小体造成细菌无法合成防御酶系,因此活性氧可以杀死细菌[11];(4)铁离子能通过SLC11A1基因转运到吞噬体,参与Fenton(芬顿)和Haber-Weiss(哈伯·韦斯)反应,产出大量活性离子杀灭微生物[12];(5)SLC11A1基因受到金属转运蛋白基因Fpn1、Dmt1和TfR的表达调控的影响,胞内金属离子的浓度降低,病原微生物的生长受到限制,有利于诱生型一氧化氮合酶(iNOS)的转录,产生更多的活性氧来发挥抗病作用[13-14]。
3 多物种SLC11A1基因多态对疾病易感性和抗性的关联
早期研究表明,SLC11A1基因具有控制鼠伤寒沙门氏菌[15]、杜氏利氏曼菌[16]以及各种分支杆菌包括麻风分支杆菌、牛分支杆菌早期感染的功能[17-18]。SLC11A1基因多态与抗病性在人及多种动物(猪、家禽等)上有大量报道,相对应的疾病有伤寒病、结核病、乳腺炎、布氏杆菌病、大肠杆菌病等[19-22]。
3.1 小鼠SLC11A1基因蛋白产物与抗病性的关联
在小鼠巨噬细胞中,SLC11A1基因表达通过含有LAMP1蛋白的溶酶体可被快迅速转移到含有活体细菌(如沙门氏菌等胞内病原微生物)的吞噬小体中发挥作用[23-24]。通过基因缺失、体外及转基因鼠体内等不同试验表明,SLC11A1基因与
小鼠对沙门氏伤寒菌、结核杆菌、利什曼菌、大肠杆菌等胞内病原微生物的抗性和易感性相关[8,24-25]。Vidal等在小鼠对病原微生物的抗性和易感性研究中发现,因为基因突变导致SLC11A1基因功能丧失的小鼠早期感染时表现为免疫力降低,在感染后期免疫功能得到恢复,这一现象证明了SLC11A1基因在巨噬细胞与病原微生物相互作用的早期免疫阶段发挥着重要作用[8]。
3.2 人SLC11A1基因变异与疾病的关联
与人结核病相关的SLC11A1基因的3个多态位点已被发现,分别为3′非编码区(3′UTR)的TGTG缺失、第543密码子位點的单碱基转换(D543N)、内含子4的G→C点突变。韩国肺结核患者与SLC11A1的3′UTR多态性显著相关[26]。SLC11A1内含子4的点突变和3′UTR区域多态性与人结核性胸膜炎紧密相关[27]。对我国汉族结核病人群的研究发现,痰涂片检测阳性的结核均与SLC11A1基因INT4和D543N位点多态性显著相关,杂合子个体较纯合子个体感染结核病的概率更高。由于汉族人3′UTR区域的TGTG缺失基因频率明显高于白种人,因此部分汉族人相比白种人更容易患结核病[28]。另有报道SLC11A1基因多态性与非结核性分枝杆菌肺病易感性存在相关性[29]。在我国北方汉族人肺结核成人患者的研究中也表明,D543N与3′UTR位点多态性可能是肺结核易感的原因[30-31]。人SLC11A1基因可能与鸟型分枝杆菌复合感染易感性有关,通过基因分型发现患者中D543N和3′UTR区域2个位点杂合子相对较多[32]。研究人的SLC11A1基因启动子区还发现了(GT)n微卫星的多态性位点,并认为它们可通过影响DNA的构象来影响基因的转录和表达水平[33]。
3.3 鸡SLC11A1基因多态与疾病的关联
Hu等对鸡SLC11A1基因与沙门氏伤寒菌感染的关联进行研究,SLC11A1基因在易感系鸡的编码区存在11处突变,其中有3处是有益突变,而位于第69位(Arg→G1n)的突变只在伤寒沙门氏菌感染易感鸡群中发现[34]。Girard-Santosuosso等对L2血清型的鸡(312只)和13周龄的商品鸡(373只)进行研究,将肠炎沙门氏菌静脉注射进这些鸡体内,试验人员在3 d后分别对生殖器官、肝脏和脾脏含沙门氏菌的数量进行分析,试验结果显示,商品代鸡SLC11A1基因的遗传多态性引起内脏组织对肠炎沙门氏菌的抵抗力有遗传上的差异[35]。Liu等在鸡SLC11A1基因组水平上发现了37处SNPs(单核苷酸多态),并在2种来航鸡的杂交后代公鸡中发现,SLC11A1基因的SNPs与接种鸡肠炎沙门氏(SE)疫苗后的抗体水平显著相关(P<0.05);在公鸡的后代中,SLC11A1基因的SNPs突变数量与脾脏的细菌附着量显著相关(P<005)。研究表明,SLC11A1基因高度保守区SNPs的多态与青年鸡接种SE疫苗及病原致病菌攻击后的免疫反应有相关性[36]。胡国顺等分析2个鸡种SLC11A1基因的SNPs与免疫性状的相关性,在如皋鸡和隐性白羽鸡SLC11A1基因的第9个外显子处发现了2处突变,在2个鸡品种中,经过免疫功能和基因型的关联分析,结果显示,BB型和AB型要显著高于AA型的H/L(异嗜性细胞与淋巴细胞比率),而AA型的淋巴细胞转化率和IgM含量显著高于BB型,说明该基因与免疫抗病性关联,可以作为抗病育种的一个优秀候选基因[37]。
3.4 猪SLC11A1基因多态与疾病的关联
吴宏梅等采用PCR-RFLP方法分析了松辽黑猪和大白猪中SLC11A1基因多态位点,发现所研究的2个品种猪SLC11A1基因位于第6内含子的NdeⅠ酶切片段多态变异和猪中性粒细胞还原力与单核细胞的细胞毒作用百分率之间显著相关[38]。刘艳冬等研究了香猪SLC11A1基因多态性与仔猪腹泻的关系,基因型与腹泻指数结果表明,不同品种猪表现出多态性不一,SLC11A1基因第6内含子区的变异影响免疫功能[39]。以上研究结果认为,猪的免疫功能受到SLC11A1基因遗传多态性的影响,在猪分子抗病育种方面能够起到一个主要候选基因的作用。
3.5 绵羊和山羊SLC11A1基因多态与疾病易感性和抗性的关联
Matthews等研究发现,绵羊SLC11A1基因多态性与伤寒沙门氏菌易感性和抗性有关[40]。Worley等研究山羊SLC11A1基因外显子10编码的蛋白质表明,在第7和第8个跨膜结构域之间有2个糖基化位点,其中1个糖基化位点在多数反刍动物中是高度保守的,由第10外显子的第16~24个碱基编码,另1个糖基化位点在山羊和绵羊中这部分序列相似,与牛、野牛及赤鹿都有很大差异,由第58~66个碱基编码。这些序列的保守性暗示了它在维持巨噬细胞和维护机体功能上的重要作用[41]。
3.6 狗SLC11A1基因与疾病易感性和抗性的关联
Altet等发现狗的SLC11A1基因序列与狗对利什曼原虫是否易感有关,发现的2处突变是在易感狗的SLC11A1基因序列中:外显子11处的完全缺失和启动子处富集G区[42]。Sanchez-Robert等对犬SLC11A1基因的研究发现,单倍型TAG-8-141与利什曼原虫易感性有关[43]。
4 牛SLC11A1基因的变异及其功能
4.1 牛SLC11A1基因的结构与变异
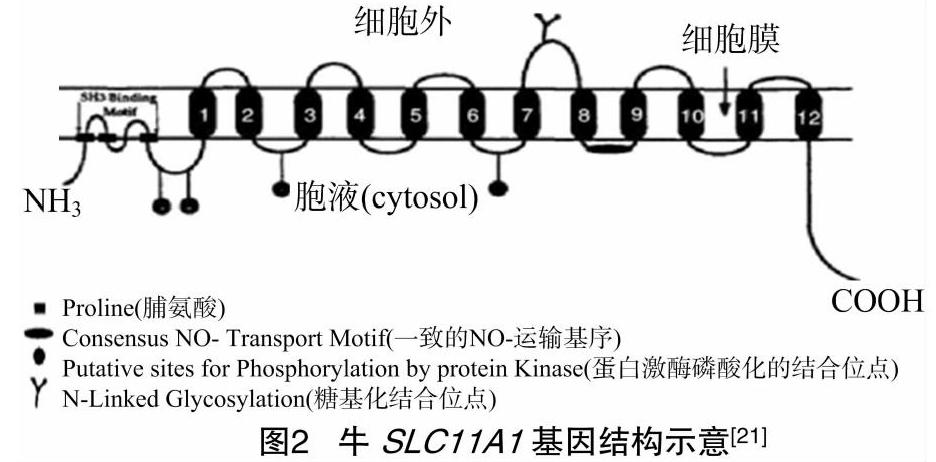
Feng等首次报道牛SLC11A1基因全长为10 925 bp,开放阅读框长1 647 bp,含有15个外显子,编码548个氨基酸;SLC11A1的氨基N端和羧基C端都位于细胞质内,在跨膜区(TMD)7和TMD8区域的细胞质外有1个糖基化位点,呈现环状结构(loop),在TMD8和TMD9之间细胞质内含有1个由20个氨基酸组成的离子转运基序(图2),在不同物种间这个离子转运基序高度保守[19]。SLC11A1蛋白N端含有多个磷酸化位点和SH3(Src基因同源性构域)结合位点。多态性研究发现,不同品种家养牛和水牛SLC11A1基因第4、5内含子和第5外显子分别有2、10、3个碱基点突变[21]。Xiao等报道,牛SLC11A1蛋白含有12个跨膜結构域[44]。
4.2 牛SLC11A1基因结构多态性与疾病的关联
对奶牛的研究发现,3′UTR有微卫星位点GT重复多态性,其中(GT)14纯合子、杂合子(GT)13/(GT)14、(GT)13/(GT)15基因型具有遗传易感性[45],且(GT)13纯合子基因型含有布氏杆菌遗传抗性。有研究对荷斯坦奶牛与瘤牛基因组进行比较表明,布鲁氏菌的抗性和敏感性受不同基因型影响差异显著,造成这些差异的原因是荷斯坦牛及瘤牛SLC11A1基因3′UTR区基因序列的遗传多态性[20]。有研究从感染牛结核分枝杆菌的牛群中取样,检测SLC11A1基因表达量,发现病畜SLC11A1基因表达量较健康牛表达量显著升高[46]。水牛SLC11A1基因3′UTR位点的多态性和布鲁氏菌易感性有关,在2007年被Capparelli等发现,并揭示出抗性型水牛中巨噬细胞和单核细胞SLC11A1的表达量显著高于敏感型水牛[47]。Ganguly等证实水牛SLC11A1基因3′UTR区GT重复多态与结核杆菌的抗性有关[48]。郭洋等研究了771头中国荷斯坦牛SLC11A1基因第10外显子、第9和第11内含子的多态性,发现不连锁的4个SNPs分别为内含子9的6067(A/G)、6358(C/T),外显子10的7155(A/G)和内含子11的7809(A/T);其中,6067(A/G)、6358(C/T)和7809(A/T)为新SNPs[49]。乳腺炎的发病率和不同基因型的关联性分析表明,SLC11A1基因的6358(C/T)和7155(A/G)对体细胞数和产奶量有显著影响(P<0.05),初步证明这2个基因型是对疾病抗性比较优良的基因型;单倍型分析结果表明,群体中共有16种单倍型的随机组合,CAAA单倍型组合被初步确定是较优良的单倍型,含有此单倍型的个体体细胞数低、产奶量高,可在奶牛乳腺炎的抗病育种筛选中作为候选基因标记[49]。Nam等应用定量PCR(RT-PCR)方法研究奶牛乳腺炎抗性与易感牛群外周血单核细胞中SLC11A1基因mRNA表达的不同,第1次报道出乳房炎抗性牛群SLC11A1基因mRNA的表达高于易感性牛群,乳腺炎高抗性牛可依据这种差异被筛选出来[50]。
Bagheri等研究了135头荷斯坦奶牛的SLC11A1基因结构和基因型的关系,发现该基因外显子11上有1个位點突变,这个点突变构成的3个基因型都与荷斯坦奶牛的乳腺炎发病率显著相关,此结果表明SLC11A1基因是一个与乳腺炎相关的抗病基因[51]。
Liu等在云南奶牛SLC11A1基因克隆及其抗结核的关联研究中确认了该基因编码蛋白抗结核的效果,通过克隆全部15个外显子和14个内含子,分析136头感染结核杆菌的荷斯坦牛和96头未感染结核杆菌牛SLC11A1基因的多态,研究表明,SLC11A1基因的多态变异和结核杆菌发病率显著相关[22]。
最近,西北农林科技大学的张涌教授团队应用基因打靶编辑技术把SLC11A1基因插入牛的胎儿成纤维细胞,通过体细胞转移,最终得到对结核杆菌病具有较强抵抗力的转基因牛[6],进一步表明SLC11A1基因是抗病育种研究中的一个重要候选基因。
5 展望
通过对小鼠、猪、鸡、人、牛的研究系统表明,溶质载体转运蛋白家族基因SLC11A1与结核杆菌、大肠杆菌等细菌抗性或易感性有关。SLC11A1基因比较保守,主要在肺脏、脾脏、血液外周白细胞等以及嗜中性粒细胞和巨噬细胞中表达,对动物的先天性免疫存在一定影响,对疾病的抗性具有非病原特异性,因此可以作为牛综合抗病的良好候选基因之一。
参考文献:
[1]Goswami T,Bhattacharjee A,Babal P,et al. Natural-resistance-associated macrophage protein 1 is an H1/bivalent cation antiporter[J]. Biochemical Journal,2001,354(3):511-519.
[2]Delaby C,Rondeau C,Pouzet C A,et al. Subcellular localization of Iron and Heme metabolism related proteins at early stages of erythrophagocytosis[J]. PLoS One,2012,7(7);e42119.
[3]Cellier M M. Developmental control of NRAMP1(SLC11A1)expression in professional phagocytes[J]. Biology,2017,6(2):28.
[4]Blackwell J M,Goswami T,Evans C A,et al. SLC11A1 (formerly NRAMP1) and disease resistance[J]. Cellular Microbiology,2001,3(12):773-784.
[5]Blackwell J M,Searle S,Mohamed H,et al. Divalent cation transport and susceptibility to infectious and autoimmune disease:continuation of the Ity/Lsh/Bcg/Nramp1/Slc11a1 gene story[J]. Immunology Letters,2003,85(2):197-203.
[6]Gao Y P,Wu H B,Wang Y S,et al. Single Cas9 nickase induced generation of NRAMP1 knockin cattle with reduced off-target effects[J]. Genome Biology,2017,18(1):13.
[7]Vidal S M,Malo D,Vogan K,et al. Natural resistance to infection with intracellular parasites:isolation of a candidate gene for Bcg[J]. Cell,1993,73(3):469-485.
[8]Vidal S M,Pinner E,Lepage P,et al. Natural resistance to intracellular infections:Nramp1 encodes a membrane phosphoglycoprotein absent in macrophages from susceptible (Nramp1 D169) mouse strains[J]. J Immunol,1996,157(8):3559-3568.
[9]Vidal S,Belouchi A M,Cellier M,et al. Cloning and characterization of a second human NRAMP gene on chromosome 12q13[J]. Mamm Genome,1995,6(4):224-230.
[10]Zhang G L,Wu H,Ross C R,et al. Cloning of porcine NRAMP1 and its induction by lipopolysaccharide,tumor necrosis factor alpha,and interleukin-1 beta:role of CD14 and mitogen-activated protein kinases[J]. Infection and Immunity,2000,68(3):1086-1093.
[11]De Voss J J,Rutter K,Schroeder B G,et al. Iron acquisition and metabolism by mycobacteria[J]. Journal of Bacteriology,1999,181(15):4443-4451.
[12]Skamene E,Schurr E,Gros P. Infection genomics:Nramp1 as a major determinant of natural resistance to intracellular infections[J]. Annu Rev Med,1998,49:275-287.
[13]Fritsche G,Nairz M,Theurl I,et al. Modulation of macrophage iron transport by Nramp1 (Slc11a1)[J]. Immunobiology,2008,212(9/10):751-757.
[14]Nevo Y,Nelson N. The NRAMP family of metal-ion transporters[J] Biochim Biophys Acta, 2006,1763(7):609-620.
[15]Lissner C R,Swanson R N,Obrien A D. Genetic control of the innate resistance of mice to Salmonella typhimurium:expression of the Ity gene in peritoneal and splenic macrophages isolated in vitro[J]. Journal of Immunology,1983,131(6):3006-3013.
[16]Crocker P R,Blackwell J M,Bradley D J. Expression of the natural resistance gene Lsh in resident liver macrophages[J]. Infect Immun,1984,43(3):1033-1040.
[17]Gros P,Skamene E,Forget A. Genetic control of natural resistanc Mycobacterium bovis in mice[J]. J Immunol,1981,127(6):2417-2421.
[18]Goto Y,Nakamura R M,Takahashi H,et al. Genetic control of resistance to Mycobacterium intracellular infection in mice[J]. Infect Immun,1984,46(1):135-140.
[19]Feng J,Li Y,Hashad M,et al. Bovine natural resistance associated macrophage protein1(Nrampl) gene[J]. Genome Res,1996,6(10):956-964.
[20]Paixo T A,Ferreira C,Borges A M,et al. Frequency of bovine Nramp1 (Slc11a1) alleles in Holstein and Zebu breeds[J]. Veterinary Immunology and Immunopathology,2006,109(1/2):37-42.
[21]Martínez R,Dunner S,Barrera G,et al.Novel variants whitin the coding region of Slc11A1 gene identified in Bos taurus and Bos indicus breeds[J]. J Anim Breed Genet,2008,125(1):57-62.
[22]Liu K H,Zhang B,Teng Z C,et al. Association between SLC11A1(NRAMP1) polymorphisms and susceptibility to tuberculosis in Chinese Holstein cattle[J]. Tuberculosis,2017,103:10-15.
[23]Gruenheid S,Pinner E,Desjardins M,et al. Natural resistance to infection with intracellular pathogens:the NRAMP1 protein is recruited to the membrane of the phagosome[J]. J Exp Med,1997,185(4):717-730.
[24]Govoni G,Gros P. Macrophage NRAMP1 and its role in resistance to microbial infections[J]. Inflammation Research,1998,47(7):277-284.
[25]Barton C H,Whitehead S H,Blackwell J M. Nramp transfection transfers Ity/Lsh/Bcg-related pleiotropic effects on macrophage activation:influence on oxidative burst and nitric oxide pathways[J]. Molecular Medicine,1995,1(3):267-279.

