棉铃虫Bt抗感品系的转录组差异表达分析
刘运泽 张丹丹 曲爱军 郑方强 萧玉涛
摘要:棉铃虫(Helicoverpa armigera)是全球性农业害虫,极大地影响作物产量。目前各地主要通过种植Bt作物进行防治,但近年来发现田间棉铃虫出现了Bt抗性。本研究对单个棉铃虫抗性品系与敏感品系进行转录组测序,并使用生物信息学方法分析得到差异表达基因。结果显示,Bt差异基因主要富集在氨肽酶、蛋白酶通路,可能对抗性产生影响。该研究为解释棉铃虫Bt抗性机理、挖掘新的抗性基因提供了重要依据。
关键词:棉铃虫;Bt抗性;转录组测序;差异基因
中图分类号:S435.622+.3:Q786文献标识号:A文章编号:1001-4942(2019)07-0005-05
Abstract Cotton bollworm (Helicoverpa armigera) is a global agricultural pest that greatly affects crop yield. At present, it is mainly controlled by planting Bt crop, but Bt resistance has emerged in recent years. In this study, one laboratorial cotton bollworm resistant strains and one sensitive strain were profiled using RNA-Seq technology. With the latest database and high-cited tools, the differentially expressed genes were screened out. The results showed that the Bt-responsive genes were mainly enriched in aminopeptidase and protease pathways which might affect the resistance. This study provided an important basis for explaining the mechanism of Bt resistance in cotton bollworm and mining new resistance genes.
Keywords Helicoverpa armigera; Bt resistance; RNA-Seq; Differentially expressed genes
棉铃虫(Helicoverpa armigera)是重要的多食性农业害虫,可以为害棉花、玉米、小麦、蔬菜、花卉等植物,严重影响农业生产[1]。为了有效减少棉铃虫危害,从1996年至今,全球商业化种植Bt棉总面积超过9.3×108 hm2[2]。害虫长期处于Bt毒素的高压选择下容易产生抗性,目前室内及田间均有棉铃虫Bt抗性的报道[3-5]。
近年来研究者们利用高通量转录组测序探索了昆虫差异基因、低表达基因、未知的编码与非编码RNA等[6,7]。对不同处理条件、不同发育阶段或不同种类的昆虫进行转录组测序,可以帮助研究者更好地了解转录本表达丰度、基因表达差异、功能基因信息、可变剪切位点等关键信息[8-11]。Zhao等利用Illumina/Solexa平台进行转录组测序找到了棉铃虫与免疫、解毒发育和代谢有关的1 022个未注释的差异基因,进行了免疫相关信号和调节途径研究[12]。Wei等研究了6个不同抗性倍数的棉铃虫品系的转录组数据,发现有9个胰蛋白相关基因出现明显下调,并且不同品系筛选的差异基因存在较大区别,推测不同品系抗性进化过程中可能产生不同的抗性机制[13]。Li 等通过 Solexa 对棉铃虫幼虫转录组进行测序,获得37 352 条 contigs,鉴定到数个可进行 RNA 干扰的备选抗性靶基因[14]。
棉铃虫等非模式昆虫,由于基因组缺失或注释不完善,目前主要采用从头拼接转录本的无参分析流程。本研究从生物信息分析角度出发,采用高质量分析软件与最新的数据库进行棉铃虫转录组分析,结合功能注释和富集分析筛选了可能与Bt抗性有关的差异表达基因,为后续的棉铃虫抗性分子生物学研究提供数据支持。
1 材料与方法
1.1 供试昆虫样品制备与测序
试验所用的敏感品系(susceptible strains,SS)棉铃虫于1996年取自中国农业科学院植物保护研究所河南新乡基地后,一直在植物保护研究所养虫间隔离饲养[4]; DU抗性品系是2017年6—9月在中国农业科学院植物保护研究所山东夏津试验基地利用3.6 g/L Cry1Ac毒素饲料筛选的高抗品系。饲养温度为(27±2)℃,相对湿度为75%±10%,光周期为 14L∶10D。
试验于2018年1月进行,首先将初孵幼虫接到24孔板中饲养,每孔4~6头,待幼虫长至4龄时(约孵化后第7天)移入指形管,待5龄时(约分装后第3天)取出,准备提取中肠组织。棉铃虫饲养期间均采用正常人工饲料,其配制方法见梁革梅等[15]的方法。
收集SS、DU品系棉铃虫的5龄幼虫活体,先将棉铃虫在冰上放置3 min,待其不活跃后,用大头针固定虫体,使用镊子、剪刀取出中肠组织。放入4℃预冷的0.7% NaCl溶液(约10 s)洗干凈内含物,接着用滤纸吸干水分,放入RNase-free EP管中,立刻液氮冷却,最后-80℃保存备用。每个品系3个生物学重复,每个重复15头棉铃虫。用Trizol试剂进行总RNA提取,RNA样品检测合格后交由北京诺禾致源科技股份有限公司进行测序。
1.2 转录本拼接
先利用FastQC、TrimGalore软件[16]对2个品系共6个样本的原始数据进行质量控制,保证不含有低质量碱基及测序接头,然后利用Trinity软件[17]对6个样本混合拼接,对拼接结果进行质量控制,为保证准确性同时采用了4种方式:计算N50值;回贴比对;利用BLAST[18]、HMMER[19]结合BUSCO[20]数据库分析组装结果中的同源基因;使用Diamond软件[21]比对非冗余蛋白数据库(Nr)。
1.3 转录本定量
采用不基于比对的Salmon软件[22]将2个品系的6个样本分别定量然后组合成一个表达矩阵。
1.4 组内样品重复性评估和组间聚类
对利用Salmon得到的表达矩阵基于R语言计算每组生物学重复之间的Pearson相关系数,并绘制热图比较组间与组内的生物学重复相关性。
1.5 表达矩阵差异分析及注释
利用edgeR[23]分析表达矩阵的原始统计值,利用calcNormFctors方法进行组间标准化,得到差异表达矩阵,接着利用其中的log2(fold change)列(以下简称logFC),将火山图代码中的logFC阈值设为mean(abs(logFC))+2×sd(abs(logFC)),即logFC绝对值的均值加两倍绝对值标准差,设置pvalue和qvalue阈值为0.05。计算DU-SS比较组的上调、下调差异基因。之后利用clusterProfiler的R包结合最新的GO、KEGG数据库进行功能注释和富集分析,结果中筛选出pvalue和qvalue小于0.05的GO条目与KEGG 通路进行统计和分析。
2 结果与分析
2.1 转录本拼接
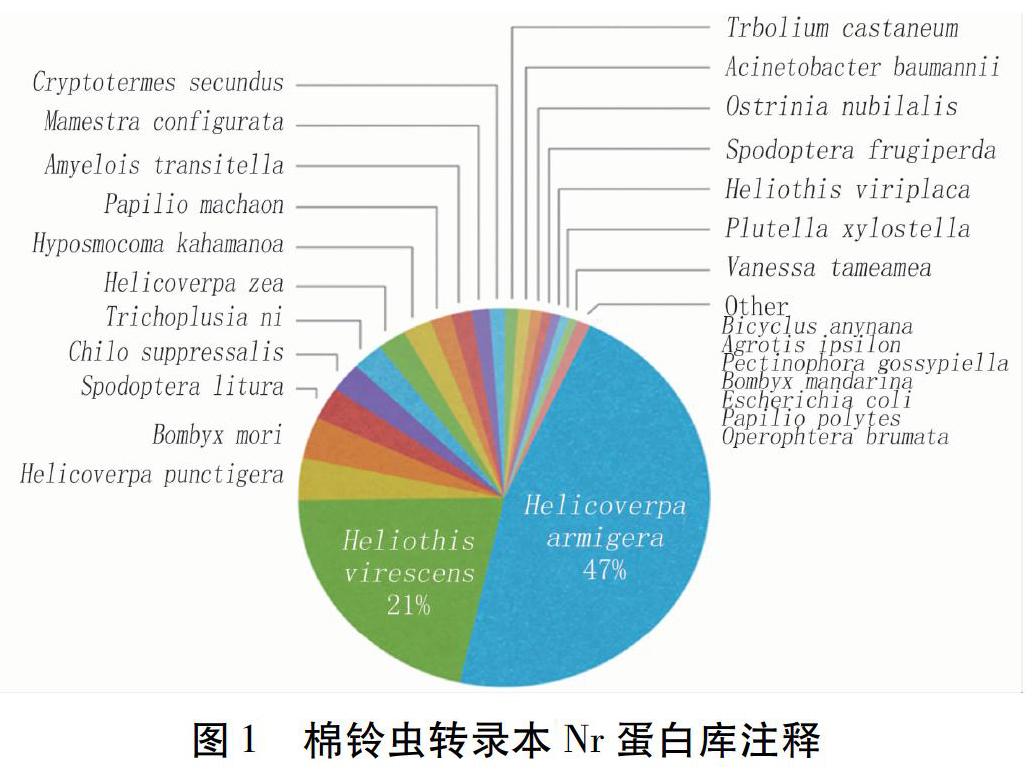
SS与DU品系的测序数据结果均得到2 000×104以上的reads,平均72×108 bp的转录组数据,过滤后碱基质量均达到Q30以上。对利用Trinity拼接得到的166 594條转录本进行质量检测,N50为1 036 bp。将SS与DU品系的测序回贴比对到拼接好的转录本,得到SS比对率为98.28%,DU比对率为98.35%,说明拼接得到的转录本中低表达、低质量或重复reads很少,拼接效果很好。BUSCO检测到昆虫保守基因的完整度为95%,一般认为这个值大于80%表示转录本拼接质量是可靠的。Nr数据库比对结果中,棉铃虫有47%的转录本序列比对到棉铃虫自身,其次与烟草夜蛾相似性达到了21%,另外还与家蚕、斜纹夜蛾、二化螟等鳞翅目昆虫序列较为相似(图1)。
2.2 组内样品重复性评估和组间聚类
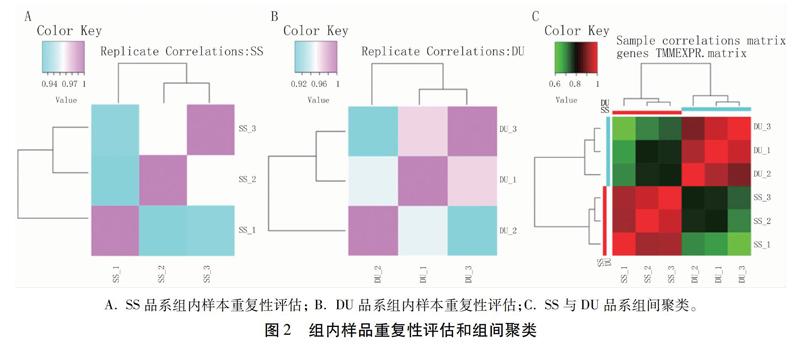
对得到的表达矩阵,计算每组3个生物学重复之间的相关性。一般而言,样本间重复的Pearson相关系数大于0.8即为存在相关性。结果显示SS与DU品系棉铃虫各自的生物学重复之间Pearson系数均在0.9附近,表示制备的生物学重复样本合格(图2A、B)。另外组间聚类显示SS与DU样本各自聚在一起,且表达差异明显,说明测序结果符合试验设计预期(图2C)。
2.3 转录本差异分析
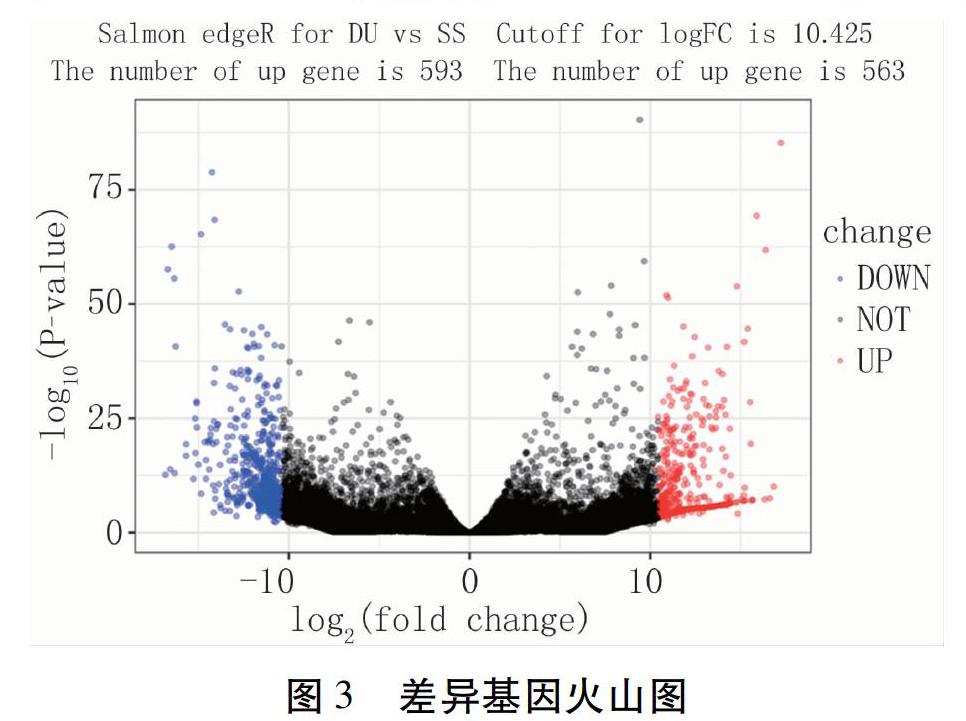
利用edgeR对Salmon表达矩阵进行差异分析,共得到1 156个差异基因,其中上调表达有593个,下调表达有563个(图3)。火山图中显示的上调及下调“离群点”表示DU-SS比较组中差异显著性更强的基因,这些差异极显著的基因更有可能导致抗性的产生。
2.4 差异基因功能注释与富集分析
DU品系与SS品系得到的1 156个差异表达基因中,有397个可以比对到Nr库并对应到RefSeq数据库。对其进行GO分析,发现了66个条目显著性富集。其中GO 生物学过程包括36个条目,细胞组分(cellular component,CC)包括10个条目,分子功能(molecular function,MF)包括20个条目。按照pvalue值从小到大挑选前20个条目进行可视化(图4A)。在分子功能这一类中,有5个基因(HaOG204148、 HaOG204182、LOC110378622、HaOG212290、HaOG216726)与氨肽酶活性有关(GO:0004177),氨肽酶如果与水解后的Bt毒素单体结合会导致中肠穿孔,因此氨肽酶活性可以作为判断抗性的指标,也说明本研究筛选得到的差异表达基因具有一定的可靠性。
另有12个基因(HaOG200516、HaOG200394、HaOG204148、HaOG204182、HaOG200489、HaOG200480、HaOG203137、HaOG215606、LOC110378622、HaOG212290、HaOG200476、HaOG216726)会影响蛋白酶活性(GO:0008233),蛋白酶的缺失会使Bt前毒素活化程度降低,导致棉铃虫对Bt毒素产生抗性。此外,有2个基因(HaOG216480、HaOG210130)响应抗菌免疫过程(GO:0002812),推测免疫反应与Bt抗性存在一定的联系。
KEGG结果同样选取pvalue前20个通路进行可视化(图4B)。其中有10个基因([JP3]HaOG212290、HaOG205566、HaOG215606、HaOG214145、 HaOG209294、HaOG208605、LOC110378622、HaOG200470、HaOG212255)在肽酶通路(ko01002)中发挥作用,支持了GO中因氨肽酶活性变化而产生抗性的推测。
3 讨论与结论
棉铃虫长期处于Bt毒蛋白的高压选择下,容易产生抗性,Bt抗性机理研究一直是防治棉铃虫的关键一步。中肠是昆虫食物消化与吸收的主要场所,也是Bt毒素发挥作用的重要组织。本研究利用以3.6 g/L的Bt毒饲料培养出的抗性品系,通过中肠转录组测序分析得到该抗性品系与敏感品系的差异基因。
本研究筛选到的棉铃虫抗感品系差异基因主要与氨肽酶、蛋白酶活性有关,参与了肽酶通路。前人研究证实Cry1Ac能与氨肽酶受体的N-乙酰氨基半乳糖(GalNAc)基团结合[24],并且氨肽酶的突变或表达量降低都会增加棉铃虫对Cry1Ac毒素的抗性[25]。中肠蛋白酶与Cry前体毒素活化关系密切。蛋白酶活性降低导致前毒素活化程度不足,不能产生足够的活化毒素,因此会产生抗性。已有研究发现烟芽夜蛾(Heliothis virescens)和欧洲玉米螟(Ostrinia nubilalis)的抗性品系中参与前毒素活化的中肠蛋白酶的活性下降[26,27]。棉铃虫中肠缺乏丝氨酸蛋白酶也会导致前毒素活化不足,从而产生Bt抗性[28]。另外有2个筛选到的基因可以影响抗菌免疫反应,前人在研究地中海粉螟(Ephestia kuehniella)时发现虫体内免疫反应会产生凝聚物,它可以与Cry1Ac结合,导致Cry1Ac与中肠受体蛋白结合减少[29],说明长期处于高浓度Bt毒素条件下的棉铃虫免疫反应较强。以上分析的差异基因未曾报道过,它们在棉铃虫抗性过程中的功能需要结合分子试验进一步验证。
本研究結果为进一步筛选与克隆关键Bt抗性基因提供了依据,也为探究棉铃虫Bt抗性机制奠定了基础。
参 考 文 献:
[1] 陆宴辉, 姜玉英, 刘杰, 等. 种植业结构调整增加棉铃虫的灾变风险[J]. 应用昆虫学报, 2018, 55(1): 19-24.
[2] Abbas M S T. Genetically engineered (modified) crops (Bacillus thuringiensis crops) and the world controversy on their safety[J]. Egyptian Journal of Biological Pest Control, 2018, 28(1): 28-52.
[3] Tabashnik B E, Wu K M, Wu Y D. Early detection of field-evolved resistance to Bt cotton in China: Cotton bollworm and pink bollworm[J]. Journal of Invertebrate Pathology, 2012, 110(3): 301-306.
[4] Guo Y Y, Li K K, Wu K M, et al. Changes of inheritance mode and fitness in Helicoverpa armigera(Hübner) (Lepidoptera∶Noctuidae) along with its resistance evolution to Cry1Ac toxin[J]. Journal of Invertebrate Pathology, 2007, 97(2): 142-149.
[5] Downes S, Mahon R. Successes and challenges of managing resistance in Helicoverpa armigera to Bt cotton in Australia[J]. GM Crops and Food, 2012, 3(3): 228-234.
[6] Qazi S S, Mittapalli O, Beliveau C, et al. Identification of odor-processing genes in the emerald ash borer,Agrilus planipennis[J]. PLoS One, 2013, 8(2): e56555.
[7] Legeai F, Derrien T. Identification of long non-coding RNAs in insects genomes[J]. Current Opinion in Insect Science, 2015, 7: 37-44.
[8] Li J, Zhu L, Hull J J, et al. Transcriptome analysis reveals a comprehensive insect resistance response mechanism in cotton to infestation by the phloem feeding insect Bemisia tabaci(whitefly)[J]. Plant Biotechnology Journal, 2016, 14(10): 1956-1975.
[9] Chang J C, Ramasamy S. Transcriptome analysis in the beet webworm, Spoladea recurvalis(Lepidoptera∶Crambidae)[J]. Insect Science, 2018, 25(1): 33-44.
[10]Ross H A, Buckley T R, Dennis A B, et al. De Novo Transcriptome analysis of the common new Zealand stick insect Clitarchus hookeri(Phasmatodea) reveals genes involved in olfaction, digestion and sexual reproduction[J]. PLoS One, 2016, 11(6): e0157783.
[11]Katsuma S, Kawamoto M, Shoji K, et al. Transcriptome profiling reveals infection strategy of an insect maculavirus[J]. DNA Research, 2018, 25(3): 277-286.
[12]Zhao Z, Wu G, Wang J, et al. Next-generation sequencing-based transcriptome analysis of Helicoverpa armigera larvae immune-primed with Photorhabdus luminescens TT01[J]. PLoS One, 2013, 8(11): e80146.
[13]Wei J, An S, Yang S, et al. Transcriptomice responses to different Cry1Ac selection stresses in Helicoverpa armigera[J]. Frontiers in Physiology, 2018, 9: 1653-1670.
[14]Li J, Li X, Chen Y, et al. Solexa sequencing based transcriptome analysis of Helicoverpa armigera larvae[J]. Molecular Biology Reports, 2012, 39(12): 11051-11059.
[15]梁革梅,譚维嘉,郭予元. 人工饲养棉铃虫技术的改进[J].植物保护,1999(2):16-18.
[16]Marcel M. Cutadapt removes adapter sequences from high-throughput sequencing reads[J]. EMBnet. Journal, 2011, 17(1): 10-12.
[17]Grabherr, Manfred G, Brian J,et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome [J]. Nature Biotechnology, 2011, 29(7): 644-652.
[18]Madden T L, McGinnis S. BLAST: at the core of a powerful and diverse set of sequence analysis tools[J]. Nucleic Acids Research, 2004, 32: 20-25.
[19]Johnson L S, Eddy R, Portugaly E. Hidden Markov model speed heuristic and iterative HMM search procedure[J]. BMC Bioinformatics, 2010, 11: 431-439.
[20]Simo F A,Waterhouse R M,Ioannidis P,et al.BUSCO:assessing genome assembly and annotation completeness with single-copy orthologs[J]. Bioinformatics, 2015, 31(19): 3210-3212.
[21]Buchfink B, Xie C, Huson H. Fast and sensitive protein alignment using DIAMOND[J]. Nature Methods, 2014, 12(1): 59-60.
[22]Patro R, Duggal G, Love M I, et al. Salmon provides fast and bias-aware quantification of transcript expression[J]. Nature Methods, 2017, 14(4): 417-419.
[23]Robinson M D, McCarthy D J, Smyth G K. edgeR:a Bioconductor package for differential expression analysis of digital gene expression data[J]. Bioinformatics, 2009, 26(1): 139-140.
[24]Sarkar A, Hess D, Mondal H A, et al. Homodimeric alkaline phosphatase located at Helicoverpa armigera Midgut, a putat ive receptor of Cry1Ac contains α-GaINAc in term inal glycan structure as interactive Epitope[J]. Journal of Proteome Research, 2009, 8(4): 1838-1848.
[25]Liang G, Wang G, Wu K, et al. Mutation of an aminopeptidase N gene is associated with Helicoverpa armigera resistance to Bacillus thuringiensis Cry1Ac toxin[J]. Insect Biochemistry and Molecular Biology, 2009, 39(7): 421-429.
[26]Oppert B,Higgins R A,Huang F,et al.Comparative analysis of proteinase activities of Bacillus thuringiensis-resistant and -susceptible Ostrinia nubilalis(Lepidoptera∶Crambidae)[J]. Insect Biochemistry and Molecular Biology, 2004, 34(8):753-762.
[27]Huang F, Oppert B, Higgins R A, et al. Susceptibility of Dipel-resistant and -susceptible Ostrinia nubilalis(Lepidoptera∶Crambidae) to individual Bacillus thuringiensis protoxins[J]. Journal of Economic Entomology, 2005, 98(4): 1333-1340.
[28]Liu C X, Xiao Y T, Li X C, et al. Cis-mediated down-regulation of a trypsin gene associated with Bt resistance in cotton bollworm[J]. Scientific Reports, 2014, 4(1): 7219-7225.
[29]Mahbubur R M, Roberts H S, Schmidt O. Tolerance to Bacillus thuringiensis endotoxin in immune-suppressed larvae of the flour moth Ephestia kuehniella[J]. Journal of Invertebrate Pathology, 2007, 96(2):125-132.

