青篱柴转录组数据SSR位点的生物信息学分析
黄建敏 李菲 刘云静

摘要:为探讨青篱柴转录组中简单重复序列(simple sequence repeat,简称SSR)位点信息,开发青篱柴分子标记技术,采用高通量测序技术获得青篱柴转录组原始试验数据,经过生物信息学软件Trinity拼接后,获得66 755条单基因簇(universal gene,简称unigene)。进一步采用生物信息学分析软件MISA对所有的unigene进行SSR位点数据挖掘。共计搜索出22 542个SSR位点,分布在18 972条unigene中,出现频率为33.77%,发生频率为28.42%,SSR平均长度为15 bp,平均分布频率是1/3.98 kb,在青篱柴转录组的SSR中,单核苷酸、三核苷酸和二核苷酸重复序列为主要重复类型,分别占总SSR的47.59%、27.95%、23.45%。青篱柴转录组数据中SSR序列共包括161种重复基元类型。其中出现频率高的基序主要有A/T、AG/CT、AAG/CTT、AT/AT、C/G、AGG/CTT。重复序列长度在10~144 bp之间,大多小于24 bp,大于24 bp的仅有0.22%(48个SSR位点)。另外,设计筛选得到15 425对SSR引物。综上表明青篱柴SSR位点多态性潜能较高,具有很大的可开发性。
关键词:青篱柴;转录组;SSR;重复类型;重复基元
中图分类号:Q811.4 文献标志码: A 文章编号:1002-1302(2019)02-0054-05
简单重复序列(simple sequence repeat,简称SSR)是指由1~6个核苷酸为重复单位组成的串联重复序列,随机、广泛、均匀地分布于真核生物基因组中,由于重复次数不同而造成简单序列长度多态性(simple sequence length polymorphism,简称SSLP)[1]。不同物种的简单重复序列差异很大,主要表现在重复次数、基序类型、长度以及在染色体上的分布情况等,从而反映出物种间高度的等位基因多样性。虽然SSR具有高度多态性,但其两端的序列一般都比较保守和专一,因此可凭借两端的保守序列设计特异性的SSR引物,通过PCR技术扩增出SSR,再利用琼脂糖凝胶电泳或聚丙烯酰胺凝胶电泳技术获得其长度多态性,即SSR分子标记。由此开发的SSR分子标记技术具有操作简单、稳定性好、分布丰富、易检测、共显性、等位基因变异多、遵循孟德尔遗传定律、进化所受选择压力小以及种族特异性强等优点。目前,SSR分子标记作为重要的遗传标记,已经在品种纯度鉴定、基因功能定位、分子辅助育种、遗传图谱构建以及亲缘关系分析等方面有广泛的应用[2-3]。
早期开发SSR分子标记的方法主要是基因组文库构建、克隆测序和通过公共数据库中表达序列标签(expressed sequence tag,简称EST)进行开发。所需费用高、时间周期长、步骤复杂、工作量大,且成功获得SSR阳性克隆和多态性引物的概率很低[4]。鉴于当前新一代高通量测序技术发展迅速,测序成本显著降低的状况。利用高通量测序技术对植物进行转录组测序,具有快速高效、花费时间合理等优点,从基因的结构和功能出发,通过新一代高通量测序技术,除了可以全面快速地获得某一特定组织或器官在特定状态下几乎所有的转录本信息,能可靠地发现大量特定组织在特定时间点的基因表达数据之外[5],还可根据测序结果开发具有基因信息的SSR分子标记[6-8],运用该方法,已在刺梨[9]、党参[10]、茯苓[11]、鱼腥草[12]、杜仲[13]等多种植物上开发出具有基因信息的SSR标记并应用于遗传分析。
青篱柴(Tirpitzia sinensis)为亚麻科青篱柴属下的一个种,属灌木或小乔木。其茎、叶能消肿止痛、接骨,在民间有用药历史,但尚未见有关药学研究报道,从民族植物学的角度是值得关注的物种。青篱柴花具有很好的观赏价值,可以对其进行引种栽培。蔡湘文在黑叶猴的觅食生物学和营养分析的研究中提到,青篱柴的叶是黑叶猴喜食的种类之一[14]。杜小浪等在重庆市彭水县进行第4次全国中药资源普查中发现了青篱柴分布新纪录,青篱柴的原分布区域位于重庆市的南面,重庆市可能是青篱柴分布的最北界,这对于植物谱系地理研究具有重要意义[15]。喻勋林等在湖南省都庞岭自然保护区植物区系的研究中将青篱柴归类为石灰岩特有(或适生)植物[16]。蒋水元等在对广西壮族自治区石灰岩特有珍惜药用植物类群的基本特征研究中,在藥用植物的分布区类型构成中,将青篱柴归为热带亚洲(印度-马来西亚)类[17]。国内外暂无单独对青篱柴的研究报道。针对青篱柴研究的空白,开展青篱柴的遗传多样性研究,揭示青篱柴资源的亲缘关系,探究其在不同地理分布区的遗传差异,揭示其物种分化过程,能为青篱柴种质资源的保护和合理利用提供理论依据。本研究首次采用二代测序技术的方法,获得大量青篱柴转录组序列信息,并对转录组数据中的SSR位点进行搜索和检测,分析其分布及结构特点,并结合SSR引物设计综合评价其可用性,以期为在分子水平上研究青篱柴遗传多样性提供基础数据。
1 材料与方法
1.1 青篱柴材料及转录组数据来源
本试验转录组测序样本来源于贵州省黔东南苗族侗族自治州施秉县云台山,采取同一株青篱柴个体上的幼嫩叶片、花和枝干组织各1.5 g,进行等量混样,迅速放入液氮,送北京诺禾致源科技股份有限公司提取mRNA,利用Illumina HiSeqTM 2000高通量测序平台对其进行转录组测序,总计产出8.71 GB数据,干净读序经Trinity组装后最终获得66 755条unigene,总长度为 89 702 690 bp,平均长度为1 344 bp,过滤后质量不低于20的碱基比例(Q20,测序错误率<1%)为 97.47%,表明测序结果良好,可对获得的数据进行进一步分析。
1.2 青篱柴转录组SSR的筛选
以组装出来的unigene作为参考序列,使用MicroSAtellite(简称MISA)软件(http://pgrc.ipk-gatersleben.de/misa/misa.html)对青篱柴转录组中的SSR位点进行搜索和检测。对应的各个核苷酸的最少重复次数分别为1-10、2-6、3-5、4-5、5-5、6-5(如1-10意思为以单核苷酸为重复单位时,其重复数至少为10才可以被检测到;2-6意思为以二核苷酸为重复单位时,其最少重复数为6)。并且对不同SSR类型在基因转录本的密度分布进行统计。同时筛选被间隔≤100 bp 碱基打断的复合型SSR。
1.3 青篱柴SSR引物设计
采用Primer 3.0(2.3.5版,默认参数)进行SSR引物设计,并针对预测到的每一个SSR位点分别设计3组引物供后期试验选择。
1.4 数据统计
将生成的文本文件导入到Excel中进行基本的统计分析。用SSR出现频率、发生频率及其分布的平均距离、重复类型、基元组成等分析青篱柴转录组SSR的分布特征,其中出现频率=搜索到的SSR总数/总unigene序列数;发生频 率= 有SSR分布的序列数/总unigene序列数;SSR分布的平均距离=序列总长度/搜索到的SSR数量。
2 结果与分析
2.1 青篱柴SSR数量与分布
由表1可知,利用MISA软件对青篱柴转录组测序获得的66 755条unigene进行检索,共找到符合条件的SSR位点有22 542个,分布于18 972条unigene中。SSR发生频率为28.42%,出现频率为33.77%。其中含单个SSR位点的unigene有14 071条,含2个及2个以上SSR位点的unigene有4 901条。复合型SSR数量为1 573个。
由表2可知,青篱柴转录组SSR位点的种类非常丰富,从单碱基到六碱基重复均有分布,对1~6 bp的SSR重复基元类型进行统计可以看出,每个重复类型所含的数量相差很大。主要重复类型是单核苷酸,占SSR总数的47.59%,其次为三核苷酸和二核苷酸,分别占27.95%、23.45%,四核苷酸、五核苷酸、六核苷酸重复的数量极少,总计占1.01%,但类型很丰富,其中四核苷酸重复基序数量高于五核苷酸、六核苷酸。从分布情况看,相应的不同重复基序长度SSR分布的平均距离差距很大,其中单核苷酸重复最多,每条SSR分布平均距离为8.36 kb,其次为三核苷酸重复,每条SSR分布的平均距离为14.23 kb,六核苷酸重復最少,每条SSR分布的平均距离为6 900.21 kb,整体上青篱柴转录组中平均每 3 979 bp 就含有1个SSR位点。
2.2 青篱柴转录组SSR的特性
青篱柴转录组SSR位点的序列总长高达330 350 bp,平均每个SSR位点长度为15 bp,其中1~6 bp各核苷酸重复基元类型的SSR位点平均长度依次分别为13、16、17、21、25、36 bp。青篱柴转录组SSR位点的重复次数分布在5~24次之间,其中重复次数以10次最多,有5 013个,占总SSR数量的22.24%,其后依次是5、6、11、7次,分别占总SSR数量的16.71%、14.29%、9.54%、7.99%。不同类型SSR重复单元的重复次数分布存在差异,单核苷酸重复以10次重复为主;二核苷酸重复以6次重复为主;三核苷酸、四核苷酸、五核苷酸、六核苷酸重复均以5次重复为主。不同类型SSR重复单元均随着重复次数的增加,位点数量呈递减的趋势。另外,随着青篱柴转录组SSR位点的重复次数增多,单核苷酸重复类型所占比例越大。其中,重复次数在10次以上的SSR位点中,单核苷酸重复类型的SSR位点所占比例高达96.23%(图1)。
在青篱柴转录组中的SSR位点长度分布于10~144 bp之间,长度为10~24 bp的重复序列最多,占99.78%;其次是25~42 bp的序列重复, 占0.19%; 大于43 bp的重复序列仅占0.03%(图2)。
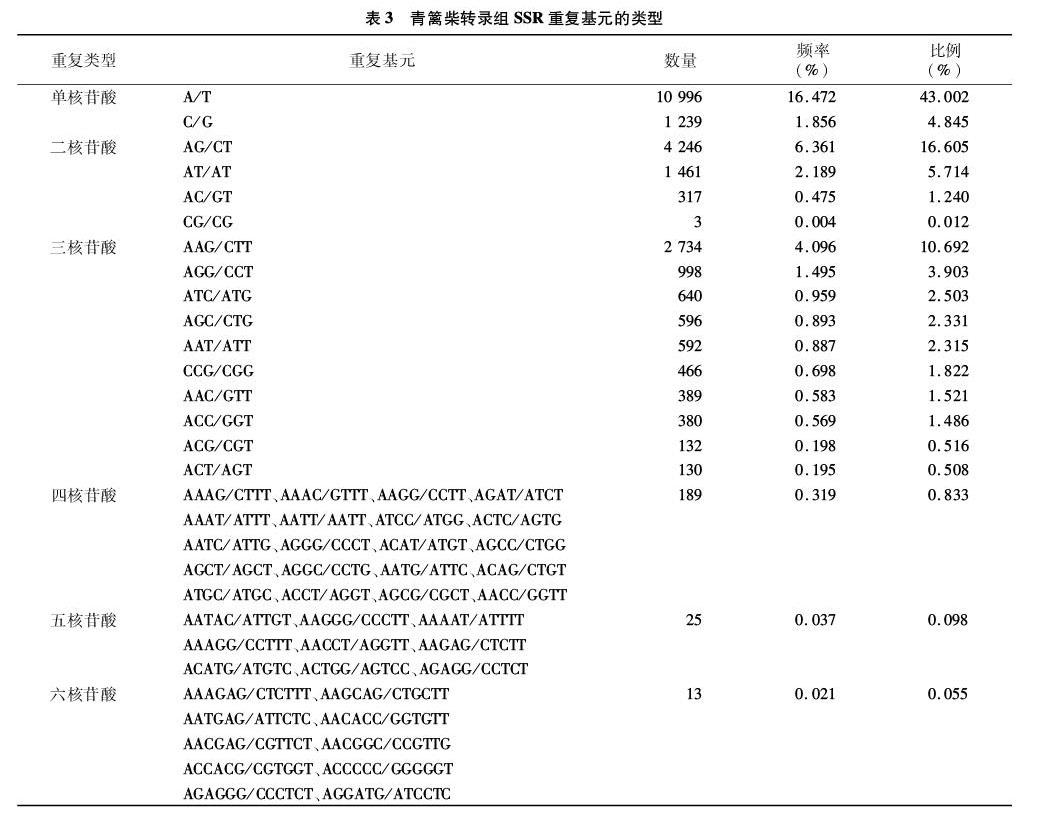
青篱柴转录组中共含有161种不同基序序列类型的SSR位点,从单核苷酸重复到六核苷酸重复依次有4、12、60、64、9、12种类型。从SSR位点重复单元出现频率(表3)来看,单核苷酸重复中A/T重复基序类型占绝对的优势,占总SSR位点数量的43.002%,二核苷酸重复中以AG/CT和AT/AT重复基序为主,分别占总SSR位点数量的16.605%、5.714%。在三核苷酸重复单元中以AAG/CTT基序类型最多,占总SSR位点数量的 10.692%,另外,四核苷酸、五核苷酸、六核苷酸重复基元类型因分布很少,包含的各重复基序类型出现频率较低。
2.3 青篱柴转录组SSR引物设计
采用Primer 3.0引物批量设计程序对22 542个SSR位点的两端序列进行引物设计,15 425条序列引物设计成功,成功率为68.43%。在设计成功的15 425对引物中,扩增产物为单碱基重复的最多,有7 482个,占48.51%;其次为三碱基和二碱基重复,分别占27.36%、17.73%。另外,PCR产物为复合型重复(含有一个以上SSR位点重复基元)的占5.34%(表4)。
2.3 青篱柴转录组SSR的可用性评价
鉴定分子标记可用性能的重要参考指标之一是其多态性,通常SSR长度是影响其多态性高低的重要因素,Temnykh等研究表明,SSR长度大于20 bp时具有高度多态性,长度在12~20 bp之间的SSR具有中度多态性,而长度在12 bp以下时多态性极低[18]。因此本研究对青篱柴转录组SSR位点进行搜索的筛选标准为单核苷酸重复至少10次,二核苷酸重复至少6次,三至六核苷酸重复至少5次。经统计发现,青篱柴转录组SSR的长度大多集中在12~45 bp之间,其中长度大于20 bp具有高度多态性的SSR共有1 739个,占总SSR的7.71%;长度在12~20 bp之间具有中度多态性的SSR共有14 756个,占总SSR的65.46%。另外,根据Zhang等的研究表明,高级重复单元类型的SSR多态性普遍比低级重复单元类型的低[19]。本试验获取的SSR主要是低级重复单元,如单核苷酸、三核苷酸、二核苷酸重复所占比例为87.27%,对青篱柴转录组SSR进行统计分析发现,长度大于20 bp的SSR全部属于单核苷酸重复,共包含508个SSR位点,表明这部分青篱柴SSR具有高度多态性潜能,可以预计在青篱柴基因功能表达差异研究方面具有很好的利用价值。
3 討论与结论
目前高通量测序技术发展迅速、测序成本显著降低,因此产生了大量的转录组测序数据,尤其是对于未知基因组序列的物种,其在转录组学中的研究越来越广泛。基于转录组测序的SSR分子标记因其具有重复性好、等位差异多、多态性十分丰富、覆盖面广等优点,目前已广泛应用于多种植物的品种选育和遗传多样性研究中。SSR广泛分布于各种真核生物的基因组中,大约每隔10~50 kb就存在1个SSR。在植物中,平均23.3 kb就有1个SSR[20]。
本研究首次通过高通量测序技术得到青篱柴转录组数据信息,经过组装与定位得到总计66 755条unigene,搜索到符合条件的SSR位点共计 22 542个,分布于18 972条unigene中,平均分布频率为 1/3.98 kb,明显高于云南松、菠菜、南方红豆杉、灯盏花、洋葱、藏茵陈、川西獐牙菜等[21-26],略低于刺梨、蓝靛果忍冬、菜薹等[27-28]。SSR位点出现频率为33.77%,明显高于红松、鱼腥草、印度南瓜、党参、金钗石斛、莕菜、野三七、茄子等,低于杜仲、芙蓉李等[29-35]。造成不同物种SSR位点的出现频率和平均分布频率差异的原因可能与转录组数据中unigene数量及长度、SSR筛选的条件以及物种本身基因组差异等有关[36]。综上说明青篱柴转录组中SSR重复位点数量相对而言比较丰富,可为SSR的开发提供重要的资源库。
不同物种之间SSR主要重复类型有所差异,很多植物的SSR主要以二核苷酸、三核苷酸重复单元类型为主。本研究发现,青篱柴转录组SSR重复基元类型主要以单核苷酸为主,占全部SSR的47.59%,其次是三核苷酸,占全部SSR的27.95%。这与红松、菜薹、茄子等相似,但与云南松、鱼腥草、洋葱、藏茵陈、刺梨等物种不同,这些植物中SSR的主要类型是三核苷酸重复。有研究表明,SSR的多态性与不同基序序列类型有一定关联,SSR的重复基序类型中普遍存在A/T优势。G/C重复基序类型在多数植物中很难见到,出现频率都很低。原因可能是打破A/T碱基对之间的氢键所需的能量低于G/C碱基对之间的氢键,A/T的波动较G/C容易[36]。但也有观点认为,由于甲基化的C残基转化为T及位于3′末端的polyA序列插入基因组后形成富含A的原始SSR,导致重复基序中A/T优势的出现[37],具体原因有待进一步研究分析。本研究也发现,青篱柴转录组SSR重复基序类型出现最多的是A/T,所占比例为43.002%。其次,AT/AT和AAT/ATT重复基序类型出现次数仍然很高,所占比例分别为5.714%、2.315%,表现出较明显的A/T优势。而G/C在所有重复基序类型中的出现频率极低,C/G和CG/CG在单核苷酸和二核苷酸中所占的比例分别仅为4.845%、0.012%,这很好地验证了上述观点。总的来讲,青篱柴转录组SSR不仅出现频率高、平均分布频率广,且类型丰富,具有较高的多态性潜能和可用性。本研究明确了青篱柴转录组SSR位点的基本特征,其结果可为开发青篱柴SSR分子标记奠定基础。与此同时,伴随着青篱柴SSR分子标记的开发和利用,对于丰富青篱柴分子标记类型,加快青篱柴功能基因资源的开发利用,建立青篱柴种质资源评价和改良机制,对青篱柴进行遗传结构、遗传分化、居群动态和遗传多样性分析等具有重要意义。
参考文献:
[1]Senan S,Kizhakayil D,Sasikumar B,et al. Methods for development of microsatellite markers:an overview[J]. Notulae Scientia Biologicae,2014,6(1):1-13.
[2]Sharma R,Maloo S R,Choudhary S,et al. Microsatellite markers:an important DNA fingerprinting tool for characterization of crop plants[J]. The Journal of Plant Science Research,2015,31(1):83.
[3]Powell W,Machray G C,Provan J. Polymorphism revealed by simple sequence repeat[J]. Trends in Plant Science,1996,1(7):215-222.
[4]Jacobsen B H,Hansen M M,Loeschcke V. Microsatellite DNA analysis of northern pike (Esox lucius L.) populations:insights into the genetic structure and demographic history of a genetically depauperate species[J]. Biological Journal of the Linnean Society,2005,84(1):91-101.
[5]Aldrich P R,Hamrick J L,Chavarriaga P,et al. Microsatellite analysis of demographic genetic structure in fragmented populations of the tropical tree Symphonia globulifera[J]. Molecular Ecology,1998,7(8):933-944.
[6]Margulies M,Egholm M,Altman W E,et al. Genome sequencing in microfabricated high-density picolitre reactors[J]. Nature,2005,437(757):376-380.
[7]Zialor C. DNA sequencing with chain terminating inhibitors[J]. Proceedings of the National Academy of Sciences of the United States of America,2014,5(1233):751-780.
[8]Nagalakshmi U,Wang Z,Waern K,et al. The transcriptional landscape of the yeast genome defined by RNA sequencing[J]. Science,2008,320(5881):1344-1349.
[9]鄢秀芹,鲁 敏,安华明. 刺梨转录组SSR信息分析及其分子标记开发[J]. 园艺学报,2015,42(2):341-349.
[10]王 东,曹玲亚,高建平.党参转录组中SSR位点信息分析[J]. 中草药,2014,45(16):2390-2394.
[11]何 海,郭继云,马毅平,等. 茯苓转录组SSR序列特征及其基因功能分析[J]. 中草药,2015,46(23):3558-3563.
[12]黎晓英,刘胜贵,王 丹,等. 鱼腥草转录组SSR位点信息分析及其多态性研究[J]. 中草药,2016,47(10):1762-1767.
[13]黄海燕,杜红岩,乌云塔娜,等. 基于杜仲转录组序列的SSR分子标记的开发[J]. 林业科学,2013,49(5):176-181.
[14]蔡湘文. 黑叶猴的觅食生物学和营养分析[D]. 桂林:广西师范大学,2004.
[15]杜小浪,慕泽泾,肖 忠,等. 重庆药用植物新记录[J]. 中国现代中药,2014,16(6):442-443,450.
[16]喻勋林,薛生国. 湖南都庞岭自然保护区植物区系的研究[J]. 中南林学院学报,1999,19(1):29-34.
[17]蒋水元,赵瑞峰,李 锋,等. 广西石灰岩特有珍稀药用植物类群的基本特征研究[J]. 广西农业生物科学,2008,27(4):381-387.
[18]Temnykh S,Park W D,Ayres N,et al. Mapping and genome organization of microsatellite sequences in rice (Oryza sativa L.)[J]. Theoretical and Applied Genetics,2000,100(5):697-712.
[19]Zhang P,Dreisigacker S,Melchinger A E,et al. Quantifying novel sequence variation in CIMMYT synthetic hexaploid wheats and their backcross-derived lines using SSR markers[J]. Molecular Breeding,2005,15(1):1-10.
[20]Marathi B,Guleria S,Singh N K,et al. Molecular diversity and segregation distortion measured by SSR markers in a new plant type based recombinant inbred line population of rice[J]. Indian Journal of Genetics and Plant Breeding,2011,71(4):297-303.
[21]蔡年辉,许玉兰,徐 杨,等. 云南松转录组SSR的分布及其序列特征[J]. 云南大学学报(自然科学版),2015,37(5):770-778.
[22]潜宗伟,陈海丽,崔彦玲. 菠菜转录组SSR位点分析及其分子标记的开发[J]. 农业生物技术学报,2016,24(11):1688-1697.
[23]李炎林,楊星星,张家银,等. 南方红豆杉转录组SSR挖掘及分子标记的研究[J]. 园艺学报,2014,41(4):735-745.
[24]陈 茵,李翠婷,姜倪皓,等. 灯盏花转录组中SSR位点信息分析及其多态性研究[J]. 中国中药杂志,2014,39(7):1220-1224.
[25]李满堂,张仕林,邓 鹏,等. 洋葱转录组SSR信息分析及其多态性研究[J]. 园艺学报,2015,42(6):1103-1111.
[26]刘 越,岳春江,王 翊,等. 藏茵陈川西獐牙菜转录组SSR信息分析[J]. 中国中药杂志,2015,40(11):2068-2076.
[27]张庆田,李晓艳,杨义明,等. 蓝靛果忍冬转录组SSR信息分析及其分子标记开发[J]. 园艺学报,2016,43(3):557-563.
[28]李荣华,王直亮,陈静芳,等. 菜薹转录组中SSR信息与可用性分析[J]. 园艺学报,2016,43(9):1816-1824.
[29]张 振,张含国,莫 迟,等. 红松转录组SSR分析及EST-SSR标记开发[J]. 林业科学,2015,51(8):114-120.
[30]王洋洋,单文琪,徐文龙,等. 印度南瓜转录组SSR信息分析及其多态性研究[J]. 园艺学报,2016,43(3):578-586.
[31]李 清,李 标,郭顺星 .金钗石斛转录组SSR位点信息分析[J]. 中国中药杂志,2017,42(1):63-69.
[32]袁阳阳,王青锋,陈进明.基于转录组测序信息的水生植物莕菜SSR标记开发[J]. 植物科学学报,2013,31(5):485-492.
[33]李翠婷,张广辉,马春花,等. 野三七转录组中SSR位点信息分析及其多态性研究[J]. 中草药,2014,45(10):1468-1472.
[34]魏明明,陈钰辉,刘富中,等. 基于转录组测序的茄子SSR标记开发[J]. 植物遗传资源学报,2016,17(6):1082-1091.
[35]方智振,叶新福,周丹蓉,等. ‘芙蓉李转录组SSR信息分析与分子标记开发[J]. 果树学报,2016(4):416-424.
[36]Biswas M K,Chai L,Mayer C,et al. Exploiting BAC-end sequences for the mining,characterization and utility of new short sequences repeat (SSR) markers in Citrus[J]. Molecular Biology Reports,2012,39(5):5373-5386.
[37]Li D J,Deng Z,Qin B,et al. De novo assembly and characterization of bark transcriptome using Illumina sequencing and development of EST-SSR markers in rubber tree (Hevea brasiliensis Muell. Arg.)[J]. BMC Genomics,2012,13(1):192.

