一株具有噬菌体抗性的芽孢杆菌BS-2的鉴定及葡萄糖流加工艺优化
戚家明 杨娜 孙杉杉 明艳超 郭亮 张东旭 徐志文
(1. 河北省农业生物技术工程技术研究中心,秦皇岛 066004;2. 河北省农业厅植物营养与海洋功能肥料重点实验室,秦皇岛 066004;3. 领先生物农业股份有限公司,秦皇岛 066004)
枯草芽孢杆菌(Bacillus subtilis)属于芽孢杆菌属,是一种好氧革兰氏阳性菌,表面生有鞭毛。在土壤和植物根际中枯草芽孢杆菌是优势种群,其可以产生芽孢在极端环境中长期存活,是一种植物根际的有益微生物,可以通过分泌拮抗物质和竞争作用防治植物病害[1]。
早期对枯草芽孢杆菌近缘种群的分类与鉴定主要是根据菌株的形态特征和生理生化特征进行区分如通过菊粉、乳糖发酵产酸等试验可区分枯草芽孢杆菌和解淀粉芽孢杆菌[2],但这种分类方法过程繁琐准确性差[3],随着分子生物学技术的发展,具有高分辨率及稳定性的基因序列分析鉴定技术逐渐成为菌株鉴定的重要手段。16S rDNA基因既具有高度保守性又具有高变性从而可以反映出不同种属间的亲缘关系,是细菌系统发育和种属鉴定的常用分类工具[4]。但枯草芽孢杆菌类群间的亲缘关系较近,16S rDNA序列相似度太高从而导致区分度较低[5],为了能够有效准确区分枯草芽孢杆菌种群间的亲缘关系,有研究在细菌的基因组中挑选了在系统进化过程中保守但又具有相对较快的变异速率的基因作为补充,如gyrA[6]、rpoB[7]及gyrB[8]基因等。
枯草芽孢杆菌在农业生产等方面具有较大的应用价值和经济价值,有许多研究对枯草芽孢杆菌的液体发酵条件进行优化,提高发酵水平达到产业化的目的[9-10]。在枯草芽孢杆菌工业发酵过程中提高发酵水平需要对接种量、pH、溶氧及消泡剂用量等一系列因素进行严格控制同时要防止噬菌体污染。
在枯草芽孢杆菌BS-0的工业发酵过程中,发现存在活菌数骤然下降,发酵液变澄清等感染噬菌体典型现象,严重影响产品质量及产量,虽然经过环境消毒以及暂停生产可以在短时间缓解噬菌体感染,但在连续生产一段时间后噬菌体感染情况再次出现。
相比于环境治理和添加抑制剂,筛选噬菌体抗性菌株是较为彻底的解决噬菌体感染问题的方法[11]。为了解决发酵生产过程中噬菌体污染问题,本研究从枯草芽孢杆菌BS-0异常发酵液中分离到2株形态不同的噬菌体S01及S02,利用这两株噬菌体对实验室保藏的芽孢杆菌菌株进行噬菌体抗性菌株筛选,对筛选得到的芽孢杆菌进行分子鉴定确定其分类地位,并对其发酵工艺进行优化。
1 材料与方法
1.1 材料
供试菌株:枯草芽孢杆菌菌株BS-0、其他供试芽孢杆菌BS-1、BS-2、BS-3及噬菌体S01、S02均由领先生物农业股份有限公司分离并保存。BS-0、BS-1、BS-2及BS-3均为土壤中筛选到具有较好生防效果的根际微生物。
主要培养基:LB固体培养基;双层平板培养基:下层培养基,牛肉膏5 g/L,蛋白胨10 g/L,葡萄糖2 g/L,NaCl 10 g/L,琼脂 18 g/L,pH 7.0,上层培养基,牛肉膏5 g/L,蛋白胨10 g/L,葡萄糖2 g/L,NaCl 10 g/L,琼脂6 g/L,pH 7.0;种子及发酵培养基:葡萄糖25 g/L,豆粉20 g/L,碳酸钙1.5 g/L,磷酸二氢钾2 g/L,蛋白胨1 g/L,酵母浸粉3 g/L,硫酸镁0.5 g/L,硫酸锰0.05 g/L,pH 7.0
1.2 方法
1.2.1 噬菌体的分离纯化 参照双层平板法[12]并改进:取5 mL异常发酵液经3 500 r/min 离心10 min,留取上清液并经过0.22 μm直径的无菌滤膜过滤除菌,取100 μL滤液进行适当稀释,取1 mL稀释液与3 mL培养至对数期前期(OD600=0.3-0.5)的枯草芽孢杆菌BS-0的菌液进行混合,放入37℃恒温培养箱中培养15 min,融化半固体培养基并冷却至45℃以下,将培养后的混合菌液与融化的半固体培养基充分混匀并立即均匀倾倒在预先准备好已凝固的下层培养基上。经凝固的培养基平板倒置于37℃培养箱中培养12 h后观察噬菌斑形态。
用灭菌牙签分别挑取形态不同的单个噬菌斑接入对数前期的枯草芽孢杆菌BS-0菌液中,培养6-8 h后离心取上清液过滤除菌,得到滤液后采用上述倾倒双层平板的方法进行纯化,次日再次挑取单个噬菌斑再次进行该试验直至平板上噬菌斑形态一致。
将纯化得到的噬菌体接入对数前期BS-0的菌液中,培养10-12 h后离心留取上清液,上清液过滤除菌后即为单一噬菌体原液,放置于4℃保存备用。
1.2.2 保藏菌株的噬菌体抗性筛选 保藏菌株进行划线活化后挑取单菌落接于液体培养基中过夜培养,取梯度稀释后的噬菌体溶液1 mL分别与3 mL菌液混匀倾倒双层平板,过夜培养观察是否出现噬菌斑。
1.2.3 噬菌体抗性菌株的分子鉴定 将筛选到的噬菌体抗性菌株从保藏的甘油管中接出进行划线培养,挑取单菌落送至生工生物工程(上海)股份有限公司北京测序部,对该菌的16S rDNA和gyrA基因进行测序,测序结果分别提交至ezbiocloud.net数据库及NCBI数据库进行比对分析,利用MEGA 7.0.26软件Neighbor-Joining 法构建系统发育树。
1.2.4 枯草芽孢杆菌发酵工艺优化
1.2.4.1 发酵种子液的制备 将菌株从保藏的甘油管中接于平板中进行活化,用接种环从平板中挑取一环接于500 mL种子液培养基(成分与发酵培养基相同)中,30℃ 170 r/min培养至对数前期。
1.2.4.2 分批发酵工艺 使用15 L发酵罐(镇江东方生物工程设备技术有限责任公司制造),500 mL种子液接入装有9 L培养基的15 L发酵罐中进行发酵。发酵过程中由溶氧电极(瑞士梅特勒公司制造)测定发酵液中溶氧值,pH值由pH电极(瑞士梅特勒公司制造)测定。发酵期间的参数控制:初始pH、通气量及搅拌转速分别为7.0、2 L/min及150 r/min,发酵期间调整通气量及搅拌转速控制溶氧在30%以上,温度恒定保持在37℃,泡沫较多时流加消泡剂消除泡沫。
1.2.4.3 流加发酵工艺 前期发酵参数同分批发酵,后期分别采取以下2种方式流加葡萄糖:(a)初始糖浓度为10 g/L,当发酵液中糖浓度低于5 g/L时,按照3g/L·h的速度流加葡萄糖,补加量10 g/L。(b)初始糖浓度为15 g/L,当发酵液中糖浓度低于5 g/L时,按照2 g/L·h的速度流加葡萄糖,补加量10 g/L。
1.2.5 发酵过程中生物量的测定 采用平皿涂布计数法[13]。
1.2.6 葡萄糖含量测定 SBA-40C生物传感分析仪测定。
2 结果
2.1 噬菌体分离及原液制备
枯草芽孢杆菌BS-0发酵液经双层平板法分离后出现2种形态不同的噬菌斑,分别将其命名为S01及S02(图1)。噬菌体S01产生边缘清晰半透明、中心全透明的同心圆形噬菌斑,内径1.1 mm、外径2.9 mm,噬菌体S02产生边缘清晰半透明圆形噬菌斑直径1.7 mm,并按照方法1.2.1制备噬菌体原液。
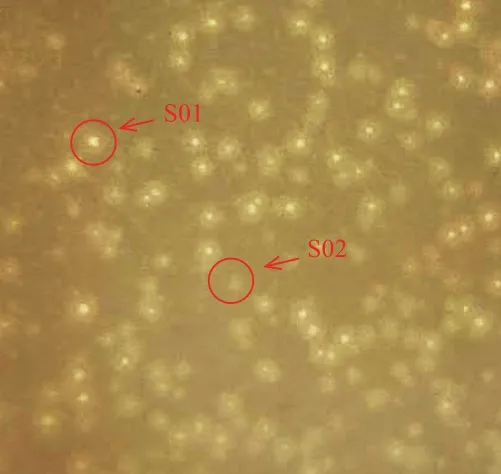
图 1 S01及S02形成的噬菌斑
2.2 噬菌体抗性菌株筛选
为了筛选噬菌体抗性菌株,从实验室的芽孢杆菌菌种库中选取了具有较好生防效果的3株芽孢杆菌(BS-1、BS-2和BS-3)进行噬菌体抗性筛选。将噬菌体S01及S02的原液按照1∶1的比例混合均匀进行复合侵染,以原始菌株BS-0作为对照。如图2所示,芽孢杆菌BS-0、BS-1及BS-3均出现了明显的噬菌斑,而芽孢杆菌BS-2并未出现噬菌斑,这表明噬菌体S01及S02无法侵染BS-2。
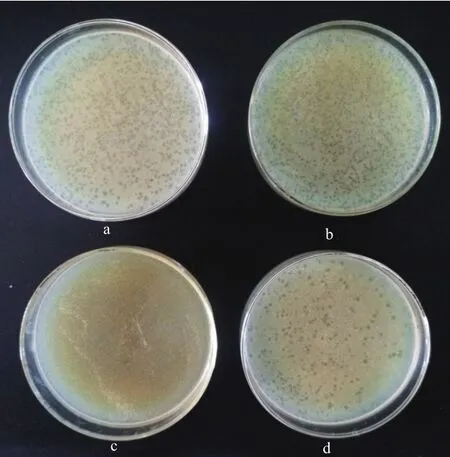
图2 噬菌体抗性菌株筛选
芽孢杆菌BS-2在发酵罐中进行发酵培养时并未出现活菌数骤然下降发酵液变澄清等噬菌体感染现象,如图3所示,对结束培养时的发酵液进行噬菌体分离,以原始菌株BS-0作为对照,在发酵液中不存在噬菌体感染情况。
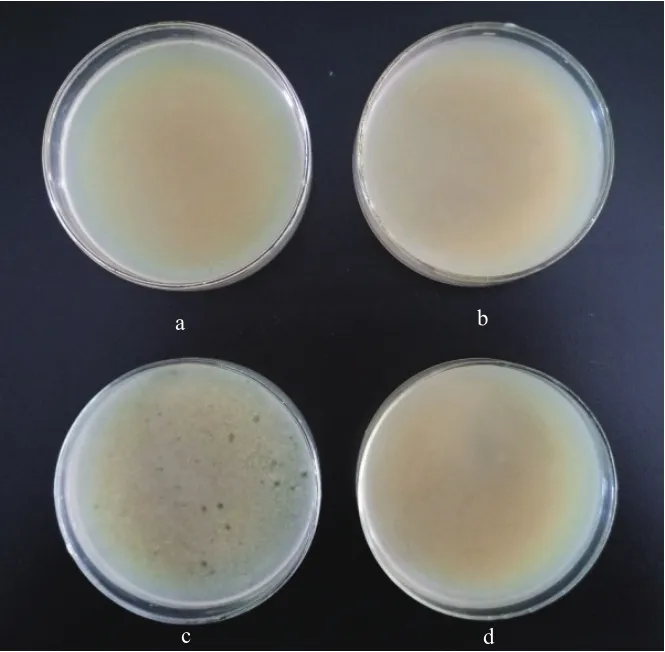
图3 BS-2发酵液中噬菌体检测
2.3 菌株BS-2的分子生物学鉴定
在前面的实验中我们得到了一株枯草芽孢杆菌的噬菌体无法侵染的芽孢杆菌BS-2,根据细胞形态及生理生化特征我们仅能将BS-2鉴定到枯草近缘种群,无法准确鉴定BS-2为枯草芽孢杆菌,故对芽孢杆菌BS-2进行分子生物学鉴定确定其分类地位。
2.3.1 基于16S rDNA 序列比对结果的系统进化分析 芽孢菌BS-2的16S rDNA基因片段序列长度为1 421 bp,GenBank登录号为 MH469709。将测序得到的序列提交至ezbicloud.net数据库与已知模式菌株的16S rDNA序列进行比对。选取同源性高于99%的菌株序列利用MEGA7软件邻位连接法构建系统发育树(相似度重复计算1 000次)。芽孢菌BS-2基于16S rDNA序列的系统发育树,如图4所示。根据比对结果与菌株BS-2同源性高于99%的菌株均为芽孢杆菌属,可以确定BS-2为芽孢杆菌属,但根据系统发育树无法准确鉴定BS-2到种。
2.3.2 基于gyrA基因序列 BLAST 结果的系统进化分析 由于根据16S rDNA 序列比对结果的系统进化分析无法准确鉴定BS-2到种,故又对芽孢杆菌BS-2的gyrA基因序列进行系统进化分析。芽孢菌BS-2的gyrA基因片段序列长度为729 bp,GenBank登录号为MH479013。将测序得到的gyrA基因序列提交至NCBI进行BLAST分析,得到Ident和Query cover均大于99%的结果100个,其中95个为枯草芽孢杆菌,选择其中13株菌的gyrA序列,同时根据16S rDNA比对结果选取其中4株同源性较高的芽孢菌的gyrA基因序列构建系统发育树(图5)。根据NCBI比对以及系统发育树,可以准确将菌株BS-2鉴定为枯草芽孢杆菌。
2.4 葡萄糖流加方式对BS-2发酵的影响
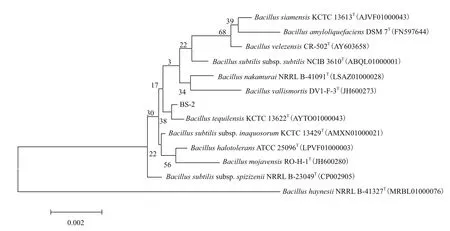
图4 基于16S rDNA序列比对结果构建的菌株BS-2的系统发育树
利用15 L发酵罐对枯草芽孢杆菌BS-2进行液体发酵,采取方法1.2.4.2所描述的分批发酵方式和方法1.2.4.3描述的2种不同的补料方式流加葡萄糖,每隔4 h收取发酵液样品进行生物量测定及葡萄糖含量测定。液体发酵期间发酵液中生物量变化曲线,如图6所示。
液体发酵期间发酵液中葡萄糖浓度变化曲线如图7所示。三种发酵方式的发酵结果统计如表1所示。
根据分批发酵生物量变化曲线,枯草芽孢杆菌BS-2发酵呈现以下特点:在0-4 h,处于延迟期,菌体生长缓慢葡萄糖消耗较慢,4-8 h出现第一个生长高峰,随后8-12 h菌数下降,12-16 h出现第二个生长高峰,16-20 h开始出现芽孢,菌数再次下降并在第20 h时芽孢率大于90%结束培养。分批发酵的初始糖浓度为25 g/L,推测该糖浓度显著高于枯草芽孢杆菌BS-2生长的最适糖浓度从而抑制其生长,故降低初糖浓度,并在发酵液中葡萄糖浓度低于5 g/L时进行补料。
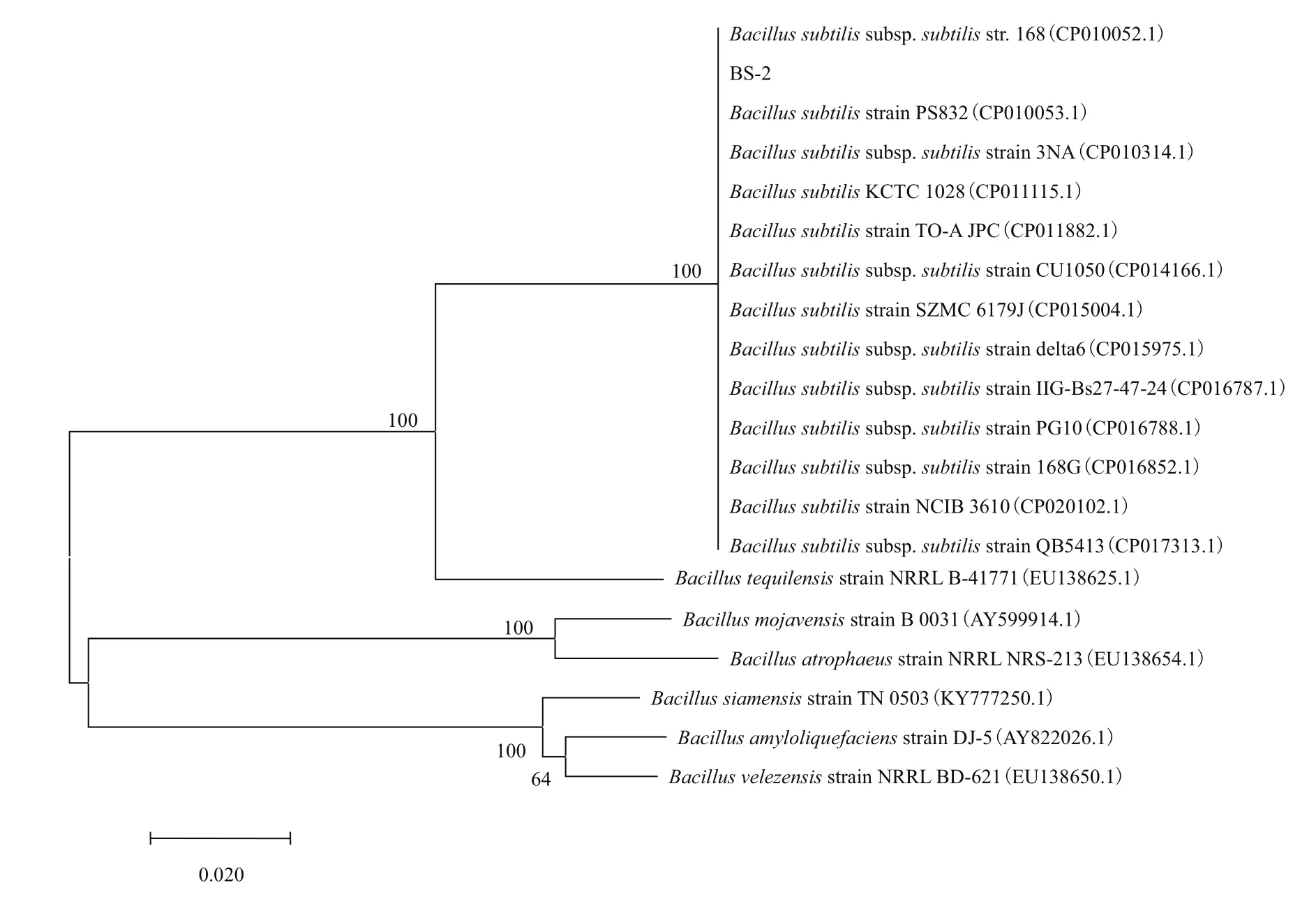
图5 基于gyrA序列比对结果构建的菌株BS-2的系统发育树
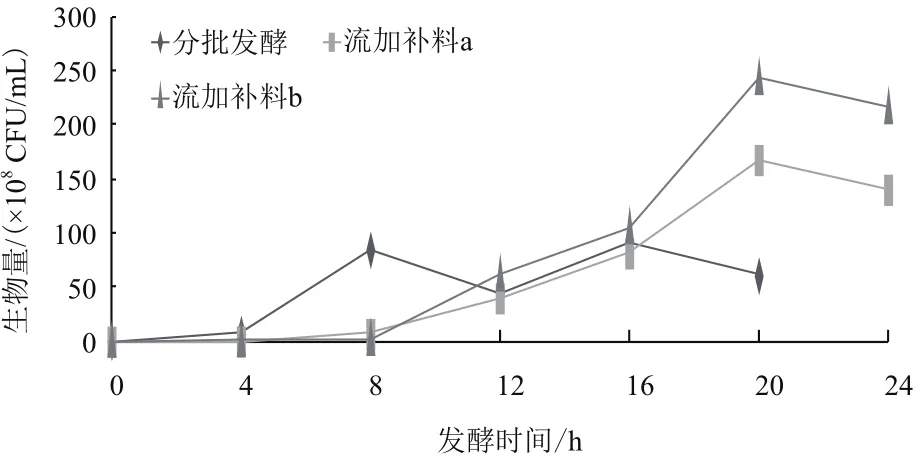
图6 枯草芽孢杆菌BS-2发酵曲线
流加补料与分批发酵相比在发酵前期0-12 h中并未出现菌数先下降后上升的现象,流加补料发酵过程中0-8 h处于延迟期,8-16 h菌体开始生长,16 h达到补料点进行流加补料,16-20 h开始快速生长在20 h达到最大值,第24 h芽孢率达到90%结束培养。结果显示初糖浓度25 g/L对于BS-2生长确实具有一定的抑制作用,在降低初糖浓度后,发酵周期延长,葡萄糖利用率及生物量显著提升。
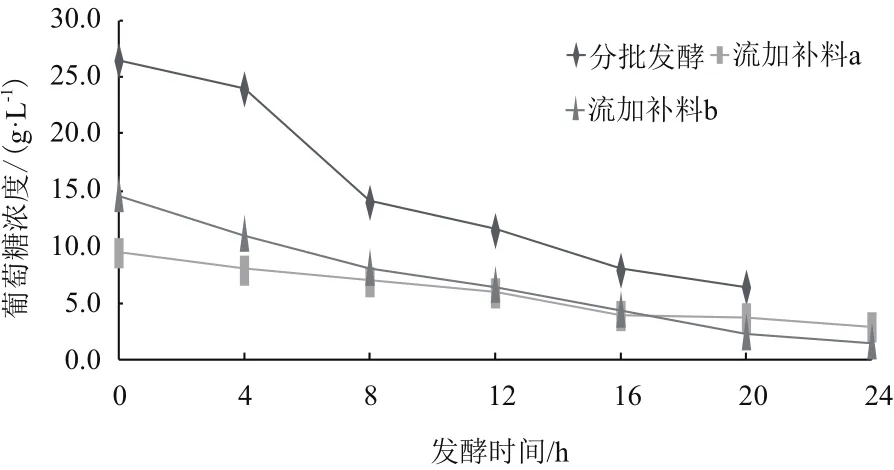
图7 枯草芽孢杆菌BS-2发酵液中葡萄糖浓度的变化
比较两种补料方式,在相同葡萄糖添加量条件下,初糖浓度15 g/L时生物量和葡萄糖利用率都高于初糖浓度10 g/L的处理,初糖浓度为15 g/L时更适宜BS-2菌体生长。
3 讨论
枯草芽孢杆菌工业化发酵生产中噬菌体感染对生产影响严重,相比于筛选能抑制噬菌体生长且不影响芽孢杆菌生长的抑制剂[14]及环境治理[15]等方法,筛选噬菌体抗性菌株的方法更加彻底和简便。

表1 三种补料方式的发酵结果
本实验从枯草芽孢杆菌BS-0异常发酵液中分离到以枯草芽孢杆菌为宿主菌的噬菌体S01及S02,并利用S01、S02对实验室芽孢杆菌库进行筛选,得到一株噬菌体无法侵染的芽孢杆菌BS-2,菌种库信息显示依据细胞形态及生理生化特征将BS-2鉴定为枯草近缘种群的芽孢杆菌,难以确定枯草芽孢杆菌的噬菌体S01、S02无法侵染BS-2的原因是BS-2并非噬菌体的宿主枯草芽孢杆菌,还是BS-2是具有噬菌体抗性的枯草芽孢杆菌。故我们对芽孢杆菌BS-2进行分子生物学鉴定以确定其分类地位。
枯草芽孢杆菌近缘种群包括莫哈维芽孢杆菌(Bacillus mojavensis)、特基拉芽孢杆菌(Bacillus tequilensis)、暹罗芽孢杆菌(Bacillussiamensis)及解淀粉芽孢杆菌(Bacillusamyloliquefaciens)等,在近缘种群内利用传统的生理生化指标及16S rDNA序列比对等手段无法进行准确区分[16],近年来在枯草芽孢杆菌分类的研究中发现有很多基因可以作为近缘种的分类工具且具有良好的区分度。有研究人员对枯草群内菌株的gyrA基因进行了测序[17],发现该基因具有较大的差异。相似性最高的两个种莫哈维芽孢杆菌与深褐芽孢杆菌间gyrA基因相似度仅为95.8%,在枯草群内的菌株具有较好的区分度[18]。根据BS-2 16S rDNA序列构建的系统发育进化树无法将BS-2与枯草群内近缘种准确区分,故又结合gyrA基因再次进行鉴定,最终确定BS-2为枯草芽孢杆菌,同时也证明枯草芽孢杆菌BS-2具有噬菌体抗性。
在摇瓶优化的基础上利用15 L发酵罐优化枯草芽孢杆菌BS-2的发酵工艺。进行分批发酵时出现了菌数先下降后上升的现象且最终发酵水平较低,过高的葡萄糖浓度对菌株的生长会有抑制作用[19],且流加发酵的工艺可以大幅度提高发酵水平[20],故降低初糖浓度并采取流加葡萄糖补料的方式进行发酵。根据发酵过程中生物量变化曲线,流加补料发酵在8-16 h中菌数持续增长,并且在16 h发酵液中葡萄糖浓度低于5 g/L时开始补加葡萄糖,16-20 h生长速度进一步加快,并且整个发酵时间延长4 h。最优的流加补料发酵方式下,葡萄糖利用率和菌体产量分别是分批发酵条件下的1.24倍和3.98倍。流加补料的方式解决了分批发酵中葡萄糖浓度较高抑制生长的问题,并且延长了菌株生长的时间,提高了葡萄糖利用率及发酵水平,葡萄糖初始浓度过高或过低都无法得到较高的生物量。
4 结论
从异常发酵液中分离到两株噬菌体S01及S02,利用噬菌体对实验室保藏的芽孢杆菌菌株进行筛选得到一株噬菌体无法侵染的芽孢杆菌BS-2,结合16S rDNA及gyrA基因序列可以确定BS-2为枯草芽孢杆菌,且其具有噬菌体抗性。经过发酵工艺优化,在15 L发酵罐培养中采取流加补料的方式,枯草芽孢杆菌BS-2发酵水平可以达到2.43×1010CFU/mL。具有噬菌体抗性的枯草芽孢杆菌BS-2可以达到较高的发酵水平,具有较强的工业化应用潜力。
致谢:本研究得到河北省产业创新创业团队(169A76114H)的资助及河北省农业生物技术工程技术研究中心李丽艳、王东、揣峻峰和周立华等同志的大力协助,在此对他们不遗余力的帮助表示感谢。

